Zarco Zavala Ilse Mariel Examen de Fisicoquímica del plegamiento de Proteínas
Anuncio
Zarco Zavala Ilse Mariel
Examen de Fisicoquímica del plegamiento de Proteínas
10 abril de 2007
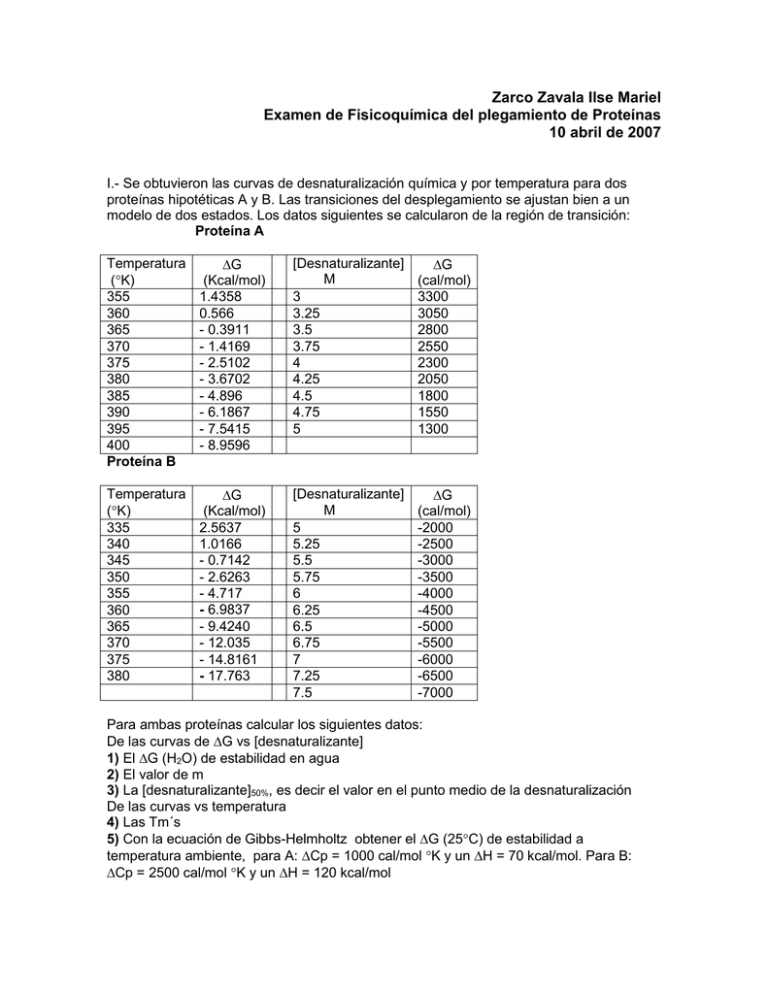
I.- Se obtuvieron las curvas de desnaturalización química y por temperatura para dos
proteínas hipotéticas A y B. Las transiciones del desplegamiento se ajustan bien a un
modelo de dos estados. Los datos siguientes se calcularon de la región de transición:
Proteína A
Temperatura
(K)
355
360
365
370
375
380
385
390
395
400
Proteína B
G
(Kcal/mol)
1.4358
0.566
- 0.3911
- 1.4169
- 2.5102
- 3.6702
- 4.896
- 6.1867
- 7.5415
- 8.9596
[Desnaturalizante]
M
3
3.25
3.5
3.75
4
4.25
4.5
4.75
5
G
(cal/mol)
3300
3050
2800
2550
2300
2050
1800
1550
1300
Temperatura
(K)
335
340
345
350
355
360
365
370
375
380
G
(Kcal/mol)
2.5637
1.0166
- 0.7142
- 2.6263
- 4.717
- 6.9837
- 9.4240
- 12.035
- 14.8161
- 17.763
[Desnaturalizante]
M
5
5.25
5.5
5.75
6
6.25
6.5
6.75
7
7.25
7.5
G
(cal/mol)
-2000
-2500
-3000
-3500
-4000
-4500
-5000
-5500
-6000
-6500
-7000
Para ambas proteínas calcular los siguientes datos:
De las curvas de G vs [desnaturalizante]
1) El G (H2O) de estabilidad en agua
2) El valor de m
3) La [desnaturalizante]50%, es decir el valor en el punto medio de la desnaturalización
De las curvas vs temperatura
4) Las Tm´s
5) Con la ecuación de Gibbs-Helmholtz obtener el G (25C) de estabilidad a
temperatura ambiente, para A: Cp = 1000 cal/mol K y un H = 70 kcal/mol. Para B:
Cp = 2500 cal/mol K y un H = 120 kcal/mol
Proteína A
Desnaturalización química
3500
G = GH2O – m desnat.
3000
2000
GH2O = 6.3 Kcal/mol
m = -1000
G
(cal/mol)
Y = 6300 – 1000x
Y =6300-1000 X
2500
1500
1000
3,0
3,5
4,0
4,5
5,0
fD = 0.5 G = 0
[desnaturalizante]50% = 6.3 M
Desnaturalizante M)
Proteína A
Desnaturalización por Temperatura
2
Y = 83.98 – 0.232x
G (kcal/mol)
0
-2
Y =83,98795-0,23138 X
-4
-6
-8
-10
350
360 Tm
370
380
390
400
T (Kº)
G = H ( 1- T/Tm) - Cp {Tm – T + T ln (T/Tm)}
Tm = 361.98 Kº
T = 298 Kº
A: Cp = 1000 cal/mol K y un H = 70 kcal/mol.
G = 6.32 Kcal/molKº
Tm = 361.98 Kº
Proteína B
Desnaturalización Química
-2000
G = GH2O – m desnat.
-3000
G (cal/mo)
Y = 8000 – 2000x
Y =8000-2000 X
-4000
-5000
GH2O = 8 Kcal/mol
m = -2000
-6000
-7000
5,0
5,5
6,0
6,5
7,0
7,5
Desnaturalizante (M)
Proteína B
Desnaturalización Térmica
5
G (Kcal/mol)
0
Y = 155.09 – 0.452x
-5
Y =155,09773-0,45216 X
-10
-15
-20
330
fD = 0.5 G = 0
[desnaturalizante]50% = 4M
340 Tm
350
360
370
380
T (Kº)
G = H ( 1- T/Tm) - Cp {Tm – T + T ln (T/Tm)}
Tm = 343.12 Kº
T = 298 Kº
B: Cp = 2500 cal/mol K y un H = 120 kcal/mol
G = 8.015 Kcal/molKº
Tm = 343.12 Kº
6) Explica los resultados comparando los datos para ambas proteínas. Qué
parámetros reflejan mejor los valores de estabilidad conformacional de una
proteína.
La proteína B es más estable que la proteína A, esto se ve reflejado por los
valores de GH2O ya que la proteína B tiene un valor de 8Kcal/mol, mientras que
la proteína A solo tiene 6.32Kcal/mol; sin embargo el punto en el proceso de
desnaturalización en el que la mitad de la proteína ha sido desplegada (tanto por
desnaturalización térmica como por desnaturalización química) es más bajo para
la proteína B con respecto a la proteína A; sin embargo el valor del G (GibbsHelmholtz sustenta la idea de mayor estabilidad conformacional para la proteína B,
este valor es más completo ya que este toma en cuenta la dependencia del H y
el S a la temperatura, factor que es corregido mediante el uso del Cp
Yo creo que no es posible asumir que ninguno de los parámetros refleja de
manera mejor la estabilidad conformacional de las proteínas ya que cada uno de
los valores provee de información de los fenómenos que ocurren dentro de
proceso de desnaturalización de la proteína y el análisis de todos en conjunto
refleja la estabilidad conformacional.
II.- La proteína p53, (proteína supresora de tumores), es un homotetrámero. Cada
monómero tiene cuatro dominios funcionales cuyas estructuras han sido definidas:
Fig.1 Secuencia de aminoácidos lineal del monómero de P53. Se muestra la posición de los 4
triptófanos. Las barras negras indican las regiones para las cuales se han obtenido las estructuras
cristalográficas.
Fig.2. Dominio de unión al DNA (core domain). Se muestra la posición del triptófano
Fig.3. Dominio de tetramerización a) dimero de dímeros b) Interfases entre monómeros. Se
muestra la localización de la Tyr en cada monómero.
El dominio central (core) que une al DNA, se aisló de manera
independiente; estudios de desnaturalización por urea, monitoreando el cambio de
fluorescencia de este dominio central, muestran que el proceso de desplegamiento
es reversible y los datos se ajustaron asumiendo un modelo de dos estados:
Dominio
DNA Core
m, kcal·mol 1·M
2.24 ± 0.008
1
[Urea]50%, M
2.66 ± 0.01
, kcal·mol
1
5.96 ± 0.20
El dominio central presenta un solo triptófano (figura 2), mientras que el monómero
completo presenta 4 triptófanos, 3 más localizados en el dominio N-terminal que
tiene una estructura desordenada (figura 1). En un trabajo posterior se estudio el
desplegamiento de la proteína en su forma tetrámerica. Cuando la
desnaturalización se siguió por cambios en la intensidad de fluorescencia, los
valores de la fluoresecencia relativa se mantuvieron constantes de 0 a 5 M de
GuHCL o urea, es decir no fue posible detectar cambios en la intensidad de
fluorescencia a ninguna concentración del desnaturalizante. La accesibilidad de
los triptófanos para la proteína tetramérica, se obtuvo mediante los ensayos de
apagamiento de fluorescencia por Yoduro. La constante de apagamiento de Stern-
Volmer, KSV obtenida de los experimentos con la p54 tetramérica fue de 0.15 M -1
(un valor de KSV 0.2 quiere decir que no hay apagamiento).
1)
Da una(s) posible(s) explicación para las diferencias observadas en la
desnaturalización seguida por cambios en la intensidad de fluorescencia
del dominio de unión y la proteína tetramérica.
Este fenómeno puede deberse a dos explicaciones: 1. que ningún cambio
significativo haya ocurrido en el ambiente de la proteína completa (Las
concentraciones de desnaturalizante usadas fueron insuficientes para llevar a
cabo la desnaturalización completa de la proteína tetramérica), mientras que la
mayor labilidad del dominio de unión aislado, haya permitido su desnaturalización
con concentraciones más bajas de desnaturalizante, con respecto a las necesarias
para la desnaturalización de la proteína tetramérica.
o 2. El cambio en estructura que se llevo a cabo en la desnaturalización, de la
proteína tetramérica, no es reflejado por la fluorescencia de los triptofanos; la
existencia del fenómeno anterior puede ser explicada por el hecho de que la
constante de apagamiento de Stern-Volmer para la p54 tetramérica fue menor a
0.2 lo cual refleja que no hay apagamiento y se relaciona con inaccesibilidad de
los triptofanos o apagamiento intrínseco de los residuos de triptofano dentro de la
proteína completa.
Por lo tanto es posible suponer que aun cuando el dominio de unión aislado sí
refleja cambios en su fluorescencia generados por su triptofano único; la proteína
completa, durante el proceso de desnaturalización y hasta llegar al estado
desplegado, se estructura de forma tal que existe un apagamiento concertado de
su fluorescencia, (aun cuando esta cuenta con 16 triptofanos!!!) que posiblemente
es mediado por los otros dominios de la proteína y/o por interacciones
intratetraméricas que apagan la fluorescencia per se y aumentan la estabilidad de
la proteína tetramérica.
2)
¿Qué experimentos propones para poder estudiar el desplegamiento de la
p53 tetramérica?
Yo propongo el uso del dicroismo circular en ensayos de desnaturalización de la
proteína, ya que este experimento daría información complementaria de la
estructura de la proteína a través del proceso de desnaturalización independiente
del ambiente de los triptofanos.
Cuando la desnaturalización del dominio central aislado es inducida por
temperatura, éste tiende a agregar, por lo tanto es un proceso irreversible. La
temperatura media de desnaturalización irreversible del dominio de unión al DNA
es de 42 C.
Otro de los dominios de la p53 que se ha estudiado de manera aislada es el
dominio de tetramerización (Fig. 3). Consta de aprox. 30 residuos y se obtiene en
forma tetramérica. Los estudios de desnaturalización térmica del dominio p53tet
dan un valor de Tm entre 75.2- 80 C.
La p53 completa, como tetrámero, también se desnaturaliza por temperatura de
manera irreversible con una Tmaparente de 73 C, sin embargo, aún a 100 C se
conserva una fracción considerable de estructura secundaria. Experimentos de
entrecruzamiento por glutaraldehído indican que la proteína completa se mantiene
como tetramero a 50 C.
3) A qué se pueden deber las diferencias entre las Tm´s del dominio central
aislado y de la proteína completa.
El incremento en la termoestabilidad de la proteína completa con respecto al
domino central aislado puede ser resultado de la formación de oligomeros, ya que
estos cuentan, un mayor número de dominios, por lo tanto son capaces de
construir una red de interacciones más amplia y estructuración más compleja que
aumentan su estabilidad con respecto al dominio aislado; este fenómeno puede
ser reflejado por:
El hecho que de que el dominio p53tet tenga un valor de Tm alto y que a una
temperatura de 50ºC la proteína mantenga su estructura tetramérica refleja que
que la perdida de estructura secundaria se lleva a cabo fuera del dominio de
tetramerización, es decir es la desnaturalización que se observa mediante en CD
pertenece a la región del C-terminal y la región N-terminal.
Al observar al dominio de unión a DNA, es posible observar que esta compuesto
principalmente por estructura de hoja- , esta clase de estructuración extendida
puede favorecer las interacciones entre monómeros, las cuales a su vez
estabilizan la conformación tetramerica.
El dominio de tetramerización (Fig. 3) se ha utilizado como modelo para
estudiar el proceso de asociación de la proteína completa. Los experimentos de
desnaturalización por GuHCl al equilibrio del dominio p53tet son completamente
reversibles, observándose una sola transición, por lo que se ajustan a un modelo
de dos estados y no se observa la acumulación de un intermediario estable. Los
datos se muestran en la tabla siguiente:
Dominio
p53tet
m, kcal·mol 1·M
4.9 ± 0.1
1
Gdisociación
kcal·mol 1
[GdnHCl]50%, M
GuH2O
kcal·mol
2.82 ± 0.01
32.4 ± 0.20 23.3
1
En la tabla se muestra también el G de disociación para el dominio de
tetramerización, obtenidos por experimentos de centrifugación al equilibrio, a
distintas concentraciones de proteína.
4) Comparando los valores de GuH2O y de disociación, calcula la contribución de
la estabilidad de cada monómero nativo respecto al estado desnaturalizado (en
kcal/mol) y qué proceso: plegamiento de los monómeros y/o asociación de
monómeros contribuye más a la estabilidad global del dominio de tetramerización.
El calculo de la contribución de la estabilidad de cada monómero; fue el siguiente
reste el G disociación al G en H20 partiendo de la premisa de que al restarlo, el
único factor que me quedaría es el de plegamiento, después dividí ese número
entre 4 para obtener así el valor para cada monómero = 2.275Kcal/mol:
Creo que el factor determinante en la estabilidad global del domino de
tetramerización es el de asociación de monómeros, ya que este proceso es el
paso limitante en la pérdida de estructura de la p53tet y mediante los otros
experimentos podemos ver que este la tetramerización le confiere un alto grado de
estabilidad conformacional a la proteína completa.
Para este dominio de tretamerización se hicieron experimentos de cinética
del desplegamiento ND, midiendo cambios a 222 nm (stop-flow CD). Los datos
obtenidos se ajustan a una ecuación de velocidad exponencial sencilla, que es lo
que se espera para un proceso unimolecular monofásico. La constante de
velocidad fue independiente de la concentración de proteína e incrementa de
manera exponencial con el aumento de la concentración de GdnHCl. En los
experimentos de replegamiento D→N, seguidos por la misma técnica, se observa
una sola fase, pero los datos se ajustan a una reacción de segundo orden. La
constante del replegamiento mostró dependencia de la concentración de proteína
pero fueron independientes de la viscosidad del medio indicando que el paso
observado en el replegamiento/asociación no está limitado por difusión.
5) Con los datos anteriores proponer un mecanismo para el plegamiento y
asociación del dominio p53tet.
El hecho de que los experimentos de replegamiento se haya ajustado a una
reacción de segundo orden nos habla de que al menos dos pasos cinéticos
(monómero desplegado y tetrámero plegado) y un intermediario transitorio
(intermediario dimérico transitorio)
se presentan durante el proceso de
plegamiento; ya que se observo solamente una fase es posible pensar que uno
de los pasos no es detectable mediante la técnica usada, esto puede deberse a
que este paso sea demasiado rápido o se lleve a cabo dentro del tiempo muerto
del experimento.
Mientras que el hecho de que la constante de replegamiento/asociación de los
monomeros dsnaturalizados no varié con la viscosidad del solvente, más sea
dependiente de la concentración de proteína p53, sugiere que el estado de
transición es dimérico.
4M
D
2D
t
1T
N