Diseño de un nuevo test NGS para el análisis ru9nario de
Anuncio

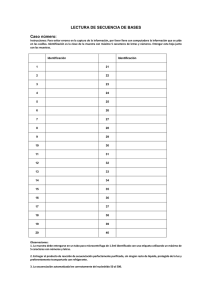
Diseño de un nuevo test NGS para el análisis ru5nario de cáncer hereditario Dr. Alberto Acedo Centro de Diagnós4co gené4co AC-­‐Gen Reading S.L. Valladolid, 5 de marzo de 2014 El cáncer: datos epidemiológicos 200.000 casos anuales Cáncer Hereditario 5-­‐10% Agregación familiar 10-­‐20% Esporádico 70% El cáncer hereditario: modelo de herencia dominante Mutaciones en línea germinal Modelo “Two hit” Knudson Cáncer de mama y ovario hereditario Riesgo CM a los 70 años población control: 7% BRCA1 BRCA2 CM 85% CM 85% CO 39% (18%-54%) CO 11% (2,1%-19%) ↑ c. colon, próstata (x3) y páncreas CM 6,9% ↑ c. próstata (x4.5) y páncreas Thompson y Easton (2001) 16% iCOGS (Nature, 2013) Cáncer de colon hereditario Polipósico No Polipósico Síndrome Riesgo de CCR Otros cánceres (riesgo) Síndrome Riesgo de CCR Otros cánceres (riesgo) Síndrome de Lynch 70-­‐85% Carcionma de ovario (9%) Gástrico (19%) Tracto urinario (10%) Intes4no y vías biliares (18%) Poliposis Adenomatosa familiar 100% en forma clásica 60% forma atenuada Duodeno (4-­‐12%) Estómago (0,5%) Hepatoblastoma (1,6% en niños < 5 años) Páncreas (2%) SNC: Turcot (<1%) Estómago (30%) Aarnio M. et al., 1995 Síndrome de Peutz-­‐Jeghers 40% Intes4no delgado (13%) Mama (54%) Páncreas(36%) Endometrio (9%) Cérvix (<0,5%) Ovario (21%) Tesaculo (<1%) Estómago (<0,5%) Duodeno (<0,5%) Poliposis Juvenil 40% Mama (20-­‐50%%) Tiroides (3-­‐10%) Estómago (<0,5%) Duodeno (<0,5%) Lynch et al. Cancer 2004; 100: 53–64. MMR=mismatch repair; FAP=familial adenomatous polyposis; AFAP=aienuated familial adenomatous polyposis; HBCC=hereditary breast and colorectal cancer; PJS=Peutz–Jeghers syndrome; FJP=familial juvenile polyposis; CD=Cowden's disease; BRRS=Bannayan–Ruvalcaba–Riley syndrome. Síndrome de Cowden Poliposis asociada al gen MYH Mama (25-­‐50%) Tirorides (3-­‐10%) >50% Duodeno (4%) HC-­‐Gen Test – Genes y SNPs incluidos GENES BRCA1 BRCA2 ATM BARD1 BRIP1 FAM175A MRE11A NBN RAD50 RAD51D RAD51C XRCC2 89SNP PALB2 STK11 CHEK2 PTEN TP53 CDH1 MUTYH MLH1 MSH2 MSH6 EPCAM PMS2 APC BMPR1A SMAD4 AXIN2 BLM ENG KIT MLH3 MSH3 PDGFRA PIK3CA PMS1 SCG5 BreastSeq ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ OvaSeq ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ColonSeq ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ PancSeq ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ ✓ Targeted Sequencing -­‐ Regiones exónicas de interés -­‐ Mejor cobertura y más homogénea -­‐ Menos variantes detectadas (falsos posi5vos y nega5vos) -­‐ Comparación rápida con bases de datos -­‐ Mul5plexado de las muestras -­‐ Reducción de costes Comprobación del diseño con Human Genome Browser Workflow: Secuenciación con Haloplex Extracción DNA librería Template Secuenciación Análisis Pureza: OD 260/289 1,8-­‐2 Integridad Comprobación Preparación de librería con Haloplex Paso 1: Diges5ón de DNA genómico librería 225ng en 45 ul (5ng/ul) (+/-­‐1) 8 reacciones de diges4ón independientes Paso 2: Hibridazión y barcoding No juntar las reacciones de diges4ón, añadir una a una en la solución de hibridación Barcode Paso 3: Captura de DNA Bio4nina + bolas de estreptavidina Paso 4: Ligación Paso 5: Amplificación y Purificación Preparación de librería con Haloplex. Punto de control 1 Evaluación de las 8 reacciones de diges5ón Sólo de la muestra control Preparación de librería con Haloplex. Punto de control 2 Validación y cuan5ficación de cada librería generada Adapter-­‐dimer -­‐10% 31.333 pmol/l Haloplex adaptador 3´específico 500pM 100pM 15pM Datos reales de secuenciación 1: ISP ac5vas Datos reales de secuenciación 2: Estadís5cas de lecturas Datos reales de secuenciación 3: Longitud de lecturas Datos reales de secuenciación 4: calidad de lecturas Alineamiento contra genoma de referencia Llamada de variantes: ¿Target o covered? Target Regions Covered Aplicación HC-­‐Gen test en cáncer de colon Tendencia y distribución municipal del cáncer colorrectal en el área de salud de León 1994-­‐2008 34% 41% Caso 1. Síndrome de Lynch Caso 1. Resultados y validación ! Gen$ Cambio$en$el$ADN$ Cambio$en$el$Aminoácido$ MLH1$ c.22_37Del$ p.I8Rfs*4$ Tipo$ mutación$ frameshift$ Valoración$ Patogénica$ Caso 2. Poliposis juvenil Colon 50 55/65 Colon 52 Colon Pulmón 65 Colon Pulmón Caso 2. Resultado complejo de evaluar ! Gen$ Cambio$en$el$ADN$ Efecto$en$la$ proteína$ Estado$ Tipo$de$ mutación$ Valoración$ BMPR1A$ c.1327C>T$ p.R443C$ Het$ Missense$ Patogénica$ Caso 2. Evidencias in silico y funcionales de patogenicidad Gen$ Cambio$en$el$ADN$ Efecto$en$la$ proteína$ Estado$ Tipo$de$ mutación$ Valoración$ BMPR1A$ c.1327C>T$ p.R443C$ Het$ Missense$ Patogénica$ ! Resultados*de*la*predicción!In!silico!del*efecto*de*la*mutación*en*la*función*de*la*proteína*BMPR1A* Mutación* PhyloP* BLOSUM*62* SIFT* Poly*Phen2* LRT* Mutation*Taster* GERP++* c.1327C>T;p.R442C. 1.00. .03.00. 1.00. 1.00. 1.00. 1.00. 5.58. El. rango. para. los. predictores. PhyloP,. PolyPhen2,. LRT. y. Mutation. Taster. es. de. 0. a. 1,. siendo. >0,95. probablemente.patogénico.y.<0,05.probablemente.benigno..Para.BLOSUM.62,.el.rango.es.de.03.a.+3,.siendo. <.0.probablemente.patogénico..GERP++,.valores.>0.probabilidad.de.residuo.limitado. ! Dr. Alberto Acedo Dra. Beatriz Sobrino Dr. Eladio Velasco AC-­‐Gen Reading Life S.L. Parque Cienafico Universidad de Valladolid Edificio CTTA 2º Planta C/Paseo de Belén nº 9 47011 Valladolid -­‐ España Alberto Acedo PhD. acedo@acgen.es Tlf: 983016257 @acedo_o Centro Autorizado de Diagnós5co Gené5co Premio de la Fundación Genoma España al Mejor Proyecto de Biotecnología.