metabolomica2010.pdf
Anuncio

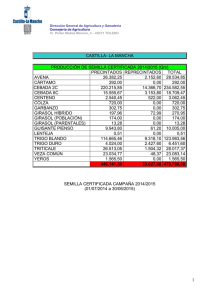
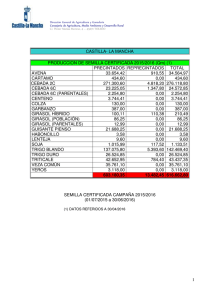
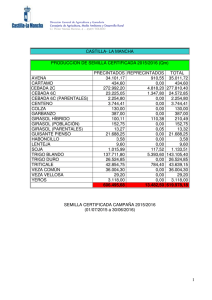
Disciplinas post-genómicas aplicadas al estudio de interacciones Hospedante – Patógeno: analisis integrado transcriptómica y metabolómica Dra Ruth Heinz Instituto de Biotecnología INTA Castelar Metabolómica • • • Disciplina postgenómica que permite analizar cambios en metabolitos originados por cambios a nivel genético, transcriptómico o proteómico Permite evaluar el efecto de un transgene en el metabolismo primario y secundario Recientemente se ha difundido el uso de varias estrategias de análisis de metabolitos para el estudio de la respuesta de plantas a patógenos, plagas y estreses abióticos Metabolómica en plantas • • • Metabolitos identificados en el reino vegetal: 100-200.000 correspondientes a metabolismo primario y secundario Técnicas que permiten realizar un “fingerprinting”: detección masiva de metabolitos en una muestra sin identificación Técnicas de análisis de perfiles: detección, cuantificación e identificación de metabolitos Estrategias de análisis metabolómico GC-MS: separación por cromatografía (GC or LC) acoplada a espectrometría de masa (MS). Compuestos termo estables y volátiles, polares y no polares ESI-MS: infusión directa por electrospray- MS . Para fingerprinting. Requiere validación posterior para identificación NMR (menor sensibilidad que GC) , Fourier transform (FT)-IR spectroscopy LC-MS: para metabolitos secundarios, de mayor tamaño Estudios de perfiles metabólicos por GC-MS Metabolismo primario: •Aminoacidos •Azúcares •Alcoholes •Acidos organicos •Acidos grasos Metabolismo secundario: alcaloides, fenilpropanos, terpenos, Estudios de perfiles metabólicos GC-MS Respuesta a infecciones con el patógeno Sclerotinia sclerotiorum Agente Antecedentes Soja y girasol cultivos oleaginosos más importantes del país. Rendimiento girasol está limitado principalmente por estreses bióticos y abióticos. La Podredumbre húmeda del capítulo (PHC) es una de las enfermedades mas devastadoras en la Argentina. En casos extremos puede causar perdidas totales La incidencia anual promedio sobre la producción de la pampa húmeda es del 10-20% ( Pérez Fernández, 2002) La base genética de la resistencia es compleja con varios QTLs involucrados en el carácter Antecedentes Agente Causal: • Sclerotinia sclerotiorum (necrotrófico) Factores de patogenicidad: • • Ácido oxálico Enzimas hidrolíticas (poligalacturonasas) Síntomas de la enfermedad: • Maceración de tejidos del capítulo • Formación de esclerocios Objetivo Estrategias Identifición y caracterización de nuevas fuentes de resistencia a PHC mediante herramientas genómicas Mapeo de QTLs: marcadores neutros SSR Caraterización fenotípica de parentales Herramientas y Metodologia Resultados obtenidos Proyección Identificación de genes candidatos: marcadores funcionales Construcción de colecciones de ADNc diferenciales Construcción de poblaciones de mapeo Análisis de perfiles transcripcionales y metabólico entre parentales R y S Selección de marcadores moleculares polimórficos Selección y aislamiento de secuencias completas de genes candidatos Caracterización fenotípica y genotípica de poblaciones F2 Craracterización estructural y funcional de genes candidatos identificación de tres QTL inéditos de resistencia a PHC en la población RHA801 x HA89 Identifiacion e de nuevos genes candiadtos; Caracterización estructural y funcional de genes De la familia GLP Integración del mapeo de QTLs y de genes candidatos en distintas poblaciones Estudio de perfiles de expresión génica de ESTs de interés Proteínas Germin-like (GLP) EF202 Actina RHA 275 RHA 801 HA 89 M 0 2 4 15 0 2 4 15 0 2 4 15 GLP HA 89 RHA 275 RHA 801 0 2 4 15 0 2 4 15 0 2 4 15 M 0 0 7 0 8 1 0 13 19 0 0 7 R1 R2 R3 R1 R2 R3 R1 R2 R3 R1 R2 R3 HA89 HA853 RHA275 RHA801 Strict Árbol filogenético Germin y GLPs Consenso estricto 5670 arboles TNT (Goloboff et al 2003) Actividad Oxalato Oxidasa Actividad Superoxido Dismutasa Ausencia de Actividad Oxalato Oxidasa Subfamilias según Carter, C., and Thornburg, R. W. (1999) consensus of 5670 trees (0 taxa excluded) PCGER1 PRGLP POPBEST PSGLP HA_3013 PNGLP BNGLP ATGLP1 PPABP20 PPABP19 PPE2 ATGLP3A ATGLP3B SAGLP pVGLP1 HVU3GLP SAGPPase HvGLP1 OSGLP10 OSGLP5 OSA6 ATGLP8 OSGLP4 zmEST ZMGLP1 GHGLP1 STGLP OSGLP8 ATGLP10 LEGLP NECTARIN OSA3 EGMA HA_9786 PSGER1 GM_27305 RHA801C1 MT_3891 OSGLP16 OSGLP3 HVGLP2 HVU4GLP OSRGLP1 TAGLP2A TAGLP2B OSGLP1 OSGLP2 OSGLP6 OSGER2 OSGER1 BUGLP2 ALGLP ATGLP7 OSGLP7 ZMGLP2 ATGLP12 ATGLP13B ATGLP13A ATGLP4 HVGER HVOXOA HVOXO1 HVOXOB TA2.8 TAGLP2 TAGLP3 TA38 TAGLP4 TAGLP1 LPOXO1 LPOXO2 LPOXO4 LPOXO3 MT_5623 MT_4265 MT_5759 PSGLP2A PSGLP2B GM_TC636 GM_TC635 GM_31794 MCGLP HA_0203 AT56.13 ATE6.14 ATE6.20 ATGLP9 ATE6.16 ATE6.18 ATK3K3.4 ATM12.15 ATM12.14 ATGLP6 ATM12.16 ATGLP2A ATK3K3.1 ATGLP2B ATK3K3.2 GLPs Gimnospermas Subfamilia 3 Subfamilia 2 Subfamilia 1 True Germins Subfamilia 1 Caracterización estructural y funcional de genes GLP en girasol Análisis transcriptómico y metabolómico: resistencia a Sclerotinia sclerotiorum en girasol Material Vegetal Estadio reproductivo R5.2 R5.6 Días post-inoculación D0 D2 R5.8 D4 R6 D12 Construcción de libraries substractivas de cDNA y aislamientos de secuencias ESTs Library substractiva RHA 801 (SSH) RHA 801 D2I-NI RHA 801 D2NI-I RHA 801 D4I-NI RHA 801 D4NI-I Distribución de secuencias según GO por Proceso biológico para las cuatro genotecas generadas. D2 I-NI D2 NI-I Distribución de secuencias según GO por Proceso biológico para las cuatro genotecas generadas. D4 I-NI 446 clones únicos D4 NI-I 295 clones únicos Entre 20 y 30% de la secuencias no tienen similitud con secuencias en bases de datos Identificación de ESTs de ADNc diferenciales • Entre 20 y 30% de la secuencias no tienen similitud con secuencias en bases de datos • Las colecciones del D2, presentan menor numero de clones únicos, señalando que los transcriptomas comparados tendrían mayor similitud • De a cuerdo con la anotación GO, los genes que mayormente se ven afectados por la infección son los involucrados en los procesos de síntesis de proteínas y proteólisis y en los procesos de respuesta a estrés oxidativo (peroxidasas y catalasass), proteínas de defensa y estrés (defensinas, acuoporinas, LTPs) enzimas de metabolismso primario de C. Analisis transcriptomico y metabolomico Material Vegetal Estadio reproductivo R5.2 R5.6 R5.8 R6 Días post-inoculación D0 D2 D4 D12 Plantas de Girasol de 3 líneas MR y una susceptible fueron crecidas a campo en la EEA INTA- Balcarce y en Castelar. Se las inoculó en el estadio R5.2 con una suspensión de ascoporas de 2500 ascosporas/ml. Se muestreó el material vegetal a los 0, 2, 4 y 12 post inoculación Curva del progreso de la enfermedad y calculo de AUDPC acumulativa En rojo y azul se encuentran representadas la línea RHA801 y la HA89 respectivamente. Estudios de perfiles metabólicos Extracción muestras de tejido objeto (incluyendo un estandar de masa y un estandar de tiempo de corrida) Derivatización Inyección en GC-MS. se inyectaron 2 volúmenes (100 y 200 l) debido a la alta concentración de los azucares. Identificación de metabolitos Cromatogramas iónicos totales obtenidos por la técnica de GC-MS de extractos de frutos de tomate (A), de flores de girasol (B) y de una mezcla estequiométrica de extractos de frutos de tomate y flores de girasol (C). Rutas metabólicas analizadas por GC-MS en flores de girasol R y S Perfiles metabólicos en capítulos de girasol inoculados con S. sclerotirum Análisis PCA y HCA en flores de girasol R y S en respuesta a la infección PCA y HCA de todos los perfiles metabólicos analizados en las flores de girasol. Los símbolos rojos y azules denotan las muestras de las líneas RHA801 y HA89 respectivamente. Correlación metabolito-metabolito en flores de girasol R y S Visualización de las correlaciones significativas metabolito-metabolito (p< 0.01) (izquierda) y la representación circular de las redes (derecha) en dos genotipos de girasol con comportamiento contrastante frente a S. sclerotiorum A. HA89 (susceptible) B. RHA801 (resistente). Análisis de actividad enzimática Integración de perfiles transcripcionales y metabólicos Carrari et al 2006. Plant Physiol. Vol. 142 1381-1396 Resumen resultados cambios metabólicos claves asociados a la interacción Girasol –Sclerotinia • • • • • • • Se valido la técnica de GC-MS para identificación y cuantificación de metabolitos en capítulos de girasol Se detectaron cambios significativos en metabolitos en estadios tempranos del proceso de infección en genotipos con respuesta contrastante al patógeno S. sclerotiorum El genotipo R presento cambios en niveles metabólicos significativos en los D 2y D4 post infección Los estudios concertados de metabolitos muestran patrones de regulación diferencial en genotipos R y S, presentando el genotipo R mayores interacciones intermódulo, lo que señalaría mayor concertación de determinadas rutas metabólicas Alcoholes (ononitol, glicerol, malitol), derivados de pared celular (xilosa, ramnosa, gluconato) carbohidratos como la trealosa. Los metabolitos relacionados con la producción de polifenoles (cafeato y clorogenato), presentan mayores cambios en el genotipo S . Metabolitos asociados con removilización de N (aumento de prolina) se han detectado en el genotipo S Grupo de trabajo Grupo genómica de girasol, IB INTA Castelar Paula Fernández Verónica Lia Lucila Peluffo Corina Fusari Sebastian Moschen Jeremías Zubrzycki Carla Filippi Federeico Ehrenbolger Veromica Nishinakamasu Norma Paniego Ruth Heinz Esteban Hopp MUCHAS GRACIAS