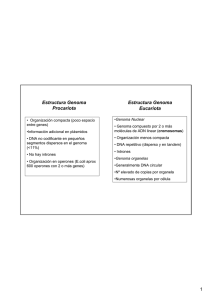
Estructura Genoma Eucariota Estructura Genoma Procariota
Anuncio

Estructura Genoma Procariota Estructura Genoma Eucariota • Organización compacta (poco espacio entre genes) • Genoma compuesto por 2 o más moléculas de ADN linear (cromosomas) •Información adicional en plásmidos • DNA no codificante en pequeños segmentos dispersos en el genoma (<11%) • No hay intrones • Organización menos compacta • DNA repetitivo (disperso y en tandem) • Intrones • Organización en operones (E.coli aprox 600 operones con 2 o más genes) 1 Estructura Genoma Procariota • DNA genómico Æ Nucleoide : empaquetamiento en forma organizada •Molécula de DNA circular única (80%) + Proteínas básicas asociadas Super-enrollamiento (agregado de giros al DNA) •DNA girasa (topoisomerasa II) •DNA topoisomerasa I 2 Nucleoide • Organización de dominios : aprox. 400 dominios: Loops super-enrollados (1040kb) •DNA genómico unido a un “core” proteico a partir del cual radian los loops •Proteínas asociadas: DNA girasa, DNA topoisomerasa I, más al menos 4 proteínas Æ HU la más abundante (Tetrámeros) 3 Variaciones en Procariotas •Algunos genoma linear Ej: Borrelia burgdorferi •Algunos genomas de tamaño mayor Ej. B.megaterium (30 x 106pb) •DNA circular o linear subgenómico Æ genoma multipartito (Brivio cholera, Borrelia) •ArchaeÆ empaquetamiento con proteínas similares a Histonas 4 Genoma eucariota nuclear • Moléculas de DNA linearÆ cromosomas 5 Características de los cromosomas • Centrómero: – DNA centromérico: secuencias repetitivas • humanos DNA alfoide: repeticiones de 171pb (1x106pb) • Levaduras : sec.mínima 125pb – Proteínas unidas al centrómero • humanos al menos 7 proteínas, una de ellas CENP-A reemplaza a histona H3 – Cinetocoro: unión de microtúbulos 6 7 •Telómeros – Indica el extremo de los cromosomas y los protege • Evita recombinación entre cromosomas • Evita la unión de extremos de cromosomas – Replicación completa de los cromosomas – Organización funcional de cromosomas en el Núcleo –DNA repetitivo •Humanos: cientos de copias de la secuencia – 5’-TTAGGG-3’ –Extensión del extremo 3’ como simple cadena –Proteínas teloméricas: •TRF1 (regula el largo de los telómeros) •TRF2 (mantiene la extensión de DNA simple cadena, T loop) 8 9 Cromatina : 50% DNA + 50% Proteínas básicas (Histonas) 1er nivel de organización: NUCLEOSOMAS ADN: aprox 200pb • ADN asociado al core 146 pb • ADN linker 10-90 pb 10 Histonas core: H2A H2B H3 H4 Histonas linker: H1 a-e, H1º, H1t y H5 11 12 Metilación y Acetilación: amino libre de Lisina Metilación: en Arginina y Histidina Fosforilación: grupos OH de Serina y Histidina 13 Modificaciones de Histonas • Modificaciones: – Acetilación/ Deacetilación de residuos Lys (HAT / HDAC) • neutralización de cargas positivas de Histonas – Metilación de Lys y Arg – Fosforilación de Ser y Thr • mitosis y meiosis: condensación de cromosomas – Ubiquitinación – ADP ribosilación • Variantes de Histonas – Reemplazan a H3 o H2A en algunos nucleosomas • CENP-A en centrómeros 14 Nucleosomas y fibra de 30nm 15 •Unión del DNA a la matriz nuclear : MAR (sec. ricas en AT) •Dominios estructurales y funcionales Dominios estructurales •Loops de ADN Dominios funcionales Loops de ADN 40100kb •Regiones sensibles a DNasa •Límites: regiones aislantes, 1-2kb (insulator) 16 Empaquetamiento en cromosomas Mitóticos 17 18 Heterocromatina /Eucromatina Heterocromatina •Principalmente DNA repetitivo (Centrómeros, Telómeros, Transposones) •Baja densidad génica •Densamente empaquetada •Sin sitios HS •Ordenamiento regular de nucleosomas •Replicación tardía en fase S Eucromatina •Sensibilidad a nucleasas •Alta densidad génica •DNA potencialmente activo transcripcionalmente •Histonas acetiladas •Metilación Lys-4 H3 •DNA no metilado •Crossing-over reducidos 19 • Heterocromatina: Modificaciones covalentes características – Histonas desacetiladas – Metilación Lys-9 H3 (unión prot HP-1) – Metilación DNA (CpG) • Heterocromatina constitutiva – Grandes regiones principalmente secuencias repetitivas • Telómeros, centrómeros, transposones • Heterocromatina Facultativa – Regiones genómicas empaquetadas en algunos grupos de células o en 1 solo cromosoma homólogo • Cromosoma X 20 Cromosomas sexuales Cromosoma Y Cromosoma X •1000-2000 genes •La mayor parte de los genes sin contraparte en el Y (solo 9 genes descriptos) •Inactivación de un cromosoma •Pequeña reg de homología con cromosoma X •95% no autosomal (seq transpuestas de X, seq de X degeneradas, Amplicon (8 bloques palindrómicos) •Pocos genes (aprox 156, la mitad pseudogenes) •60 genes específicos de macho que se expresan en testículo (gen SRY) 21 Heterocromatina Facultativa Inactivación del cromosoma X 22 23 Inactivación del cromosoma X Modificación del estado de la cromatina (actividad de acetilasas, metilasas). Inicio en un locus específico Xic y progresión a todo el cromosoma. Sitio de iniciación Xic: produce un ARN no traducido ARN Xist. ARN Xist: permanece unido al ADN, recubrimiento del cromosoma Xi. Señal para el inicio de modificaciones de la cromatina (hipoacetilación, metilación del ADN) Gen Tsix: Transcripto antisentido Regulador negativo de Xist: Regula la elección del cromosoma X que permanece activo Región Xpr: sensado del número de cromosomas X. Coordina la expresión reciproca de Xist/ Tsix Metilación del ADN, bajos niveles de acetilación de Histonas, bajos niveles de metilación de Lys-4 de histona H3, y altos niveles de metilación de Lys-9 de histona H3, asociados a silenciamiento génico. Adicionalmente, los nucleosomas del Xi presentan una variante de histona llamada macroH2A. 24 Imprinting Genes cuya expresión está determinada por su origen parental. Expresión exclusiva del alelo materno por imprinting del paterno o viceversa Asimetría en la función de los genomas parentales Mecanismo de imprinting: Iniciado durante gametogénesis en forma diferencial en espermatogénesis y ovogénesis. Genes “imprinted” en general agrupados, regulación coordinada a partir de centros IC que determinan regiones de metilación diferencial DMR Metilación del ADN en CpG en los promotores de los genes Æ inactivación Demetilación Æ ARN antisentidoÆ inactivación Formación de células germinales primordiales: Remoción de todo imprinting Los patrones de metilación genómica de ovocitos y espermatozoides depende de la actividad de ADN metiltransferasas Dnmt 25