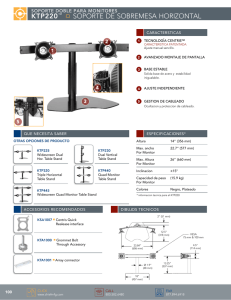
Regresión log´ıstica y cangrejos herradura
Anuncio

Regresión logı́stica y cangrejos herradura
11 de enero de 2009
Empezamos leyendo los datos.
> table.4.3 = read.table("..//data/crab.txt", col.names = c("C",
+
"S", "W", "Sa", "Wt"))
La variable respuesta que nos interesa es si las dichosas hembras de los cangrejos
herradura (cuya fidelidad no es precisamente la mayor de sus virtudes) tienen, al
menos, un satélite. Como predictor solamente vamos a utilizar la anchura
del caparazón. Construimos la variable a partir de la variable original que nos da
el número de satélites.
> (table.4.3$Sa.bin = ifelse(table.4.3$Sa > 0, 1, 0))
[1]
[38]
[75]
[112]
[149]
1
1
0
1
1
0
0
0
0
1
1
1
1
1
1
0
1
1
1
0
1
1
0
0
0
0
1
1
1
0
0
1
0
0
0
0
1
0
1
1
0
1
0
1
0
0
1
1
1
1
0
1
0
1
0
0
1
1
0
1
1
1
0
1
1
0
1
0
1
1
1
1
0
0
1
1
1
0
1
0
1
1
1
1
1
1
1
0
1
0
0
1
0
1
0
1
1
0
1
1
1
1
0
0
1
1
0
1
1
1
1
0
0
1
0
1
0
1
1
0
1
1
1
0
0
0
1
1
1
1
1
1
1
1
1
1
1
0
1
1
1
0
1
1
1
1
0
1
1
0
0
1
1
0
1
1
0
1
1
1
0
Ya podemos ajustar el modelo de regresión logı́stica con la maravillosa función
glm.
> (crab.fit.logit = glm(Sa.bin ~ W, family = binomial, data = table.4.3))
Call:
glm(formula = Sa.bin ~ W, family = binomial, data = table.4.3)
Coefficients:
(Intercept)
-12.3508
W
0.4972
Degrees of Freedom: 172 Total (i.e. Null); 171 Residual
Null Deviance:
225.8
Residual Deviance: 194.5
AIC: 198.5
Construimos una variable que discretiza los valores originales de la variable W que
nos da la anchura del caparazón.
> table.4.3$W.fac = cut(table.4.3$W, breaks = c(0, seq(23.25, 29.25),
+
Inf))
Calculamos la proporción de cangrejos que tienen al menos un satélite para cada
grupo de anchura de caparazón. En definitiva estamos estimando la probabilidad
del valor uno en la variable respuesta.
> prop = aggregate(table.4.3$Sa.bin, by = list(W = table.4.3$W.fac),
+
mean)$x
1
1
1
1
1
0
0
1
1
0
1
1
1
Calculamos la anchura media del caparazón en cada grupo de anchura de caparazón.
> plot.x = aggregate(table.4.3$W, by = list(W = table.4.3$W.fac),
+
mean)$x
Ponemos los ejes y las etiquetas.
plot(y = table.4.3$Sa.bin, x = table.4.3$W, xlab = expression(paste("Width, ",
italic(x), "(cm)")), ylab = expression(paste("Proportion having satellites,",
{
pi
}, "(x)")), axes = F, type = "n")
axis(2, at = seq(0, 1, 0.2))
axis(1, at = seq(20, 34, 2))
lines(y = prop, x = plot.x, pch = 16, type = "p")
ind = order(table.4.3$W)
lines(x = table.4.3$W[ind], y = predict(crab.fit.logit, type = "response")[ind],
type = "l", lty = 3)
0.8
0.6
0.4
0.2
0.0
Proportion having satellites,π(x)
1.0
>
+
+
+
+
>
>
>
>
>
+
22
24
26
28
30
32
34
Width, x(cm)
Veamos un resumen del ajuste. Los errores estándar están basados en la distribución asintótica. En la columna etiquetada z value tenemos el estadı́stico de
Wald.
> summary(crab.fit.logit, correlation = F)
Call:
glm(formula = Sa.bin ~ W, family = binomial, data = table.4.3)
Deviance Residuals:
Min
1Q
Median
-2.0281 -1.0458
0.5480
3Q
0.9066
2
Max
1.6941
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -12.3508
2.6287 -4.698 2.62e-06 ***
W
0.4972
0.1017
4.887 1.02e-06 ***
--Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 225.76
Residual deviance: 194.45
AIC: 198.45
on 172
on 171
degrees of freedom
degrees of freedom
Number of Fisher Scoring iterations: 4
El estadı́stico del cociente de verosimilitudes de que el coeficiente es nulo lo
podemos obtener como
> crab.fit.logit$null.deviance - crab.fit.logit$deviance
[1] 31.30586
Ası́ como los intervalos de confianza para los parámetros.
> library(MASS)
> confint(crab.fit.logit)
2.5 %
97.5 %
(Intercept) -17.8100090 -7.4572470
W
0.3083806 0.7090167
Estimamos las probabilidades de que la respuesta sea igual a uno.
> crab.predict = predict(crab.fit.logit, type = "response", se = T)
Representamos los intervalos de confianza.
>
>
+
+
>
>
>
+
>
+
ind = order(table.4.3$W)
plot(table.4.3$W[ind], crab.predict$fit[ind],
xlim = c(20, 33), ylab = "Probabilidad de
italic(x), "(cm)")))
axis(2, at = seq(0, 1, 0.2))
axis(1, at = seq(20, 32, 2))
lines(table.4.3$W[ind], crab.predict$fit[ind]
lty = 3)
lines(table.4.3$W[ind], crab.predict$fit[ind]
lty = 3)
3
axes = F, type = "l",
satélite", xlab = expression(paste("Anchura,
- 1.96 * crab.predict$se[ind],
+ 1.96 * crab.predict$se[ind],
Probabilidad de satélite
0.2
0.4
0.6
0.8
1.0
20
22
24
26
28
Anchura, x(cm)
4
30
32