33466784.B-gal biologica II 2011
Anuncio

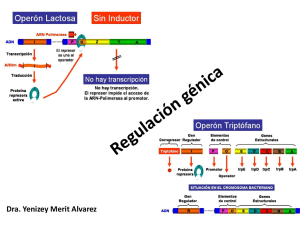
TPNº 1 – Química Biológica II – 2011 Regulación de la Expresión Genética INTRODUCCION En las Clases Teóricas hemos aprendido sobre los genes, cómo se replican y cómo se transmiten. También hemos visto que la información contenida en los genes se transcribe a ARN mensajeros y luego es traducida a proteínas. Además, la célula regula su funcionamiento, pero… ¿Cómo decide la célula que proteínas necesita producir en cada momento y qué cantidad de cada proteína es necesario sintetizar? En los organismos pluricelulares con diferentes órganos y tejidos está claro que existe un reparto de las funciones, de manera que las células de diferentes tejidos llevan a cabo distintas misiones y para ello necesitan sintetizar diferentes tipos de proteínas. De forma que un hepatocito (célula del hígado) produce una colección de proteínas diferentes a las de una neurona. Por consiguiente, en los diferentes tejidos de un individuo pluricelular adulto, se están expresando distintos grupos de genes. En los organismos unicelulares, como las bacterias, también es necesario regular la expresión de los genes adaptándola a las necesidades ambientales. Es importante para la economía celular que la expresión de los genes sea regulada según las circunstancias celulares. Un buen ejemplo de esta situación en bacterias es la regulación de las enzimas implicadas en el metabolismo de los azúcares. Las bacterias pueden emplear para obtener energía distintas fuentes de carbono, como la glucosa, lactosa, galactosa, maltosa, ramnosa y xilosa. Existen proteínas transportadoras cada uno de estos azúcares en la bacteria y enzimas capaces de metabolizarlos para obtener energía. Lógicamente, sería un derroche energético producir simultáneamente todas las enzimas necesarias para metabolizar los diferentes azúcares mencionados. Por consiguiente, es mucho más económico para la célula producir solamente las enzimas necesarias en cada momento. Por ejemplo, si en el medio en el que vive la bacteria la principal fuente de carbono es la lactosa, solamente se expresan los genes necesarios para metabolizar dicho azúcar, mientras que los otros genes no se expresan. Por lo tanto, es esencial que existan mecanismos de regulación de la expresión génica, de manera que los genes se expresen cuando sean necesarios. Dentro de ellos podemos distinguir en las bacterias dos categorías principales: Mecanismos de regulación asociados a cambios genotípicos. Dentro de esta categoría podemos incluir a: I) variaciones hereditarias no asociadas con transferencia de material genético, las cuales corresponden a diversos tipos de mutaciones; II) variaciones hereditarias asociadas con transferencia de material genético entre una bacteria donante y otra receptora, las cuales se producen por transformación, conjugación o transducción. Mecanismos de regulación para la adaptación al medio: son cambios moleculares y fenotípicos que ocurren sin modificación del material hereditario (sin variación del genotipo). Dichos mecanismos pueden darse por regulación a nivel de la transcripción o de la traducción, regulando la síntesis de proteínas. Uno de los mecanismos mejor conocido es la regulación a nivel de la transcripción. A continuación mencionaremos algunos conceptos importantes sobre este tipo de regulación. Algunos procesos metabólicos son necesarios para el funcionamiento normal de las células. Por consiguiente, los genes que codifican para las enzimas necesarias para ese metabolismo celular básico se están expresando continuamente, es decir, se expresan de forma constitutiva o continua. Por tal motivo, a este tipo de genes se los denomina genes constitutivos. Esto no significa que la actividad de los genes que se están expresando continuamente no esté regulada, simplemente están sometidos a un tipo de regulación diferente que hace que se estén expresando siempre. Los genes constitutivos codifican para sistemas enzimáticos constitutivos, que se necesitan siempre para la actividad normal de la célula. Por otro lado, existen genes que se expresan solamente en determinadas situaciones y que, por consiguiente, codifican para enzimas que solamente se necesitan en momentos concretos. A este tipo de genes se les llama genes adaptativos y a las enzimas codificadas por ellos, sistemas enzimáticos adaptativos. Se denominan así pensando en que se expresan cuando la célula se adapta a una determinada situación ambiental. En algunos libros de texto se denomina a este tipo de genes, “genes regulados”, sin embargo, esta denominación no es demasiado buena, ya que así parece que son los únicos genes cuya expresión está regulada. 1 Sistemas Inducibles y Sistemas Represibles Sistemas inducibles: son aquellos en los cuales la presencia del sustrato de la enzima provoca la síntesis de la misma. A este efecto del sustrato se le denomina inducción positiva. El compuesto que desencadena la síntesis de la enzima se denomina inductor. La mayoría de los sistemas inducibles se corresponden con procesos catabólicos, por ejemplo, el operón lactosa, el operón arabinosa, el operón maltosa. Se trata de sistemas enzimáticos encargados de degradar la lactosa, arabinosa, maltosa, etc. Sistemas represibles: cuando el producto final de la reacción que cataliza la enzima impide la síntesis de la misma. Este fenómeno recibe el nombre de inducción negativa. Al compuesto que impide la síntesis de la enzima se lo denomina correpresor. Los sistemas represibles se corresponden con procesos de síntesis o anabolismo, por ejemplo el operón triptófano y el operón histidina. Se trata de las rutas metabólicas que conducen a la síntesis de triptófano y a la síntesis de histidina. Elementos del Operón Jacob, Monod y colaboradores analizaron el sistema genético para el metabolismo de la lactosa en Escherichia coli, de manera que los resultados de sus estudios permitieron establecer el modelo genético del operón que permite comprender como tiene lugar la regulación de la expresión génica en bacterias. Jacob y Monod recibieron en 1965 el Premio Nobel por estas investigaciones. “Operón es un grupo de genes estructurales cuya expresión está regulada por los mismos elementos de control (promotor y operador) y los mismos genes reguladores”. Los principales elementos que constituyen un operón son los siguientes: • • • • Los genes estructurales: llevan información para la síntesis de los polipéptidos. Se trata de los genes cuya expresión está regulada. Los operones bacterianos suelen contener varios genes estructurales consecutivos, se los denomina poligénicos o policistrónicos. Pero existen algunos operones bacterianos que tienen un solo gen estructural. En su mayoría los operones eucarióticos contienen un sólo gen estructural, denominados monocistrónicos. El promotor (P): es una región del ADN con una secuencia que es reconocida por la ARN polimerasa para comenzar la transcripción, y a su vez es un elemento de control. Se encuentra antes de los genes estructurales. Abreviadamente se lo designa con la letra P. El operador (O): se trata de otro elemento de control, es una región del ADN con una secuencia que es reconocida por la proteína reguladora. El operador se sitúa normalmente entre la región promotora y los genes estructurales. Abreviadamente se le designa por la letra O. El gen regulador (i): secuencia de ADN que codifica para la proteína reguladora que reconoce la secuencia de la región del operador. El gen regulador por lo general está cerca de los genes estructurales del operón, pero no inmediatamente al lado. Abreviadamente se le denomina gen i. Elementos de control Elementos del Operón en bacterias. Moléculas difusibles Genes Estructurales Gen regulador Promotor Operador Proteínas reguladoras Efectores Inductores Codifican para polipéptidos Codifica para proteína reguladora La regulación de la expresión génica más frecuente es la que involucra el inicio de la transcripción. Las proteínas reguladoras o factores de transcripción influencian la unión de la ARN-polimerasa a los distintos promotores y ejercen control induciendo o reprimiendo la transcripción luego de unirse a secuencias específicas. Los promotores de genes distintos varían en su secuencia y esto se refleja en un mayor o un menor nivel de la transcripción. 2 Control Positivo y Control Negativo Control positivo: se dice que un sistema está bajo control positivo cuando el producto del gen regulador activa la expresión de los genes, es decir actúa como un activador. Control negativo: se dice que un sistema está bajo control negativo cuando el producto del gen regulador reprime o impide la expresión de los genes, es decir actúa como un represor. EL OPERÓN LACTOSA: Sistema Inducible y de Control Negativo El operón lactosa, que abreviadamente se denomina operón lac, es un sistema inducible que está bajo control negativo, es decir que la proteína reguladora (producto del gen i), es un represor que impide la expresión de los genes estructurales en ausencia del inductor. El inductor del sistema es el sustrato, la lactosa. Los genes estructurales del operón lactosa son los siguientes: • • • El gen Z+: codifica para la β-galactosidasa que cataliza la hidrólisis de la lactosa en glucosa y galactosa. El gen Y+: codifica para la galactósido permeasa que transporta β-galactósidos al interior de la célula bacteriana. El gen A+: codifica para la tiogalactósido transacetilasa que cataliza la transferencia del grupo acetil del acetil Coenzima A al 6-OH de un aceptor tiogalactósido. Este gen no está relacionado con el metabolismo de la lactosa. En los estudios del operón lactosa se utiliza como inductor un análogo sintético de la lactosa que es el isopropil tiogalactósido (IPTG). El IPTG tiene la ventaja que no necesita ser transportado por la galactósido permeasa para entrar en la bacteria y por otro lado no es metabolizado por la enzima, por lo que actúa como inductor permanente. El operón lac en E. coli es inducible, de manera que en ausencia de lactosa (inductor), la proteína represora producto del gen i se encuentra unida a la región operadora, impide la unión de la ARN-polimerasa a la región promotora y, como consecuencia, no se transcriben los genes estructurales (Fig 1). Fig 1: Estado del operón lactosa en ausencia de inductor. 3 La presencia del inductor (lactosa) hace que se expresen los genes estructurales del operón, necesarios para metabolizar la lactosa (Fig 2). La lactosa se une a la proteína reguladora, la que cambia su conformación y se suelta de la región operadora dejando acceso libre a la ARN-polimerasa para que se una a la región promotora y se transcriban los genes estructurales. También es conveniente notar que los tres genes estructurales del operón lactosa se transcriben juntos en un mismo ARNm, es decir que los ARN mensajeros de bacterias suelen ser policistrónicos, poligénicos o multigénicos. Sin embargo, en eucariontes los mensajeros suelen sen monocistrónicos o monogénicos, es decir, corresponden a la transcripción de un solo gen estructural. En el trabajo práctico de expresión y purificación de proteínas recombinantes veremos como la regulación del operon lac sirve para controlar la expresión de proteínas de una manera muy eficiente. Fig 2: Estado del operón lactosa en presencia del inductor. OBJETIVO DEL TRABAJO PRACTICO En el transcurso del Trabajo Práctico determinaremos la transcripción (o expresión) de dos operones de la bacteria E. coli mediante una estrategia muy utilizada, en la cual la secuencia de ADN correspondiente al promotor del gen a estudiar se fusiona con los genes estructurales del operón lactosa. Esta técnica permite seguir la expresión del gen en estudio, determinando la actividad β-galactosidasa, la cual es cuantificable mediante un ensayo sencillo y sensible. La actividad β-galactosidasa es usualmente ensayada por un método espectrofotométrico, en el cual la enzima hidroliza a un sustrato sintético cromogénico (homólogo a la lactosa), el ONPG (o-nitrofenil-β-D-galactopiranosil-D-glucosa), el cual al hidrolizarse da o-nitrofenol, compuesto de color amarillo cuya absorbancia se lee a 420 nm. Este método fue descripto por Miller en el año 1992. Se determinará la expresión del operón sdhCDAB que codifica para la enzima del ciclo de Krebs, succinato deshidrogenada (Fig 3), esta enzima tiene dos subunidades catalíticas (SdhA, SdhB) más dos subunidades de membrana (SdhC, SdhD). Además se determinará la expresión del operón copA que codifica para la enzima CopA, presente en la membrana de la célula bacteriana, y transporta de manera activa el cobre a través de la membrana, con hidrólisis de ATP (Fig 4). 4 Operon sdh CDAB P O P O sdhD sdhC sdhA sdhB lacY lacA A lacZ B Fusión sdh- lacZ Fig 3: A) Esquema del operón sdhCDAB y B) de la fusión del promotor sdh con el operón lac. P O P O copA A lacZ lacY lacA B Fig 4: A) Esquema del operón copA y B) de la fusión del promotor copA con el operón lac. PARTE EXPERIMENTAL Se utilizarán cultivos de las cepas bacterianas que contienen las fusiones de cada uno de los promotores con el operón lac. Las bacterias crecerán durante 2 horas a 37 ºC en medio rico, LB (Luria Broth), con agitación y luego cada cultivo se dividirá en dos. Una mitad se incubará durante una hora con sulfato de cobre 500 μM y la otra mitad se usará como control sin cobre. Transcurrido el tiempo de incubación se determinará la actividad βgalactosidasa en cada fracción. Cepas de E. coli a utilizar: TSDH00 (MC4100 λ[Φ (PsdhC-lacZ)]) (contiene la fusión del promotor sdh con el sistema lac). WOII260E (BW25113 λ[Φ (PcopA-lacZ)]) (contiene la fusión del promotor copA con el sistema lac). Medición de la actividad β-galactosidasa En primer lugar se determina la absorbancia del cultivo a 560 nm (la cual debe estar entre 0,3 y 0,8), si esto no es así se deben realizar las diluciones necesarias para obtener dicha absorbancia. Luego, en un tubo se agrega: • 900 µl de buffer Z + • 100 µl del cultivo bacteriano (A560nm entre 0,3-0,8) 5 • • 15 µl de cloroformo 15 µl de SDS (dodecilsulfato sódico) al 0,1% Se agita vigorosamente y se incuba 15 min a temperatura ambiente • Luego se adiciona 200 µl de ONPG (4 mg ml -1) Se incuba a 28°C hasta la aparición del color amarillo (IMPORTANTE: medir este tiempo con cronómetro). Si la reacción es lenta, se incuba durante un período máximo de 20 min. • La reacción se detiene adicionando 500 µl de Na 2CO3 1 M que desnaturaliza a la enzima y aumenta la intensidad del color. Se determina la absorbancia a 420 y a 550 nm. El buffer Z tiene altas concentraciones de β-mercaptoetanol el cual protege a la enzima cuando es liberada al medio cuando las células se permeabilizan por el agregado de cloroformo y el SDS. La reacción se detiene por la desnaturalización de la enzima por el agregado del Na2CO3. La actividad enzimática se expresa como UNIDADES MILLER (UM) y se calcula según la siguiente fórmula. A420 − (1,75 x A550) UM= x 1000 T x V x A560 Donde T es el tiempo de reacción en min, V es el volumen de cultivo ensayado en ml (0,1 en nuestro caso). 1,75 es el factor que corrige la absorbancia a 550 nm (A 550). El uso de ésta fórmula permite normalizar la actividad enzimática para la absorbancia del cultivo (A 560) y para la absorbancia residual después de la permeabilización (A 550). Soluciones para determinar actividad de β-galactosidasa - ONPG 4 mg ml-1 en agua bidestilada (guardado en heladera). - Na2CO3 1 M en agua bidestilada (guardado en heladera). -Buffer Z: Na2HPO4 0,06 M; NaH2PO4.2H2O 0,04 M; KCl 0,01 M; MgSO 4.7H2O 0,001 M; βmercaptoetanol 0,05 M, pH 7. - SDS 0,1 %. - Cloroformo. 6