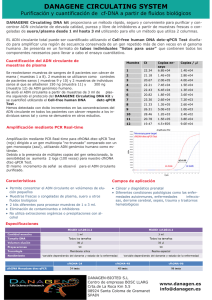
Superenrrollamiento
Anuncio
ADN y RNA caracterización y métodos de estudio Separación por centrifugación de equilibrio en gradiente de densidad de en CsCl Separación por centrifugación de equilibrio en gradiente de densidad de en CsCl 2 DNAs, uno lineal y otro circular, de igual composicion, tienen distinta densidad?? δ Electroforesis en gel de agarosa Buffer: TBE = Tris Borate EDTA o TAE tris Acetato Concentración: 0.7% a 2%. 0.7% DNA grandes (5–10kb) 2% fragmentos pequeños (0.2–1kb). Para muy pequenos = polyacrylamide gel Revelado Estimación PM con uso de patrones Superenrrollamiento (ADN circular) Superenrrollamiento (ADN circular) Lk (numero de enlace) = Tw + Wr desenrollo Tw = número de vueltas de la hélice Wr = grado de retorcimiento o superenrrollamiento Lk define la topología del ADN. Para acomodar del desenrrollamiento, el DNA se retuerce -2 vueltas. El superenrrollamiento (-) (o righthanded) introduce una tensión que favorece el desenrrollamiento Tw= 12 vueltas Wr= 0 vueltas Lk=12 vueltas Tw= 14 vueltas Wr= -2 vueltas Lk=12 vueltas Lk solo cambia por ruptura de un enlace fosfodiester (topoisomerasas) Los agentes intercalantes (bromuro de etidio) desenrollan el ADN introducen superenrrollamientos (+) expuestos diferentes tiempos a topoisomerasas El grado de superenrrollamiento afecta la densidad del ADN Ni la sedimentación con ClCs ni la electroforesis en gel de agarosa puede discernir entre ADN superenrollado (+) o (-) Obtención de DNA plasmídico ADN genómico: lineal (o circular) gran tamaño lenta renaturalizacion menor estabilidad ADN plasmídico: generalmente circular(superenrrollado) pequeño tamaño facil renaturalizacion mayor estabilidad Obtención de DNA plasmídico ADN genómico: adherido a la membrana esencial para la supervivencia y normal funcionamiento ADN plasmídico: libre resistencia a antibióticos, producción de toxinas, etc. Obtención de DNA plasmídico Lisis alcalina: buffer + EDTA (quelante de Ca+2 y Mg+2) 0.2M NaOH + SDS 1% (pH 12.0-12.5) Ruptura de membrana celular Desnaturalización selectiva de DNA genómico y proteínas Lisis de RNA Neutralización con alta [sales]: AcK 3M Renaturalización y agregado insoluble de DNA genómico Precipitación de complejos SDS-proteínas K-SDS-membrana y RNA de alto peso molecular: Los 3 mayores contaminantes macromoleculares co-precipitan y pueden ser removidos por ultracentrifugación EDTA quelante de iones y desestabilizante de membrana Obtención de DNA plasmídico Concentrado del DNA plasmídico: Precipitación con alcohol y bajas temperaturas Eliminación de sales y electrolitos pequeños: Lavado con etanol Electroforesis en gel de agarosa Detección con bromuro de etidio Técnicas basadas en la renaturalización del DNA Southern blot (DNA) Se usan enzimas de restricción para cortar el ADN en pequeños fragmentos. Se realiza una electroforesis en gel de agarosa para separar por tamaño. Si algunos de los fragmentos quedaron grandes (> 15 kb), se trata ton HCl para despurinizar y romperlo en pedazos mas chicos para que sea mas eficiente la transferencia del gel a la membrana. Se trata el gel con NaOH para desnaturalizar el dsADN, esto favorece la unión a la membrana y destruye cualquier ARN residual Southern blot (DNA) Se coloca una hoja de nitrocelulosa o nylon sobre el gel, presionando. El ADN con carga (-) se une a la membrana con carga (+) Calentar la membrana a 80 °C por 2 horas o exponerla a UV para fijar el ADN. Exponer la membrana a la sonda radiactiva o fluorescente para hibridizar. Para asegurar la union específica de la sonda, se usa ADN de salmon para bloquear la superficie de la membrana. Se lava la sonda con buffer y se revela la membrana Southern blot (DNA) Northen blot (RNA) Hibridización de ADN Secuenciación: Método de Sanger (1975) Secuenciación automática