al DNA
Anuncio
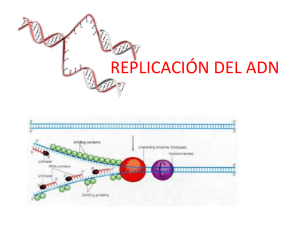
¿Cuál de estas 3 posibilidades de replicación es la que ocurre? Marcaje del DNA bacteriano con N15 Experimento de Meselson-Stahl (1958) 1. Bacteria crecida en N14 2. Bacteria crecida en N15 3. Incubación en presencia de N14 4. Centrifugación de generaciones sucesivas CONCLUSIÓN: La Replicación es semi-conservativa La replicación es catalizada por: DNA polimerasa • Las DNA polimerasas añaden dNTPs complementarios a una cadena molde • Se necesita un 3’OH libre para que las DNA polimerasas actúen (cebador) • Las DNA polimerasas requieren Mg2+ como cofactor Se produce un ataque nucleofílico del 3’OH al fosfato del dNTP entrante Reacción básica de la replicación de DNA DNA polimerasa (DNA)n+1 + PPi DNAn + dNTP Mg2+ dNTP DNAn molde PPi (DNA)n+1 ¿Dónde comienza la replicación? ¿En una molécula circular de doble cadena, en qué sentido ocurre la replicación? ¿Además de la polimerasa hay otras moléculas involucradas en replicación? ? La REPLICACIÓN comienza en una secuencia llamada ORIGEN y es BIDIRECCIONAL Hay DOS Horquillas de replicación en sentido opuesto • Cada cadena de DNA original sirve de molde para una cadena nueva • Los precursores son desoxiribonucleósidos 5’ TRIfosfato (dNTPs: dATP, dTTP, dGTP, dCTP) • Las cadenas de DNA originales se separan y se sintetiza la complementaria de cada una de manera simultánea • La replicación comienza en una secuencia llamada Origen •La DNA polimerasa requiere de un cebador que proporcione el 3’OH para la catálisis •Se requiere la función de otras enzimas además de la DNA polimerasa durante la replicación Origen de Replicación bacteriano OriC En bacteria hay un solo origen de replicación Secuencias repetidas en tandem (13 pb) Cajas DnaA (9 pb) 1. Activación por metilación de A en GATC 2. Unión de DnaA “abre el DNA” 3. Unión de DnaB y DnaC- actividad helicasa ATP dependiente 4. Unión de SSB para mantener separadas las cadenas de DNA DNA B (helicasa) Rompe los puentes de hidrógeno entre las bases y separa las dos hebras La HELICASA es un hexámero que utiliza ATP para romper puentes de hidrógeno en el DNA Proteínas de unión a cadena sencilla (SSB) mantienen las hebras separadas La unión de SSB es cooperativa y ayuda a la polimerasa facilitando su actividad 5. Síntesis de Cebador de RNA 6. Polimerización de DNA 1. Primasa 2. Polimerasa RNA DNA polimerasas de bacterias Exonucleasa Pulgar Palma La estructura de las polimerasas asemeja un mano que agarra el DNA y lo sujeta firmemente mientras va tejiendo la nueva hebra Actividad de polimerasa 5’ 3’ Actividad de exonucleasa 3’ 5’ La fidelidad de la DNA polimerasa III es de 109 El cromosoma bacteriano tiene DOS horquillas de replicación activas Cada horquilla tiene una cadena que crece de manera continua y otra discontinua Ambas cadenas crecen en dirección 5’ 3’ Esquema de la horquilla que va en contra-sentido de las manecillas del reloj Hebra “guía” Fragmentos de Okazaki Hebra “retrasada” El crecimiento de ambas cadenas es en sentido 5’ 3’ En cada horquilla de replicación, la DNA pol III es la que replica las dos hebras al mismo tiempo. La replicación de la hebra retrasada se interrumpe cada 1000 nt aproximadamente Subunidades de la DNA polimerasa III de E. coli Sub # por holoenzima Mr Función 2 132,000 Actividad polimerasa 2 27,000 Exonucleasa 3’5’ 2 10,000 Se requiere para la union de DnaB 2 71,000 1 52,000 1 35,000 ’ 1 33,000 1 15,000 1 12,000 4 37,000 Unión estable al molde, dimerización del núcleo Complejo que carga las subunidades al DNA Pinzas que forman una rueda (abrazadera) sobre el DNA y aseguran óptima procesividad Núcleo de la polimerasa Modelo del dímero La DNA pol III es altamente procesiva gracias a las subunidades que forman una abrazadera El complejo γ monta a la abrazadera sobre el DNA en cada hebra proteínas (abrazadera) complejo La DNA polimerasa III es muy procesiva gracias a la abrazadera (subunidades β) Video DNA polimerasas en E. coli Pol I Pol II pol III gen pol A polB polC PM (kDa) 103 90 130 mol/cél. 400 100 10 Vmax 20 nt/s 3 nt/s 750 nt/s 3' exonucl. + + + 5' exonucl + no no 10000 500000 procesiv. 3-200 DNA polimerasa I La DNA pol I rellena los espacios entre fragmentos de Okazaki Utiliza actividad exonucleasa 5’3’ para eliminar cebador de RNA DNA ligasa La DNA ligasa sella un enlace fosfodiester cuando ya no falta ningún nulceótido por añadir La replicación causa superenrollamiento Las topoisomerasa Girasa alivia la tensión Terminación de la replicación Topoisomerasa IV participa en la terminación de la replicación de moléculas circulares. Cómo se asegura la célula que su DNA se replica solo una vez? Patrones de metilación del DNA Las enzimas Dam metilasas regulan el inicio en el origen de replicación