Diapositiva 1 - Genética y Biología Molecular
Anuncio

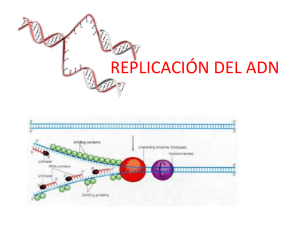
Objetivos del tema El alumno... V. METABOLISMO DEL DNA Revisará los diferentes procesos metabólicos en los que el DNA es la molécula central, i.e., la replicación, la reparación y la recombinación. Describirá la enzimología de los diferentes procesos y destacará la importancia de la fidelidad de la replicación y de la necesidad de conservar la estructura y función del DNA. 1. Replicación del DNA 1.1. Comprenderá la importancia de los eventos que rigen la duplicación del material genético. 1.2. Describirá el mecanismo semiconservativo de replicación de DNA basado en la estructura de la doble hélice. 1.3. Asociará la replicación semidiscontinua del DNA con la estructura de la doble hélice. 1.4. Distinguirá entre la cadena continua y discontinua en el DNA molde. 1.5. Explicará la función de un origen de replicación. 1.6. Describirá las características generales mínimas de un origen de replicación. 1.7. Distinguirá entre un origen de replicación eucarionte y un origen de replicación procarionte. 1.8. Comprenderá la replicación bidireccional del DNA a partir de un origen. 1.9. Describirá la función de la proteína DnaA en el reconocimiento y unión al origen de replicación procarionte, así como su función en la apertura de la zona de origen. 1.10. Describirá la función de helicasa (DnaB) en la apertura de la doble hélice durante la replicación, así como la función de la proteína DnaC. 1.11. Describirá la función de la proteína SSB durante la replicación. Conoci- Compren- Aplicamiento sión ción X X X X X X X X X X X Objetivos del tema El alumno... 1.12. Comprenderá la importancia de la existencia de un extremo 3’OH para la síntesis de DNA y reconocerá la importancia de primasa en la formación de la horquilla de replicación. 1.13. Distinguirá a las dos principales DNA polimerasas procariontes. 1.14. Describirá las funciones de las diferentes de polimerasa III. 1.15. Comprenderá los conceptos de procesividad y fidelidad y los asociará a las subunidades responsables. 1.16. Describirá la formación de fragmentos de Okazaki. 2. Topología del DNA Conoci- Compren- Aplicamiento sión ción X X X X X 1.17. Reconocerá las actividades enzimáticas en polimerasa y su función en la maduración de los fragmentos de Okazaki. 1.18. Comprenderá la función de ligasa en la maduración de los fragmentos de Okazaki. 1.19. Comparará el reconocimiento del origen de replicación procarionte y eucarionte. 1.20. Describirá la función de las principales DNA polimerasas eucariontes (alfa, delta, epsilon). 1.21. Comparará la terminación de la replicación en eucariontes y procariontes. 1.22. Describirá la función de las DNA telomerasas en la replicación del DNA eucarionte. 2.1. Relacionará la topología del DNA con la manera en que afecta los procesos de replicación, reparación y recombinación. 2.2. Describirá la función de las DNA topoisomerasas y DNA girasa durante la replicación del DNA. 2.3. Comprenderá el mecanismo catalítico de las topoisomerasas. 2.4. Distinguirá entre las topoisomerasas tipo I y tipo II. X 2.5. Describirá la función de topoisomerasa IV durante la terminación de la replicación en procariontes. X X X X X X X X X Replicación del DNA Con base en la estructura del DNA..... 1. La doble hélice de DNA con su apareamiento entre cadenas complementarias sugiere que cada cadena sirve como molde para la síntesis de una cadena hija 2. Hay conservación en el contenido A/T y G/C en todas las células de un mismo organismo Posibles mecanismos de replicación del DNA Hebras parentales Estructura del DNA Doble Hélice Replicación Semiconservadora Cadenas complementarias Conservadora Dispersa al azar ¿Pruebas experimentales? Hebras hijas Experimento de Meselson y Stahl Alimentar cultivos de E. coli con fuente de 14N (ligero) o 15N (pesado) y purificar el DNA Purificar el DNA centrifugarlo en un gradiente de CsCl DNA con 14N DNA con 15N Migración El DNA que tiene 15N migrará más en el gradiente de CsCl, mientras que el DNA que tiene 14N migrará menos. Experimento de Meselson y Stahl Comprobación del mecanismo semiconservador de replicación E. coli se cultiva por muchas generaciones en un medio con N “pesado” Los resultados son consistentes con el mecanismo de replicación semicoservadora Luego se pasa a un medio con N “ligero” solo por una generación Se deja otro ciclo de replicación DNA bacteriano durante replicación ¿Es la replicación del DNA unidireccional o bidireccional? En bacterias, que tienen genoma circular cerrado Replicación del DNA de E. coli por 1.5 generaciones en presencia de nucleótidos marcados Autoradiografía Demostración experimental Experimento de Gyurasits y Lake DNA de Bacillus subtilis Pulso de baja intensidad en el inicio Pulso de alta intensidad Resultados del experimento de Gyurasits y Lake • Horquillas en ambas direcciones muestran marca fuerte, lo que indica que fueron activas durante el segundo pulso. Ambas horquillas surgen de un punto inicial que es el origen de replicación. Las horquillas se mueven en direcciones opuestas. • Evidencia de que la replicación del DNA es bidireccional y a partir de un origen de replicación Reacción química de la replicación del DNA DNA polimerasa DNAn + dNTP (DNA)n+1 + PPi • Cada cadena de DNA sirve como molde para la síntesis de una cadena nueva • Los substratos son desoxiribonucleósidos 5’ fosfato (dNTPs: dATP, dTTP, dGTP, dCTP) (nucleótidos) • Las cadenas de DNA se separan y se sintetiza la hebra complementaria de cada una, de manera simultánea • Las DNA polimerasas añaden dNTPs al molde de la cadena madre en dirección 5’ a 3’ La DNA polimerasa requiere un 3’OH libre para poder polimerizar Se produce un ataque nucleofílico del 3’ OH al fosfato del dNTP entrante dNTP DNAn molde PPi (DNA)n+1 Pero si… La horquilla de replicación es bidireccional Las cadenas de DNA son antiparalelas Y la DNA polimerasa solo sintetiza en dirección 5’ a 3’ Entonces…. ¿Qué ocurre en la horquilla de replicación para que se puedan sintetizar las dos hebras de manera complementaria y antiparalela? Mecanismos para explicar la síntesis de las cadenas complementarias Síntesis continua requiere que las DNA polimerasas sintetisen DNA de 5’->3’ y de 3’ -> 5’ Síntesis discontinua las dos cadenas nuevas se sintetizan en fragmentos, en dirección de 5’->3 Síntesis semidiscontinua una cadena se sintetiza en forma continua y la otra en forma discontinua, en dirección de 5’->3. La replicación del DNA es semidiscontinua Experimentos de Reiji Okazaki Durante la replicación del DNA se sintetizan fragmentos que varían entre 1000 a 2000 nt en E. coli y entre 100 a 200 nt en eucariotes ¿Explicación? Replicación semidiscontinua: Una hebra se sintetiza en forma continua y la otra discontinua Cadena adelantada o guía (leading)/ Cadena retrasada (lagging) 5’ 3’ 5’ * * 3’ 3’ 3’ 5’ 5’ 3’ 5’ 5’ 3’ Como la dirección de síntesis de la cadena discontinua es opuesta a la dirección de apertura de la horquilla, se comienza una nueva cadena. En cambio, la cadena continua simplemente se sigue alargando. Como la DNA polimerasa no puede polimerizar una hebra nueva de DNA si no cuenta con un cebador que tenga un 3’OH libre, es necesario que estos cebadores sean sintetizados previamente a que la DNA polimerasa actúe Hebra “guía” continua Fragmentos de Okazaki Hebra “retrasada” discontinua El crecimiento de ambas cadenas es en sentido 5’ 3’ Los cebadores son sintetizados por una DNA primasa La DNA primasa emplea ribonucleótidos para sintetizar los cebadores Generalidades de la Replicación de DNA • • • • • Semiconservadora A partir de un origen de replicación Bidireccional Semidiscontinua Requiere de cebadores de RNA La replicación se divide en tres fases • Inicio • Alargamiento • Terminación El inicio de la replicación comienza en el: Origen de Replicación En bacteria hay un solo origen de replicación (OriC en E. coli) Secuencias repetidas en tandem (13 pb) Cajas DnaA (9 pb) 1. Activación por metilación de A en GATC 2. Unión de DnaA “abre el DNA” 3. Unión de DnaB y DnaC- actividad helicasa ATP dependiente 4. Unión de SSB para mantener separadas las cadenas de DNA DnaA, DnaB, DnaC y HU en OriC DnaA reconoce OriC y es la que inicia Se unen 30 monómeros de DnaA en el OriC Luego le siguen otras proteínas Proteína DnaA • La proteína DnaA es un monómero de 52 kDa. Tiene una muy alta afinidad por ATP (Kd = 0.03 M) y lo hidroliza lentamente a ADP en una forma dependiente de DNA. Tiene un dominio de unión a ATP. • Se une con alta afinidad y de forma cooperativa a las cajas dnaA de oriC y se ha calculado una estequiometría de 30 subunidades de dnaA por oriC. Proteína DnaB. Helicasa Monómeros de 50 kDa que forman un homohexámero. Tiene dominios que se requieren para: • Interacción con la proteína DnaC • Unión a DNA de cadena sencilla • Unión a DNA de cadena doble • Activación de la primasa • Hidrólisis de ATP La helicasa rodea una de las hebras del DNA duplex y se desplaza logrando la apertura de la doble hélica por exclusión estérica. Una hebra es excluida del canal interno, mientras que la otra hebra es retenida en el interior del anillo. DnaB. Helicasa Rompe los puentes de hidrógeno entre las bases Reconocimiento del oriC y formación del primosoma 2. La unión del complejo HUATP-DnaA desestabiliza la doble hélice en la región P1 1. La proteína HU causa torsiones en el DNA y facilita su unión a la proteína DnaA 3. Unión de DnaB a la región abierta del DNA. 4. Unión de DnaB y DnaC Proteína de unión a DNA de cadena sencilla (SSB, Single-strand DNA binding proteins) Las proteínas SSB se une al DNA de cadena sencilla con alta afinidad y así previene que se vuelva a formar el híbrido DNADNA. Proteínas de unión a cadena sencilla (SSB) La unión de SSB es cooperativa y ayuda a la polimerasa facilitando su actividad Estructura de las SSB • Una vez abierto el DNA, comienza la síntesis (Alargamiento) DnaG Primasa La primasa es una proteína monomérica de 60 kDa que sintetiza oligo-ribonucleótidos (cebadores o primers) de 10 a 12 unidades de longitud, usando rNTPs. Es dependiente de molde y la secuencia corresponde al origen de replicación. El extremo 3‘ del último ribonucleótido es extendido como DNA por la acción de la DNA polimerasa. Se forma una unión covalente entre RNA y DNA DNA primasa RNA polimerasa que sintetiza los cebadores de RNA Hay tres polimerasas en Escherichia coli La DNA polimerasa III cataliza la reacción de polimerización durante la replicación Con un alto grado de fidelidad La actividad de DNA polimerasa cataliza la adición de un dNTP al extremo 3’-OH de un polidesoxinucleótido complementario al DNA molde, por un mecanismo de desplazamiento nucleofílico. Propiedades de las DNA Polimerasas FIDELIDAD Y PROCESIVIDAD La FIDELIDAD se refiere al seguimiento exacto de la secuencia que sirve como molde. En promedio, las DNA pols, 1 error por cada 108 nts La actividad de exonucleasa 3’ 5’ de la DNA polimerasa contribuye a la fidelidad pues tiene actividad correctora (proofreading). Competencia cinética entre la actividad de Polimerasa y de Exonucleasa La PROCESIVIDAD se refiere a la capacidad de una DNA polimerasa de elongar una cadena de DNA por muchos nucleótidos antes de disociarse del complejo que forma con el sustrato. La DNA polimerasa I tiene una procesividad baja. Es distributiva. La DNA polimerasa III tiene una procesividad alta. Actividad de exonucleasa 3’ 5’ Subunidades de la DNA polimerasa III de E. coli Sub # por holoenzima Mr Función 2 132,000 Actividad polimerasa 2 27,000 Exonucleasa 3’5’ 2 10,000 Se requiere para la union de DnaB 2 71,000 2 52,000 1 35,000 ’ 1 33,000 1 15,000 1 12,000 4 37,000 Unión estable al molde, dimerización del núcleo Abrazadera que carga las subunidades al DNA Pinzas que forman una rueda sobre el DNA y aseguran óptima procesividad Núcleo de la polimerasa Abrazadera complejo proteínas Pinza rodante La DNA pol III es altamente procesiva gracias a las subunidades Modelo del dímero para la DNA Polimerasa Función en la horquilla de replicación La DNA pol III es la que replica las dos hebras a la vez Corrige errores con actividad exonucleasa 3’5’ La replicación de la hebra retrasada se interrumpe cada 1000 nt approx DNA polimerasa I La DNA pol I rellena los espacios entre fragmentos de Okazaki Utiliza actividad exonucleasa 5’3’ para eliminar los cebadores de RNA Función de la DNA polimerasa I en la replicación Función de la DNA ligasa en la replicación 1. Formación del intermediario Enzima ATP 2. Transferencia del adenilo al 5’-P 3. Formación del enlace fosfodiéster La DNA ligasa de E. coli es una enzima de 75 kDa. Es muy lábil. Terminación de la replicación en procariontes Las dos horquillas de replicación se aproximan a la misma región que contiene las cajas Ter. Son secuencias de 22 pb, tambien llamados sitios de terminación. Están presentes en tandem (seis) en forma invertida. A estas secuencias se unen las proteínas TUS (TBP). La presencia de estas proteínas de unión a DNA causa que se detenga el avance de las horquillas. Tus: termination utilization substance TBP: Termination binding protein. Proteína de 36 kDa que afecta la actividad de la DNA helicasa (DnaB). TERMINACIÓN. Desenrrollamiento y Síntesis reparativa. Las dos hebras duplex, productos de la replicación están enrrolladas La topoisomerasa IV contribuye a la desnaturalización y descatenación de las hebras. Mutantes en el gene de TopoIV exhiben cromosomas que no se han separado totalmente. Ocurre síntesis reparativa para llenar los huecos ¿Cómo se asegura la célula que su DNA se replica sólo una vez? Porque la replicación sólo inicia cuando el DNA está completamente metilado . Así, cuando la hebra nueva, recién sintetizada no está todavía metilada, la replicación no se puede iniciar nuevamente. Las enzimas Dam metilasas regulan el inicio en el origen de replicación Los cromosomas eucariontes tienen múltiples origenes de replicación No todos los orígenes de replicación se activan al mismo tiempo REPLICACIÓN DE DNA EN EUCARIONTES En eucariontes, la replicación comienza en muchos sitios a lo largo de los cromosomas. Los orígenes de replicación de metazoarios no están definidos por una secuencia específica, como el oriC Sino que consisten en: • Sitios de inicio de alta frecuencia • Sitios de inicio de baja frecuencia Los orígenes de replicación se establecen durante la fase G1 del ciclo celular y dependen de varios parámetros: Estructura nuclear Estructura de la cromatina Secuencia de DNA Modificaciones en el DNA Permite modificar el número y localización de los orígenes de replicación DNA polimerasas en células eucariotas Proliferating-Cell Nuclear Antigen (PCNA) • PCNA es una proteína de 29 kDa. • Forma un trímero alrededor del DNA. • Incrementa la procesividad de la DNA pol delta hasta 40 veces. •Se ha demostrado su interacción in vitro con más de 50 proteínas. Entre ellas: • Ciclina D1, cdk2 y el inhibidor de cdks Comparación de la replicación del DNA en E. coli y en células humanas E. coli Cél humanas Cantidad de DNA, pb/ cél. 3.9 106 109 Velocidad avance horquilla m/min 30 3 Velocidad de replicación, nt / seg 850 60-90 Número de orígenes de replicación / célula 1 103-104 Tiempo 1 replicación genómica (h) 0.27 8 Tiempo 1 división celular (h) 0.33 24 Los orígenes de replicación más estudiados son de levadura Origen de replicación en eucariotas Complejo prereplicativo Terminación de la replicación en eucariontes El dilema de los cromosomas lineales Durante la terminación en procariontes, hay hidrólisis del cebador pero el extremo 3’ de la cadena funciona para cebar la síntesis que así completa la cadena. Sin embargo, en los cromosomas lineales de eucariontes, después de eliminar al cebador no hay forma de completar la síntesis. Esto implica que los cromosomas se irían acortando después de cada ronda de duplicación Terminación en eucariontes El dilema de los cromosomas lineales. La Solución. Telomerasa. Enzima que adiciona secuencias cortas que se repiten en los extremos de los cromosomas. Telómeros. Las secuencias repetidas varían entre especies: Tetrahymena TTGGGG Humano TTAGGG Paramecium TTGGGG Trypanosoma TTAGGG Arabidopsis TTTAGG La telomerasa es una DNA polimerasa que utiliza RNA como molde La telomerasa está compuesta por dos subunidades: • Subunidad catalítica (proteína) • Subunidad de RNA asociada Funciona como molde para alargar una de las cadenas. Mecanismo de acción de la telomerasa Mecanismo de acción de la telomerasa El resultado: La telomerasa está presente en células embrionarias, pero en células somáticas su actividad es muy baja. Topología del DNA • Tiene implicaciones en los procesos de Replicación, Recombinación y Reparación La replicación causa superenrollamiento La replicación causa superenrollamiento DNA duplex parental relajado DNA circular Replicación Región Región superenrollada superenrollada Las proteínas DnaB y DNA Girasa resuelven el problema Proteína DnaB DNA Girasa En eucariontes, durante la replicación hay modificaciones en los octámeros de histona Las histonas parentales se distribuyen proporcionalmente entre las nuevas cadenas El patrón de nucleosomas se hereda a las cadenas hijas Las topoisomerasas Las topoisomerasas Tipo I cortan 1 sola hebra Las topoisomerasas Tipo II cortan las 2 hebras Topoisomerasa IV Girasa Topoisomerasa IV participa en la terminación de la replicación de moléculas circulares Decatenación