Tema 5.3. Replicación del ADN
Anuncio
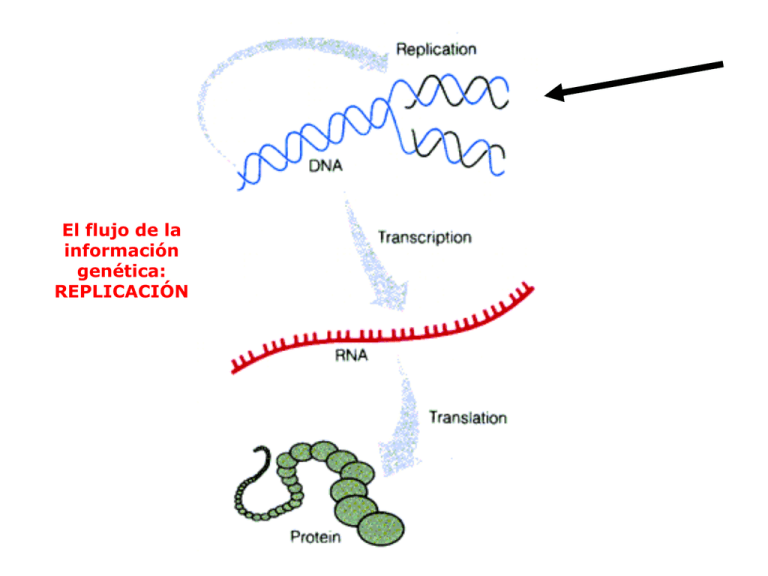
El flujo de la información genética: REPLICACIÓN La replicación según Watson & Crick g a c t 5’…gatccgacttagacc…3’ 3’…ctaggctgaatctgg…5’ 5’…gatccgacttagacc…3’ 5’…gatccgacttagacc…3’ 3’…ctaggctgaatctgg…5’ 3’…ctaggctgaatctgg…5’ 5’…gatccgacttagacc…3’ 3’…ctaggctgaatctgg…5’ Distribución semiconservativa El experimento de Meselson y Stahl g El experimento de Meselson y Stahl El experimento de Meselson y Stahl El experimento de Meselson y Stahl Mecanismo de la replicación DNA polimerasa (A. Kornberg y col. 1958) 103 kdaltons Reacción: (DNA)n residuos + dNTP (DNA)n+1 residuos + PPi Requerimientos: los 4 dNTPs (dATP, dGTP, dTTP, dCTP) Presencia de Mg2+ Cadena cebadora con grupos 3’-OH libres Molde de DNA de cadena sencilla Elongación en dirección 5’ a 3’ Mecanismo de la replicación Actividad 3’ a 5’ exonucleasa de la DNA polimerasa I Actividad 5’ a 3’ nucleasa de la DNA polimerasa I Estructura de la DNA polimerasa I (fragmento Klenow) Proteasa Exonucleasa 5’-3’ Exonucleasa 3’-5’ Polimerasa Fragmento grande (Klenow) Corrección de errores en la replicación por medio de la actividad 3’ a 5’ exonucleasa de la DNA polimerasa I Clases de DNA polimerasas en organismos procarióticos Nombre Función DNA polimerasa I Elimina el cebador y lo sustituye Rellena los huecos en la hebra retrasada DNA polimerasa II Repara el DNA, no necesaria en replicación DNA polimerasa III Enzima principal en la replicación del DNA Requerimientos: los 4 dNTPs (dATP, dGTP, dTTP, dCTP) Presencia de Mg2+ Cadena cebadora con grupos 3’-OH libres Molde de DNA de cadena sencilla Elongación en dirección 5’ a 3’ Actividad exonucleasa 3’-5’ Marcaje radiactivo del DNA de una bacteria: visualización de estructuras theta (θ) Primera ronda de replicación Autoradiografía Interpretación Autoradiografía Interpretación Segunda ronda de replicación Una copia del ADN imposible La copia correcta del ADN: fragmentos de Okazaki Sitios específicos de origen de la replicación (E. coli) Iniciación de la Replicación en E. Coli La replicación necesita un cebador Arquitectura propuesta para la DNA polimerasa III Subunidad catalítica Actividad Exonucleasa 3’-5’ Pinza deslizante Coordinación en la síntesis de la hebra guía y la hebra retardada del ADN Coordinación en la síntesis de la hebra guía y la hebra retardada del ADN Proteínas que intervienen en la horquilla de replicación Clases de DNA polimerasas en organismos eucarióticos Nombre Función DNA polimerasa α Subunidad primasa Unidad DNA polimerasa Polimerasa iniciadora Sintetiza el cebador de RNA Añade unos 20 nucleótidos al cebador DNA polimerasa β Repara el DNA DNA polimerasa δ Enzima principal en la replicación del DNA Proteína Función Tamaño (kD) Moléculas por célula Mecanismo de la DNA ligasa ADN relajado y enrollado ADN relajado y enrollado: Topoisómeros Estructura de la Topoisomerasa I ADN Mecanismo de la topoisomerasa I Topoisomerasa II Mecanismo de la Topoisomerasa II Topoisomerasa II o DNA girasa es blanco de varios antibióticos: NOVOBIOCINA impide unión de ATP a la girasa Ácido Nalidíxico y CIPROFLOXACINA dificultan rotura y empalme de cadenas de DNA Tratamiento de infecciones de las vías urinarias al inhibir la replicación bacteriana. Flujo de información en virus con ARN Replicación de genomas de ARN que no pasan por DNA Replicasa Replicasa Hebra sentido Ejerce de mRNA Híbrido RNA-RNA Replicasa o RNA polimerasa dependiente de RNA Hebra antisentido Ejerce de molde para las hebras sentido Hebra sentido Empaquetada en viriones Flujo de información en los retrovirus: del ARN al ADN Copia e integración del genoma de un retrovirus Zidovudina, Azidotimidina o AZT El primer medicamento antirretroviral aprobado