Genética 3º Biología
Anuncio

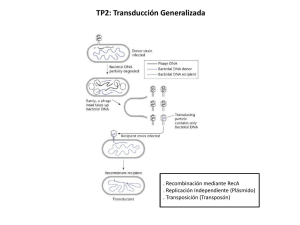
División de Genética Genética 3º Biología PRÁCTICA 4 Mutagénesis con transposones en Salmonella enterica ATENCIÓN: Salmonella enterica (serovar Typhimurium) es una bacteria inofensiva no patógena de humanos, pero ser recomienda tomar medidas higiénicas si se va a estar posteriormente en contacto con una persona inmunodeprimida o un bebé sin flora bacteriana desarrollada. En esta práctica. realizaremos varios procedimientos básicos de la Genética bacteriana: 1. 2. 3. 4. 5. 6. 7. Transducción con bacteriófagos Titulación de un lisado de bacteriófagos Selección de resistencia a antibióticos Mutagénesis con transposones compuestos Réplica de colonias bacterianas Identificación de auxótrofos inducidos por transposición Identificación de fusiones de operones Introduccion. Para realizar una mutagénesis cons transposones en bacterias, es necesario disponer de: • • • • • Elemento transponible, capaz de causar mutaciones por inserción en cualquier gen. Sistema de selección de inserción del transposón. Molécula portadora del transposón, que puede ser cromosoma, plásmido o fago. Sistema apropiado de introducción del transposón en la bacteria que queremos mutagenizar: transformación, conjugación o transducción, a través de un vehículo o vector. Sistema de contraselección de vehículo introductor del transposón en la célula receptora. En esta práctica. utilizaremos los siguientes elementos, en el mismo orden arriba mencionado: • • • • • Transposón MudJ, seleccionable porque confiere resistencia al antibiótico kanamicina y que contiene el operón lac de E. coli sin promotor. Es un derivado del fago Mu que es incapaz de transponerse por sí mismo porque carece de gen de la transposasa. La transposasa la aporta un fago Mu ayudador adyacente, Mud1. La molécula portadora del transposón es el cromosoma bacteriano de una estirpe de S. enterica, concretamente el gen hisD, dentro del operón de síntesis del aminoácido histidina. El sistema de introducción en este caso es la transducción, empleando como vector el bacteriófago atemperado P22, perteneciente a la familia del fago λ. Esta estirpe de P22 es capaz de hacer transducción generalizada en Salmonella: la mutación int impide su integración en el cromosoma, la mutación HT aumenta su frecuencia de transducción; con este fago, el 50% de las cápsidas son partículas transductantes. En una transducción hacen falta dos estirpes bacterianas, una donadora y otra receptora. Los genotipos de las estirpes a utilizar son: o Donadora: TT10288: hisD9943::MudJ (Knr) his-9941::Mud1 (Apr) o Receptora: TT1704: ∆his o Bacteriófago: P22: int7 HT Como sistema de contraselección, emplearemos la incapacidad de replicación (no hay origen de replicación) y la imposibilidad de recombinación del fragmento cromosómico donde está situado el transposón, ya que emplearemos una receptora con una deleción del operón de la histidina. Adicionalmente, emplearemos el operón lac para obtener fusiones de operón. Cuando el transposón se inserte en un gen en la orientación adecuada, los genes lac se expresarán a partir del promotor del gen mutado. Así, podemos monitorizar la expresión de este gen siguiendo la expresión de lac en medios que lo detectan por cambio de color. Día O (no han estado presentes los alumnos). 1 El profesor amablemente ha inoculado la estirpe TT10288 a 30ºC. Día 1. Preparación de un lisa do de P22 de la estripe donadora TT10288 1. Añadir a un tubo estéril 0.5 ml de cultivo de TT10288. 2. Añadir 2 ml de NB 3. Añadir 25 µl de fagos. 4. Incubar a 30º toda la noche. Preparación de la estirpe receptora TT1704 1. Añadir 2 ml de LB a un tubo de cultivo. 2. Picar una colonia con el asa estéril e inocular el medio anterior. 3. Incubar a 30º toda la noche. Día 2 Preparación del lisado 1. Transferir 1 ml del lisado a un tubo eppendorf (está turbio, ya que P22 es un fago atemperado). 2. Centrifugar 1 min. a máxima velocidad, para eliminar las bacterias, en su mayoría lisógenas. 3. Recoger el sobrenadante en un tubo nuevo. 4. Añadir 3 gotas de cloroformo. 5. Tapar bien el tubo y agitar enérgicamente en el vórtex durante un minuto. ATENCIÓN: El cloroformo es tóxico. Evitar contacto con la piel y no inhalar los vapores 6. Volver a centrifugar para precipitar el cloroformo. El lisado ya está preparado para su utilización. Titulación de lisado Para hacer una transducción, debemos saber la concentración de fagos en el lisado, es decir, titular el lisado. Como inicialmente desconocemos la concentración de fagos del lisado, debemos preparar diluciones del lisado apropiadas para poder contar las calvas de lisis de forma cómoda y con validez estadística. Alrededor de 100 calvas por placa es lo ideal Se dispone de 6 tubos de dilución, tampón de dilución de fagos, cultivo de TT1704 (puede ser empleada cualquier estirpe susceptible de ser lisada por P22) y lisado de fagos de TT10288. Añadir 5 ml de tampón de dilución a cada tubo. Cada alumno debe calcular como preparar diluciones del lisado de 10-7 y 10-8. Preparación de la titulación. 1. Añadir en cada tubo de agar de cobertera, preparado anteriormente con 3 ml de agar y mantenido en el termobloque a 50º: a. 0.1 ml de cada una de las diluciones antes mencionadas. b. 0.1 ml de cultivo de estirpe receptora. 2. Mezclar por rotación entre las manos, para evitar la formación de burbujas. 3. Verter el contenido de cada tubo en una placa de LB, de forma que el agar de cobertera cubra toda la caja. Evitar la formación de burbujas. Dejar soldificar el agar durante 10 minutos; marcar las cajas convenientemente. 4. Incubar las cajas a 37º hasta el día siguiente. Transducción y mutagénesis. 1. Mezclar 0.2 ml de lisado con 0.2 ml de cultivo de TT1704 en un tubo eppendorf. 2. Incubar los tubos en el baño a 42ºC durante 1 hora. 3. Sembrar 0.1 ml del contenido de tubo en cada una de dos placas de LB con kanamicina. 4. Incubar en la estufa a 37º hasta el día siguiente. Día 3. Titulación del lisado de fagos. La presencia de un fago individual se manifiesta como una calva de lisis, que es el hueco que dejan las bacterias lisadas por el fago. 1. Elegir la caja de Petri que contenga alrededor de 100 calvas de lisis. Contar el número de calvas. 2 2. Calcular la concentración de fagos: unidades formadoras de calva por mililitro de lisado (ufc/ml). Hay que tener en cuenta el factor de dilución y el volumen empleado. Un lisado óptimo para transducción tiene 1091010 ufc/ml. Mutagénesis. Las colonias que crecen en el medio LB-kanamicina son resistentes a kanamicina porque han recibido una inserción del transposón MudJ en el cromosoma. 1. Contar el número de colonias resistentes a kanamicina en la placa de mutagénesis. 2. Calcular la frecuencia de inserción por unidad formadora de placa. Identificación de mutantes auxótrofos inducidos por inserción y de fusión de operones. Si la inserción ha ocurrido en un gen de la ruta biosintética de algún compuesto que puede ser suplementado en el medio, tendremos un mutante auxótrofo. Para identificar auxótrofos, tenemos que replicar los clones obtenidos a medio mínimo. Si la inserción ha ocurrido en un gen en la orientación correcta, cuando ese gen se exprese la bacteria será lac + (S. enterica es lac – de forma natural) y en medio LB+X-gal y dará una colonia de color azul. Replicaremos las colonias con inserciones a este medio, a la vez que a medio mínimo. 1. 2. 3. Picar con un palillo una colonia aislada de una de las placas de mutagénesis. Transferir la colonia picada, usando el mismo palillo y sin picar dos veces, a 1. Primero: placa de medio mínimo con histidina (la estirpe receptora es auxótrofa de histidina debido a la deleción). 2. Segundo: placa de medio completo, LB+X-gal. 3. Tercero: tirar el palillo en las bolsas dispuestas a tal efecto. Repetir el proceso hasta completar la placa, aproximadamente 120 colonias. Incluir la estirpe receptora y la donadora como controles Incubar las placas a 37ºC hasta el día siguiente. Día 4 Identificación de mutantes auxótrofos inducidos por inserción y de fusión de operones. Observar cuidadosamente las placas. 1. Calcular la frecuencia de inducción de auxótrofos respecto del número de inserciones. 2. Calcular la frecuencia de fusiones lac + respecto del número de inserciones. Identificación de bacterias libres de fagos y de bacterias lisogénicas Aislar colonias de alguno de los transductantes en placas indicadoras EBU. Determinación de las auxotrofías (COLECTIVO). Para determinar de qué compuesto son auxótrofos nuestros mutantes, los replicaremos a un conjunto de 9 medios a los que se les ha añadido una combinación de compuestos de los que comúnmente se pueden aislar auxótrofos en bacterias: aminoácidos, bases nitrogenadas y algunas vitaminas. Los medios contienen los suplementos de su fila y columna correspondiente, además de histidina. Medios Medios 5 6 7 8 9 1 Adenosina Val Phe Gln Timina 2 Guanosina Leu Tyr Arg Ser 3 Uracilo Ile Trp Pro Glu 4 Met Lys Thr Tiamina Gly Día 5. Identificación de bacterias libres de fagos y de bacterias lisogénicas Las colonias, o sectores de colonias, de color claro están libres de fagos, mientras que las de color oscuro son lisógenos. Determinación de las auxotrofías (COLECTIVO). El crecimiento en las placas suplementadas con una determinada combinación de compuestos identifica la auxotrofía que presentan los mutantes. Responde a estas preguntas en la hoja del examen: 3 1 ¿Cuál ha sido el título de tu lisado? 2 ¿Cuál ha sido la frecuencia de obtención de auxótrofos? 3 ¿Cuál ha sido la frecuencia de obtención de fusiones lac +? 4 ¿Has obtenido auxótrofos en la práctica? En casi afirmativo, cuántos y de qué compuesto son. 4