inicio de la traducción eucarionte
Anuncio

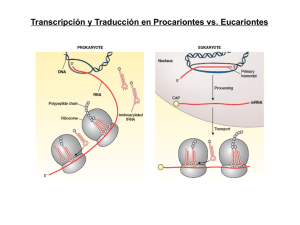
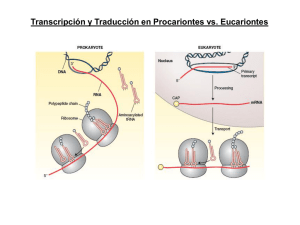
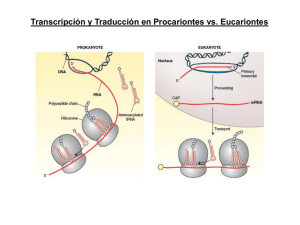
Transcripción y Traducción en Procariontes vs. Eucariontes Etapas de la traducción Iniciación Elongación Terminación Para el inicio de la traducción: Las subunidades ribosomales deben estar separadas La subunidad pequeña debe reconocer al mRNA El tRNA aminoacilado (fmet-tRNAmet o met-tRNA) debe colocarse en la posición P de la subunidad ribosomal pequeña Debe ocurrir el reconocimiento codón-anticodón de inicio Ocurre hidrólisis de al menos una molécula de GTP Factores de iniciación de la traducción IFs (bacteria) o eIFs (eucariontes) La hidrólisis de GTP es catalizada por IF2 INICIO DE LA TRADUCCIÓN PROCARIONTE IF1 IF2 IF3 50S 30S [Mg2+] AUG 3 3 1 GDP 2 fMet + Shine-Dalgarno 3 1 fMet GTP 2 3 1 AUG complejo de inicio de la traducción fMet GTP + 2 fMet GTP 2 3 1 FACTORES DE INICIO DE LA TRADUCCIÓN (PROCARIONTES) Factor Función IF1 Previene la unión de tRNAs en el sitio A de la subunidad 30S IF2 GTPasa que interacciona con 3 componentes claves durante iniciación: la subunidad 30S, IF1, y fMet-tRNAif-Met) IF3 Se une a 30S y evita re-asociación con 50S. Participa en el reconocimiento codon-anticodon f-met IF3 5’ aa-tRNA La región Shine Dalgarno interacciona con el rRNA 16S en la subunidad 30S y coloca esta subunidad sobre el AUG de inicio de la traducción (sitio P) IF1 E P A 3’ INICIO DE LA TRADUCCIÓN EUCARIONTE 60S eIF1A eIF2 eIF3 eIF4 eIF5 40S [Mg2+] + Met 3 4 Met GTP 1A + 2 AUG 4 5 1A 2 3 4 Escaneo para buscar el AUG GDP Met GTP 5 2 3 1A AUG 3 Met GTP 5 2 4 3 1A AUG complejo de inicio de la traducción Met GTP 2 3 1A Se forma un complejo circularizado con los extremos 5’ y 3’ del mRNA que recluta a la subunidad 40S LEJOS del AUG de inicio Los factores eIF4 NO existen en bacteria Ocurre un escaneo de la región 5’ no traducible (5’UTR) hasta encontrar el primer AUG de inicio eIF4A: helicasa; utiliza ATP para deshacer estructura secundaria en el mRNA y permitir paso de 40S Para regenerar eIF2•GTP que se une a tRNA-met para iniciar traducción, se requiere el factor eIF2B eIF2B intercambia GDP x GTP en eIF2 eIF2•GDP Resumen inicio de la traducción en eucariontes Complejo 43S Participación de mas de 20 subunidades protéicas diferentes MUY diferente de procariontes eIF3-eIF4G La regulación del inicio de la traducción en eucariontes es compleja y MUY estricta Complejo 48S Elongación ribosoma mRNA tRNAs-aa factores de elongación GTP GTP x aa Factores de elongación Bacteria EF-Tu EF-Ts EF-G Eucariontes eEF-1 eEF-1 eEF-2 1. Posicionamiento del aa-tRNAaa elongador correcto (EF-Tu/eEF-1) 2. Hidrólisis de GTP y cambio conformacional. Para regenerar EF-Tu•GTP se requiere EF-Ts Formación del enlace peptídico Bacteria Eucariontes Actividad peptidil transferasa del RNAr 23S o 28S (RIBOZIMA) 3. Ataque nucleofílico amino del aa2 al carboxilo del aa1 4. Posicionamiento del péptido sobre el tRNA del sitio A Translocación Bacteria Eucariontes EF-Tu EF-Ts EF-G eEF-1 eEF-1 eEF-2 5. Entrada de EF-G/eEF-2 6. Hidrólisis de GTP 7. Cambio conformacional y desplazamiento ciclos de elongación Terminación ribosoma mRNA factores de terminación GTP proteína subunidades ribosoma mRNA tRNA libre Factores de terminación Bacteria Eucariontes RF1 RF2 RF3 RRF IF3 EF-G eRF1 eRF3 eRF1 utiliza agua para hidrolizar el péptido Modelo de terminación propuesto para bacteria Moleculas que unen el mismo sitio en el ribosoma El gasto energético del proceso de traducción Se hidrolizan 2 GTP´s por cada aminoácido incorporado La hidrólisis promueve cambios conformacionales Cargado de tRNA con su aminoácido 1 ATP /aa TRADUCCION Iniciación Elongación Terminación 1 GTP (1er aminoácido) 2 GTPs /aa 1 GTP Resumen RIBOSOMA Centro peptidil transferasa (PTC) A: aceptor P: peptidil E: salida (exit) Centro activador de GTPasa Centro decodificador