Capítulo 19. Inter Simple Sequence Repeats (ISSRs)
Anuncio

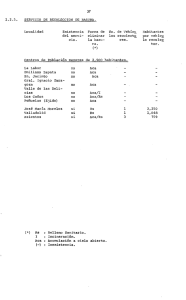
Inter Simple Sequence Repeats (IISRs) 567 Capítulo 19 Inter Simple Sequence Repeats (ISSRs) Andrea González y Xitlali Aguirre Los ISSRs son un tipo de marcador genético que nos permite obtener los niveles de variación en las regiones microsatélite que se encuentran dispersas en varios genomas, particularmente el nuclear. Estas regiones consisten en repeticiones en tandem de motivos simples como (CT)n ó (CA)n, ubicadas entre secuencias no repetitivas del genoma nuclear eucarionte. Los motivos repetidos, llamados también SSRs (simple sequence repeats) pueden ser penta-, tetra-, tri- y dinucleótidos. La longitud de las secuencias de microsatélites tiende a ser altamente variable entre individuos debido a las altas tasas de mutación que experimentan, ya que cuando el DNA se replica durante la meiosis, la DNA polimerasa puede “tartamudear” hacia adelante o hacia atrás en las unidades repetidas, eliminando o agregando unidades a la cadena. Las cadenas resultantes pueden entonces presentar menos o más unidades de repetición (o pares de bases) que las cadenas parentales (Zietkiewicz et al., 1994; Wolfe, 2000). Los ISSRs son marcadores semiarbitrarios amplificados por la reacción en cadena de la polimerasa (PCR) a partir de la presencia de un oligonucleótido o primer complementario a un microsatélite, diseñado para unirse a los motivos repetidos de di y trinucleótidos (evitando los mononucleótidos presentes en el cloroplasto). Los primers de ISSRs consisten en un motivo repetido de di- o trinucleótidos complementario a la secuencia del microsatélite. En ocasiones es posible agregar a esta secuencia un par de nucleótidos extras arbitrarios en 567 568 Las herramientas moleculares el extremo 3’ o en el 5’, que jugarán el papel de “anclas” asegurando así que la amplificación inicie siempre en el extremo 5’ o en el 3’ del microsatélite, respectivamente (Zietkiewicz et al., 1994; Bornet y Branchard, 2001; Pradeep, 2002). Cuando dos secuencias repetidas se presentan dentro de una distancia amplificable y con una orientación invertida, el primer complementario a ellas (uno por reacción de PCR; véase el capítulo 17 de este libro) puede inducir la amplificación del segmento de ADN intermedio. La molécula generada, con un tamaño particular (peso molecular), se considera un “locus”, que representa el segmento de ADN entre los microsatélites. Y se ha visto que los ISSRs frecuentemente amplifican de 25 a 50 bandas en una sola reacción. Este patrón característico de productos de PCR se considera la “huella digital genética” de cada uno de los individuos analizados. El polimorfismo entre individuos de la misma población puede detectarse, ya que el análisis es sensible a la presencia/ausencia del elemento genómico reconocido por el primer y a la longitud de la secuencia intermedia amplificada (Zietkiewicz et al., 1994). Figura 1. Amplificación con un primer (CA)n anclado en el extremo 5’ con tres nucleótidos extras. Se amplifica el segmento intermedio entre dos secuencias de microsatélite en orientación invertida Las bandas de ISSRs son consideradas marcadores dominantes. La presencia de la banda representa el genotipo dominante (homócigo o heterócigo), mientras que su ausencia representa el genotipo homócigo recesivo. Se asume que existen dos alelos por locus. La ausencia de una banda puede deberse a varios factores: 1) La no existencia de un sitio de unión completo al primer debido a una mutación, 2) Rearreglos estructurales en el cromosoma durante la meiosis, 3) Inserciones o deleciones suficientemente grandes como para aumentar o Inter Simple Sequence Repeats (IISRs) 569 disminuir el tamaño de la banda, de manera que se identifica como un locus diferente. Ventajas y desventajas Las ventajas que ofrece esta técnica se centran principalmente en la alta variación que detecta, así como en su reproducibilidad debida principalmente a las altas temperaturas de alineación utilizadas en la PCR. Asimismo, no son necesarias altas concentraciones de ADN. Por otro lado, para diseñar los primers no es necesario conocer la secuencia del genoma del organismo en estudio. Pueden visualizarse tanto en geles de agarosa como de acrilamida. Finalmente, son sencillos de montar, rápidos, eficientes y poco costosos. En cuanto a las desventajas, la homología de las bandas es incierta. Y dado que son marcadores dominantes, no permiten el cálculo de ciertos parámetros que exigen distinguir a los heterócigos de los homócigos dominantes (como FIS y FIT; Wright, 1965). Asimismo, para estimar la heterocigosis poblacional es necesario asumir a priori que la población se encuentra en equilibrio de Hardy-Weinberg, por lo que las estimaciones de heterocigosis y estructuración genética pueden sesgarse un poco, aunque existen correcciones estadísticas (Lynch y Milligan, 1994). Aplicaciones Los ISSRs pueden ser utilizados como marcadores en la resolución de múltiples problemas biológicos. Los polimorfismos que presentan, además de su heredabilidad, permiten aplicarlos en la identificación de individuos, distinción de variedades intraespecíficas (particularmente en especies con importancia económica), identificación de paternidad y maternidad, mapeo genético, evaluación de diversidad y subdivisión genética en poblaciones, reconstrucciones filogenéticas, introgresión e hibridización, y distinción de individuos con origen clonal y sexual (Zietkiewicz et al., 1994; Wolfe, 2000; Pradeep et al., 2002). Ejemplo Al empezar a trabajar con ISSRs es recomendable probar diferentes primers en una condición estándar y después optimizar aquellos que dieron resultados positivos. Esta optimización incluye modificar las condiciones iniciales de 570 Las herramientas moleculares la PCR, particularmente la temperatura y tiempo de alineación, así como la concentración de MgCl2. Por ejemplo, en algunos estudios de genética de poblaciones con especies del género Agave (Aguirre, 2004; González, 2004) se amplificaron ISSRs con los siguientes primers: 841 842 846 857 GAG AGA GAG AGA GAG AYC GAG AGA GAG AGA GAG AYG CAC ACA CAC ACA CAC ART ACA CAC ACA CAC ACA CYT Y=C o T R=A o G Para una reacción de 25 µl se agregaron 2.5 µl de amortiguador para PCR 10X, 0.5 µl de dNTP mix (mezcla de los 4 dideoxinucleótidos a 10 mM), 0.2 µl de Taq polimerasa a 5 U/µl; 2 µl de DNA a 15 ng/µl; el volumen de MgCl2 (concentración inicial de 30 mM) depende de la concentración óptima para cada primer y para completar los 25 µl de la reacción, se agregó el volumen necesario de agua ultrapura correspondiente (tabla 1) Tabla 1. Condiciones óptimas de amplificación para algunos primers. Primer Temperatura de alineación Tiempo de Alineación[MgCl2] (mM) 841 842 846 857 60º C 60º C 52º C 52º C 45” 35” 45” 45” 2.1 2.3 0.96 1.5 No. de ciclos 30 35 30 30 Una vez que se visualizaron las bandas en geles de agarosa al 2% teñidos con bromuro de etidio, se hizo un listado de todas las bandas o loci ampificados en la población. Con estos datos se elaboró una matriz de presencia/ausencia de las bandas por individuo. Esto puede hacerse a mano, o bien por medio de programas como el 1D 3.5 de Kodak. Una vez obtenida la matriz, los datos permitieron obtener frecuencias alélicas, y parámetros clásicos de la genética de poblaciones como heterocigosis poblacional, por especie, estimaciones de la estructuración, distancias genéticas, árboles de similitud y análisis jerárquicos de la varianza molecular. Estos cálculos pueden realizarse con diversos programas como el TFPGA (Miller, 1997) o el Arlequin (Schneider et al., 2000). Inter Simple Sequence Repeats (IISRs) 571 Bibliografía Aguirre X. 2004. Genética de poblaciones de Agave cupreata y Agave potatorum: aportaciones para el manejo y la conservación de dos especies mezcaleras. Tesis de licenciatura, Facultad de Ciencias, UNAM. 73 pp. Bornet B. y Branchard M. 2001. Nonanchored inter simple sequence repeat (ISSR) markers: reproducible and specific tools for genome fingerprinting. Plant Molecular Biology Reporter 19:209-215 González A. 2004. Biología reproductiva y genética de poblaciones del Agave garciaemendozae. Tesis de licenciatura, Facultad de Ciencias, UNAM. 88 pp. Lynch M. y Milligan B.G., 1994. Analysis of population genetic structure with RAPDs markers. Molecular Ecology 3:91-99 Miller M.P., 1997. Tools For Population Genetic Analyses (TFPGA) 1.3: A Windows program for the analysis of allozyme and molecular population genetic data. Computer software distributed by author. Pradeep R., Sarla N. y Siddiq E.A., 2002. Inter simple sequence repeats (ISSR) polymorphism and its aplication in plant breeding. Euphytica 128(1):9-17 Schneider S., Roessli D., y Excoffier L., 2000. Arlequin ver. 2.000. A software for population genetics data analysis. Genetics and Biometry Laboratory, University of Geneva, Switzerland. Wright S., 1965. The interpretation of population structure by F-statistics with special regard to systems of mating. Evolution 19:395-420 Wolfe A., 2000. ISSR Resource Website. http://www.biosci.ohio-state.edu/~awolfe/ ISSR/ISSR.html Zietkiewicz E., Rafalski A. y Labuda D., 1994. Genome fingerprinting by simple sequence repeats (SSR)-anchored polymerase chain reaction amplification. Genomics 20:176-183