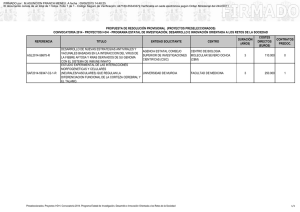
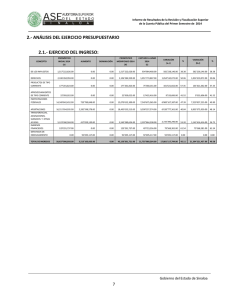
Series de tiempo: Caracterización empírica y selección de técnicas
Anuncio

Series de tiempo: Caracterización empírica y selección de
técnicas de pronóstico basada en Redes Neuronales Artificiales
Ma. Guadalupe Villarreal Marroquín, Mauricio Cabrera Ríos*
División de Posgrado en Ingeniería de Sistemas, FIME-UANL
{lupita@yalma.fime.uanl.mx, mauricio@yalma.fime.uanl.mx}
*Autor Responsable
Resumen
En este trabajo se propone un método para ayudar a las personas con poco entrenamiento estadístico a elaborar un
pronóstico de series de tiempo caracterizando primero su serie y posteriormente, en base a esta caracterización,
seleccionando la técnica más adecuada de pronóstico. El método utiliza redes neuronales artificiales para realizar la
selección. Se puso especial cuidado en que tanto los métodos estadísticos como las redes neuronales artificiales utilizadas
pudieran ser instauradas en MS Excel, por lo que los requerimientos de paquetería computacional son modestos. El método
propuesto se basa parcialmente en las ideas presentadas por (Gupta et al., 2000) en el terreno del secuenciamiento de tareas
en plantas de manufactura.
Palabras claves
Redes Neuronales Artificiales, Métodos de Pronóstico, Series de Tiempo, Caracterización de Series de Tiempo
1. Introducción
Nuestra vida cotidiana está llena de decisiones. Las decisiones que tomamos a diario dependen en gran medida tanto de la
información que tenemos a la mano como de la que podemos proyectar a futuro. El área de pronósticos trata precisamente
de generar información estimada sobre el comportamiento de un fenómeno observable a futuro. En muchos casos, esta área
se basa en el comportamiento histórico de tal fenómeno organizado en una serie de tiempo. Es razonable esperar que una
toma de decisiones más acertada resulte de mejores pronósticos.
A pesar de que una gran cantidad de personas elaboran pronósticos diariamente, existen tres problemas que se pueden hallar
de manera común en esta práctica: (1) no contar con el entrenamiento estadístico necesario, (2) no contar con paquetería
computacional especializada, o (3) no tener tiempo suficiente para hallar el método más adecuado.
Estos tres problemas motivaron los estudios que se incluyen en este trabajo. El primer estudio consistió en una exploración
de métodos tradicionales de pronósticos por medio de MS Excel. Este primer estudio proveyó una base de conocimiento
para el segundo, el cual demuestra un método basado en redes neuronales artificiales (RNAs) para seleccionar la mejor
técnica de pronóstico de entre aquellas utilizadas en el primer estudio a partir de una serie de tiempo arbitraria. El método
propuesto incluye también una sencilla caracterización de series de tiempo.
Este trabajo resulta entonces en un método que no depende de software estadístico especializado y que permite caracterizar
y seleccionar la técnica más adecuada de pronósticos de una manera repetible. Tanto las técnicas de pronóstico como las
RNAs utilizadas son convenientemente instaurables en MS Excel. Un impacto probable es que, a través de la aplicación de
este método, las personas que generen pronósticos lo hagan consistentemente mejor y más rápido. En consecuencia, sería
posible tomar mejores decisiones en las aplicaciones pertinentes aún en casos de contar con un entrenamiento estadístico
modesto.
1
2. Método propuesto
La Figura 1 presenta un diagrama de flujo con el método propuesto. En este método, la parte de selección se basa en las
ideas presentadas en (Gupta, et al., 2000) en el terreno de secuenciamiento de tareas en plantas de manufactura. El objetivo
del método en este trabajo es determinar la mejor técnica de pronóstico de entre varias conocidas a partir de una serie de
tiempo caracterizada por dos parámetros sencillos de obtener.
Normalizar los datos a la escala [ −1,1]
Caracterizar la serie de tiempo
Someter a la red neuronal artificial
Establecer un orden entre los métodos y
determinar el mejor
Figura 1. Método propuesto para la selección de la mejor técnica de pronóstico entre varias conocidas.
El método propuesto se apoya en la capacidad de predicción de las RNAs al utilizar una de ellas para predecir el desempeño
de varias técnicas de pronóstico cuantificado por el error cuadrado medio o MSE. La capacidad predictiva de las RNAs ha
sido estudiada y matemáticamente demostrada para funciones analíticas en (Hornik et al., 1989) y la factibilidad de usar el
MSE como medida de desempeño en pronóstico se encuentra documentada en (Hillier y Lieberman, 2001).
Los pasos del método, partiendo de que se tiene una serie de tiempo, son como sigue:
1.
Normalizar los datos a la escala [-1,1]: los datos de la serie de tiempo se normalizan para que caigan en un rango
de [-1,1] con el objetivo de eliminar efectos de dimensionalidad.
2.
Caracterizar la serie de tiempo: se determinan dos parámetros que caracterizan la serie: el número de períodos de
la serie de tiempo (t) y el grado del primer polinomio (n) que se ajuste a los datos de la serie con un coeficiente de
determinación, R ≥ 80% .
2
Los dos parámetros que se necesitan para esta caracterización son fácilmente obtenibles. El parámetro t resulta de
un conteo directo de datos y el parámetro n de una prueba secuencial de aproximación ejecutable en MS Excel.
Ambos se pueden obtener a partir de una gráfica de dispersión de Datos vs. Período (Figura 2). Para encontrar n,
basta usar la opción de ajustar una línea de tendencia de tipo polinomial a los datos y presentar en pantalla tanto el
valor de R2 como la expresión polinomial. El objetivo es determinar el orden del polinomio de manera creciente a
fin de detectar el primero con el que se obtenga un R 2 ≥ 80% (ver Figura 2). Como se podrá observar, no se
necesita entrenamiento estadístico para lograr esta caracterización. Es importante señalar que esta caracterización
es original, por lo que los resultados de este estudio sirven además para probar qué tan adecuada resulta. En el
caso de estudio que se presenta posteriormente se ofrecen resultados para su evaluación.
2
n
1.50
y = -0.0079x 3 + 0.1499x 2 - 0.6164x + 0.0467
R2 = 0.8959
1.00
observación
0.50
0.00
1
2
3
4
5
6
7
8
9
10
11
12
t
-0.50
-1.00
-1.50
pe ríodo
Serie de tiempo
Ajuste polinomial
Figura 2. Observación vs. Período y Ajuste de una línea de tendencia de tipo polinomial a los datos.
3.
Someter a la red neuronal artificial: se utiliza una red neuronal artificial previamente entrenada, donde se usan
como entradas t y n y como salida la predicción del error cuadrado medio para cada uno de los métodos de
pronóstico.
Una RNA, en el contexto del método propuesto, es esencialmente un modelo matemático no lineal que contiene
parámetros conocidos como “pesos”. Mientras más pesos tenga una RNA, mayor no linealidad se podrá representar
con ella. Para encontrar estos pesos se utilizan algoritmos de optimización que minimizan una función de errores
cuadrados de aproximación. En el área de redes neuronales, varios de estos algoritmos de optimización se
clasifican bajo el nombre de “algoritmos de retropropagación”. Por otro lado, encontrar un conjunto de pesos que
permitan una aproximación adecuada a datos conocidos se le denomina “entrenamiento”.
La RNA que se propone en esta etapa se representa como se muestra en la Figura 3. Esta RNA cuenta con 3 capas:
una capa de entrada que recoge los valores de t y n, una capa oculta que procesa esta información, y una capa de
salida por la que se obtiene el MSE predicho para un método de pronóstico en particular.
Capa de entrada
n
t
1
vij
Capa oculta
...
1
wj
Capa de salida
MSE
Figura 3. RNA con tres capas, entrenada por retropropagación del error
3
En la Figura 3,
vi , j es un peso de la conexión que llega a la j-ésima neurona de la capa oculta procedente de la
i-ésima neurona de la capa de entrada y w j es el peso de la conexión que llega a la neurona de salida procedente de
la j-ésima neurona de la capa oculta. Estos pesos son utilizados para ponderar las salidas generadas por las
neuronas, y representan la memoria de la RNA. Los pesos que se aplican a las conexiones provenientes de
neuronas que vemos con valor constante de uno se denominan sesgos.
4.
Establecer un orden entre los métodos y escoger el mejor: por último, se ordenan los métodos de acuerdo a su
MSE predicho y se escoge el mejor, donde éste será el que tenga el MSE más pequeño.
El método resultante en el punto 4 es el que se debe utilizar entonces para generar los pronósticos. Como se dijo
anteriormente, todos los métodos de pronóstico que se utilizaron en este trabajo fueron codificados en MS Excel y se
encuentran disponibles por parte de los autores. La creación de las RNAs que se utiliza para la selección en este método se
describe a continuación.
3. Obtención de las RNAs a utilizar en el método propuesto
Las RNAs que se requieren para el método de selección propuesto deben ser capaces de predecir el desempeño de cada
método de pronóstico. Como se ha explicado previamente, para construir una RNA se requiere de una fase de
entrenamiento. Adicionalmente, para corroborar que el modelo tenga una capacidad predictiva adecuada también se utiliza
una fase de validación.
Para poder construir las RNAs necesarias se generaron 54 series de tiempo para entrenamiento. Cada una de las series de
tiempo fue preprocesada, de acuerdo con el método propuesto, para que los datos cayeran en el intervalo de [-1,1]. Las
series utilizadas en entrenamiento tuvieron como parámetros t∈{12, 36,60} períodos y aproximación polinomial dada por
n ∈{1, 2, 3, 4, 5, 6}, lo cual resulta en un total de 3 x 6 = 18 combinaciones de t y n. Para cada una de estas combinaciones
se generaron 3 réplicas por medio de una perturbación aditiva aleatoria, de ahí que haya un total de 54 series de tiempo.
Para validación se utilizaron además 22 series; 12 de ellas resultan de todas las posibles combinaciones de series de tiempo
con t∈{24,48} y aproximación polinomial de n∈{1, 2, 3, 4, 5, 6}; 8 de las restantes comprenden series publicadas
(Makridakis y Wheelwright, 1998) y dos más provienen de una compañía de telecomunicaciones que incluyen series de
tiempo de t∈[12,70] períodos y aproximación polinomial n∈[1,6].
Se consideraron 8 métodos de pronóstico tradicionales que se codificaron completamente en MS Excel: (1) método ingenuo,
(2) promedio, (3) promedios móviles, (4) suavizado exponencial simple, (5) ARIMA (0, 1,1), (6) regresión lineal, (7)
suavizado exponencial doble y (8) ARIMA (0, 2, 2) [3, 4].
Se entrenaron y validaron RNAs con dos y tres neuronas en la capa oculta en una etapa inicial, con resultados prometedores
pero con una tendencia de disminución del error de validación al aumentar el número de neuronas en la capa oculta. Por esta
razón en una segunda etapa se construyeron RNAs con cuatro hasta siete neuronas en la capa oculta. En esta capa es vital
determinar un número adecuado de neuronas puesto que un número excesivo de ellas provoca una flexibilidad excesiva del
modelo, que a su vez puede traducirse en una pérdida de capacidad de predicción. A este fenómeno se le denomina
sobreentrenamiento.
El entrenamiento de la RNA se pudo abordar con el optimizador incluido en MS Excel (Excel Solver). Dado que este
optimizador es de tipo local para un problema no lineal como el que se presenta al tratar de minimizar la suma de errores
cuadrados o SSE, se cuidó de inicializar los pesos en diferentes valores para aumentar la probabilidad de tener una solución
satisfactoria. Los pesos se inicializaron nueve veces. Las primeras cinco, todos los pesos tomaban el mismo valor del
siguiente conjunto {−1, −0.5, 0, 0.5,1} , las otras cuarto veces, se inicializaron con valores aleatorios entre -1 y 1. Al final
se mantuvieron los pesos que resultaron en el menor SSE de validación independiente del error de entrenamiento, pues es
precisamente la capacidad de predicción lo que debemos preservar en este caso.
El error de validación se utilizó entonces como criterio para determinar el número de neuronas en la capa oculta, ésto es
para seleccionar en nuestro caso entre dos y tres neuronas en la etapa inicial y de ente dos a siete en la segunda (donde las
4
primeras dos son la misma de la primera etapa). Por supuesto a menor error de validación corresponde una mejor capacidad
de predicción. La Tabla 1 muestra los errores de validación obtenidos para cada RNA tratada en la primera etapa (dos y tres
neuronas en la capa oculta).
Método de pronóstico
Ingenuo
Promedio
Promedio móvil
ARIMA(0,1,1)
Suavizado exponencial simple
Regresión lineal
ARIMA(0,2,2)
Suavizado exponencial doble
SSE(dos neuronas)
0.950
2.965
0.260
0.680
0.924
2.552
1.409
1.501
SSE(tres neuronas)
0.954
3.028
0.223
0.948
0.954
1.881
1.210
1.245
Tabla 1. Suma de errores cuadrados (SSE) de validación para RNAs con dos y tres neuronas en la capa oculta.
En la Tabla 1 se puede ver que una red no domina totalmente a la otra, por lo cual se recurrió a una prueba de hipótesis para
dos medias donde nuestra hipótesis nula ( h0 : µ1 = µ 2 ) es que no hay una diferencia significativa entre el desempeño de
las redes, contra la alternativa de que sí hay, con un nivel de significancia de
un valor de
α = 0.01 . El estadístico de prueba resulta con
t14 = 0.244 , el cual se debe comparar con un valor de tablas de t0.005,14 = 2.947 . Dado que t14 < t0.005,14 ,
no se puede rechazar la hipótesis nula, por lo que concluimos que en el 99% de nuestros casos no hay una diferencia
significativa en utilizar una red con dos o tres neuronas en la capa oculta. Considerando este resultado se decidió en una
primera etapa utilizar las redes de dos neuronas en la capa oculta, ya que son modelos más simples que los de tres neuronas
ocultas. En una segunda etapa como ya se menciono se decidió utilizar RNAs con cuatro hasta siete neuronas en la capa
oculta para reducir el error de validación, sin embargo al mismo tiempo también se comparan con las RNAs de la primera
etapa. La Tabla 2 muestra los errores de validación obtenidos para las RNAs con dos hasta siete neuronas en la capa oculta.
Método
de pronóstico
Ingenuo
Promedio
Promedio móvil
ARIMA(0,1,1)
Suavizado
exponencial simple
Regresión lineal
ARIMA(0,2,2)
Suavizado
exponencial doble
SSE
(2neuronas)
0.950
2.965
0.260
0.680
SSE
(3neuronas)
0.954
3.028
0.223
0.948
SSE
(4neuronas)
0.949
3.017
0.288
0.964
SSE
(5neuronas)
0.929
3.448
0.294
0.326
SSE
(6neuronas)
0.855
2.961
0.216
0.282
SSE
(7neuronas)
0.854
3.028
0.232
0.413
0.924
0.954
0.866
1.055
1.086
1.045
2.552
1.409
1.881
1.210
0.874
0.704
1.764
1.505
0.884
1.092
1.373
1.073
1.501
1.245
0.882
1.111
1.180
2.228
Tabla 2. Suma de errores cuadrados (SSE) de validación para RNAs con dos hasta siete neuronas en la capa oculta.
En la Tabla 2 se pueden observar los errores de validación para cada red, donde al igual que en la primera etapa ninguna
supera a otra en todos los métodos, pero dado que la red para cada uno de los métodos tradicionales de pronóstico es
independiente entonces decidimos aquí elegir redes diferentes para cada uno, por lo cual en la Tabla 2 se puede observar en
negritas la RNA con menor SSE por método. Esto último quiere decir que para el método Ingenuo se utilizará la RNA con
siete neuronas en la capa oculta; para los métodos de Promedio, Promedio Móvil y ARIMA(0,1,1) se utilizará la RNA con
seis neuronas en la capa oculta; y para el caso de Suavizado Exponencial Simple, Regresión Lineal, ARIMA(0,2,2) y
Suavizado Exponencial Doble se utilizará la RNA con cuatro neuronas en la capa oculta.
Las RNAs seleccionadas se pueden ahora utilizar para predecir el MSE de los ocho métodos de pronóstico para series de
tiempo caracterizadas por una combinación específica de n y t.
5
4. Resultados de la aplicación del método propuesto
Para demostrar la utilización de las RNAs obtenidas anteriormente en el contexto del método propuesto se utilizaron las
series de validación. Cabe aclarar que los datos de validación no se encuentran memorizados en la RNA, por lo que se
preserva la objetividad de la evaluación.
La Tabla 3 muestra el MSE real para 22 series de tiempo con combinaciones de n y t particulares, la Tabla 4 el MSE
predicho por las RNAs seleccionada para cada método de pronóstico en la etapa inicial, la Tabla 5 para cada método de la
segunda etapa, esto para una serie de tiempo con características particulares.
SERIE
n
1
2
3
4
5
6
1
2
3
4
5
6
1
2
5
1
1
1
4
2
1
4
t
24
24
24
24
24
24
48
48
48
48
48
48
24
24
24
24
24
24
48
48
67
67
Ingenuo Promedio
0.05
0.39
0.09
0.27
0.22
0.48
0.20
0.43
0.14
0.34
0.16
0.36
0.07
0.27
0.09
0.27
0.13
0.30
0.10
0.26
0.10
0.27
0.11
0.23
0.01
0.44
0.02
0.39
0.41
0.08
0.13
0.32
0.09
0.53
0.08
0.38
0.41
0.01
0.01
0.40
0.40
0.01
0.03
0.30
MÉTODO DE PRÓNOSTICO
Suavizado
Promedio ARIMA exponencial Regresión
simple
móvil
(0,1,1)
lineal
0.10
0.05
0.05
0.04
0.11
0.08
0.08
0.06
0.18
0.16
0.17
0.11
0.18
0.14
0.14
0.10
0.15
0.11
0.11
0.07
0.13
0.13
0.13
0.08
0.07
0.05
0.05
0.05
0.08
0.07
0.07
0.06
0.10
0.09
0.09
0.07
0.09
0.07
0.07
0.06
0.09
0.07
0.07
0.07
0.06
0.07
0.07
0.05
0.09
0.01
0.01
0.01
0.15
0.01
0.02
0.10
0.29
0.20
0.08
0.08
0.13
0.10
0.13
0.06
0.15
0.08
0.09
0.06
0.12
0.07
0.08
0.05
0.14
0.12
0.01
0.01
0.12
0.01
0.33
0.00
0.04
0.05
0.01
0.01
0.07
0.07
0.03
0.03
ARIMA
(0,2,2)
0.03
0.07
0.12
0.13
0.09
0.14
0.05
0.05
0.08
0.07
0.09
0.07
0.00
0.00
0.09
0.07
0.07
0.05
0.01
0.00
0.01
0.03
Suavizado
exponencial
doble
0.04
0.08
0.17
0.13
0.11
0.12
0.05
0.06
0.08
0.08
0.08
0.07
0.00
0.00
0.08
0.17
0.25
0.08
0.01
0.00
0.01
0.03
ARIMA
(0,2,2)
0.06
0.07
0.07
0.08
0.08
0.09
0.06
0.07
Suavizado
exponencial
doble
0.07
0.07
0.08
0.08
0.09
0.10
0.05
0.06
Tabla 3. MSE real de series de tiempo para validación
SERIE
n
1
2
3
4
5
6
1
2
t
24
24
24
24
24
24
48
48
Ingenuo Promedio
0.09
0.43
0.09
0.42
0.11
0.40
0.15
0.38
0.19
0.35
0.21
0.32
0.08
0.26
0.08
0.26
MÉTODO DE PRÓNOSTICO
Suavizado
Promedio ARIMA exponencial Regresión
simple
móvil
(0,1,1)
lineal
0.15
0.10
0.09
0.04
0.15
0.10
0.15
0.06
0.15
0.11
0.16
0.07
0.15
0.12
0.16
0.08
0.15
0.12
0.16
0.11
0.15
0.13
0.15
0.12
0.08
0.07
0.06
0.04
0.08
0.07
0.08
0.06
6
3
4
5
6
1
4
48
48
48
48
67
67
0.08
0.08
0.08
0.09
0.09
0.09
0.26
0.26
0.26
0.26
0.26
0.26
0.08
0.08
0.08
0.08
0.07
0.07
0.07
0.07
0.07
0.07
0.06
0.06
0.09
0.09
0.08
0.08
0.05
0.07
0.06
0.06
0.06
0.06
0.04
0.06
0.07
0.08
0.08
0.09
0.06
0.07
0.06
0.07
0.07
0.08
0.05
0.06
Tabla 4. MSE predicho por las RNAs con dos neuronas en la capa oculta para cada serie de tiempo utilizada para
validación
SERIE
n
1
2
3
4
5
6
1
2
3
4
5
6
1
4
t
24
24
24
24
24
24
48
48
48
48
48
48
67
67
Ingenuo Promedio
0.09
0.44
0.09
0.42
0.11
0.41
0.15
0.39
0.17
0.36
0.20
0.33
0.08
0.26
0.08
0.26
0.08
0.26
0.08
0.25
0.08
0.25
0.08
0.25
0.08
0.26
0.08
0.26
MÉTODO DE PRÓNOSTICO
Suavizado
Promedio ARIMA exponencial Regresión
móvil
(0,1,1)
simple
lineal
0.13
0.06
0.09
0.05
0.17
0.10
0.15
0.07
0.18
0.12
0.16
0.09
0.18
0.13
0.16
0.09
0.17
0.13
0.15
0.09
0.16
0.12
0.15
0.08
0.07
0.07
0.06
0.04
0.08
0.07
0.08
0.06
0.08
0.07
0.09
0.06
0.08
0.07
0.09
0.06
0.08
0.07
0.09
0.06
0.08
0.07
0.09
0.06
0.07
0.07
0.05
0.05
0.07
0.07
0.07
0.06
ARIMA
(0,2,2)
0.06
0.08
0.10
0.10
0.10
0.10
0.07
0.07
0.07
0.08
0.08
0.08
0.07
0.08
Suavizado
exponencial
doble
0.06
0.11
0.11
0.10
0.10
0.10
0.06
0.06
0.06
0.06
0.06
0.06
0.06
0.06
Tabla 5. MSE predicho por las RNAs seleccionadas para cada método de pronóstico tradicional (ver Tabla 2) para cada
serie de tiempo utilizada para validación
En la Tabla 3 observamos el MSE obtenido de evaluar cada una de las series de tiempo usando cada método de pronóstico.
En las Tablas 4 y 5 podemos observar el MSE predicho por las RNAs seleccionadas en la primera y segunda etapa para
cada serie de tiempo utilizada para validación. Si observamos a detalle las Tablas 3, 4 y 5 vemos que algunas series de
tiempo tienen el mismo MSE para diferentes métodos, esto se debe a que algunos métodos son casos particulares de otros.
Así sucede, por ejemplo, con el método de promedio y el método de promedios móviles. Adicionalmente, los MSE reales de
ambos métodos presentan patrones similares. Cuando el MSE predicho es el mismo en una serie en particular para
diferentes métodos se considera a éstos en el mismo lugar de preferencia. Por ejemplo en la Tabla 4 para la serie con n=4 y
t=67 el modelo ARIMA(0.1.1), el método de Regresión Lineal y Suavizado Exponencial Doble tienen el mismo MSE y
ocupan ambos el primer lugar de preferencia. Queda entonces a criterio del usuario elegir uno si es necesario.
De los resultados de las Tablas 3 y 4 se extrajo la información presentada en la Tabla 6 y de las Tablas 3 y 5 se extrajo la
información presentada en la Tabla 7, que es un conteo de las veces en las que el mejor método de los ocho considerados
(basados en el MSE) se encontró entre los tres mejores predichos por la RNA. Esta información sirvió para determinar
estadísticamente qué tan buena fue la capacidad de predicción de las RNAs utilizadas en ambas etapas.
Real
Predichos
por la RNA
1
1
2
14
16
7
3
Otro
Total
12
7
49
Tabla 6. Número de métodos predichos por las RNAs de la primer etapa vs. Real
Real
Predichos
por la RNA
1
1
2
3
Otro
Total
17
15
10
7
49
Tabla 7. Número de métodos predichos por las RNAs de la segunda etapa vs. Real
De las Tablas 6 y 7 podemos concluir que en 42 de 49 casos, las RNAs predijeron la mejor técnica real de pronóstico dentro
de las primeras tres mejores, para las dos etapas del estudio. Sin embargo, las RNAs estudiadas en la segunda etapa
mostraron un mejor desempeño pues predijeron en 17 casos el primer método real, mientras que las RNAs de la primera
etapa lo hicieron en 14 casos. Con esta información podemos obtener un intervalo de confianza de 95% para proporciones
(θ), donde 0.751 < θ < 0.962, por lo tanto para el 95% de todos los casos podemos asegurar que del 75% al 96% de éstos,
las RNAs predecirán la mejor técnica real dentro de las primeras 3.
Se hace notar que en las Tablas 6 y 7 se toman 49 casos, siendo solamente 22 las series utilizadas para validar. La razón
obedece a lo explicado anteriormente, esto es que si el MSE real se repite 3 veces en el mejor lugar, se considera como tres
series en las cuales éste es primero.
5. Conclusiones y Trabajo a futuro
En este trabajo se propuso un método para seleccionar la mejor técnica de pronóstico tradicional para series de tiempo de
entre varias conocidas. Además se propuso una caracterización de series de tiempo cuyos resultados preliminares resultan
prometedores por su precisión y simpleza. Se describió también la construcción de las RNAs que se deben utilizar con el
método propuesto. Los resultados de validación permiten concluir que las RNAs predijeron satisfactoriamente la mejor
técnica dentro de las tres mejores técnicas en términos del MSE. Esta conclusión se corroboró con un buen nivel de
confianza obteniendo una prueba de hipótesis para proporciones, donde ésta comprende las series en las cuales su mejor
MSE real coincide con uno de los mejores tres MSE predichos por la RNA. Además se pudo observar que para una serie de
tiempo con características particulares puede haber más de un método de pronóstico con el mismo MSE tanto en los casos
reales como los predichos, lo que nos habla de que las RNAs utilizadas tienen buena capacidad de aproximación.
Tanto contar con un método fácil de implementar en MS Excel como con una caracterización precisa y simple de una serie
de tiempo son contribuciones de este trabajo que tienen importantes implicaciones de carácter práctico, pues con la
instauración del método propuesto se pueden esquivar los tres problemas identificados inicialmente con la generación de
pronósticos: (1) falta de entrenamiento estadístico, (2) falta de paquetería computacional adecuada y (3) falta de tiempo para
encontrar la mejor técnica.
Como una de las extensiones de este trabajo se tratará de ampliar el entrenamiento de las RNAs para abarcar un mayor
número de neuronas en la capa oculta de las redes utilizadas para probar la metodología propuesta. Con esto se pretende
verificar si se puede mejorar aún más la precisión al aumentar el número de neuronas en la capa oculta. Se pretende además
probar si el ordenamiento de los métodos depende del tamaño y la precisión de las RNAs o bien si el ordenamiento es en
realidad robusto ante variaciones de precisión. Un comportamiento robusto del ordenamiento podría llevarnos a economizar
8
el número de parámetros (pesos) que se utilizan en las RNAs, y por tanto, llevar a instauraciones computacionales más
eficientes.
Posteriormente, se planea también investigar la factibilidad de incorporar parámetros adicionales a la caracterización
propuesta a fin de mejorarla.
Referencias
Gupta J.N.D., Sexton R.S., and Tunc E.A., “Selecting Scheduling Heuristics Using Neural Networks”, Journal on
Computing, Vol. 12, No.2, Spring 2000, pp.150-162.
Hillier F.S. and Lieberman G.J., Introduction to Operations Research, 7th Edition, McGraw Hill, 2001, pp. 1009-1052
Hornik K., Stinchcombe M. and White H., “Multilayer feedforward networks are universal approximatiors”, Neuronal
Networks, Vol.2, No.5, 1989, pp.359-366.
Makridakis S., Wheelwright S.C., and Hyndman R.J., Forecasting Methods and Applications, 3rd Edition, John Wiley
&Sons, Inc., 1998, pp.42-45, 373.
9