CARACTERÍSTICAS. COMPONENTES BÁSICOS
Anuncio

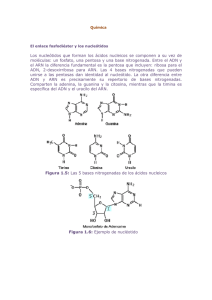
Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA 35 Tema 35.1 LOS ÁCIDOS NUCLEICOS: CARACTERÍSTICAS. COMPONENTES BÁSICOS: NUCLEÓTIDOS Y POLINUCLEÓTIDOS. TIPOS. ESTRUCTURA DE LOS ÁCIDOS NUCLEICOS. FUNCIONES E IMPORTANCIA BIOLÓGICA. GUIÓN - ÍNDICE 1. LOS ÁCIDOS NUCLEICOS 1.1. 1.2. 2. COMPONENTES BÁSICOS: ESTRUCTURA Y FUNCIÓN 2.1. 2.2. 3. 3.2. 3.3. Tel.: 91 564 42 94 El ADN 3.1.1. Estructura del ADN El ARN 3.2.1. ARNm 3.2.2. ARNt 3.2.3. ARNr 3.2.4. Otros ARN Ácidos nucleicos en mitocondrias y cloroplastos FUNCIONES E IMPORTANCIA BIOLÓGICA 4.1. 4.2. 4.3. CEDE - C/ Cartagena, 129 - 28002 Madrid Nucleótidos Polinucleótidos TIPOS DE ÁCIDOS NUCLEICOS: ESTRUCTURA Y FUNCIÓN 3.1. 4. Características generales Organización del genoma Características generales Replicación en eucariotas Replicación en procariotas © C E D E – www.cede.es Tema 35.2 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA 1. LOS ÁCIDOS NUCLEICOS Todas las células contienen la información necesaria para realizar distintas reacciones químicas mediante las cuales las células crecen, obtienen energía y sintetizan sus componentes. Esta información está almacenada en el material genético, el cual puede copiarse con exactitud para transmitir dicha información a las células hijas. Sin embargo estas instrucciones pueden ser modificadas levemente, es por eso que hay variaciones individuales y un individuo no es exactamente igual a otro de su misma especie (distinto color de ojos, piel, etc.). De este modo, podemos decir que el material genético es lo suficientemente maleable como para hacer posible la evolución. La información genética o genoma, está contenida en unas moléculas llamadas ácidos nucleicos. Existen dos tipos de ácidos nucleicos: ADN y ARN. El ADN guarda la información genética en todos los organismos celulares, el ARN es necesario para que se exprese la información contenida en el ADN; en los virus podemos encontrar tanto ADN como ARN conteniendo la información (uno u otro nunca ambos). 1.1. CARACTERÍSTICAS GENERALES Los ácidos nucleicos son macromoléculas formadas por C, H, O, N y P, y que están constituidas por unidades denominadas nucleótidos. En la célula, la información requerida para la replicación, indispensable para la continuación de la vida, se halla en el ADN o ácido desoxirribonucleico. Para que dicha información pueda expresarse se requiere otra molécula, el ARN o ácido ribonucleico. Ambos ácidos nucleicos están formados por la repetición de unas unidades denominadas nucleótidos. Cada nucleótido esta a su vez formado por tres moléculas distintas: − Una base nitrogenada. Existen 5 bases diferentes en los ácidos nucleicos: 2 purinas: adenina y guanina y 3 pirimidinas: citosina, timina y uracilo. − Una pentosa, que puede ser la D-ribosa o la 2-desoxi-D-ribosa. − Un ácido fosfórico, que en la cadena de ácido nucleico une dos pentosas a través de una unión fosfodiester. Esta unión se hace entre el C-3’ de la primera pentosa y el C-5’ de la segunda. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.3 Pentosas: Ácido ortofosfórico: Figura 1. Componentes estructurales de los ácidos nucleicos: bases nitrogenadas, pentosas y ácido ortofosfórico La unión entra la base nitrogenada y el azúcar se realiza por un enlace N-glicosídico entre un grupo amino de la base y el C-1 de la pentosa, originándose un nucleósido que se denominará: adenosina, guanosina, citidina, timidina o uridina, en función de la base nitrogenada presente (ver figura numeración y nomenclatura de nucleósidos). CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.4 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Figura 2 Figura 3. Numeración y nomenclatura de los nucleósidos El nucleósido se une al ácido fosfórico por un enlace fosfo-éster entre un OH del ácido y el C-3 o el C-5 del azúcar, originándose así el nucleótido, que se denominará con el nombre del nucleósido seguido de la palabra 3’-fosfato o 5’-fosfato. Ejemplo: Adenosín 5’-monofosfato. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.5 Figura 4 Los nucleósidos quedarán enlazados entre sí por los grupos fosfato que se unirán a la posición 3 de un azúcar y a la 5 de otro (enlaces fosfodiéster). CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.6 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Figura 5. Nucleótidos unidos en una cadena de ADN. De Karlson, 1973 Por tanto, los ácidos nucleicos están formados, como ya se ha dicho anteriormente, por la polimerización de muchos nucleótidos, los cuales se unen de la siguiente manera: 3’-pentosa-5’-fosfato--3’-pentosa-5’fosfato-----. A continuación daremos una idea general de éstos para posteriormente detallarlos por separado: Las diferencias a grandes rasgos entre ADN y ARN son: − En el ADN nunca aparece la base uracilo, mientras en el ARN nunca aparece timina. − La pentosa del ARN es la ribosa, mientras la del ADN es la 2-desoxi-D-ribosa. − El ADN se localiza en la cromatina en células eucariontes, mientras el ARN aparece en el nucléolo y en el citoplasma. − El ADN es la molécula portadora de la herencia, mientras el ARN participa en el proceso de síntesis de proteína con las “instrucciones” dictadas por el ADN. A nivel estructural las diferencias son considerables, por lo que se verán a continuación, pero en general, el ADN es bicatenario y el ARN monocatenario. En general y salvo excepciones que se analizarán, se puede decir que las diferencias fundamentales entre los dos ácidos nucleicos son las expresadas en la siguiente tabla. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Localización Bases Pentosa Estructura Tema 35.7 Funciones ADN Cromatina A,G,C,T 2-Desoxi-D-ribosa Bicatenaria Portador de la herencia ARN Nucléolo y Citoplasma A,G,C,U Ribosa Monocatenaria Síntesis de Proteínas Tabla 1 Figura 6. Nucleósidos presentes en los ácidos nucleicos. De Karlson, 1973 CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.8 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Figura 7. Ejemplos de nucleótidos 1.2. ORGANIZACIÓN DEL GENOMA El genoma es el conjunto de genes de una especie. Con la excepción de ciertos virus, que contienen un genoma de ARN, el resto de los genomas utiliza el ADN como depositario de la información genética. Sin embargo, debemos señalar algunas diferencias significativas en cuanto a la organización del genoma en procariontes y eucariontes. • En primer lugar, el ADN procarionte se presenta como una única molécula circular, en tanto el ADN eucarionte es de estructura lineal. Además las células eucariotas poseen usualmente más de una molécula de ADN en sus núcleos. Cada molécula corresponde a un cromosoma, cuyo © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.9 número es constante para todas las células de una misma especie (con excepción de los gametos). • El ADN eucarionte, por otro lado, se halla asociado íntimamente a diferentes proteínas, entre las cuales las histonas juegan el papel más importante en lo que respecta al empaquetamiento del ADN. Esta asociación a histonas no se verifica en el ADN procarionte, por lo cual se lo ha denominado “ADN desnudo”. • En las células eucariotas los cromosomas están confinados en el compartimiento nuclear, donde tiene lugar la transcripción, mientras que la traducción se localiza en el citoplasma; por lo tanto, ambos procesos se encuentran separados espacial y temporalmente. Esto permite que los transcriptos de ARN experimenten en el núcleo un proceso de maduración previo a la traducción. En las células procariotas, donde no existe la envoltura nuclear, el ADN está en contacto directo con el citosol, y los procesos de transcripción y traducción no se hallan separados en espacio ni en tiempo. Los ARNs no son sometidos a modificaciones postranscripcionales. • Los eucariontes tienen genomas mucho más grandes que los procariontes. Aunque el valor C (cantidad haploide de ADN de una especie) es muy variable entre los mismos eucariontes (ver tabla 2), es siempre notablemente mayor que el de los procariontes. No necesariamente un mayor valor C refleja una mayor complejidad genética (véase en tabla 2 valores de Salamandra y Homo sapiens). • En los procariontes se da una máxima economía de información: en casi todos los casos cada cromosoma contiene una sola copia de cualquier gen particular, y, con excepción de las secuencias reguladoras y señaladoras, prácticamente se expresa todo el ADN. En cambio, en toda célula eucariota parecería haber un gran exceso de ADN, o por lo menos de ADN cuyas funciones se desconocen por completo. Por ejemplo, la estimación del ADN “innecesario” en los seres humanos llega a ser tan elevada como el 95% del genoma. 4 x 106 E. coli Drosophila 1,4 x 108 Homo sapiens 2,87 x 109 Salamandra 8 x 1010 Tabla 2. Valor de C en las diferentes especies (expresado en pares de bases) 2. COMPONENTES BÁSICOS 2.1. NUCLEÓTIDOS Además de los nucleótidos detallados en el apartado anterior como constituyentes de los ácidos nucleicos, algunos nucleótidos cumplen funciones por sí mismos. Así, por ejemplo: CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.10 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA a) Nucleótidos que intervienen en las transferencias de energía: Se trata de moléculas que captan o desprenden energía al transformarse unas en otras. Así, el ATP desprende energía cuando se hidroliza, transformándose en ADP y fosfato inorgánico (Pi). Por el contrario, el ADP almacena energía cuando reacciona con el fosfato inorgánico y se transforma en ATP y agua. De esta forma se transporta energía (unas 7 kilocalorías por mol de ADP/ATP) de aquellas reacciones en las que se desprende (exergónicas) a aquellas en las que se necesita (endergónicas). Ejemplos de nucleótidos transportadores de energía: − AMP (adenosina-5'-monofosfato). − ADP (adenosina-5'-difosfato). − ATP (adenosina-5'-trifosfato). − GDP (guanosidina-5'-difosfato). − GTP (guanosidina-5'-trifosfato). Figura 8 © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.11 Figura 9 b) Nucleótidos que intervienen en los procesos de óxido-reducción. Estas moléculas captan electrones de moléculas a las que oxidan y los ceden a otras moléculas a las que a su vez reducen. Así, el NAD+ puede captar 2e- transformándose en su forma reducida, el NADH, y éste puede ceder dos electrones a otras sustancias, reduciéndolas y volviendo a transformarse en su forma oxidada, el NAD+. Así, se transportan electrones de aquellas reacciones en las que se desprende a aquellas en las que se necesitan. Ejemplos de nucleótidos transportadores de electrones: − NAD+ /NADH (Nicotinamida-adenina-dinucleótido) oxidado y reducido, respectivamente. − NADP+ /NADPH (Nicotinamida-adenina-dinucleótido-fosfato), oxidado y reducido. − FAD/FADH2 (Flavina-adenina-dinucleótido), oxidado y reducido. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.12 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Figura 10 c) Nucleótidos reguladores de procesos metabólicos. Algunos nucleótidos cumplen funciones especiales como reguladores de procesos metabólicos, por ejemplo el AMPc (adenosina3',5'-monofosfato) o AMP cíclico, en el que dos OH del fosfato esterifican los OH en posiciones 3 y 5 de la ribosa formando un ciclo. Este compuesto químico actúa en las células como intermediario de señalización celular. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.13 Figura 11 2.2. POLINUCLEÓTIDOS Dos nucleótidos van a poder unirse entre sí mediante un enlace ésterfosfato (fosfoéster). Este enlace se forma entre un OH del ácido fosfórico de un nucleótido y el OH (hidroxilo) del carbono número 3 del azúcar del otro nucleótido con formación de una molécula de agua. La unión de otros nucleótidos dará lugar a un polinucleótido. Es de destacar que en toda cadena de polinucleótidos el nucleótido de uno de los extremos tendrá libre el OH del azúcar en posición 3, éste será el extremo 3' de la cadena. El ácido fosfórico del nucleótido que se encuentre en el extremo opuesto también estará libre, éste será el extremo 5'. Esto marca un sentido en la cadena de polinucleótidos. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.14 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA 5’ 3’ Figura 12 3. TIPOS DE ÁCIDOS NUCLEICOS: ESTRUCTURA Y FUNCIÓN 3.1. EL ADN El ADN es el portador de la información genética y a través de ella puede controlar, en forma indirecta, todas las funciones celulares. Encontramos ADN en el núcleo de las células animales y vegetales, en los organismos procariontes, en organoides como los cloropastos y mitocondrias, como así también en algunos virus, a los que llamamos ADN-virus. 3.1.1. Estructuras del ADN Se considera estructura primaria del ADN la secuencia de nucleótidos anteriormente descrita. Considerando que en un ADN existen 4 nucleótidos diferentes, el número de moléculas distintas de ADN que puede haber es de 4n, siendo n el número de nucleótidos que contiene dicho ADN. Si tenemos en cuenta que dicho número puede ser del orden de millones, podemos inmediatamente comprender que el número de moléculas diferentes que se pueden formar es, prácticamente, ilimitado. La estructura secundaria consiste en la ordenación espacial de la primaria, que desde 1953 se conoce gracias a los trabajos de Watson y Crick, apoyados en los estudios de difracción de rayos X de Franklin y Wilkins. De todos es conocida la estructura en doble hélice del ADN, que se produce gracias a la unión a través de puentes de hidrógeno entre las dos cadenas, con la presencia de dos puentes en la pareja adenina-timina y de tres en la guanina-citosina (figura 13). La conformación más común © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.15 del ADN es la B (figura 15), hélice dextrógira que fue la descubierta por Watson y Crick y que presenta las características siguientes: − La hélice completa da una vuelta cada 10.4 pares de bases. − Una vuelta de hélice mide 3.4 nm. − Los nucleótidos adyacentes están desfasados unos de otros 34.6º. − El diámetro de la doble hélice es de 2.37 nm. Hoy en día se conocen también otras configuraciones del ADN, el A-ADN y el Z-ADN. Figura 13. Puentes de hidrógeno en las parejas A-T y C-G Figura 14. Representación bidimensional y tridimensional de una doble hélice de ADN CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.16 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Como se puede apreciar en la figura 14, en la doble hélice las dos hebras son antiparalelas, es decir, mientras una tiene el extremo 5’ libre, en el extremo complementario de la otra hebra queda libre el 3’. La hélice Z, levógira y más irregular, fue descubierta por Alexander Rich et al. Además de ser levógira, se puede diferenciar de la anterior en que mientras la B posee dos surcos que se alternan a lo largo de su trazado, uno mayor y otro menor, en la Z todos los surcos son iguales y existen 12 pares de bases cada vuelta de hélice. En la hélice A, también dextrógira, los pares de bases tienen una inclinación distinta. Mientras en la hélice B dichos pares de bases están horizontales y el eje vertical los atraviesa por el centro, en el modelo A están inclinados unos 20º respecto a un eje vertical. Cada vuelta completa contiene 11 pares de bases. De todas formas, en los últimos años se ha comprobado que tanto las estructuras A como las B constituyen modelos “aproximados”, con cierta variabilidad en la conformación. Figura 15. Diferencia entre configuración Z, levógira, más irregular y con un sólo tipo de surco muy profundo, y la B, dextrógira, más regular y con dos surcos distintos. De Darnell, 1988. La configuración A parece producirse a partir de la deshidratación de la B, que sería, por tanto, la estructura estable en solución. Debe ser la estructura que presentan los ARN de doble cadena, así como los híbridos ADN-ARN. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.17 La configuración Z es favorecida por la metilación de los residuos de citosina en la posición 5 de la misma. Se han detectado estas conformaciones en regiones de la cromatina que no se expresan, por lo que podría estar relacionada con la regulación de la biosíntesis de proteínas. Las dos hebras del ADN son antiparalelas, como se puede apreciar en la figura 14, es decir, una tiene el extremo 5’ terminal libre, mientras en la otra el extremo libre es el 3’. Para que el ADN se replique, debe desnaturalizarse, es decir, las cadenas tienen que separarse unas de otras. Esto puede conseguirse en el laboratorio al aumentar la temperatura, lo que hace que se rompan los puentes de hidrógeno entre los pares de bases. Teniendo en cuenta que los pares G-C están unidos por tres puentes de hidrógeno, mientras las parejas A-T sólo lo están por dos, podemos imaginar que la temperatura de desnaturalización, también llamada temperatura de “fusión” del ADN aumentará con la proporción G+C/A+T que contenga y será, por consiguiente, específica de cada ADN. Debemos considerar que dada la estructura de un ADN bicatenario, éste siempre cumplirá que el número de adeninas será igual al de timinas y que el número de guaninas será igual al de citosinas, ya que estas bases se presentan siempre apareadas. Matemáticamente se puede expresar como: A = T; G = C; es decir: A + G = C + T, y, por lo tanto A + G/C + T = 1, es decir Purinas/Pirimidinas = 1 Otra igualdad que se cumple en cualquier ADN bicatenario será: A + C = G + T, es decir: A + C/G + T = 1 Sin embargo el cociente G+C/A+T será variable para cada ADN y, como se ha indicado, de él dependerá la temperatura de desnaturalización. Existen otros factores que pueden influir en este proceso. La presencia de sales en una disolución, por ejemplo, desestabiliza la doble hélice y permite temperaturas de desnaturalización menores. La renaturalización, es decir, la reorganización de la doble hélice a partir de hebras sueltas, es más complicada, pero puede producirse bajo ciertas condiciones de laboratorio y con un enfriamiento muy lento, lo que será la base de muchos de los trabajos que se están realizando en la actualidad en el campo de la Ingeniería Genética. Hasta ahora hemos hablado de cadenas dobles de ADN y abiertas por los extremos. Sin embargo, existen también moléculas circulares, cerradas, como ocurre en las bacterias y en muchos virus, al igual que en mitocondrias y en cloroplastos. Este tipo de moléculas sufre una renaturalización más sencilla que la de eucariotas con el ADN abierto, ya que aunque se desnaturalicen quedan anudadas unas a otras y por lo tanto, las parejas A-T y G-C quedan enfrentadas. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.18 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA El ADN es siempre bicatenario excepto en algunos virus. Robert Sinsheimer, trabajando con el virus φ × 174, que infectaba a Escherichia coli, comprobó que su ADN no cumplía las reglas que se citaron anteriormente y que debían ser universales para cualquier ADN, por ejemplo la de Purinas/pirimidinas = 1. También observó que una solución de este ADN era mucho menos viscosa que la de los ADNs conocidos. Por último, los grupos aminos del ADN de dicho virus reaccionaban fácilmente con el formaldehido, mientras esto no ocurre en otros ADN. De esta forma se detectó la existencia de ADN de cadena simple, si bien para la replicación, y ya en el interior de la bacteria, aparecía ADN vírico de cadena doble, para la formación de la nueva cadena se utiliza un enzima presente en la célula huésped. Figura 16. A la izquierda, estructura en collar de perlas (De Dualde, 1987). A la derecha, estructura en solenoide, de Alberts et al. 1992. La estructura terciaria supone el enrollamiento del ADN y moléculas de histonas, estructura conocida como “collar de perlas” por el aspecto que presenta y que origina la cromatina. Las “cuentas del collar” son los nucleosomas, formados por 8 moléculas de histonas, 4 de las cuales, 2 H3 y 2 H4, forman un tetrámero y las otras (H2A y H2B), originan dos dímeros. Al octámero de histonas se enrolla un fragmento de 146 pares de nucleótidos de ADN que da casi 2 vueltas alrededor de aquellas (ver figura 7). Entre dos nucleosomas el fragmento de ADN posee un total de 54 pares de nucleosomas, pudiéndose denominar ADN espaciador. Aunque estas cantidades varían en las diferentes especies (Stryer, 1988). Esta estructura se denomina también de los 100 Å o de los 10 nm al ser éste el grosor que presenta. Existe además otra histona, la H1, que se asocia al ADN espaciador y que sostiene la estructura. Según Stryer (1988) existe una molécula de H1 que interacciona con la H2, en un modelo similar al de la figura. Según Rawn (1989) existen dos moléculas de H1 por cada nucleosoma, de posición desconocida. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.19 Figura 17. Modelo de empaquetamiento de la cromatina Pero esta fibra de 10 nm representa sólo un primer nivel en la estructura de la cromatina. A continuación se produce un enrollamiento de esta misma estructura originando una superior conocida con el nombre de estructura en solenoide o de los 300 angstroms y que supone un enrollamiento helicoidal de la estructura terciaria que da una vuelta cada 6 nucleosomas, por lo que cada vuelta del solenoide consta de aproximadamente 1200 pares de bases. Para la condensación en cromosomas parece, a la luz de los últimos descubrimientos, existir un eje proteico no histónico, al que se adhieren las fibras de 30 nm que formará bucles o lazos que volverán a condensarse hasta originar el cromosoma metafásico (figura 17). La única molécula de ADN presente en cada cromátida parece estar dividida en dominios llamados microválvulas que al microscopio electrónico de barrido tienen el aspecto de mazorcas de maíz y que se suceden en orden lineal y disposición helicoidal alrededor de una serie de proteínas no histónicas. 3.2. EL ARN El ácido ribonucleico se forma por la polimerización de ribonucleótidos. Estos a su vez se forman por la unión de: a) Un grupo fosfato. b) Ribosa, una aldopentosa cíclica. c) Una base nitrogenada unida al carbono 1’ de la ribosa, que puede ser citosina, guanina, adenina y uracilo. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.20 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA En general los ribonucleótidos se unen entre sí, formando una cadena simple, excepto en algunos virus, donde se encuentran formando cadenas dobles. La cadena simple de ARN puede plegarse y presentar regiones con bases apareadas, de este modo se forman estructuras secundarias del ARN, que tienen muchas veces importancia funcional, como por ejemplo en los ARNt (ARN de transferencia). Se conocen tres tipos principales de ARN y todos ellos participan de una u otra manera en la síntesis de las proteínas. Ellos son: El ARN mensajero (ARNm), el ARN ribosomal (ARNr) y el ARN de transferencia (ARNt). El tamaño de los ARNs es muy variable, siendo los mayores algunos de los ribosomales y el más pequeño, el ARN-transferente. 3.2.1. ARNm El ARN mensajero, que transmite el mensaje del ADN está formado por una cadena sencilla de nucleótidos, es decir, sólo posee estructura primaria. En su extremo 3’ presenta una secuencia terminal a base de la repetición de entre 200-250 adeninas (poli A), debido a la existencia de un enzima que no copia sino que sólo añade residuos de adeninas a este extremo. Esta unión no puede ser hidrolizada por muchas ribonucleasas y aparece ya en el ARN-hn (ARN recién formado) antes de su maduración a mensajero. Los ARNm son moléculas lineales de cadena simple en donde residen las instrucciones para la elaboración de un producto proteico. Los ARNm eucariotas y procariotas son esencialmente iguales en el sentido que presentan, una vez listos para la traducción, una secuencia continua de codones que se extienden desde el principio al fin del mensaje genético dictando con exactitud la secuencia lineal de aminoácidos de una cadena polipeptídica en particular. Esta secuencia se lee en la dirección 5' → 3'. El principio de la secuencia presenta un codón iniciador (casi siempre AUG), y el final de la secuencia o región codificadora es un codón de terminación o stop (UGA, UAA, UAG). Además de la región codificadora presenta sectores extra en los extremos 5' y 3' del mensajero denominados secuencias directora y seguidora respectivamente. Estas regiones no se traducen en proteína aun cuando forman parte del ARNm transcrito del gen. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.21 Figura 18. Correspondencia entre las regiones del gen eucariota, su transcrito maduro y la cadena polipeptídica Existen diferencias entre los ARNm procariota y eucariota: ARNm eucariota 1. La mayoría de los ARNm eucariotas presentan la secuencia del mensaje interrumpida, es decir coexisten a lo largo de la molécula sectores que codifican para la proteína llamados exones, con secuencias intercaladas sin información denominadas intrones. Figura 19. Estructura en mosaico del ARNm transcrito primario eucariota 2. Los ARNm transcriptos primarios sufren una serie de modificaciones antes de salir al citoplasma como ARNm maduro. Algunos cambios consisten en el agregado de moléculas en los extremos 5' y 3' llamados capping y poliadenilación. Capping: es la denominación del proceso por el cual se adiciona en el extremo 5' del ARNm una molécula de 7 metil-guanosina (un nucleótido metilado) a la que se conoce como cap (del inglés, capuchón). Esta molécula se agrega al ARNm naciente cuando éste alcanza, aproximadamente, los 30 nucleótidos de longitud. Por ser esta modificación simultánea a la transcripción se la considera co-transcripcional. La cap impide la degradación del ARNm inmaduro por nucleasas y fosfatasas nucleares. También participa en la remoción de intrones y en el inicio de la traducción. Poliadenilación: consiste en el agregado de alrededor de unas 250 adenosinas o cola poli A, en el extremo 3' del ARNm. El ARNm presenta una secuencia de nucleótidos específica CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.22 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA AAUAAA conocida como señal de poliadenilación. Una nucleasa corta al pre-ARNm a unos 20 nucleótidos después de la señal. Una vez liberado el pre-ARNm, la enzima poliA-polimerasa le agrega las adenosinas. Por otra parte, la ARN pol II continúa transcribiendo un tramo más del molde, para finalmente disociarse del gen. Este último tramo de ARN es totalmente infructuoso, pues resulta rápidamente degradado por nucleasas y fosfatasas. Las funciones de la cola poli A son: proteger el extremo 3' de la degradación y ayudar a los ARNm a salir del núcleo. Carecen de cola poli A los ARNm de histonas, ya que no presentan la señal de poliadenilación. Otra excepción la ejemplifican algunos genes que presentan más de una señal de poliadenilación. Cuando así ocurre, el mismo gen puede ser transcripto en dos productos diferentes, dependiendo de cuál sea la señal reconocida. Otra modificación significativa afecta a la secuencia codificadora, que sufre un acortamiento producto de la eliminación de los intrones, quedando como producto final los exones empalmados en secuencia continua. Este tipo de procesamiento se llama empalme (splicing) y fue descubierto en 1977. Para que se lleve a cabo este tipo de maduración del pre-ARNm se necesita una batería de ribonucleoproteínas nucleares: las RNPpn (snRNP, del inglés, small nuclear ribonucleoprotein). Estas partículas ricas en uridinas y diversas proteínas se denominan U1, U2, U4, U5, U6 y se combinan de a una por vez en los extremos de cada intrón (lindantes a sendos exones). El complejo resultante del ensamblado de las distintas RPNpn se denomina espliceosoma. La actividad enzimática residiría en los ARNpn del espliceosoma. Éstos serían los responsables de reconocer las secuencias señalizadoras de corte, escindir a los intrones y empalmar a los exones entre ellos, produciendo una molécula de ARNm maduro. Mecanismo molecular del splicing El corte de intrones y el empalme de exones deben ser muy exacto, pues de lo contrario un error pequeño, causaría un corrimiento del marco de lectura del ARNm. Los intrones son clivados del transcripto primario por las RNPpn que reconocen cortas secuencias conservadas dentro del intrón y en los límites con los exones. Estas secuencias, muy similares en todos los intrones estudiados, son: − Secuencia GU en el extremo 5' o sitio dador. − Secuencia AG en el extremo 3' del intrón o sitio aceptor. − Secuencias conocida como sitio de ramificación que se localiza en el interior del intrón. Estas secuencias participan en las reacciones de corte y empalme que ocurren en dos etapas: © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.23 En la primer etapa, una RNPpn reconoce al sitio dador y otra se enlaza al sitio de ramificación. El extremo 5' es clivado y ligado a un nucleótido(A) del sitio de ramificación. En la segunda etapa, se da el reconocimiento del extremo 3’ por parte de otra RNPpn y se produce el corte en el extremo 3' del intrón, seguido por el empalme de los dos exones. Así se libera el ARNm maduro del espliceosoma. El intrón queda eliminado en forma de lazo y será degradado posteriormente en el núcleo. Figura 20. Acción del espliceosoma Como citáramos anteriormente, la presencia del capuchón en el extremo 5' es requerida para que las RNPpn trabajen, no así la cola poli A. 3. Los ARNm maduros son monocistrónicos, es decir el sector codificador dicta la secuencia para una sola cadena polipeptídica. Figura 21. Estructura de la molécula de ARNm maduro eucariota ARNm procariota 1. Los ARNm procariotas presentan las secuencias codificadoras continuas, pues carecen de intrones. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.24 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA 2. No sufren modificaciones post-transcripcionales. 3. Muchos ARNm procariotas son policistrónicos, es decir que una sola molécula de ARNm contiene información para varias proteínas. Este ARNm contiene codones para la terminación y para la iniciación de la traducción entre las secciones codificadoras de proteínas, de modo que se traduce como varias moléculas de proteína distintas. Figura 22. Estructura de la molécula de ARNm procariota 3.2.2. ARNt Los ARNt son moléculas “adaptadoras” ya que interactúan por un lado con la cadena polinucleotídica (ARNm) y por el otro con los aminoácidos que formarán parte de la cadena polipeptídica, así alinean a los aminoácidos siguiendo el orden de los codones del ARNm. El ARN transferente que transporta los aminoácidos durante la síntesis de proteínas es una molécula en forma de hoja de trébol, la explicación de cuya estructura aparece en la siguiente figura. Se trata de moléculas pequeñas, que contienen entre 70 y 80 nucleótidos. Se unen al aminoácido por el extremo 3' libre y en su estructura tridimensional destacan tres lazos o bucles, formados por bases no apareadas y que muchas veces pueden presentar bases “raras”, es decir, distintas a las 4 que caracterizan a los ARNs, como ocurre en el ARN-t de la figura, que contiene dihidrouridina, ionosina, timina y pseudouridina. Muy frecuentemente las bases “raras” del transferente se deben a metilaciones de después de la síntesis del ARN-t. En el bucle opuesto al extremo de inserción al aminoácido se encuentra el anticodón, triplete complementario al del codón del mensajero y por el que se une al mismo. Es corriente que en el centro de la molécula haya una secuencia UUCG que sufra transformaciones en los dos uracilos, de forma que el primero se metila originando una timina y el segundo pasa a pseudouridina, dando lugar a un fragmento que es muy similar en la mayoría de los ARN-t, como es el del bucle de la derecha de la representación de la izquierda de la figura. Estas zonas comunes, unidas a la similitud de la estructura tridimensional en la mayoría de los ARN-t, pueden hacer pensar en la existencia de lugares que poseen una misma función durante la traducción de las proteínas. En esta molécula se distinguen básicamente dos extremos: © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.25 − En el extremo 3' o extremo aceptor de la molécula se encuentra el trinucleótido CCA que representa el sitio de unión donde se liga el aminoácido. Esta unión es catalizada por una enzima aminoacil ARNt sintetasa específica y apropiada para cada uno de los 20 aminoácidos. − En el otro extremo de la “ele” se localiza un triplete de nucleótidos conocido como anticodón, cuya secuencia varía en cada tipo de ARNt. Cada anticodón es complementario de un codón del ARNm, aunque podría acoplarse a más de un codón (sinónimo). Por lo hasta aquí expuesto, podemos concluir que el ARNt debe poseer varias propiedades específicas: • Debe ser reconocido por una aminoacil ARNt sintetasa que lo una al aminoácido correcto. • Debe tener una región que actúe como sitio de unión para el aminoácido. • Debe tener una secuencia complementaria (anticodón) específica para el codón del ARNm correcto. Figura 23. Estructura del ARN-t. A la izquierda, estructura primaria en forma de hoja de trébol. A la derecha, diagrama mostrando la estructura espacial de dicha molécula. Existen 64 combinaciones posibles en que las 4 bases pueden ordenarse en tripletes o codones. De ellos, 3 son codones stop. Los 61 tipos restantes codifican para los 20 aminoácidos, existiendo para varios de ellos codones sinónimos (ver código genético). CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.26 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Los anticodones de los ARNt reconocen a los codones del ARNm, sin embargo no hay 61 tipos distintos de ARNt porque no se requiere la complementaridad exacta entre los 3 nucleótidos del codón y el anticodón. En muchos casos, mientras coincidan las 2 primeras bases del codón, hay suficiente “balanceo” en la tercera posición para permitir el acoplamiento con más de un tipo de nucleótido en el anticodón, de manera que existen aproximadamente 31 tipos de ARNt. Por lo tanto, no existe el mismo número de ARNs-t que de codones posibles (64), no sólo por la existencia de 3 codones que no codifican ningún aminoácido (codones de terminación), sino también porque el mismo anticodón puede reconocer más de un codón de los que codifican un determinado aminoácido debido al balanceo, fenómeno consistente en un apareamiento atípico en el que el tercer nucleótido del codón puede unirse al tercero del anticodón sin ser las típicas uniones A-U o G-C. La ionosina, por ejemplo es una base rara que puede interaccionar con adenina, con citosina o con uracilo y que es frecuente en esta posición. De esta manera anticodones con I en la tercera posición pueden aparearse con codones que en dicha posición tengan una de las tres bases mencionadas y que codifiquen para el mismo aminoácido. 3.2.3. ARNr Los ARNr, junto a proteínas, son los componentes de los ribosomas. Los ribosomas y los ARNt intervienen en la traducción de la información codificada en el ARNm por lo cual los primeros son considerados fábricas de proteínas. El ARN ribosomal posee una estructura terciaria que se produce al unirse espontáneamente con las proteínas ribosomales, originando las subunidades 30S y 50S de los ribosomas procariontes y las 40S y 60S de los eucariontes. Por tanto, cada ribosoma consta de dos subunidades: la subunidad mayor y la subunidad menor. El S indica unidades Svedberg que son unidades de sedimentación, directamente proporcionales al tamaño. La subunidad mayor contiene una depresión en una de sus superficies, en la cual se ajusta la subunidad menor. El ARNm se inserta en el surco formado entre las superficies de contacto de las subunidades. Dentro de cada ribosoma hay dos huecos llamados sitios A (aminoacídico) y P (peptídico), para el ingreso de los ARNt unidos a sus correspondientes aminoácidos. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.27 Figura 24. Sitios aminoacídico y peptídico del ribosoma Las subunidades ribosómicas trabajan conjuntamente para la síntesis proteica. La subunidad menor aloja al ARNm sobre el cual se acomodan los ARNt para que puedan unirse los aminoácidos que transportan. La subunidad mayor cataliza la unión peptídica de los aminoácidos gracias a la acción de la peptidil transferasa (enzima que forma parte de la estructura de esta subunidad). Si bien cumplen idéntica función, los ribosomas procariotas y eucariotas se diferencian en su tamaño, coeficiente de sedimentación, y en el tipo y número de moléculas de ARNr y proteínas que los forman: Figura 25. Comparación entre los ribosomas procariotas y eucariotas CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.28 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Los de procariontes son de menor tamaño, 70S y están formados por las dos subunidades antes mencionadas: 50S y 30S. Los de eucariontes son 80S y constan de una subunidad grande, 60S, y otra pequeña, 40S. En la subunidad grande de los ribosomas procariontes aparecen dos tipos de ARNs ribosomales, el 23S, de unos 2900 nucleótidos y el 5S, de sólo 120. El total de proteínas es de 34 que se nombran L1, L2... L34. En la subunidad pequeña de procariontes existe un solo ARN, 16S, con unos 1500 nucleótidos y 21 proteínas (S1...S21). La subunidad grande de un ribosoma eucarionte está formado por dos ARNs unidos, uno 28S y otro 5.8S, con 4800 y 160 nucleótidos respectivamente; y un tercer ARN separado de los anteriores, un ARN 5S como el de procariontes. El total de proteínas es de 49 (L1...L49). La subunidad pequeña de los eucariontes contiene un solo ARN de unos 1900 nucleótidos (18S) y 33 proteínas (S1...S33). 3.2.4. Otros ARN − Los ARN pequeños forman complejos con proteínas específicas dando lugar a la formación de partículas ribonucleoproteicas (RNP). Las RNP localizadas en el núcleo se denominan RNPpn: partícula ribonucleoproteica pequeña (sn RNP/s: small −pequeña−/n: nuclear RNP: ribonucleoproteica). Varios RNPpn conocidos como U1 a U6 participan en el procesamiento de los ARNm. Estas partículas forman un complejo multienzimático denominado espliceosoma, encargado de realizar cortes y empalmes en los ARNm transcritos primarios (splicing). Las RNP localizadas en citoplasma se denominan RNPpc: partícula ribonucleoproteica pequeña citoplasmática (scRNP/s: small/c: cytosolic-citosol-/RNP: ribonucleoproteica). La función más conocida de cualquier RNPpc es la que desempeña el complejo ARN/proteínas que componen la partícula de reconocimiento de la señal o SRP. Estas partículas participan en el reconocimiento de secuencias específicas de las proteínas de secreción, membranares y lisosomales, deteniendo su traducción hasta que el ribosoma en donde se están sintetizándose contacte con la membrana del retículo endoplasmático granular, en donde finalizan su traducción. − El ARN nucleolar, se encuentra asociado a diferentes proteínas formando el nucléolo. Una vez formado se fragmenta dando lugar a los diferentes tipos de ARNr. − Existen, además de los ARNs citados, otros de mayor tamaño, denominados ARN-hn o ARN heterogéneo nuclear que serán los que en un posterior proceso de maduración den origen al ARN definitivo. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.29 − Por otro lado, las ribozimas, son ARN con función catalítica. 3.3. ÁCIDOS NUCLEICOS EN MITOCONDRIAS Y CLOROPLASTOS Mitocondrias y cloroplastos contienen ácidos nucléicos. El ADN mitocondrial se encuentra en la matriz y contiene información para sintetizar un pequeño número de proteínas, trece, después de transcribirse a ARN-m mitocondrial y con la participación de ribosomas mitocondriales. En cambio, la mayoría de las proteínas existentes en la mitocondria, sobre el 95%, se han formado a partir de ADN nuclear por ribosomas citoplasmáticos, siendo posteriormente transportados a la mitocondria. El ADN mitocondrial humano es una molécula circular cerrada que ha sido secuenciada y que consta de 16569 pares de bases, siendo de un tamaño similar al de muchos invertebrados pero muy inferior al de levaduras o algunas plantas, que llegan a contener unos 78000 pares de bases. Las proteínas que codifican son, sin embargo, muy similares, lo que indica que la transcripción o el código genético son diferentes. El ADN mitocondrial transcribe para la formación, además de los 13 ARNs-m mitocondriales, de dos ARNs ribosomales que forman parte de los ribosomas de la mitocondria: 12S y 16S, y de 22 ARNs-t, más pequeños que los citoplasmáticos (entre 59 y 75 nucleótidos), para sintetizar las 13 proteínas. Se propusieron varias hipótesis para tratar de explicar el menor número de transferentes mitocondriales que citoplasmáticos. La que resultó verdadera fue que tenían una forma distinta de reconocer los codones. El código genético perdía así parte de su “universalidad”, debido a cambios en la posición de “balanceo” para los anticodones que se unirán a codones que codifican para algunos de los aminoácidos. Además existen codones de terminación, que por tanto no se unen a ARN-t, que no lo son en el núcleo: AGA y AGG. El ADN de los cloroplastos está menos estudiado. Las proteínas del cloroplasto pueden, como las mitocondriales, codificarse a partir de ADN nuclear o ADN del cloroplasto. El ADN del cloroplasto es mucho mayor que el mitocondrial, existiendo además múltiples copias por orgánulo. Pueden tener más de 100000 pares de bases y codifican para un mayor número de proteínas, además de al menos 27 ARNs-t y 5 ARNs-R (23S, 16S, 5S, 4.5S y 27S). Es curioso que en algunas plantas, como la espinaca existen repeticiones invertidas de las regiones que codifican para ARN-r, por lo que aparecen dos copias de éste. 4. FUNCIONES E IMPORTANCIA BIOLÓGICA En cada apartado de los tratados anteriormente se ha ido detallando las funciones que desempeñan cada uno de los ácidos nucleicos y de sus componentes estructurales. Además, dada su importancia biológica como portadores de la información genética solo quedaría por citar los principales mecanismos a partir de los cuales el ADN desempeña esta importante actividad: replicación, transcripción y traducción del mensaje genético. Asimismo, resulta interesante recalcar la importancia del código genético a la hora de entender y descifrar el mensaje genético. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.30 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica). Esta duplicación del material genético se produce de acuerdo con un mecanismo semiconservador, lo que indica que las dos cadenas complementarias del ADN original, al separarse, sirven de molde cada una para la síntesis de una nueva cadena, complementaria de la cadena molde, de forma que cada nueva doble hélice contiene una de las cadenas del ADN original. Gracias a la complementariedad entre las bases que forman la secuencia de cada una de las cadenas, el ADN tiene la importante propiedad de reproducirse idénticamente, lo que permite que la información genética se transmita de una célula madre a las células hijas y es la base de la herencia del material genético. Aunque los principios generales de la duplicación o replicación del ADN son sencillos y pueden considerarse como consecuencia directa de su estructura, el proceso requiere una maquinaria compleja que contiene una gran cantidad de enzimas y proteínas que actúan en conjunto. La duplicación del ADN se produce previo desenrollamiento de las dos cadenas de la doble hélice, usando cada una como molde para sintetizar las nuevas cadenas. Figura 26. Esquema de la duplicación del ADN La molécula de ADN se abre como una cremallera por ruptura de los puentes de hidrógeno entre las bases complementarias liberándose dos hebras y la ADN polimerasa sintetiza la mitad complementaria añadiendo nucleótidos que se encuentran dispersos en el núcleo. De esta forma, cada nueva molécula es idéntica a la molécula de ADN inicial. La replicación empieza en puntos determinados: los orígenes de replicación. Las proteínas iniciadoras reconocen secuencias de nucleótidos específicas en esos puntos y facilitan la fijación de otras proteínas que permitirán la separación de las dos hebras de ADN formándose una horquilla de replicación. Un gran número de enzimas y proteínas intervienen en el mecanismo molecular de la replicación, formando el llamado complejo de replicación o replisoma. Estas proteínas y enzimas son homólogas en eucariotas y arqueas, pero difieren en bacterias. La transcripción consiste en la síntesis de moléculas de ARN a partir de ADN que se utiliza como molde. Se realiza con la ayuda de enzimas denominadas ARN-polimerasas. En bacterias existe una sola ARN-polimerasa, mientras en eucariontes existen tres diferentes, I, II y III que sintetizan ARNs diferentes, de mayor a menor tamaño respectivamente. También existen polimerasas específicas en mitocondrias y en cloroplastos. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.31 La transcripción es tan sólo el primer paso de la expresión genética. Los ARN obtenidos, ARN mensajeros, son, como su nombre lo indica, meros transportadores de información que aún debe ser decodificada, información que debe traducirse en la síntesis de una proteína. La traducción constituye el segundo paso de la expresión genética propuesto en el dogma central de la biología molecular. Figura 27. Dogma central de la biología molecular ¿De qué manera la estructura de una molécula de ARNm lleva implícita la información para construir una proteína? La información reside en la secuencia de bases y está “escrita” en un código propio al que llamamos código genético. Muchas de las características del código genético fueron anticipadas en forma teórica a partir del descubrimiento del ARNm. Pero en el año 1961, Nirenberg y Matthaei desarrollaron una técnica que abrió el camino para que en los siguientes cuatro años el código genético fuera enteramente descifrado. Podemos pensar al código genético como un idioma. Los idiomas utilizan una cierta cantidad de letras, éstas se combinan para formar palabras, y cada palabra tiene un significado, designa a un objeto particular. En el código genético están presentes todos estos elementos: las letras son las 4 bases que forman las cadenas de ARN (A, U, C, G); las palabras son siempre agrupaciones de 3 letras o tripletes de bases, llamadas codones en la molécula del ARNm, y los objetos designados por dichas palabras son cada uno de los 20 aminoácidos que componen las proteínas. ¿Por qué las palabras-codones se forman con 3 bases? Si cada palabra constara de 1 base, habría 4 palabras, y si se formara con 2, las palabras posibles serían 16. En ninguno de los casos alcanzarían para designar a todos los aminoácidos. Pero se pueden obtener 64 combinaciones diferentes si las bases se combinan de 3 en 3; 64 codones son más que suficientes para nombrar a los 20 aminoácidos. ¿Cuál es la función de los 44 codones restantes? Así como en cada idioma existen palabras distintas con un mismo significado, el código genético emplea codones diferentes para nombrar a un mismo aminoácido. La mayoría de los aminoácidos están codificados por más de 1 codón: existen codones sinónimos. La presencia de codones sinónimos en el código genético habla de una degeneración, característica que, lejos de resultar desventajosa, reporta a las células sus beneficios. Los codones sinónimos son muy similares entre sí, por lo tanto la sustitución de una sola base en un codón frecuentemente da por resultado otro codón que especifica al mismo aminoácido. Por otra parte, aminoácidos de propiedades semejantes son codificados por codones semejantes. Si en una proteína se reemplaza un aminoácido hidrófobo por otro de iguales características a consecuencia de una mutación, las chances de que el cambio sea viable son mayores. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.32 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Esta flexibilidad del código no debe confundirse con ambigüedad: el código no es ambiguo, en tanto cada codón especifica a uno y sólo a un aminoácido. Así no da lugar a error en el momento de ser traducido. Los codones que codifican aminoácidos suman en total 61. Los 3 codones que no especifican ningún aminoácido, UGA, UAG y UAA, actúan como señales de terminación en la traducción o síntesis de proteínas. Son llamados codones de terminación o stop. La tabla del código que se halla a continuación es válida para los seres vivos más diversos: el hombre, las bacterias, las levaduras, las plantas. El código genético es universal, lo cual da prueba de que todos los organismos comparten un mismo origen. Una de las contadas excepciones a la universalidad del código es el ADN mitocondrial, en el cual algunos codones son leídos de manera diferente. Figura 28. El código genético © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.33 RESUMEN Figura 29. Mapa conceptual del tema Los ácidos nucleicos son macromoléculas formadas por C, H, O, N y P, y que están constituidas por unidades denominadas nucleótidos. Un nucleótido es una molécula formada por la unión de una pentosa: ribosa o desoxirribosa, un ácido fosfórico y una de las 4 bases nitrogenadas siguientes: Adenina, guanina, citosina, timina o uracilo. Existen dos ácidos nucléicos diferentes: el ADN y el ARN, que se diferencian en su estructura, localización celular y funciones. A nivel de estructura primaria el ADN está formado por nucleótidos de 2-desoxirribosa y nunca presenta uracilo entre sus bases. El ARN tiene ribosa como azúcar y nunca contiene timina (puede aparecer como elemento minoritario en algún ARN transferente). La estructura secundaria también es diferente. El ADN posee estructura en doble hélice y es, excepto en algunos virus, bicatenario. El ARN que salvo en algunos virus es monocatenario, presenta una estructura distinta según se trate de ARN mensajero, transferente o ribosomal. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.34 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA La doble hélice de ADN sufre una serie de enrollamientos, en unión a histonas, que permiten su condensación en cromatina y en cromosomas. El ADN eucariota aparece en el núcleo celular, formándose a partir de un proceso denominado replicación. El ARN eucariota se sintetiza en el nucléolo y en la cromatina nuclear descondensada gracias a una transcripción del ADN, para madurar y salir al citoplasma con posterioridad. Los retrovirus realizan un proceso de transcripción inversa por el que sintetizan una molécula de ADN bicatenario a partir de su ARN, ya en el interior de la célula infectada. El ADN es la molécula que contiene los factores hereditarios, mientras las distintas moléculas de ARN- van a participar en la traducción del mensaje genético del ADN, traduciéndolo a proteínas que participarán en la adquisición de un fenotipo determinado. El dogma central de la Genética molecular, con las excepciones debidas a descubrimientos posteriores, como la existencia de una transcripción inversa en retrovirus, dice que el ADN posee capacidad de autoduplicación y que es capaz de transcribirse a ARN, molécula que codificará una proteína determinada. Todos estos procesos son catalizados por enzimas específicas. Enz. →→→ ↑ ↓ ↑ ADN ⇒⇒ ARN ⇒⇒ Proteínas ↓ ←←← Enz. Enz. El mecanismo de traducción se produce gracias al código genético que se caracteriza por: − El código genético consta de 64 codones o tripletes de bases. 61 codones codifican aminoácidos. − 3 codones funcionan como señales de terminación. − El código no es ambiguo, pues cada codón especifica a un solo aminoácido. − El código es degenerado, ya que un aminoácido puede estar codificado por diferentes codones. − Es universal, debido a que sus mensajes son interpretados de la misma forma por todos los organismos. − Utiliza un marco de lectura establecido al inicio de la traducción y no lo modifica. − No se produce solapamiento de codones. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA Tema 35.35 BIBLIOGRAFÍA COMENTADA • ALBERTS, B.; BRAY, D.; LEWIS, J.; RAFF, M.; ROBERTS, K.; WATSON, J. (2004). Biología molecular de la célula. 3ª ed. Barcelona: Omega. • DARNELL, J.; LODISH, H.; BALTIMORE, D. (1988). Biología celular y molecular. Barcelona: Labor. Los libros de Alberts y Darnell son, probablemente, los mejores y más actualizados sobre biología celular. Son muy útiles al tratar todos los aspectos de este tema desde un punto de vista ligado a la célula y con una profundidad y un grado de actualización máximos. • FENSELFELD, G. (1895). ADN. Barcelona: Investigación y Ciencia, 111, 24-34. • DARNELL, J. (1985). ARN. Barcelona: Investigación y Ciencia, 111, 36-49. • MONTOYA, J.; ATTARDI, G. (1986). ADN mitocondrial humano. Barcelona: Investigación y Ciencia, 118, 60-69. • RENNIE, J. (1993). Los nuevos giros del ADN. Barcelona: Investigación y Ciencia, 200, 6875. Los artículos de Investigación y Ciencia del 85 y el 86 representan un resumen de lo que en este momento se conocía sobre los temas que indican los títulos. Más actual es el artículo del 93 que es una recopilación de los últimos descubrimientos sobre la estructura del ADN y del ARN. • RAWN, J.D. (2004). Bioquímica (II). Madrid: McGraw-Hill Interamericana de España (4ª edición). • STRYER, L. (2007). Bioquímica. Barcelona: Reverté (6ª edición). • LEHNINGER, A.L. (2006). Principios de bioquímica. Barcelona: Omega, última edición. El Rawn, el Stryer y el Lehninger son tres excelentes libros de Bioquímica que con su modernidad y su claridad en las explicaciones son muy útiles para profundizar en temas de Bioquímica. El Rawn dedica a la Genética molecular los temas 22-30, más de 300 páginas, todas ellas en el segundo tomo. • KARLSON, P. (1973). Manual de bioquímica. Barcelona: Marín. El Karlson es un libro más antiguo y por tanto algo desfasado, pero es muy útil para adquirir conocimientos generales sobre el tema. CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94 © C E D E – www.cede.es Tema 35.36 Profesores de Enseñanza Secundaria BIOLOGÍA Y GEOLOGÍA • MENSUA (Coord.) (1989). Biología C.O.U. Madrid: Anaya. • PANADERO, J.E.; VANRELL, J.; GRANDMONTAGNE, C. (1989). Biología C.O.U. Madrid: Bruño. • JIMENO, A.; BALLESTEROS, M.; PARDO, A.; UGEDO, L. (1990). Biología. C.O.U. Ed. Santillana. • TEIXIDÓ, F.; VELASCO, T. y otros (2009). Biología 2 Bachillerato. Mc Graw Hill. Los libros de texto de COU y Bachillerato presentan esquemas muy útiles para el alumno así como un tratamiento de los temas muy actualizado. PÁGINAS WEB DE INTERÉS • http://www.ehu.es/biomoleculas/cibert.htm En esta página web encontrareis información relacionada con todas las biomoléculas, tanto orgánicas como inorgánicas. Además, trata también los átomos e isótopos. • http://www.um.es/molecula Aula virtual de Biología, dedicada a la enseñanza de Biología para 2º de Bachillerato. © C E D E – www.cede.es CEDE - C/ Cartagena, 129 - 28002 Madrid Tel.: 91 564 42 94