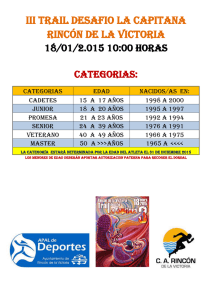
Estudio de los mecanismos que regulan la expresión y función del
Anuncio

Estudio de los mecanismos que regulan la expresión y función del ligando de muerte TRAIL en células T normales y leucémicas. Tesis Doctoral presentada por Jorge C. Morales Camino para optar al título de Doctor por la Universidad de Granada. Directora de la Tesis: María del Carmen Ruiz-Ruiz. Instituto de Biopatología y Medicina Regenerativa Universidad de Granada Granada, mayo 2008 Editor: Editorial de la Universidad de Granada Autor: Jorge Carlos Morales Camino D.L.: GR.1255-2008 ISBN: 978-84-691-4418-3 2 AUTORIZACIÓN PARA LA PRESENTACIÓN DE LA TESIS Dª. MARIA DEL CARMEN RUIZ RUIZ, PROFESORA DEL DEPARTAMENTO DE BIOQUÍMICA, BIOLOGÍA MOLECULAR 3 E INMUNOLOGIA DE LA UNIVERSIDAD DE GRANADA. CERTIFICA: que la presente tesis titulada “ESTUDIO DE LOS MECANISMOS QUE REGULAN LA EXPRESIÓN Y FUNCIÓN DEL LIGANDO DE MUERTE TRAIL EN CÉLULAS T NORMALES Y LEUCÉMICAS”, de la que es autor JORGE CARLOS MORALES CAMINO, superó el programa de Doctorado “Inmunología Celular y Molecular” y que ha sido dirigida bajo su dirección en el Departamento de Bioquímica y Biología Molecular 3 e Inmunología, Universidad de Granada. Revisado el presente trabajo, la directora considera que tiene la calidad científica necesaria para ser defendido ante el Tribunal que se designe al efecto, por lo que: AUTORIZA la presentación de la referida Tesis para su defensa y mantenimiento de acuerdo con lo previsto en la legislación vigente. Y para que conste y surta sus efectos en el expediente correspondiente, expido la presente certificación en Granada, 24 de Abril de 2008. Dra. Maria del Carmen Ruiz Ruiz 3 4 5 6 Esta tesis ha sido posible gracias a la ayuda y apoyo de mucha gente, tanto en el terreno personal como en el laboral…y para no seguir un orden estricto, iré dando las gracias conforme me vengan a la mente. Principalmente se lo agradezco a mis padres, a mi familia en general, porque me han aguantado los continuos cambios de humor y me han dando ánimo los últimos…29 años. Pero si hablamos de aguante, me parece que a quien más debería agradecer en esta tesis (y me estoy planteando hacerle un altar, la verdad) es a mi jefa. La Mari, antes conocida como Mari Carmen, la de ratos que hemos echado discutiendo, gritando, riendo, diseñando experimentos y, últimamente, soltándome broncas por ser tan coñazo. No me imagino haber hecho este trabajo sin ti, mezcla de jefa, mentora y hermana mayor. Si alguna vez pensaba en que esto no era lo mío, solo ver tu esfuerzo me levantaba el ánimo para seguir repitiendo experimentos. Me has educado casi tanto como persona como investigador, aunque la verdad, creo que en lo segundo tus enseñanzas han sido más productivas…. Cuando te conocí, tan joven, sonriente y al mismo tiempo tan seria y formal, ¡quien no querría hacer la tesis contigo! Bueno, creo que voy a parar, que esto se está pareciendo a una declaración más que a unos agradecimientos. De todas manera, no puedo quejarme del laboratorio en el que me ha tocado trabajar, entre Enrique, Ana e Ignacio, siempre he tenido una palabra amable, una broma en el momento justo o la ayuda que he necesitado cuando no he sabido como seguir....y entre mis compañeros, bueno, a algunos le cortaba la cabeza, pero a esos los incluiré en los agradecimientos para que “no se sientan mal”. Si es que sois más amigos que compañeros de trabajo: Irene, ¿con quien discutiré, iré al cine, de viaje o le lloraré a las tres de la madrugada cuando nos vayamos cada uno por un lado?; Ester, compañera de fiestas, siempre con la sonrisa, no olvides que siempre serás mi “favorita”; Zulema, es imposible ser más buena conmigo; Mª José, eres el mejor equipo que se puede tener, tanto en el laboratorio como delante de una caña; Diana, has llegado para quedarte con mi puesto como alborotador del laboratorio,¡eres mi alma gemela!; Raquel, tú y yo somos la prueba de que dos personas pueden ser amigos ¡sin tener ni una sola opinión en común!; a Api y Sara les agradezco en bloque, porque son una unidad, las risas, abrazos y las mil tonterías, ¡si es que he tenido mil madres en este laboratorio!; Juandi (esta es la única vez que te llamaré como te gusta), gracias por las fiestas que nos hemos pegado y las tardes juntos en el laboratorio, al principio de los tiempos; gracias a Domingo por no haber intentado matarme después de todas las cosas que te digo siempre, ¡lo tuyo si que es paciencia!; Claudia, no te puedes imaginar la tranquilidad que me transmite el mero hecho de escuchar tu voz; Gustavo, gracias por 7 ser tan buen amigo y por haberme ayudado al principio, cuando no sabía ni lo que era una pipeta; Lucía, me entran ganas de adoptarte todos los días y por ultimo, a Osmany y Girish, esto…um…gracias… Cómo me enrollo…bueno, por supuesto le agradezco a Abelardo y a Javier por haberme ayudado en los seminarios, con los reactivos, planteamiento de experimentos y hasta adopción en sus laboratorios. Y a toda la gente que ha trabajado con ellos, gracias por enseñarme técnicas y por vuestra amistad Men, Menchu, Gema, Rocío, Rosario, Griselda, Jose Antonio, Jose Manuel, Marién, Andreina, Celina y David. Y por último pero no por ello menos importante, a todos mis amigos que aunque no hayan estado nunca en el laboratorio, han tenido que escuchar la palabra “TRAIL” hasta la saciedad, en especial a Eva, que tiene que aguantarme todos los días en casa y que es la que mejor me da las palmaditas en la espalda cuando más lo necesito. 8 9 10 ÍNDICE 11 12 I. Introducción 1. Apoptosis ………………………………………………………………………….. 19 1.1 Apoptosis y Equilibrio ……………………………………………………….... 19 1.2 Maquinaria de muerte celular: Caspasas ……………………………………... 20 1.3 Rutas de inducción de apoptosis ……………………………………………..... 23 1.3.1 Ruta intrínseca ………………………………………………………... 23 1.3.2 Ruta extrínseca ……………………………………………………….. 29 1.4 Regulación de la apoptosis …………………………………………………..... 32 2. TRAIL y sus receptores …………………………………………………………. 35 2.1 TRAIL …………………………………………………………..…………….. 35 2.2 Receptores de TRAIL ……………………………..………………………...... 35 2.3 Regulación de la expresión de TRAIL ………………………………………... 37 2.4 Papel fisiológico de TRAIL ………………….………………………………... 39 3. TRAIL en terapia antitumoral ……………………….………………….….….... 41 3.1 Regulación de la expresión de FLIP …………………….……………...……... 41 3.2 Ruta de MAPK ……………………………………….…………………...….. 43 3.3 Ruta de PI3K ………………………………………..….……………………... 44 3.4 Ruta de NF-κB ………………………………………....…………………….... 46 3.5 Regulación del ciclo celular……………….....……………….……………….. 48 3.5.1 Proteínas reguladoras del ciclo celular (CDKs) ……….……………... 48 3.5.2 Organización del citoesqueleto …………..…………..………..…….... 48 4. Epigenética ……………………….……...………………….…...………………... 49 4.1 Metilación del ADN ……………………...…………………………….……... 50 4.2 “Código de Histonas” ………………………….……………….……………... 51 4.2.1 Metilación de histonas ………….………………………………….…... 52 4.2.2 Ubiquitinación de histonas ………………………...…………………... 52 4.2.3 Fosforilación de histonas …………………..…………………………... 53 13 4.2.4 Poli-ADP-ribosilación de histonas ….…………………………………. 53 4.3 Acetilación de histonas …………………..……………………………..……... 54 4.3.1 Histona acetil transferasas (HAT) …………………..…..……………... 54 4.3.2 Histona deacetilasas (HDAC) …………………..…………….………... 55 4.4 Histona deacetilasas y cáncer …………………..……………………………... 60 4.4.1 HDAC y proliferación celular …………………..…..…..……………... 60 4.4.2 HDAC y diferenciación hematopoyética …………..………..…..……... 60 4.4.3 HDAC, angiogénesis y metástasis. ………………………….…..……... 61 4.4.4 HDAC y factores de transcripción …………………..…………..……... 61 4.5 Inhibidores de histona deacetilasas (HDACi) ………….…………..…..……... 63 4.6 HDACi y TRAIL …………………………………………………..…..……... 65 II. Materiales y Métodos ………………………….……...………………….…...…... 69 III. Objetivos ………..……………….……...………………….…...………………... 73 IV. Resultados 1. Regulación de la expresión de TRAIL en células T activadas …………..…….. 87 1.1 Activación del promotor de TRAIL en células T en respuesta al tratamiento simultáneo con ionomicina y PMA ......………..…..……………............................ 87 1.2 Regulación de la expresión y función de TRAIL por ciclosporina A en células T activadas ………………………………….….…………..…..……..…… 89 2. Mecanismos que regulan la resistencia a TRAIL en linfocitos T primarios humanos …...……………………….………….…....……………....... 91 2.1 Resistencia de linfocitos T primarios en reposo y activados a la inducción de apoptosis por TRAIL ……………………….......…….….…..……… 91 2.2 Expresión de proteínas involucradas en la ruta de señalización de 14 TRAIL en linfocitos T en reposo y activados ……………………....…………..….. 93 2.3 Efecto de diferentes moduladores de la sensibilidad a TRAIL en la resistencia de linfocitos T primarios humanos ……….……………....…………… 96 2.4 La inhibición de NF-κB sensibiliza a linfocitos T activados a la apoptosis mediada por TRAIL ………….....…………….…………..…..………… 98 2.5 BAY 11-7085 regula la expresión de receptores de TRAIL y de genes anti-apoptóticos en linfocitos T activados ….………………………...…… 104 3. Regulación de la sensibilidad a TRAIL en células T leucémicas …………..… 107 3.1 Efecto de diferentes agentes moduladores de la sensibilidad a TRAIL en células T leucémicas ………….……………………...….….…………..…..… 107 3.2 Regulación por VPA y LY 294002 de factores implicados en la ruta de señalización de TRAIL en líneas leucémicas T ………….……...….…. 111 3.3 Potenciación por HDACi de la apoptosis mediada por TRAIL en líneas leucémicas T sensibles ………….……..………….….…………..…..… 113 3.4 HDACi potencian la activación de caspasas y las señales mitocondriales mediadas por TRAIL en líneas leucémicas T …………...…….…. 118 3.5 Regulación por HDACi de proteínas involucradas en la ruta de señalización de TRAIL en células T leucémicas ………….…………………….... 121 V. Discusión …...………..………………………………….…………......…....…… 133 VI. Conclusiones …...…...……………………………………….….....……….….... 149 VII. Bibliografía ………………………….................................................………….153 VIII. Publicaciones…………………………….................................................……. 191 15 16 I. INTRODUCCIÓN 17 18 1. APOPTOSIS 1.1 Apoptosis y Equilibrio El concepto del Yin y el Yang surgido de la filosofía oriental nos recuerda la naturaleza dual del Universo. Dos fuerzas opuestas pero complementarias que rigen las reglas de la vida y que nos muestran que nada existe en estado puro. El concepto de muerte unida a la vida por lazos que se cruzan tejiendo una compleja trama se encuadrara dentro de este pensamiento. Pero no es necesario ponernos transcendentales…al fin y al cabo, los organismos pluricelulares han seguido esta norma desde su aparición sin ni siquiera tener consciencia de ello. La vida para todos los organismos metazoicos requiere la eliminación de células innecesarias, infectadas o dañadas. La renovación de tejidos, la angiogénesis, así como la homeostasis del sistema inmunológico, requieren de manera constante la muerte de algunas células para mantener la vida del organismo completo. Pero no somos organismos perfectos. Todos los procesos vitales llevan asociados errores y la regulación del plan de muerte celular no es una excepción. Enfermedades neurodegenerativas y autoinmunes son el resultado de un plan imperfecto en la búsqueda del equilibrio entre la vida y la muerte celular. Y la patología más común por ausencia de muerte celular es el cáncer. Las células neoplásicas acumulan alteraciones genéticas que les permiten sobrevivir, a pesar de la ausencia de factores de supervivencia y las condiciones de hipoxia y estrés oxidativo, y que con el tiempo determinan la expansión del tumor hasta formar una masa de células que han perdido su función, que sufren una proliferación y una diferenciación celular alteradas y que presentan una movilidad y capacidad invasiva incrementadas (Frisch and Screaton, 2001; Ionov et al., 2000; Reed, 1999; Tschopp et al., 1999). La eliminación de las células no funcionales, o disfuncionales en el caso de las diferentes patologías, es llevado a cabo por un mecanismo de muerte celular programada denominado apoptosis (Kerr et al., 1972) caracterizado por un conjunto de cambios bioquímicos y morfológicos (Wyllie et al., 1980) que incluye la condensación del citoplasma así como la compactación de la cromatina dando lugar a densos agregados que se deslocalizan para situarse junto a la membrana nuclear y que posteriormente serán degradados por endonucleasas en fragmentos oligonucleosomales de 180 pares de bases o múltiplo de éstos. De forma paralela tiene lugar la dilatación del 19 retículo endoplasmático dando lugar a formación de vesículas que confieren a la célula el fenotipo típico en forma de “burbujas” (zeosis). Por último, la célula se fragmenta en los denominados cuerpos apoptóticos que son fagocitados por macrófagos y otras células circundantes gracias a la exposición en superficie de marcadores como la fosfatidilserina, normalmente en la cara interna de la membrana plasmática de la célula íntegra (Hengartner, 2000), evitando así la propagación del material intracelular y su exposición al sistema inmune que conllevaría al desarrollo de una respuesta inflamatoria. IN D U C C IO N D E A P O P T O S IS CELULA NORM AL C o n d e n s a c ió n d e la c r o m a tin a D is m in u c ió n d e l v o lu m e n c e lu la r R E C O N O C IM IE N T O Y F A G O C IT O S IS CELULA EN A P O P T O S IS F r a g m e n ta c ió n n u c le a r F o r m a c ió n d e e s tr u c tu r a s p r o tu b e r a n te s D iv is ió n d e la c é lu la e n c u e r p o s a p o p tó tic o s CUERPO S A P O P T O T IC O S Figura 1. Cambios morfológicos de la célula durante el proceso de muerte por apoptosis 1.2 Maquinaria de muerte celular: Caspasas Las CASPASAS, Cysteine ASPartyl-specific proteASES, son las proteasas encargadas de llevar a cabo los cambios morfológicos citados para las células apoptóticas (Cryns and Yuan, 1998). Son una familia de cisteín-proteasas que tienen especificidad de corte en residuos de aspártico (Asp). Estas enzimas se encuentran expresadas de forma constitutiva en forma de proenzimas o zimógenos inactivos que requieren de proteolisis para activarse (Thornberry and Lazebnik, 1998). Todas las caspasas presentan características similares en cuanto a su estructura y especificidad por el sustrato. Los aminoácidos de la enzima que se unen al sustrato, denominados S4-S3- 20 S2-S1, reconocen una secuencia tetrapeptídica en las proteínas diana denominada P4-P3P2-P1 a la que cortan en el extremo carboxilo de P1. Esta secuencia se caracteriza por la absoluta necesidad del residuo de aspártico en la posición P1, mientras que el residuo en P2 es variable y en posición P3 se suele encontrar un residuo de glutamina. El residuo en P4 es siempre un aminoácido hidrofóbico, alifático o un residuo de aspártico. Los aminoácidos de los sitios S3 y S1 son prácticamente idénticos en las distintas caspasas, mientras que los que participan en la unión al sustrato, S4 y S2, varían significativamente originando distintas especificidades por el sustrato en las posiciones P4 y P2. Las caspasas están constituidas por un dominio N-terminal de longitud variable, seguido de una subunidad larga de unos 20 kDa y una corta de 10 kDa en la región Cterminal. Su actividad proteolítica reside en un residuo de cisteína que forma parte de una secuencia pentapeptídica conservada QACXG y se encuentra en la subunidad grande, mientras que los residuos que forman el sitio de unión para el sustrato se localizan tanto en la subunidad grande como en la pequeña, aunque el residuo dominante para la especificidad de sustrato se localiza en la subunidad pequeña. Su activación requiere de la escisión de estas tres subunidades para formar un tetrámero, integrado por dos subunidades largas y dos cortas, que presentará dos sitios activos de catálisis (Wolf and Green, 1999). Esta proteolisis tiene lugar en dos fases: en la primera se separa la subunidad grande junto con el prodominio, de la subunidad pequeña, y en la segunda fase este prodominio es escindido. La presencia de aspártico en los sitios de separación de las subunidades se relaciona con la capacidad de las caspasas para autoactivarse o ser activadas por otras caspasas dentro de la cascada apoptótica. Hasta la fecha han sido descritas 12 caspasas en humanos, denominadas caspasas -1 a -10, además de las caspasas-12 y -14 (Pistritto et al., 2002). La enzima denominada en un primer momento caspasa-13 fue posteriormente identificada como un homólogo bovino de la caspasa-4 humana (Koenig et al., 2001) y las caspasas -11 es una enzima murina homóloga a la caspasas -4 humana (Lamkanfi et al., 2002). Recientemente se ha descrito la denominada caspasa-15 en otras especies. Según su función dentro de la cascada de señalización apoptótica, las caspasas pueden clasificarse en iniciadoras o efectoras. Las caspasas iniciadoras son las primeras en activarse tras un estímulo apoptótico gracias a la presencia en su prodominio de motivos de interacción proteína-proteína que les permiten asociarse a proteínas adaptadoras con dominios homólogos. Existen dos 21 tipos de motivos de interacción con proteínas: dominios efectores de muerte (DED), presentes en las caspasas -8 y –10, y dominios de activación y reclutamiento de caspasas (CARD), en las caspasas -2 y –9. El reclutamiento de las caspasas iniciadoras por proteínas adaptadoras determinó la aparición de la teoría de activación por un mecanismo de autocatálisis por proximidad (Salvesen, 1999). Estudios más recientes han demostrado, sin embargo, que la activación de caspasas iniciadoras no requiere cortes proteolíticos sino un cambio conformacional que tiene lugar tras la homodimerización de las formas proenzimáticas monoméricas (Boatright et al., 2003). El conjunto de caspasas efectoras está integrado por las caspasas -3, -6 y -7, carentes de dominios DED y CARD, que se activan por procesamiento mediado por caspasas iniciadoras. Las caspasas efectoras son las encargadas de degradar los distintos sustratos celulares que provocan los cambios morfológicos y bioquímicos característicos de la muerte por apoptosis. Figura 2. Esquema de capasas. Con la flecha gruesa se muestra el primer corte proteolítico entre la subunidad grande y la pequeña. Las flechas finas indican otros sitios adicionales de corte. Los segmentos sombreados indican las regiones que corresponden a los bucles que constituyen la cavidad catalítica. (Adaptado de Shy, Y. Mol Cell 2002). Además de las caspasas implicadas en apoptosis existen otros miembros de esta familia, caspasas -1, -4, -5, -11 y –13, que participan en la respuesta inflamatoria. 22 1.3 Rutas de inducción de apoptosis Existen dos rutas interconectadas de activación del proceso apoptótico: - Ruta intrínseca o mitocondrial, disparada por distintos tipos de estrés como es el caso de la radiación y del tratamiento con drogas genotóxicas que causan daños en el ADN. - Ruta extrínseca, mediada por receptores de muerte de la superfamilia del factor de necrosis tumoral (TNF) y fundamental en procesos fisiológicos como la muerte celular inducida por activación (AICD) o la citotoxicidad mediada por linfocitos T CD8+ o células NK. 1.3.1 Ruta intrínseca La mitocondria juega un papel fundamental en esta ruta apoptótica al actuar como reservorio de proteínas de localización intermembranal cuya salida al citoplasma por permeabilización de la membrana mitocondrial externa (PMME) constituye el inicio de un conjunto de reacciones en cadena que va a tener como consecuencia la entrada en apoptosis de la célula en respuesta a estímulos tan diversos como la deprivación de factores de crecimiento, la sobreexpresión de c-Myc, el tratamiento con drogas quimioterapéuticas, actinomicina D, radiación ultravioleta (UV), estaurosporina, o glucocorticoides (Duan et al., 2003; Han et al., 2001; Munk et al., 2007; Sutherland et al., 2006; Wang et al., 2007; Zhou et al., 2005). Esta PMME suele ir acompañada de una pérdida del potencial de membrana mitocondrial. Dos mecanismos han sido propuestos para explicar la PMME: - La formación del poro de permeabilidad transitoria (PPT), que permitiría el paso de agua y moléculas de bajo peso molecular provocando la pérdida del equilibrio iónico así como el incremento en el volumen de la matriz mitocondrial por exceso de hidratación que desencadenaría la ruptura de la membrana mitocondrial externa. Este poro está formado, entre otros componentes, por el transportador de nucleótidos de adenina (ANT) a nivel de la membrana mitocondrial interna y el canal de aniones voltaje dependiente (VDAC) de la membrana externa. En condiciones fisiológicas es necesario para el 23 mantenimiento de la función energética mitocondrial (Mattson and Kroemer, 2003). - La formación de canales proteicos constituidos por la dimerización de distintos componentes pro-apoptóticos de la familia de Bcl-2 que actuarían directamente sobre la membrana mitocondrial externa, o bien de largos poros derivados de la asociación de dichas proteínas pro-apoptóticas de la familia de Bcl-2 con distintos componentes lipídicos (Green and Kroemer, 2004). Existe una tercera posibilidad surgida de la asociación de los dos modelos anteriores, en la que miembros pro-apoptóticos de la familia de Bcl-2, como Bax, interaccionarían con proteínas del PPT como VDAC para permitir la salida de citocromo C a través de la membrana mitocondrial externa (Shimizu et al., 1999), o ANT para formar canales iónicos en la membrana mitocondrial interna (Marzo et al., 1998). Figura 3. Mecanismos propuestos para la PMME. a) Apertura del PPT; b) Canal de Bax; c) Canal Bax/ANT; d) Poro lipídico o complejo proteolipídico. La figura a) pertenece a mecanismos de la primera clase, que implican al PPT y a la membrana mitocondrial interna. Las figuras b), c) y d) pertenecen a la segunda clase de mecanismos que implican a las proteínas pro-apoptóticas de la familia Bcl-2. 24 Como consecuencia de la PMME en la mitocondria tienen lugar los siguientes eventos: Destrucción de la cadena de transporte electrónico. Este proceso determina la pérdida de la capacidad celular para llevar a cabo la fosforilación oxidativa y, por tanto, provoca una caída en la producción de ATP, de manera que la célula no podrá llevar a cabo sus funciones metabólicas (Bradbury et al., 2000). Alteración del potencial de oxidación-reducción (redox). La mitocondria es la principal productora de anión superóxido en la célula. Existen distintos mecanismos que amortiguan la generación de especies reactivas de oxígeno (ROS) evitando su acción citotóxica, pero la alteración del potencial redox provoca la inactivación dichos mecanismos o disminuye su capacidad, incrementándose la peroxidación lipídica que originará la ruptura de las membranas celulares (Le Bras et al., 2005). Liberación de proteínas del espacio intermembrana de la mitocondria al citosol. Se trata de proteínas con capacidad para activar caspasas y nucleasas o neutralizar inhibidores citosólicos de la cascada apoptótica. Las más relevantes son: - AIF ( Apoptosis Inducing Factor). Flavoproteína de localización mitocondrial con capacidad de traslocación al núcleo en respuesta a estímulos apoptogénicos produciendo la fragmentación del ADN y la compactación de la cromatina (Wang et al., 2002b). - Endonucleasa G. Actúa de forma conjunta a AIF en la fragmentación del ADN nuclear. En ambos casos esta fragmentación es independiente de la actividad caspasa (Li et al., 2001). - Citocromo C. Por asociación con la proteína adaptadora Apaf-1, y en presencia de ATP, el citocromo c liberado de la mitocondria conlleva a la formación del complejo denominado apoptosoma donde se recluta y se activa la caspasa-9 iniciadora. (Zou et al., 1999). - Smac/Diablo. Proteína con capacidad de unión e inactivación de proteínas antiapoptóticas de la familia IAP (proteínas inhibidoras de apoptosis) que a su 25 vez mantienen inactivas a la caspasa-9 y a las caspasas efectoras –3 y -7 (Du et al., 2000). - Omi/HtrA2. Serín-proteasa con capacidad de unión a los IAPs de forma análoga a la proteína Smac. En ambas proteínas, Smac y Omi, se ha descrito la capacidad de inducción de apoptosis independiente de su interacción con las proteínas IAPs (Liston et al., 2003). Célula normal Célula apoptótica Mitocondria apoptosoma Núcleo Núcleo Figura 4. Activacióm mitocondrial tras el estímulo apoptótico. Las proteínas “sólo BH3” se unen a Bcl-2 y dejan el camino libre para que las proteínas tipo Bax formen canales en la membrana mitocondrial que permitan la liberación de citocromo c, Smac/DIABLO, AIF y Omi, entre otros factores. Con la salida del citocromo c se forma el apoptosoma, Smac/Diablo y Omi se unen a los IAPs, pudiéndose así activarse la caspasa-9 y caspasas efectoras. (Adaptada de Adams, J. Genes&Dev 2003). Como ya se ha mencionado anteriormente, proteínas de la familia de Bcl-2 juegan un papel fundamental en la activación y regulación de la ruta mitocondrial. La proteína antiapoptótica Bcl-2, que da nombre a toda la familia, recibió dicha denominación tras haber sido identificada inicialmente en linfomas B foliculares que presentaban la translocación t(14;18). Al translocarse el gen que codifica Bcl-2 desde el cromosoma 18 al 14, se ubica tras el promotor de la cadena pesada de las inmunoglobulinas, de forma que Bcl-2 se transcribe de forma constitutiva, bloqueando la muerte por apoptosis (Cleary and Sklar, 1987). Existe una veintena de proteínas homólogas que constituyen 26 la familia de proteínas Bcl-2, caracterizadas por presentar al menos uno de los cuatro dominios de homología denominados BH (BH1-BH4) (Ottilie et al., 1997). Distinguimos tres grupos en los que pueden clasificarse las proteínas de esta familia: Grupo I: Miembros anti-apoptóticos Grupo II: Miembros pro-apoptóticos tipo Bax Grupo III: Miembros pro-apoptóticos “BH3-only” Miembros pro-apoptóticos tipo Bax Este grupo se encuentra constituido por las proteínas Bax, Bak y Bok/Mtd, que presentan los dominios BH1-BH3. Bax y Bak presentan una amplia distribución mientras que Bok se localiza únicamente a nivel de los tejidos reproductores (Hsu et al., 1997; Krajewski et al., 1996), lo que determina que tan sólo los dos primeros puedan considerarse esenciales para el correcto desarrollo de la apoptosis celular, como se ha observado en distintas modelos murinos deficientes para Bax y Bak (Lindsten et al., 2000). En respuesta al estímulo apoptótico, estas proteínas forman canales de permeabilización en la membrana mitocondrial o interaccionan con las proteínas formadoras de canales provocando la PMME. Mientras que Bak y Bok se encuentran constitutivamente unidos a la membrana mitocondrial en células sanas (Griffiths et al., 1999), Bax requiere del estímulo apoptótico para modificar su extremo C-terminal hidrofóbico y traslocarse a la mitocondria, en cuya membrana externa se inserta de manera estable (Borner, 2003; Nechushtan et al., 1999). Por otra parte, la acción de estas proteínas pro-apoptóticas parece extenderse hasta el retículo endoplasmático, donde median la apoptosis en respuesta a estímulos que provocan la salida de calcio (Scorrano et al., 2003). Miembros pro-apoptóticos “BH3-only” A este subgrupo pertenecen proteínas como Bad, Bim, Blk, Hrk y Bid (Huang and Strasser, 2000), emparentadas entre sí y con el resto de miembros de la familia Bcl-2 tan sólo por presentar el dominio BH3. Parecen actuar como sensores y mediadores de la 27 apoptosis que permiten la transmisión de señales de muerte bien por unión y neutralización de los miembros anti-apoptóticos de la familia Bcl-2 (Cory and Adams, 2002) o por activación directa de Bax y Bak (Wei et al., 2000), pero que son incapaces de inducir apoptosis en ausencia de estos últimos (Zong et al., 2001). Bid se presenta como una proteína fundamental en la inducción de apoptosis al conectar las dos rutas de inducción de apoptosis, intrínseca y extrínseca, como más adelante explicaremos (Luo et al., 1998). Miembros antiapoptóticos Comparten con el resto de proteínas de la familia la presencia de dominios BH, de los que se requieren más de tres para llevar a cabo su función anti-apoptótica. Pertenecen a este grupo las proteínas Bcl-2, Bcl-XL, Bcl-w, Bfl1-1, Bcl-w y Mcl-1. Los dominios BH1-BH4 intervienen en las interacciones con otras proteínas de la familia y con moléculas localizadas en la cara citoplasmática de las membranas intracelulares como la membrana mitocondrial externa, retículo endoplasmático y envuelta nuclear. Los dominios BH1-BH3 forman un surco hidrofóbico estabilizado por BH4 (Petros et al., 2001) que le permite interaccionar con el dominio BH3 de los miembros proapoptóticos “BH3-only” y los “tipo Bax” de esta misma familia (Sattler et al., 1997). Bcl-2 es una proteína de 26 kDa que posee los cuatro dominios típicos BH1-BH4. Se localiza fundamentalmente en la cara citoplasmática de la membrana externa mitocondrial constituyendo una estructura en forma de poro que permite regular el flujo de sustancias entre este orgánulo y el citoplasma en respuesta a posibles daños. Bcl-XL es una de las proteínas más estrechamente relacionada con Bcl-2, tanto en su estructura como en su función. Posee los cuatro dominios BH y su peso molecular es de 30 kDa. Su sobreexpresión puede mediar una resistencia significativa a la muerte celular por apoptosis dependiente de deprivación de factor de crecimiento (Zinkel et al., 2006). Ambas proteínas actúan como inhibidores de la apoptosis formando heterodímeros con Bax y Bak e inhibiendo su función, evitando así la salida de citocromo c y otros factores apoptogénicos de la mitocondria (Yang et al., 1997). Por otra parte, su capacidad anti-apoptótica incluye la unión al extremo C-terminal de la proteína Apaf-1, evitando con ello la asociación y activación de la caspasa-9 (Hu et al., 1998). 28 Mcl-1 es una proteína de 37 kDa ampliamente expresada en distintos tipos de tejidos y que guarda una gran homología con Bcl-2, aunque presenta una capacidad antiapoptótica reducida respecto a dicha proteína cuando se une a distintos miembros proapoptóticos de la familia, como Bax, Bak, Bim, Puma y Noxa (Chen et al., 2005; Wang et al., 1998b). Aun así, Mcl-1 sufre una rápida degradación en respuesta a distintos estímulos apoptóticos, lo que revela su papel en la regulación de la supervivencia celular (Cuconati et al., 2003; Chao et al., 1998; Nijhawan et al., 2003). 1.3.2 Ruta extrínseca En situaciones tanto fisiológicas como patológicas, la célula puede entrar en apoptosis en respuesta al estímulo desencadenado por la unión de los denominados ligandos de muerte a sus receptores específicos. Se trata de proteínas transmembrana tipo II pertenecientes a la superfamilia del factor de necrosis tumoral (TNF), como FasL/CD95L o TRAIL (TNF-Related Apoptosis Inducing Ligand), que presentan en su región extracelular los denominados dominios de homología a TNF (THD) a través de los cuales se asocian a sus receptores (Bodmer et al., 2002). Estos ligandos son expresados por distintas células del sistema inmunológico como linfocitos T, células NK, monocitos y células dendríticas, participando y permitiendo la eliminación de células que han cumplido su función, células infectadas, autorreactivas y metaplásicas (Greil et al., 1998; Perlman et al., 2001; Singh et al., 1999; Yang et al., 2001; Zhang et al., 2004). Los receptores pro-apoptóticos a los que se unen estos ligandos son proteínas transmembrana tipo I de la superfamilia de TNF y reciben el nombre de receptores de muerte. Se caracterizan por presentar en su región intracelular los denominados dominios de muerte (DD) a través de los cuales va a transmitir el impulso apoptótico. Existen además receptores anti-apoptóticos o “decoy” en los que la unión del ligando no transmite la señal de apoptosis y entre los que podemos incluir receptores solubles (Gruss and Dower, 1995). Ambos tipos de receptores presentan en su región extracelular unos dominios ricos en cisteínas (CRDs) que se asocian con las regiones THD de los ligandos de muerte por medio de puentes disulfuro formando multímeros funcionales (Banner et al., 1993). 29 Ligandos Receptores Figura 5. Ligandos y receptores de la familia de TNF. En gris se representan los dominios ricos en cisteínas (CRDs); con un rectángulo negro se representan los dominios transmembrana; y finalmente, con un rectángulo sombreado se representan los dominios de muerte. La unión del ligando de muerte a su receptor pro-apoptótico va a tener como consecuencia la trimerización del receptor y el reclutamiento de proteínas citoplasmáticas adaptadoras con dominios DD homólogos al del receptor, para formar así el denominado complejo inductor de señales de muerte (DISC). Además de dominios DD, algunas de estas proteínas adaptadoras, como FADD, presentan dominios efectores de muerte (DED), que van a permitir el reclutamiento y activación de las caspasas iniciadoras -8 ó -10 a nivel del DISC (Kischkel et al., 2000). 30 FasL/CD95L Fas/CD95 FADD Bax, Bak DISC procaspasa-8, -10 Mitocondria Fragmentación de Bid tBid Casp-8*, -10* Bcl-2 Citocromo c Apaf-1 dATP/ATP Casp-3*, -7* Apoptosoma procaspasa-9 Casp-9* APOPTOSIS Figura 6. Señalización de apoptosis inducida por el sistema FasL/Fas en células tipo I y tipo II En función de la necesidad de la ruta intrínseca para la inducción efectiva de la apoptosis tras la activación de los receptores de muerte, las células pueden clasificarse en tipo I si no requieren de la vía mitocondrial o en tipo II si requieren la participación de este orgánulo (Scaffidi et al., 1998). En las células tipo I, las caspasas iniciadoras activadas a nivel del DISC a su vez activan directamente a las caspasas efectoras -3 y -7 que provocarán finalmente la muerte celular por apoptosis. En las células tipo II, la caspasa-8, además de cortar y activar a las caspasas efectoras, corta a la proteína “BH3-only” Bid. Tras el corte por la caspasa, los fragmentos C-terminal de 15 kDa y N-terminal de 7 kDa se asocian provocando un cambio en la conformación del denominado cBid ( Bid cortado), que se traslocará a la membrana mitocondrial provocando su permeabilización y así la liberación de factores apoptogénicos al citoplasma (Li et al., 1998; Lutter et al., 2001; van Gurp et al., 2003). Esta PMME está mediada, como ya se ha explicado, por la interacción de cBid con Bcl2 y Bcl-XL inactivando la función de estas proteínas anti-apoptóticas. Además cBid se une y activa directamente a Bax y a Bak (Eskes et al., 2000; Wei et al., 2000). Se ha postulado también las hipótesis de la homooligomerización de cBid para formar canales en la membrana externa mitocondrial, aunque este hecho solo se ha demostrado in 31 vitro(Grinberg et al., 2002), así como de su capacidad para desestabilizar la bicapa lipidica de la membrana, lo que determinaría la salida al citoplasma de elementos del espacio intermembrana (Oh et al., 2005). Sea cual fuere el mecanismo de acción de cBid, una vez liberados los factores pro-apoptóticos de la mitocondria se induce la formación del apoptosoma y la activación de la caspasa-9 iniciadora, que en este caso será la principal responsable de la activación de las caspasas efectoras (Zou et al., 1999). El requerimiento del corte de Bid y la participación de la mitocondria en las células tipo II parece deberse a una activación insuficiente de caspasa-8 en el DISC como para generar la suficiente activación de caspasas efectoras que desencadenen la muerte de la célula. Por otra parte, y de forma independiente a la colaboración entre ambas rutas, en los dos tipos de células la señal apoptótica se ve incrementada y prolongada gracias a la capacidad de la caspasa-3 efectora de activar a la caspasa-8 por un mecanismo de retroalimentación tanto a través de la caspasa -6 como directamente, siendo este último el principal mecanismo responsable de la potenciación de la apoptosis en algunas modelos de células tumorales resistentes la inducción de esta forma de muerte celular (Yang et al., 2006). 1.4 Regulación de la apoptosis. Debido al elevado numero de señales que pueden inducir el inicio de la muerte celular por apoptosis es necesaria la existencia de mecanismos que regulen de manera estricta la entrada en este proceso, de manera que no ocurra de forma accidental, y solo tenga lugar en aquellas situaciones en las que sea necesario para conservar la homeostasis del organismo. Alteraciones en los mecanismos de control de la apoptosis modifican la sensibilidad o resistencia de las células a la inducción de este tipo de muerte celular y pueden desencadenar patologías como el cáncer. Entre estos mecanismos podemos señalar el balance de receptores pro- y anti-apoptóticos (Zhang et al., 2000) y los niveles intracelulares de proteínas antiapoptóticas como FLIP o los miembros de las familias de IAP y Bcl-2. IAPs. Se han descrito hasta 8 proteínas inhibidoras de la apoptosis en humanos, con capacidad para evitar la actividad de las caspasas -3, -7 y -9 (Deveraux et al., 1997; 32 Shiozaki and Shi, 2004): XIAP, cIAP1, cIAP2, IAP asociado a melanoma (ML-IAP), proteína inhibidora de la apoptosis neuronal (NAIP), survivina y bruce (Wright and Duckett, 2005). Figura 7. Mecanismo de acción de XIAP Estas proteínas inhibidoras de la apoptosis con capacidad de unión e inactivación de caspasas tanto iniciadoras como efectoras llevan a cabo su acción a través de los denominados dominios BIR (Baculoviral IAP Repeat), 80 aminoácidos plegados alrededor de una atómo de Zinc y que reciben su nombre de la identificación por primera vez de estas proteínas en células infectadas por baculovirus incapaces de sufrir apoptosis mediada por caspasas. Pero es el dominio contiguo RING el encargado de la destrucción de la caspasa, pues actúa como una ligasa de ubiquitina que promueve la degradación por el proteasoma del propio IAP asociado a la enzima (Yang and Li, 2000). Junto al dominio RING, tanto en c-IAP1 como en c-IAP2 se localiza un dominio 33 CARD que sugiere que estas IAPs podrían regular directa o indirectamente el procesamiento de las caspasas a través de interacciones por dicho dominio. FLIP.- Flice Inhibitory Protein o FLIP de origen viral fue descubierto por primera vez en linfocitos T citotóxicos donde el virus trataba de mantener con vida a la célula infectada para lograr con éxito su etapa de replicación, evitando la apoptosis mediada por receptores de muerte (Bertin et al., 1997; Thome et al., 1997). En humanos, encontramos dos isoformas de la proteína obtenidas por procesamiento alternativo del ARNm y denominadas por su longitud FLIP largo (FLIPL), de 55 kDa, y FLIP corto (FLIPS), de 26 kDa (Hu et al., 1997). Ambas isoformas se caracterizan por presentar en su estructura dos dominios DED, necesarios para cumplir su función inhibidora de apoptosis, pues a través de éstos pueden asociarse por interacciones homeotípicas con la proteína adaptadora FADD y ser reclutadas en el DISC en lugar de las caspasas iniciadoras -8 y -10 (Goltsev et al., 1997). Si bien ésta constituye la estructura básica de la isoforma FLIPS, en el caso de FLIPL encontramos también un dominio pseudocaspasa inactivo que va a ser degradado a nivel del DISC como si de la auténtica caspasa -8/-10 se tratara, dando lugar a una fracción de 43 kDa que quedaría anclada al DISC impidiendo el reclutamiento de la auténtica caspasa al tiempo que se libera un fragmento de 12 kDa homólogo al de la caspasa iniciadora, evitando el inicio de la cascada de señales apoptóticas (Krueger et al., 2001; Scaffidi et al., 1999). Figura 8. Estructura de las isoformas de c-FLIP y de v-FLIP en comparación con procaspasa -8 34 2. TRAIL Y SUS RECEPTORES 2.1 TRAIL TRAIL (TNF-Related Apoptosis Inducing Ligand) es una proteína transmembrana tipo II perteneciente a la superfamilia de TNF (Wiley et al., 1995). Con 32 kDa de peso molecular, está formada por una fracción intracelular seguida de un dominio extracelular que presenta un 28% de homología con el de CD95L (Wiley et al., 1995). Este dominio extracelular puede ser liberado al medio, pudiendo así aparecer este ligando de muerte en forma soluble (Mariani and Krammer, 1998a). De igual forma, se ha descrito que linfocitos T activados pueden liberar al medio TRAIL como una proteína completa mediante vesículas (Monleon et al., 2001). Al igual que el resto de ligandos de muerte, TRAIL se une a sus receptores específicos en forma de homotrímero, cuya estabilidad y actividad biológica requieren de la presencia de un átomo de Zinc unido al residuo Cys230 localizado a nivel de los CRDs (Bodmer et al., 2000), regiones extracelulares ricos en cisternas que se asocian los ligandos de muerte por medio de puentes disulfuro formando multímeros funcionales (Banner et al., 1993). 2.2 Receptores de TRAIL TRAIL pude unirse específicamente a cuatro tipo de receptores transmembrana denominados TRAIL-R1 a -R4, cuyos genes codificantes se localizan en la región p2221 del cromosoma 8, lo que sugiere que evolucionaron de un precursor común (DegliEsposti, 1999), teoría avalada por la homología superior al 50% de las secuencias nucleotídicas de los mismos. (Degli-Esposti et al., 1997a). TRAIL puede unirse también a un receptor soluble denominado osteoprotegerina (OPG) con menor afinidad que al resto de receptores(Truneh et al., 2000), y que parece estar implicado en la inhibición de la osteoclastogénesis, así como en la resistencia a la apoptosis mediada por TRAIL en ciertos tumores, como en cáncer de próstata, por secuestro del ligando de muerte antes de su acción en la membrana de la célula tumoral (Holen et al., 2002). 35 Los receptores de TRAIL en membrana pueden ser clasificados en pro- y antiapoptóticos, en función de su capacidad para inducir la activación de la cascada apoptótica. Los receptores TRAIL-R1/DR4 (Pan et al., 1997b) y TRAIL-R2/DR5 (Walczak et al., 1997) son denominados pro-apoptóticos pues presentan en su región intracelular los dominio de muerte (DD) necesarios para transmitir la señal de apoptosis tras la unión del ligando (Cha et al., 2000). TRAIL Figura 9. Esquema de la estructura de los receptores de TRAIL. Los receptores TRAIL-R3/DcR1 (Degli-Esposti et al., 1997b) y TRAIL-R4/DcR2 (Degli-Esposti et al., 1997a) son conocidos como receptores anti-apoptóticos o “señuelo” debido a su incapacidad para inducir apoptosis tras la unión del ligando. TRAIL-R3 carece de dominio intracitoplasmático y transmembrana, anclándose a la membrana a través de un lípido glicosil-fosfatidil-inositol (Degli-Esposti et al., 1997b), y por lo tanto carece de DD. TRAIL-R4, por el contrario, sí que presenta regiones citoplasmática y transmembrana, pero posee un DD truncado que lo incapacita a la hora de ensamblar la maquinaria necesaria para la activación del proceso apoptótico, pero que le permite activar la ruta de supervivencia de NFκB tras la unión a TRAIL (Degli-Esposti et al., 1997a), hecho que también se ha descrito tras la unión del ligando de muerte a sus receptores pro-apoptóticos (Chaudhary et al., 1997). 36 El perfil de expresión de receptores pro- y anti-apoptóticos en membrana puede modular la susceptibilidad celular a la inducción de apoptosis vía TRAIL. Junto con la capacidad antes mencionada de activación de la ruta de NFκB, los receptores antiapoptóticos se postularon en un primer momento como los responsables de la resistencia a la muerte por el ligando, al secuestrarlo e impedir su unión a receptores pro-apoptóticos (Pan et al., 1997a). Sin embargo, este mecanismo de resistencia no aparece en todos los casos, como demuestran estudios más recientes donde se describen tipos celulares resistentes a TRAIL que no expresan receptores anti-apoptóticos (Kim et al., 2000). Otro mecanismo de acción anti-apoptótica conferido a los receptores TRAIL-R3 y -R4 consistiría en la formación de heterodímeros con el receptor pro-apoptótico TRAILR2 (Munoz-Pinedo et al., 2002), alterando el balance de señalización. 2.3 Regulación de la expresión de TRAIL. El gen codificante del ligando de muerte se encuentra localizado en el cromosoma 3, se compone de aproximadamente unas 20 kb y consta de cinco exones de 222, 138, 42, 106 y 1245 pb, separados por cuatro intrones de 8.2, 3.2, 2.3 y 2.3 kb respectivamente (Gong and Almasan, 2000; Wang et al., 2000a). Figura 10. Gen de TRAIL. H. Sapiens cromosoma 3, clon hRPK.44_A_1 (Accesion No. AC007051) (Gong, B. et al. Biochem. Biophys. Res. Commun. 278, 745-752 (2000) Su promotor carece de caja TATA o CAAT típicas, aunque contiene dos secuencias Sp1 que parecen ser las reguladoras del inicio de transcripción. Además contiene en la región proximal otros sitios de unión a factores reguladores de la transcripción como secuencias AP-1 o GAS (Gong and Almasan, 2000; Wang et al., 2000a). Numerosos trabajos han descrito la regulación de la expresión de TRAIL por interferones de tipo I 37 (IFN-α e IFN-β) o de tipo II (IFN-γ), tanto en células del sistema inmune (células T, NK, monocitos y células dendríticas) (Griffith et al., 1999; Kayagaki et al., 1999; Smyth et al., 2001) como en otros modelos de células normales y tumorales. Se ha propuesto la existencia de dos sitios ISRE cercanos al sitio de inicio de transcripción como responsables de la regulación de la expresión de TRAIL por interferón (Clarke et al., 2004). Figura 11. Secuencia del del promotor de TRAIL. También se ha demostrado que la activación de células T con anticuerpos antiCD3/anti-CD28, fitohemaglutinina (PHA), ésteres de forbol/ionomicina y en general cualquier estímulo que imite la activación del receptor de la célula T para el antígeno (TCR), conduce a un rápido aumento en la expresión de TRAIL tanto a nivel de ARNm 38 como de proteína en la membrana celular, permaneciendo la célula T activada resistente a la acción de dicho ligando (Jeremias et al., 1998). Esta inducción de TRAIL en linfocitos T activados parece estar regulada por el factor de transcripción NF-κB (Siegmund et al., 2001). De hecho se ha descrito la presencia de dos sitios de unión para dicho factor, en la región proximal del promotor de TRAIL, necesarios para la inducción de este ligando en respuesta a la activación de linfocitos T (Baetu et al., 2001). Artículos recientes también han propuesto una posible regulación de la expresión de TRAIL por NFAT, pero la contribución exacta de este factor de transcripción aún no se ha caracterizado (Liu et al., 2006). 2.4 Papel fisiológico de TRAIL Poco se conoce acerca del papel fisiológico de TRAIL in vivo, pero distintos estudios parecen indicar su participación en diferenciación, en la hematopoyesis y el mantenimiento de la homeostasis del sistema inmunitario y en la eliminación de tumores. TRAIL en diferenciación celular.- Osteoclastos y células intestinales. La diferenciación de los osteoclastos hasta constituirse como una célula madura parece estar relacionada con dos receptores considerados “decoy” de ligandos de muerte. El primero de estos receptores es RANK, ya que la unión de este receptor a su ligando, RANKL determina la activación y supervivencia de este tipo celular vía NF-κB (Fuller et al., 1998; Jilka et al., 1999). El otro receptor implicado en la diferenciación es OPG (osteoprotegerina), receptor soluble con capacidad de unión a TRAIL (Emery, 1998), así como a RANKL (Lacey et al., 1998). Este hecho parece indicar que TRAIL puede jugar un papel en el metabolismo óseo, sobre todo tras estudios recientes que muestran como TRAIL puede inhibir la osteoclastogénesis in vitro (Zauli et al., 2004). Por otra parte, es conocida la actividad de TRAIL a nivel de la proliferación y diferenciación del epitelio intestinal, ya que interviene en la regulación de la inflamación a nivel del tracto digestivo y la progresión de ciertos tumores (Strater et al., 2002). Durante el proceso de diferenciación celular desde las criptas a las vellosidades intestinales, se observa un incremento en la expresión de TRAIL así 39 como de sus receptores, que no provocan toxicidad en este tipo celular pero puede favorecer la renovación en caso de células infectadas o transformadas (Zauli and Secchiero, 2006). TRAIL en hematopoyesis.- Maduración y eritropoyesis. Los distintos linajes celulares presentan diferencias en cuanto al sistema formado por TRAIL sus receptores. Si bien los precursores CD34+ no presentan expresión alguna del ligando ni sus receptores (Zauli and Secchiero, 2006), se ha descrito que, tanto en monocitos como neutófilos, la maduración celular provoca la progresiva expresión de distintos receptores de TRAIL que van a favorecer su delección tras la entrada en senescencia (Lum et al., 2005; Renshaw et al., 2003). Por otro lado parece que el ligando de muerte interviene en el proceso de eritropoyesis puesto que los eritroblastos inmaduros presentan receptores pro-apoptóticos y son sensibles a TRAIL (Secchiero et al., 2004). TRAIL en la homeostasis del sistema inmunitario Se ha descrito que TRAIL media la apoptosis de linfocitos CD8+ una vez realizada su función (Zerafa et al., 2005), evitándose con ello la expansión de dichas células y el desarrollo de problemas de autoinmunidad, Estudios realizados en modelos murinos deficientes en TRAIL muestran que estos ratones sufren defectos en la apoptosis de timocitos que acaban traduciéndose en una mayor susceptibilidad a desarrollar enfermedades autoinmunes (Lamhamedi-Cherradi et al., 2003). TRAIL en supresión de tumores TRAIL es expresado por distintas células del sistema inmunitario, como linfocitos T, células NK, monocitos y células dendríticas (Griffith et al., 1999; Liu et al., 2001; Mariani and Krammer, 1998a). La estimulación de dichas células a través de interferones o del TCR, en el caso de los primeros, induce la expresión de este ligando y así se favorece o se estimula su capacidad de inducción de apoptosis sobre células tumorales (Takeda et al., 2002). Pero lo más relevante de este ligando de muerte, y lo que lo diferencia de otros ligandos de muerte de la familia de TNF, es su capacidad para inducir apoptosis de manera selectiva en células tumorales y no en células normales (Wiley et al., 1995). Si 40 bien existe un gran número de tumores resistentes a la apoptosis mediada por TRAIL (Griffith and Lynch, 1998), su estudiada efectividad en la erradicación de tumores en modelos murinos acompañada de la ausencia de citotoxicidad en humanos (Ashkenazi et al., 1999) confieren a TRAIL propiedades como un prometedor agente terapéutico en la lucha contra el cáncer. 3. TRAIL EN TERAPIA ANTITUMORAL La capacidad de TRAIL para inducir apoptosis selectivamente en células transformadas y la necesidad de terapias antitumorales menos agresivas que las utilizadas actualmente, han propiciado el desarrollo de numerosos estudios dirigidos al empleo de este ligando en la erradicación de tumores. En la actualidad, anticuerpos frente a los receptores pro-apoptóticos TRAIL-R1 y -R2 se encuentran en ensayos clínicos de fase I y de fase II tras comprobar su capacidad para inducir apoptosis en líneas tumorales y tumores primarios in vitro (Georgakis et al., 2005; Vulfovich and Saba, 2005). Este hecho es extensible al empleo del propio TRAIL recombinante, en forma de homotrímero soluble unido a un átomo de Zinc, tras haberse establecido su tolerancia por parte de algunas células no transformadas, como hepatocitos o células neuronales previamente descritas como sensibles al ligando en ciertos estudios (Kelley et al., 2001). Dada la existencia de un gran número de modelos tumorales resistentes a TRAIL, la mayor parte de los estudios se han centrado en el desarrollo de terapias combinadas con agentes que rompan la resistencia a este ligando. Entre dichos agentes podemos mencionar drogas genotóxicas, inhibidores de histona deacetilasas, inhibidores del proteasoma, así como compuestos inhibidores de distintas rutas de supervivencia. 3.1 Regulación de la expresión de FLIP Una de las piezas claves en la sensibilización de distintos tumores a la acción de TRAIL radica en la regulación de la proteína FLIP. Competidor natural del reclutamiento de caspasa-8 a nivel del DISC, FLIP se presenta como la barrera más 41 apical en la ruta de apoptosis mediada por TRAIL. Desde el descubrimiento de esta proteína (Irmler et al., 1997), distintos agentes se han propuesto como moduladores de la resistencia a TRAIL al provocar un descenso en los niveles de FLIP. Entre éstos, destacamos: Doxorubicina (DRX), antraciclina que inhibe a la enzima topoisomerasa II y además actúa como agente intercalante de la doble cadena de ADN, impidiendo de este modo la síntesis de ácidos nucleicos y con ello la progresión tumoral. Se emplea en el tratamiento de leucemia linfática aguda, leucemia mieloide aguda, linfoma de Hodgkin y carcinoma de mama entre otros. En algunos modelos, como en cáncer de próstata, se ha demostrado una sensibilización a TRAIL por doxorubicina mediada por el descenso en los niveles de FLIP (Kelly et al., 2002). Cicloheximida (CHX), inhibidor de la síntesis proteica en organismos eucariotas producido por la bacteria Streptomyces griseus. Actúa interfiriendo en la actividad peptidil transferasa del ribosoma 60S, bloqueando la elongación de la traducción. Su capacidad para sensibilizar a la muerte por apoptosis mediante regulación de FLIP se ha demostrado en distintos tipos de cáncer como el carcinoma renal (Brooks and Sayers, 2005) y CLL (Kang et al., 2003). Actinomicina D, antibiótico polipeptídico aislado de las bacterias de suelo del género Streptomyces que inhibe el proceso de transcripción. También en ciertos modelos, como leucemia linfática crónica B, se ha descrito la sensibilización a TRAIL por este compuesto a través de la regulación de FLIP (Olsson et al., 2001). Otros agentes que comienzan a ser empleados en la sensibilización de tumores a TRAIL por inducir una reducción en los niveles de FLIP, entre otros factores antiapoptóticos, son los denominados inhibidores de histona deacetilasa (HDACi) (Hernandez et al., 2001), cuyo mecanismo de acción aún no completamente caracterizado será explicado en capítulos posteriores. La expresión de FLIP se encuentra regulada a través de distintas vías de supervivencia celular como son la vía de la proteina quinasa II dependiente de calcio/calmodulina (CaMKII), la vía de las proteinas quinasas activadas por mitógenos (MAPK), la vía de la fosfatidil-inositol 3’ quinasa (PI3K), o a través del factor de 42 transcripción NF-κB. El empleo de inhibidores específicos de estas rutas provoca el descenso de los niveles de FLIP y así la sensibilización a TRAIL en distintos modelos tumorales (Xiao et al., 2005). 3.2 Ruta de MAPK Las proteinas quinasas activadas por mitógenos transmiten señales implicadas tanto en proliferación celular como en apoptosis a través de la fosforilación de sustratos como fosfolipasas, otras quinasas, factores de transcripción o proteínas del citoesqueleto. Existen tres subfamilias de MAPK, de las cuales sólo dos se encuentran bien caracterizadas: ERK1 y ERK2 Quinasas activadas por factores de crecimiento o a través de la proteina quinasa C (PKC), intervienen en mitosis, meiosis, diferenciación, angiogénesis y apoptosis, así como en la activación de factores de transcripción relacionados con la supervivencia celular como NF-κB (Hagemann and Blank, 2001; Johnson and Lapadat, 2002; Lee and McCubrey, 2002). La actividad ERK1/2 se encuentra aumentada en ciertos tumores, convirtiéndose en diana de drogas inhibidoras que presentan escasa o nula toxicidad en células no transformadas, como el PD184352 o PD98059, actualmente en ensayos clínicos (Sebolt-Leopold, 2000); o la bisindolilmaleimida, inhibidor más apical de la ruta a nivel de PKC. En ambos casos se ha demostrado la capacidad de estos agentes de sensibilizar a TRAIL en distintos modelos tumorales (Dai et al., 2003; Ohtsuka and Zhou, 2002) (Sarker et al., 2001) JNK y p38 La quinasa c-Jun NH2-terminal en asociación con p38, activadas al igual que ERK1/2 por quinasas MKK de la ruta de las MAPK, están por el contrario relacionadas con la inducción de apoptosis a través de la activación de miembros pro-apoptóticos de la familia de Bcl-2 como Bax o Bim, de la inhibición de miembros anti-apoptóticos como Bcl-2 o Bcl-XL, así como a través de la proteína supresora de tumores p53 (Corazza et al., 2006; Lee et al., 2005; Shimada et al., 2003) 43 Esta ruta puede ser activada por estímulos pro-apoptóticos como el propio TRAIL, que favorece la traslocación de c-Jun y Fos al núcleo y la formación de AP-1, factor de transcripción que determina la síntesis de ligandos pro-apoptóticos como TNFα o FasL (Corazza et al., 2006; Sah et al., 2003). Por otro lado, estudios recientes muestran como la inhibición de JNK podría favorecer la apoptosis debido a la potenciación de la actividad de p38 (Hsu et al., 2007), quinasa para la que se ha descrito su implicación en la inducción de apoptosis (Amran et al., 2007; Lim et al., 2006), de forma que inhibidores de la JNK como SP-600125 podrían sensibilizar a TRAIL. Estímulo Respuesta biológica Factores de Crecimiento Crecimiento Desarrollo (FLIP) Estrés, TNFα Inflamación Apoptosis Figura 12. Ruta de las MAPK. 3.3 Ruta de PI3K La ruta de supervivencia iniciada por PI3K, activada por la unión de estímulos de supervivencia como EGF a sus receptores específicos (Poulaki et al., 2002) o bien directamente por el protooncogen Ras (Rodriguez-Viciana et al., 1996), determina la activación de la quinasa Akt que se encuentra incrementada en un elevado número de tumores, inhibiendo la actividad de TRAIL (Lane et al., 2007; Oka et al., 2006). 44 Akt, regulada en condiciones no patológicas por la proteína tirosina fosfatasa inhibidora de tumores PTEN (Li et al., 1997), bloquea señales intracelulares apoptóticas por fosforilación de distintos sustratos: • BAD, que pierde así su capacidad de unión e inhibición de Bcl-XL . • Caspasa -9, inhibiendo su activación. • MDM2, favoreciendo su unión a p53 y así la degradación de esta proteína supresora de tumores. • IKK, potenciando la ruta de supervivencia mediada por NF-κB. El compuesto LY294002, que bloquea esta ruta de supervivencia inhibiendo de forma específica a PI3K, se ha descrito como un agente sensibilizador de la apoptosis mediada por TRAIL en diversos modelos como en células de endotelio vascular (Alladina et al., 2005), al inducir un descenso en los niveles de FLIP. Figura 13. Ruta de PI3K/Akt 45 3.4 Ruta de NF-κB El papel de este factor de transcripción en el mantenimiento de la supervivencia celular y desarrollo del cáncer es de una gran relevancia, como se puede deducir de su implicación en las vías previamente citadas, activadas por distintos estímulos. Tanto la vía de las MAPK como la de PI3K acaban confluyendo en la activación de NF-κB, que a su vez regula la expresión de distintas proteínas relacionadas con la resistencia a la apoptosis junto con FLIP, IAP1 e IAP2 (Wang et al., 1998a), XIAP (Stehlik et al., 1998), Bcl-XL (Chen et al., 1998) o el receptor anti-apoptótico TRAIL-R3 (Bernard et al., 2001). Pero la acción de NF-κB, en el contexto de la regulación de la apoptosis mediada por TRAIL, no se limita a la mediación de estímulos y vías de supervivencia sino que es mucho más compleja ya que este factor de transcripción también puede intervenir positivamente en la cascada apoptótica, como demuestra su capacidad para regular la expresión de TRAIL y de sus receptores de muerte (Baetu et al., 2001). Además, el propio TRAIL es capaz de inducir la activación de NF-κB. Si bien en un primer momento se describió este efecto como consecuencia de la unión del ligando de muerte al receptor anti-apoptótico TRAIL-R4 (Degli-Esposti et al., 1997a), poco después se amplió esta actividad a los receptores pro-apoptóticos TRAIL-R1 y -R2 (Chaudhary et al., 1997). En definitiva, el concepto de equilibrio con el que comienza este trabajo vuelve a aparecer en forma de una complicada madeja de señales intracelulares, de manera que los denominados dominios de muerte de los receptores de TRAIL parecen mediar dos tipos de señales opuestas, pro-apoptóticas y de supervivencia. La activación de NFκB por unión de TRAIL a sus distintos receptores requiere el reclutamiento de la protein quinasa RIP a través del adaptador FADD (Lin et al., 2000; Varfolomeev et al., 1996). RIP fosforila y activa a IKK, complejo IkB quinasa compuesto por tres subunidades: IKK1/IKKα, IKK2/IKKβ, ambas subunidades catalíticas con actividad quinasa, e IKKγ/NEMO, subunidad inhibitoria que es separada del resto del complejo por la acción de RIP, liberando así a las subunidades con actividad quinasa de IKK (Mercurio et al., 1997) (Karin and Delhase, 2000). NFκB se describe como un grupo de factores de transcripción diméricos (Karin and Lin, 2002) cuyo heterodímero activo más conocido es el compuesto por las subunidades 46 p65/p50. Asociados al inhibidor IκB, dichos heterodímeros se encuentran secuestrados en el citoplasma (Huang and Miyamoto, 2001). La fosforilación de IκB por parte de IKK activo determina la ubiquitinación del inhibidor, que es degradado a nivel del proteasoma, liberándose NFκB que se trasloca al núcleo donde determina la trascripción de genes relacionados con supervivencia y proliferación (Ehrhardt et al., 2003) TRAIL TRAIL-R1/2 PROTEASOMA UBIQUITINACIÓN SEÑAL DE LOCALIZACIÓN NUCLEAR NUCLEO Figura 14. Ruta de activación de NF-κB. Adaptada de Hoffmann A, et al. Science 2002 Una vez establecida la importancia de NF-κB y su estrecha relación con TRAIL, distintos grupos han demostrado la sensibilización de tumores a este ligando mediante terapias combinadas con inhibidores de distintos puntos de la ruta de NF-κB, como MG132 y bortezomib, inhibidores del proteasoma que impiden la degradación de IκB (Kyritsis et al., 2007; Li et al., 2007); BAY 11-7085, inhibidor de la fosforilación de IκB (Huerta-Yepez et al., 2004); o SN50, inhibidor de la traslocación de NF-κB al núcleo (Kasuga et al., 2004). 47 3.5 Regulación del ciclo celular 3.5.1 Proteínas reguladoras del ciclo celular (CDKs) La correcta progresión del ciclo celular se encuentra regulada por la denominadas quinasas dependientes de ciclinas (CDKs), cuya actividad está a su vez controlada por fenómenos de fosforilación/defosforilacion y por la unión a las citadas proteínas ciclinas en respuesta a la estimulación por diversos factores como pueden ser factores de crecimiento. De forma general, la unión de las ciclina D a las CDK4 y 6 favorece la progresión de la fase G1, mientras que el complejo CDK2-ciclina E permite la entrada en fase S, CDK2-cliclina A en G2, CDK1-ciclina A en mitosis y el retorno a G0 requiere de CDK1-ciclina B (Collins and Garrett, 2005). En células no transformadas, la progresión del ciclo de una fase a la siguiente pasa por una serie de “puntos de control” donde se diagnostica la correcta ejecución del proceso y se intentan subsanar las posibles anomalías. De no ser esto posible, la célula es eliminada por apoptosis. Las células tumorales desarrollan mecanismos para evitar estos puntos de control íntimamente ligados con las funciones de las CDKs, lo que ha convertido a estas enzimas en dianas farmacológicas en la lucha contra el cáncer. Entre los distintos inhibidores de las CDKs, la roscovitina se presenta como un posible agente pro-apoptótico en combinación con TRAIL. Se ha demostrado su efecto sensibilizador al ligando en ciertos modelos tumorales, mediado posiblemente por su capacidad para regular los niveles de proteínas pro-apoptóticas como Bak o de factores anti-apoptóticos como Mcl-1 y XIAP (Hahntow et al., 2004; Kim et al., 2004). 3.5.2 Organización del citoesqueleto. El mantenimiento de la integridad del citoesqueleto se encuentra íntimamente ligado a la correcta progresión del ciclo celular. Los microtúbulos que componen el citoesqueleto tienen su origen en polímeros dinámicos que intervienen en la movilidad de la célula, el tráfico intracelular, así como en el crecimiento y la división celular. La organización del citoesqueleto puede verse alterada por distintos agentes como el nocodazol, inhibidor de la polimerización de microtúbulos, o el paclitaxel o taxol, 48 inhibidor de la despolimerizacion, produciéndose en ambos casos una parada de ciclo en fase G2/M y la inducción de apoptosis en la célula (Wang et al., 2000b). Esta inducción de la cascada apoptótica parece asociada a la capacidad de regulación de distintas proteínas señalizadoras de la muerte celular por parte de estos agentes inhibidores de la dinámica de microtúbulos. Se ha descrito el efecto de estos agentes en la transcripción de proteínas pro-apoptóticas como TRAIL-R1 y -R2, Bax y Bak, que ven incrementada su expresión (Shankar et al., 2005a), al tiempo que se reducen los niveles de Bcl-XL, FLIP o XIAP (Liu and Stein, 1997; Park et al., 2004). Por tanto, estos inhibidores también pueden ser útiles en estrategias de terapias combinadas con TRAIL que potencien la acción de este ligando sobre ciertos modelos tumorales (Nimmanapalli et al., 2001). 4. EPIGENÉTICA La epigenética se define como el conjunto de cambios heredables en la expresión de los genes no asociados a modificaciones en la secuencia del ADN. La accesibilidad transitoria de distintas secuencias de ácidos nucleicos, regulada en gran medida por factores epigenéticos, determina la expresión de genes que codifican proteínas imprescindibles en procesos como pluripotencialidad, diferenciación y maduración celulares. Así mismo, los cambios epigenéticos modulan la capacidad transcripcional de la célula durante procesos inherentes a la correcta progresión del ciclo celular como son la mitosis, o en su caso la apoptosis (Prigent and Dimitrov, 2003). Por tanto, disfunciones en esta forma de control de la información genética pueden desembocar en distintas patologías. Así, existe una íntima relación entre epigenética y cáncer. Los fenómenos de activación de oncogenes y de inactivación de genes supresores de tumores asociados a la aparición y el desarrollo de un tumor (Galm et al., 2006) son fruto en muchas ocasiones de alteraciones en las modificaciones epigenéticas durante el ciclo celular. Y es por ello que los factores implicados en dichas modificaciones epigenéticas pueden ser dianas importantes en la lucha contra el cáncer. 49 Las dos principales mecanismos epigenéticos, junto con la impronta genética, son la metilación del ADN y la modificación de histonas por metilación, acetilación o fosforilación (Klose and Bird, 2006; Strahl and Allis, 2000). 4.1 Metilación del ADN La modificación epigenética más extendida en humanos es la metilación de citosinas que preceden a guanosinas en la secuencia del ADN (dinucleótidos CpG) (Galm et al., 2006). Esta metilación tiene lugar por unión covalente de un grupo metilo al carbono 5′ del anillo de citosina resultando una 5-metilcitosina, proceso llevado a cabo por unas enzimas denominadas ADN metiltransferasas (DNMTs). La metilación del ADN inhibe la expresión génica bien directamente bloqueando la unión de factores de transcripción, o provocando la unión de las denominadas “proteínas de unión a CpG metilado” (MBP) que silencian la expresión de los genes por remodelación de la estructura de la cromatina a la que se unen (Klose and Bird, 2006). En el ADN de mamíferos encontramos 5-metilcitosinas en aproximadamente el 4% del ADN genómico y esta metilación basal parece contribuir al mantenimiento de una gran cantidad de ADN en un estado transcripcional inerte. Existen unos pequeños tramos de ADN ricos en CpG (de unos 0.5Kb) llamados islas CpG, localizadas próximas a la las regiones promotoras y que se encuentran normalmente no metiladas para permitir la expresión génica. Figura 15. Patrón de metilación de las islas CpG en células tumorales y normales. 50 En la célula transformada los patrones de metilación del ADN se encuentran profundamente alterados, presentando un espectro de genes hipermetilados específico para cada tejido y origen del tumor, por lo que pueden actuar como biomarcadores para la detección del cáncer, así como para predecir la susceptibilidad y respuesta a la terapia (Baylin and Ohm, 2006). Generalmente, son los promotores de distintos genes relacionados con la regulación del ciclo celular y apoptosis los que se encuentran hipermetilados en células tumorales (Widschwendter and Jones, 2002), mientras que el resto del ADN se presenta hipometilado (Weber et al., 2005). Así, tanto en tumores hematopoyéticos como sólidos se ha descrito el silenciamiento por hipermetilación del ADN de genes que codifican moléculas de adhesión, proteínas que regulan la proliferación, la diferenciación, la reparación del ADN, la muerte por apoptosis, la angiogénesis, metástasis del tumor e inhibidores de quinasas dependientes de ciclinas como p15, p27 o p21 (Gomyo et al., 2004). 4.2 “Código de Histonas” La condensación del genoma eucariótico en forma de cromatina requiere de la organización del ADN en unidades denominadas nucleosomas (Khorasanizadeh, 2004), en los que encontramos 146 pares de bases enrolladas sobre un núcleo proteico en forma de octámero compuesto por dos copias de cada una de las subunidades histonas H3, H4, H2A y H2B. Figura 16. Representación esquemática de un nucleosoma. Adaptado de Ruijter A.J.M.,Biochem. J, 2003 51 Estas histonas presentan un núcleo central altamente conservado, de estructura globular (Luger et al., 1997), mientras que las colas aminoterminales contienen secuencias variables particularmente ricas en lisina y arginina. Dichas secuencias actúan como diana de distintas enzimas responsables de modificaciones post-traduccionales tales como metilación, fosforilación, ubiquitinación, poli-ADP-ribosilación y acetilación, que van a influir en el grado de compactación de la cromatina (Spotswood and Turner, 2002). Esto ha permitido acuñar el término “código de histonas” para hacer referencia a las modificaciones en la expresión de distintos genes en función de su accesibilidad determinada por alteraciones reversibles de las histonas. 4.2.1 Metilación de histonas La metilación de histonas, si bien es un proceso que no altera la carga total de la proteína, incrementa su carácter básico e hidrofobicidad al tiempo que modifica su afinidad por moléculas aniónicas como el ADN (Baxter and Cooke, 1995), de forma que el nucleosoma ve modificada su estructura y función (Rice and Allis, 2001). Este proceso está mediado por las histona metiltransferasas (HMTs) (Kouzarides, 2002), con capacidad de metilar los residuos de lisina K4, K9, K27, K36 y K79 de la histona H3 y K20 de la H4, o bien las argininas R2, R17 y R26 de la H3 y R3 de la H4. Este hecho favorece la liberación de la estructura eucromática del ADN facilitándose la transcripción por unión de la ARN polimerasa II a los residuos metilados K4, K36 y K79 de la H3 (Gerber and Shilatifard, 2003). 4.2.2 Ubiquitinación de histonas La ubiquitina es una proteína altamente conservada de 76 aminoácidos cuya unión a otras proteínas determina la proteolisis de las mismas a nivel del proteasoma (Adams, 2004), aunque la ubiquitinación a nivel del residuo de lisina K123 de la histona H2B parece no constituir una señal directa para su degradación, sino tan solo una señal dentro del código de las histonas para la expresión genética (Osley, 2004), en tanto que actúa como un elemento regulador de la metilación de los residuos K4 y K36 de la H3 (Krogan et al., 2003). 52 Figura 17. Efectos de la ubiquitinación (Ub) de H2B sobre la metilación (M) de H3. Adaptado de Zhang Y.Genes & Dev. 2003 4.2.3 Fosforilación de histonas En mamíferos, la fosforilación de histonas en respuesta a determinados estímulos tiene lugar sobre todo en los residuos S10 y S28 de la histona H3 a través de la vía de las MAPK, de Akt y de IKK-1 (Anest et al., 2003; Hazzalin et al., 1997; Salvador et al., 2001). Se trata de un proceso dual pues está implicado en la relajación de la cromatina, permitiendo la transcripción de genes como c-jun, c-fos y c-myc que regulan la síntesis de proteínas necesarias durante la interfase (Prigent and Dimitrov, 2003); y al mismo tiempo también se ha descrito su participación en la regulación del ciclo celular durante los procesos de mitosis y meiosis (Gurley et al., 1998) aunque su papel durante la condensación de la cromatina asociada a la división celular no se conoce con exactitud. 4.2.4 Poli-ADP-ribosilación de histonas Al igual que muchas otras proteínas, las histonas pueden ser modificadas por unión de sucesivos monómeros de ADP-ribosa (poli-ADP-ribosa o PAR) por las PAR polimerasas (PARPs). La acción de PARP en la regulación de la transcripción radica en su capacidad para alterar la estructura de la cromatina favoreciendo su condensación y reprimiendo, por tanto, la transcripción (Huletsky et al., 1989), o bien permitiendo la asociación de complejos que incluyen distintos factores de transcripción a nivel de distintos promotores (Kraus et al., 2003). 53 4.3 Acetilación de histonas La progresión del ciclo celular desde el punto de vista epigenético es fruto del correcto equilibrio entre la acetilación y la deacetilación de histonas con una relevancia de proporciones infinitamente mayores respecto al resto de modificaciones a las que hemos hecho referencia. El proceso de acetilación es llevado a cabo por las histona acetil transferasas (HAT), enzimas que actúan sobre el grupo amino de los residuos de lisina de las histonas (Johnson and Turner, 1999) neutralizando su carga positiva y con ello su capacidad de unión al ADN, mientras que el proceso contrario está mediado por las histona deacetilasas (HDAC), que eliminan dichos grupos acetilo favoreciendo la compactación de la cromatina. Así, una situación de hipoacetilación de las histonas llevado a cabo por las HDAC se correspondería con la disminución en la accesibilidad al ADN. Por el contrario, la hiperacetilación provocada por la acción de las HAT, o por inhibición de las HDAC, se traduciría en un incremento de la transcripción (Maison et al., 2002). 4.3.1 Histona acetil transferasas (HAT) Se trata de complejos multiproteicos de elevado peso molecular en los que además de la subunidad catalítica encontramos una subunidad encargada de controlar la selectividad de sustrato del complejo (Carrozza et al., 2003). Esto se traduce en que, en función de las proteínas que conformen la región reguladora de este complejo, las HATs van a reclutarse en diferentes regiones de distintos promotores por interacción con proteínas activadoras que determinarán la acetilación de las histonas circundantes. En humanos, podemos clasificar las HATs en tres familias en función de su homología de secuencia y la similitud en sus funciones biológicas (Roth et al., 2001). Familia GNAT.- En esta familia distinguimos las HATs Gcn5 y ATF-2, caracterizadas por presentar un dominio con un átomo de Bromo que le permite interaccionar con grupos que presenten las lisinas acetiladas, actuando como complejos co-activadores. Familia MYST.- En la que encontramos las enzimas MOZ, que actúa como coactivador, y Tip60, que interviene en procesos como la reparación del ADN y 54 apoptosis (Carapeti et al., 1999). Estructuralmente, se caracterizan por presentar un dominio catalítico de unos 250 aminoácidos que incluye una región rica en cisteína, una región de unión a Zinc y un grupo Cromo amino-terminal para el reconocimiento de grupos lisina metilados (Utley and Cote, 2003). Familia CBP/p300.- Conjunto de enzimas que comparten en su estructura la presencia de un residuo catalítico típico de las HATs, un dominio de Bromo y una región rica en cisteína que permite la interacción proteína-proteína (Marmorstein and Roth, 2001), pudiendo actuar como modulador global de la transcripción. Esta enzima interviene activamente en procesos tumorales, debido a su capacidad de acetilación de la proteína supresora de tumores p53 (Liu et al., 1999). 4.3.2 Histona deacetilasas (HDAC) Las HDAC son enzimas con capacidad catalítica para eliminar el grupo acetilo de distintas proteínas, como p53, E2F o tubulina (Hubbert et al., 2002), pero particularmente de las histonas (Gray and Ekstrom, 2001). Las HDAC actúan formando un complejo con distintas proteínas encargadas del reclutamiento y remodelación de la estructura de la cromatina. De forma general, elevados niveles de acetilación de histonas se asocian a un incremento de la actividad transcripcional, mientras que la hipoacetilación se asocia con la represión génica (Wade, 2001). La principal señal para la represión de la transcripción mediada por las HDAC se localiza en el propio ADN, a través de los grupos metilo de las citosinas de las islas CpG (Klose and Bird, 2006). A estas regiones, como hemos citado previamente, se asocian las “proteínas de unión a CpG metilado” (MBP) que son las responsables directas del anclaje de las HDAC al ADN. Una vez ancladas al ADN, la eliminación del grupo acetilo de las histonas tiene lugar por un sistema basado en la modificación de la carga de la proteína. El dominio catalítico de las HDAC, de unos 390 aminoácidos, presenta dos residuos de histidina adyacentes y separados unos 30 aminoácidos de dos residuos de tirosina, y un átomo de Zinc que va a retirar el grupo acetilo de la histona por ruptura del enlace convalente (Buggy et al., 2000; Finnin et al., 1999). 55 Figura 18. Compactación y relajación de la cromatina por acción de las HATs y las HDACs. Adaptado de Hess-Stumpp et al, IJBCB, 2007 En la actualidad, se han descrito hasta 18 HDAC diferentes que pueden clasificarse en tres familias, las HDAC “clásicas” de clase I y II, que difieren en su localización, y las HDAC clase III que presentan un mecanismo de acción diferente al de las HDAC clásicas. HDAC clase I.- De amplia distribución en distintas líneas celulares y tejidos y de localización estrictamente nuclear (Fischle et al., 2001), presentan un mecanismo de acción típico mediado por el átomo de Zinc. A esta familia de proteínas pertenecen las HDAC1, 2, 3 y 8. HDAC1 y HDAC2. Con secuencias con un 82% de homología entre sí, ambas enzimas presentan capacidad de asociación in vivo a diferentes complejos de 56 unión al ADN, entre los que distinguimos los denominados Co-REST, Sin3, y NuRD (Zhang et al., 1999). En el caso de estos dos últimos, junto con las proteínas que le dan nombre encontramos otros integrantes como RbAp46 o RbAp48, que se unen a la histona H4 directamente. La actividad de estos complejos está regulada por fosforilación, de forma que un estado de hiperfosforilación se asocia con un incremento en la capacidad de deacetilación de la enzima al tiempo que disminuye la asociación al ADN para evitar una actividad excesiva. (Heinzel et al., 1997; Taplick et al., 2001). HDAC3. De idéntico dominio catalítico pero tan solo un 68% de homología con las HDAC1 y HDAC2, la región C-terminal no conservada de la HDAC3 es imprescindible para ambas actividades deacetilasa y represora de la transcripción. Pero lo que la separa del resto de las HDAC es el complejo al que se une, el cual le confiere individualidad y especificidad. La actividad de HDAC3 requiere de las presencia de SMRT (silencig mediator for retinoic acid and thyroid hormone receptors) o N-CoR (nuclear receptor co-repressor), cuya unión a la enzima les permite actuar como co-represores de la transcripción. A través de estos complejos, la HDAC3 puede asociarse a otras HDACs como son la HDAC4, 5 y 7 (Fischle et al., 2001; Yang et al., 2002), aunque también se ha observado su asociación a las HDAC9 y 7 a través de otras proteínas. HDAC8. De estructura similar a la HDAC3, está compuesta por un dominio catalítico asociado a una señal de localización nuclear (NLS), aunque se desconoce el complejo regulador al que puede encontrarse asociado. HDAC clase II.- De complejo catalítico similar al de HDACs de clase I, difieren de éstas en cuanto a su localización, limitada a un menor número de tipos celulares, pero donde encuentran expresión tanto nuclear como citosólica (Verdin et al., 2003) (Fischle et al., 1999). A esta familia enzimática pertenecen las HDAC4, 5, 6, 7, 9 y 10. HDAC4, HDAC5 y HDAC7. Comparten una estructura similar que incluye el dominio catalítico próximo a la región C-terminal, mientras que hacia la región N-terminal se localizan el NLS y los dominios de unión a CtBP (roteínas de unión al extremo C-terminal) y MEF2 (myocite enhancer factor 2). Estas HDACs se localizan fundamentalmente en células musculares, donde regulan el papel de MEF2 durante la diferenciación, de forma que su asociación con las HDAC 57 se traduce en una incapacidad de unión al ADN y por tanto de actuar como factor de transcripción, que es recuperada con la disociación del complejo tras la fosforilación de HDAC por CaMK (McKinsey et al., 2000). Las HDAC4, 5 y 7 presentan sitios de unión a SMRT y N-CoR, favoreciendo su asociación a la HDAC3 y con ello el reclutamiento de esta enzima al ADN (Guenther et al., 2000). HDAC6. Esta HDAC presenta dos rasgos estructurales que la separa del resto de enzimas de la familia. El primero de ellos es la presencia de dos dominios catalíticos dispuestos en tándem (Marks et al., 2001) y el segundo, un dominio Cterminal que actúa como señal para la ubiquitinación y que le confiere una mayor predisposición a la degradación. Estas peculiaridades estructurales parecen asociadas a su función como tubulina deacetilasa regulando la movilidad celular dependiente de microtúbulos (Hubbert et al., 2002). Este hecho determina la presencia de una secuencia de localización citosólica (NES), aunque también se ha descrito su localización a nivel nuclear, asociada a la HDAC11. HDAC9. Aunque aparece como variante por procesamiento alternativo del ARN y por tanto está emparentada con las HDAC4, 5 y 7, la HDAC 9 difiere del resto de proteínas de la familia en la localización del dominio catalítico, que se encuentra dispuesto en el extremo N-terminal. Debido a su analogía con las HDAC4, 5 y 7, presenta capacidad de unión y reclutamiento al ADN de la HDAC3, y también se ha descrito su interacción con MEF2, de lo que se deduce su posible implicación en la diferenciación del tejido muscular. HDAC10. Su capacidad de unión a las HDAC1, 2, 3, 4, 5 y 7 parece indicar que su papel es predominantemente el reclutamiento de otras HDACs al ADN, en lugar de la deacetilación por sí misma, aunque la presencia de dos sitios de unión a Rb sugieren la posibilidad de su intervención en la regulación del ciclo celular. HDAC11. La clasificación de la HDAC11 no se encuentra completamente establecida. Si bien filogenéticamente parece situarse próxima a las HDAC3 y 8, no se ha establecido ningún tipo de asociación a los complejos descritos como Sin3 o SMRT, lo que parece indicar que presenta una función bioquímica diferente al resto (Gao et al., 2002). Esto ha llevado a que sea clasificada como HDAC clase IV. 58 Figura 19. Clasificación y realación filogenética de las HDACs. Adaptado de Hess-Stumpp et al, IJBCB, 2007 HDAC clase III o Sirtuinas. Difieren de las HDAC de clase I y II en su mecanismo de deacetilación de histonas, al emplear un grupo NAD+ como aceptor de grupos acetilo (Landry et al., 2000), y por no ser susceptibles de inhibición por compuestos como el TSA (Tricostatina A) (Frye, 1999). En humanos estas HDAC reciben el nombre de SIRT1-7 y son responsables del silenciamiento de los telómeros, jugando un importante papel en la formación de la heterocromatina (Palladino et al., 1993). 59 4.4 Histona deacetilasas y cáncer Las HDAC regulan la expresión y actividad de numerosas proteínas que juegan un papel en el inicio y en la progresión del cáncer. De entre los procesos en los que interviene destacamos los siguientes 4.4.1 HDAC y proliferación celular La proteína p21, inhibidora de CDKs, se presenta como uno de las dianas de la acción de HDACs con mayor relevancia para la progresión del ciclo celular. Su expresión durante el desarrollo se asocia a una hiperacetilación de las histonas H3 y H4 a nivel del promotor (Richon et al., 2000), de forma que la acción de las HDACs, en especial la HDAC1, determina un descenso en la expresión de este inhibidor de la proliferación que puede desembocar en una división celular incontrolada, como se ha observado en distintos modelos tumorales (Ocker and Schneider-Stock, 2007). 4.4.2 HDAC y diferenciación hematopoyética La progresión desde células precursoras pluripotenciales hasta células hematopoyéticas maduras requiere de la interrelación de una compleja gama de moléculas. El bloqueo o desregulación de alguno de los pasos de este proceso de diferenciación puede determinar la proliferación de células leucémicas y aquí pueden jugar un papel importante las HDAC. Un ejemplo son los casos de leucemia mieloide aguda (AML) asociados a la formación de la proteína de fusión AML-ETO, como consecuencia de la translocación cromosómica t(8;21). Esta proteína de fusión se une a las HDAC1, 2, 3, 5, 6 y 9 con afinidad variable (Hiebert et al., 2003) y así se asocia al ADN provocando un descenso, por deacetilación, en los niveles expresión de ciertos genes como el c-FMS, factor de diferenciación fundamental para macrófagos (Follows et al., 2003). 60 4.4.3 HDAC, angiogénesis y metástasis Entres los genes implicados en la progresión tumoral que son regulados por acetilación y deacetilación de histonas, encontramos algunos relacionados con el proceso de angiogénesis y con el control de la adhesión, migración e invasión celular. Se ha demostrado que la situación de hipoxia localizada en el centro del tumor induce la expresión de HDAC1, y con ello, la represión de p53 así como la inducción del factor inducible por hipoxia-1α (HIF-1α) y el factor de crecimiento de endotelio vascular (VEGF). De este modo HDAC1 regula la angiogénesis (Kim et al., 2001). Por otra parte, las HDACs clase I regulan directamente la expresión de la Ecadherina, proteína fundamental en el proceso de adhesión y cuya pérdida determina la invasión epitelial, primera fase de la metástasis (Peinado et al., 2004). De la misma forma, las HDACs se encuentran implicadas en la regulación de la expresión del receptor de quimioquinas CXCR4 en leucemia linfática aguda (ALL), que interviene en la migración de estas células transformadas hacia hígado, bazo, cerebro y nódulos linfáticos (Crazzolara and Bernhard, 2005). 4.4.4 HDAC y factores de transcripción Cada día son más las proteínas no histonas descritas que son susceptibles de modificación por acetilación y deacetilación por las HATs y HDACs, respectivamente, alterando su función, capacidad de interacción proteína-proteína, estabilidad y localización (Glozak et al., 2005). Entre estas proteínas, la acetilación de distintos factores de transcripción parece jugar un importante papel durante la oncogénesis. En respuesta a diferentes tipos de daño en el ADN se produce la acetilación de p53 por el complejo p300/CBP, incrementándose así la capacidad de unión de este gen supresor de tumores a secuencias específicas situadas en ciertos genes, y por tanto su acción como factor de transcripción. La activación de p53 promueve la parada del ciclo celular a través de p21, en el caso de daños provocados por agentes alquilantes, e induce apoptosis por activación de Bax tras daños en la maquinaria de reparación de ácidos nucleicos (Gu et al., 1997; Sakaguchi et al., 1998). En linfoma folicular de células B, la traslocación t(14;18) determina la sobreexpresión de Bcl-2 regulada por los factores de transcripción Sp1 y C/EBP . La 61 interacción de la HDAC2 con dichos factores de transcripción permite su deacetilación y así se favorece su unión al promotor del gen anti-apoptótico (Duan et al., 2005). Figura 23. Activación de la transcripción de genes antiapoptóticos por acetilación de NF-κB. Adaptdo de Kagoshima et al, Biochemical Society 2003 NF-κB interviene en el desarrollo de una gran diversidad de procesos oncológicos en humanos. La actividad y estabilidad de este factor de transcripción también se encuentra regulada por procesos de acetilación y deacetilación a distintos niveles (Basseres and Baldwin, 2006). Una vez activado, NF-κB se trasloca al núcleo donde la unidad p65 es acetilada por p300/CBP en los residuos K218, K221 y K 310, favoreciendo su unión al ADN y así la transcripción de diversos genes anti-apoptóticos, e impidiendo su asociación con la proteína inhibidora IκB. Una vez cumplida su función, es deacetilado por HDAC1, 2 y 3, facilitando el exporte nuclear. Pero esta asociación acetilación-activación no es estricta en el caso de NF-κB, ya que la acetilación de los residuos K122 y K123 produce el efecto contrario, reduciéndose la afinidad del factor de transcripción por el ADN (Kiernan et al., 2003). En estas condiciones, la inhibición de la actividad HDAC puede ser beneficiosa para el tratamiento de tumores, siendo este hecho aún más evidente si tenemos en cuenta que en cada uno de los procesos relacionados con la oncogénesis descritos anteriormente, la actividad HDAC se traduce en la progresión del tumor. De ahí que la inhibición de estas enzimas se presente como una diana potencial en la terapia antitumoral. 62 4.5 Inhibidores de histona deacetilasas (HDACi) Se han descrito un gran número de inhibidores de la actividad enzimática de las distintas HDACs (HDACi), tanto de origen natural como derivados sintéticos, que inducen parada de ciclo celular, diferenciación o apoptosis en células tumorales en cultivo y modelos animales (Johnstone, 2002). Dichos inhibidores presentan diferentes tipos de estructuras por lo que podemos distinguir los siguientes grupos: Ácidos hidroxámicos La tricostatina A o TSA fue el primer hidroxamato descrito con capacidad de inhibición de una amplia gama de HDACs (Yoshida et al., 1990). Pero es el vorinostat o SAHA, inhibidor de HDACs de clases I y II, el único ácido hidroxámico aprobado actualmente por la “Food and Drug Administration” para ser utilizado en el tratamiento del linfoma T cutáneo, habiendo superado los ensayos de fase II en los que ha demostrado su seguridad y eficacia y presentando una respuesta positiva en el 25% de los casos (Duvic and Vu, 2007). Otros miembros de esta familia de estructura similar al Vorinostat y que se encuentran en fase I de ensayos clínicos son el LBH-589 o panobinostat en el tratamiento de diferentes tipos de leucemias (Giles et al., 2006), el ITF2357 para el tratamiento del mieloma múltiple y leucemia mieloide aguda (Golay et al., 2007), y el PXD-101 o bellinostat, que presenta una buena tolerancia y una eficacia antitumoral considerable en pacientes con un avanzado estado de oncogénesis (Marson et al., 2007). Péptidos cíclicos A esta familia estructural pertenecen los HDACi apicidina y depsipéptido. Este último se caracteriza por inhibir eficazmente a las HDAC1 y 2, al tiempo que presenta un efecto muy leve sobre las HDAC4 y 6 (Santini et al., 2007). Actualmente se encuentra en fase II de ensayos clínicos en pacientes con linfoma T cutáneo con un porcentaje de respuesta positivo del 36%, así como para el tratamiento de linfomas T 63 periféricos y distintos tumores sólidos o hematológicos en combinación con otros agentes antitumorales (Piekarz et al., 2007). Benzamidas A este grupo pertenecen compuestos como N-acetildinalina (CI-994), MGCD-0103 y MS-275, inhibidor específico de las HDACs de clase I ante todo de las HDAC 1 y 3 (Hu et al., 2003). Actualmente en fase I de ensayos clínicos, este compuesto de actividad comparable al TSA ha demostrado actividad antitumoral en neonatos y adultos con cáncer de mama y riñón. Figura 24. Estructura de los distintos HDACi. Adaptado de Minucci and Pelicci, Nature Reviews, 2006 Ácidos alifáticos El ácido valproico (VPA) y el butirato sódico (NaB) son los representantes más destacados de esta familia estructural, habiéndose probado sus efectos terapéuticos en 64 síndromes mielodisplásicos (Kuendgen et al., 2004). En el caso del VPA, junto con su capacidad de inhibición enzimática, se ha demostrado también su implicación en la degradación de la HDAC2 (Kramer et al., 2003). 4.6 HDACi y TRAIL Los HDACi, al promover la hiperacetilación de histonas, afectan a la expresión de un 20% de los genes conocidos (Glaser et al., 2003), entre ellos genes implicados en las rutas de señalización de apoptosis. Diferentes autores han demostrado la inducción por HDACi de un elevado número de genes pro-apoptóticos como caspasa-8, caspasa-3, Bid, Bim, Bax o Bak en distintos modelos de células tumorales. También se ha observado la represión en la expresión de otros tantos genes de carácter anti-apoptótico como Bcl-2, Bcl-XL o XIAP en respuesta al tratamiento con HDACi (Lindemann et al., 2004; Shankar et al., 2005b). Figura 25. Efecto selectivo de los HDACi sobre células transformadas. Adaptado de Minucci and Pelicci, Nature Reviews, 2006 65 Entre los factores pro-apoptóticos regulados por HDACi se encuentran el ligando de muerte TRAIL y su receptor de muerte TRAIL-R2 (DR5). Mediante estudios con ARN de interferencia se ha demostrado que la regulación de dichos genes es necesaria para la inducción de apoptosis por HDACi en ciertos modelos de células leucémicas, como es el caso de la leucemia mieloide, y que el efecto de estos inhibidores parece ser selectivo sobre células tumorales y no sobre células normales (Nebbioso et al., 2005). Además, en tumores hematológicos y ciertos tumores sólidos se ha descrito la sensibilización a TRAIL mediante el uso de determinados HDACi a concentraciones no tóxicas (Zhang et al., 2003). En algunos casos se ha observado que el tratamiento simultaneo, pero no secuencial, de ambos agentes determina un cierto sinergismo en la inducción de apoptosis, de lo que se deduce que ésta no se debe a la represión de proteínas antiapoptóticas mediada por los HDACi, sino que se requiere la coactivación de ambas rutas apoptóticas, extrínseca por parte de TRAIL e intrínseca por acción de los HDACi (Rosato et al., 2003). 66 II. OBJETIVOS 67 68 El ligando de muerte TRAIL, cuya expresión se incrementa en la superficie de células T en respuesta a señales de activación, parece mediar en gran parte la actividad citotóxica de estas células. Aunque se ha demostrado la regulación de la expresión de TRAIL por Interferones o por activación de NF-κB no se sabe nada acerca de la implicación de otros factores de la transcripción que podrían estar jugando un papel adicional e importante en su regulación, como es el caso de NFAT, factor que interviene en las señales de activación de los linfocitos T y para el cual se han encontrado varios sitios de unión en el promotor de TRAIL. La ausencia de toxicidad de TRAIL para la mayoría de células no transformadas es el hecho más importante a tener en cuenta a la hora de considerarlo como un agente antitumoral. También los linfocitos T activados que expresan TRAIL en membrana parecen ser resistentes a la acción de este ligando, siendo éste un requisito esencial para que TRAIL pueda funcionar como mediador de la respuesta antitumoral de los propios linfocitos T. Sin embargo, el mecanismo implicado en la resistencia de células normales a TRAIL no está completamente caracterizado. En la actualidad se están empleando distintos agentes en terapias combinadas con TRAIL a fin de mejorar la sensibilidad de ciertos modelos tumorales a este ligando de muerte. Pero los efectos que dichos agentes pueden tener sobre la resistencia de células normales no se conocen y en la mayoría de las ocasiones no han sido analizados. Existe por tanto la necesidad de realizar un estudio paralelo y comparativo sobre la capacidad de numerosos agentes para potenciar la apoptosis en células normales y en células tumorales, estudio que nos dará a conocer la inocuidad o los efectos adversos de estos agentes al mismo tiempo que nos ayudará a caracterizar los mecanismos de resistencia a TRAIL específicos de las células no transformadas. Los inhibidores de HDAC (HDACi) constituyen un nuevo grupo de fármacos que han demostrado una gran efectividad en la inhibición de la proliferación y supervivencia de células tumorales. Estos efectos antitumorales parecen deberse principalmente a su capacidad para regular genes relacionados con el ciclo celular y apoptosis. Al igual que en el caso de TRAIL, las células no transformadas presentan una alta tolerancia a estos inhibidores aunque su mecanismo de acción selectivo sobre células tumorales no se conoce con precisión. Es por ello que, la posibilidad de una terapia combinada con HDACi a dosis apropiadas y TRAIL puede constituir una estrategia muy útil en el tratamiento de tumores, especialmente de aquellos más resistentes a TRAIL. Los 69 estudios realizados hasta el momento con HDACi se centran en los efectos concretos de uno o un par de estos inhibidores sobre un determinado modelo tumoral. Pero el desarrollo de todo el potencial de esta terapia requiere aún de un estudio comparativo de la efectividad de HDACi pertenecientes a distintas familias estructurales sobre células tumorales y células normales, así como de un análisis de los genes diana específicos de cada uno de estos compuestos, teniendo en cuenta la posible diversidad en su mecanismo de acción. OBJETIVOS En base a los antecedentes y planteamientos expuestos nos propusimos los siguientes objetivos para la realización de esta tesis doctoral: 1.- Estudio de la regulación de la expresión de TRAIL en linfocitos T en respuesta a la activación. Implicación del factor de transcripción NFAT. 2.- Caracterización de los mecanismos implicados en la resistencia a TRAIL de linfocitos T primarios tanto en estado de reposo como activados. 3.- Análisis comparativo de la regulación de la respuesta a TRAIL en células T primarias y en células leucémicas T. 4.- Estudio de la modulación por inhibidores de HDAC de la apoptosis mediada por TRAIL en células T leucémicas. 70 III. MATERIALES Y MÉTODOS 71 72 1.- Cultivos celulares Las muestras de sangre utilizadas procedentes de donantes sanos fueron recogidas en tubos con citrato. Las células mononucleares se obtuvieron por centrifugación en gradiente de Ficoll- Histopaque (Sigma-Aldrich, St. Louis, MO), eliminando los monocitos del cultivo por adhesión a frascos de plástico durante 1 hora a 37 ºC. Los linfocitos T de sangre periférica fueron aislados por selección negativa mediante un sistema de unión indirecta a partículas magnéticas formado por un grupo de anticuerpos frente a CD14, CD16, CD19, CD36, CD56, CD123 y CD235a asociados a biotina (Human Pan T Cells Isolation Kit II, Miltenyi Bistec, GmbH). La pureza de los linfocitos T fue superior al 95% de células CD3 positivas tras el análisis por citometría de flujo con un anticuerpo marcado de forma directa (OKT3-FITC). Las células T en reposo se resuspendieron en medio RPMI 1640 (Cambrex, Bio Sciece, Bélgica) enriquecido con suero bovino fetal (GIBKO, California, USA) al 10%, L-glutamina 1 mM, 100 U/ml de penicilina y 100 μg/ml de estreptomicina (Cambrex) (medio completo), y se mantuvieron en un incubador a 37 ºC y 5% de CO2. La activación de linfocitos T se realizó mediante cultivo a la concentración de 2x106 células/ml en presencia de 5 μg/ml PHA (fitohemoglutinina-M, Roche, Indianápolis, USA) y 1 μg/ml anti-CD28 durante 20 horas. Tras lavar con PBS, los linfocitos se cultivaron en medio completo suplementado con 25 U/ml de IL-2 durante 5 días. La separación de las subpoblaciones de linfocitos T activados se realizó por un sistema de partículas magnéticas asociadas a un anticuerpo frente al CD8 (Human CD8 MicroBeads, Miltenyi Bistec, GmbH) según el protocolo desarrollado por el fabricante. Las líneas de células T leucémicas Jurkat, CEM-6, MOLT-4 y HPB-ALL , así como las líneas tumorales de cáncer de mama SKBr3 y BT474 y la de cáncer de cervix Hela, se mantuvieron en medio completo RPMI 1640 y en un incubador de idénticas características que los linfocitos T primarios. Las muestras de sangre de pacientes con linfoma o con leucemia de células T fueron proporcionadas por el Hospital Virgen de Las Nieves de Granada, y los linfocitos de sangre periférica fueron obtenidos por centrifugación en gradiente de Ficoll- Histopaque 73 (Sigma-Aldrich) y resuspendidas en medio RPMI 1640 completo, para ser tratadas y analizadas de forma inmediata. 2.- Reactivos y Anticuerpos TRAIL humano recombinante, obtenido como se ha descrito previamente (MacFarlane et al., 1997), fue donado amablemente por el Dr. Abelardo López Rivas (CABIMER, Sevilla). PMA, ionomicina, PHA, doxorubicina, LY 294002, nocodazol, ácido valproico (VPA), tricostatina A (TSA), butirato sódico (NaB), MS-275, BAY 117085, sulfosalazina y anticuerpos anti-tubulina y anti-actina se obtuvieron de SigmaAldrich (St. Louis, MO). Vorinostat (SAHA), apicidina y bisindolilmaleimida (BIS) de Calbiochem (La Jolla, CA). Velcade (bortezomib) de Janssen-Cilag SA (Madrid, España). IFN-α de PeproTech EC Ltd. (London, UK). PD 98059 de New England Biolabs (Beverly, MA). El inhibidor de caspasas Z-VAD de Bachem (Bubendorf, Suiza). Partenolide, CAPE, anticuerpo monoclonal anti-cFLIP NF6 y anticuerpos frente a los receptores de TRAIL y frente a TRAIL (2E5) humanos fueron de Alexis Biochemicals (San Diego, CA). Anticuerpos monoclonales frente a CD28 y fretne a la subunidad p50 de NF-κB fueron adquiridos de eBioscience (San Diego, CA); anticuerpo monoclonal anti-caspasa-8 de Cell Diagnostica (Münster, Alemania); anticuerpo monoclonal anti-FADD de Transducction Laboratorios (Lexington, KY); anticuerpos monoclonales anti-PARP, anti-XIAP, anti-c-IAP2 y anti-Bax de BD Biosciences (San Jose, CA). Anticuerpo monoclonal anti-p65 de Santa Cruz Biotechnology Inc. (Santa Cruz, CA). Anticuerpo monoclonal anti-caspasa-9 y los inhibidores de caspasas Z-IETD-FMK y Z-LEHD-FMK de R&D Systems (Minneapolis, MN). Anticuerpo monoclonal anti-Bcl-2 de Dako (Glostrup, Denmark). Anticuerpo policlonal de conejo anti-caspasa-3 de Stressgen (Ann Arbor, MI). Anticuerpo policlonal de conejo anti-histona H4 acetilada de Upstate Biotechnology (Lake Placid, NY). Anticuerpo monoclonal anti-Bid de Cell Signaling Technology (Boston, USA). Anticuerpo monoclonal anti-citocromo C de ZYMED Laboratories (filial de Invitrogen, Carlsbad, CA). 74 3.- Determinación de células apoptóticas Las células apoptóticas hipodiploides fueron detectadas por citometría de flujo de acuerdo con el protocolo publicado (Gong, 1994). De forma breve, las células se lavaron con buffer salino (PBS), se resuspendieron en 100 μl del mismo y se fijaron durante 5 minutos en 900 μl de una solución de etanol al 70 % frío. Después de lavar el exceso de etanol, se resuspendieron en 250 μl de PBS, se añadió 250 μl de una solución de extracción de ADN (0.2 M NaHPO4, 0.1 M ácido cítrico pH 7.8) y se incubaron 10 minutos a 37 ºC. Finalmente, para la tinción del ADN se incubaron en 200 μl de PBS conteniendo ARNasa a 100 μg/ml y ioduro de propidio a 40 μg/ml, durante 30 minutos a 37 ºC en oscuridad. El análisis del pico sub-G1 del ciclo celular se realizó midiendo la fluorescencia del ADN en el detector FL2 o en FL3 del citómetro de flujo (FACScan, Becton Dickinson) utilizando el programa Cell Quest (BD Biosciences). Otro método empleado para la cuantificación de la muerte por apoptosis fue la detección del fosfolípido fosfatidilserina en la cara externa de la membrana celular mediante la proteína Anexina-V-FLUOS (Roche), que se une al fosfolípido en presencia de Ca++. Las células se suspendieron en un tampón de incubación compuesto por Hepes NaOH 10 mM pH 7.4, NaCl 140 mM y CaCl2 5 mM al que se añadió Anexina-VFLUOS a la concentración indicada por el fabricante durante 15 minutos a temperatura ambiente y en oscuridad. La externalización de la fosfatidilserina se detectó en la región de emisión correspondiente a FL1 del citómetro de flujo. 4.- Análisis por citofluorometría de TRAIL y sus receptores Para la detección del ligando de muerte y sus receptores en superficie, las células control y tratadas se lavaron y posteriormente resuspendieron en PBS, donde se incubaron con el anticuerpo primario monoclonal correspondiente (5 μg/ml) a 4 ºC durante 30 minutos. Tras lavar con PBS para eliminar los restos del anticuerpo primario que no se hayan unido al receptor, las células se incubaron con un anticuerpo de cabra frente a ratón, conjugado con isotiocianato de fluoresceína (FITC) (Caltag laboratories, Invitrogen) durante 30 minutos a 4º C. Tras esta segunda incubación, las células se 75 lavaron de nuevo, se resuspendieron en PBS y se analizaron en el detector FL1 del citómetro FACScan. 5.- Análisis por citofluorometría del nivel de acetilación de histonas Las pruebas para determinar los niveles de acetilación de histonas se llevaron a cabo en células Jurkat y CEM-6 según el protocolo descrito por Ronzoni y colaboradores (Ronzoni et al., 2005). Las células se incubaron a la concentración de 106 /ml durante 4 ó 24 horas con los distintos HDACi, se recogieron, se lavaron con PBS frío y se fijaron en 500 μl de una solución al 1% de formaldehído en PBS durante 20 minutos a 4 ºC. Tras la fijación, se lavaron con 1 ml de PBS conteniendo albúmina sérica (Sigma) al 1% y se permeabilizaron por incubación en 100 μl de PBS con Tritón X-100 al 0.1% durante 10 minutos a temperatura ambiente. A continuación, se lavaron de nuevo con BSA 1% en PBS, se realizó un bloqueo mediante incubación con 250 μl de suero de cabra (Sigma) al 10% en PBS durante 30 minutos a 4º C, se centrifugaron y se incubaron durante 1 hora a temperatura ambiente con el anticuerpo policlonal de conejo anti-H4-Ac a una concentración de 0.1 μg/ml en 100 μl de PBS-BSA 1%. El exceso de anticuerpo primario se eliminó mediante lavado con PBS-BSA 1% y el marcaje con el anticuerpo de cabra frente a conejo marcado con FITC se realizó por incubación con dicho anticuerpo a una concentración de 1:500 en 100 μl de PBS-BSA 1% durante 1 hora a temperatura ambiente en oscuridad. Tras lavar las muestras con 1 ml de PBSBSA 1%, se resuspendieron en 200 μl de PBS y los niveles de acetilación de histonas se analizaron en la región de emisión correspondiente a FL1 en un citómetro FACScan. 6.- Inmunodetección de proteínas por Western Blot Las células control y tratadas fueron lavadas con PBS y lisadas en soluciones de distinta composición en función de la localización de la proteína a detectar. Para la detección de proteínas citosólicas, las células (2-5x106 según el tipo) se incubaron durante 30 minutos en hielo con 100 μl de buffer de lisis (150 mM NaCl, 50 76 mM Tris-ClH y NP-40 al 1%). Los sobrenadantes citosólicos se separaron por centrifugación durante 10 minutos a 13000g y se les añadió tampón de carga. Para el análisis de proteínas localizadas en el núcleo, como PARP o la histona H4, se lisaron las membranas celulares mediante congelación y descongelación de las células resuspendidas en PBS, se añadió tampón de carga y se procedió a la sonicación de la muestra para romper el ADN. La separación de fracción membranosa y citosólica para detectar la liberación de citocromo c de la mitocondria y la translocación de Bax en líneas celulares, se logró lisando las células en frío durante 5 minutos con 50 μl de buffer compuesto por KCl 80 mM, sacarosa 250 mM, digitonina 500 μg/ml, PMSF 0.1 mM, DTT 1 mM e inhibidores de proteasas, en PBS y centrifugando 5 minutos a 13000g. Las proteínas del sobrenadante (fracción citosólica) y las del pellet (fracción membranosa) se mezclaron con tampón de carga y estas últimas fueron sonicadas para romper el ADN. Para la detección de factores de transcripción importados al núcleo se llevó a cabo el lisado de 5x106 células en 300 μl de un buffer hipotónico (10 mM de HEPES pH 7.6, KCl 10 mM, EDTA 0.1mM, EGTA 0.1mM, DTT 1mM, PMSF 0.5 mM, Na2MoO4 10mM e inhibidores de proteasas) en presencia de NP-40 al 0.6%. Los núcleos de esta manera obtenidos se incubaron en 50 μl de un buffer de elevado contenido salino (20 mM HEPES pH 7.6, KCl 0.4 mM, EDTA 1 mM, EGTA 1 mM, DTT 1 mM, PMSF 0.5 mM, Na2MoO4 10 mM e inhibidores de proteasas) durante 30 minutos en una plataforma giratoria a 4 ºC. El lisado se centrifugó a 13000 g durante 10 minutos separándose así las membranas de los extractos nucleares, solubles en el sobrenadante. En todos los casos las proteínas se cuantificaron por el método Bradford (Sigma) antes de añadir el tampón de carga, buffer Laemmli compuesto por Tris 2 M pH 6.8, urea 6 M, B-mercaptoetanol 6%, azul de bromofenol 0.003% y SDS al 3%. Las proteínas se separaron en geles de SDS-poliacrilamida (Bio-Rad) al 7.5%, 10% ó 12%, en función del tamaño de la proteína a detectar, en el sistema Mini Protean (BioRad) durante 80 minutos a 140 voltios. Una vez separadas las proteínas por tamaño se transfirieron desde el gel a una membrana de PVDF (Millipore) por transferencia semiseca empleando el sistema Trans-Blot SD (Bio-Rad) durante 50 minutos a 50 mA. La membrana se bloqueó con una solución de PBS / 0.1% Tween 20 (PBSt) con 5% de leche en polvo durante 1 hora a temperatura ambiente, tras lo que se lavó con PBSt 77 antes de incubar durante 1 hora a temperatura ambiente o toda la noche a 4 ºC con el anticuerpo frente a la proteína a detectar, diluido en PBSt con 1% de leche. Tras la incubación, se lavó la membrana 5 minutos tres veces con PBSt y finalmente se incubó durante otra hora a temperatura ambiente con el anticuerpo secundario (frente a Ig de ratón o de conejo según el caso) marcado con peroxidasa de rábano (HRP), preparado en solución de bloqueo. Tras otros tres lavados de 5 minutos con PBSt, la membrana se reveló por quimioluminiscencia empleando el reactivo ECL (Amersham Biosciences), quedando la señal recogida en placas fotográficas (AGFA). 7.- Retrotranscripción (RT) y PCR Se extrajo ARN total empleando el reactivo Trizol (Invitrogen) y siguiendo el protocolo recomendado. Brevemente, se resuspendieron las células controles y tratadas en un volumen de 1 ml de Trizol por cada 5-10x106 células. Se añadió 200 μl de cloroformo y tras mezclar ambas fases, se centrifugó a 12000 g durante 15 minutos a 4 ºC. Se recogió la fase acuosa y se mezcló con 500 μl de isopropanol, dejando incubar durante 10 minutos a temperatura ambiente antes de centrifugar otros 10 minutos a 12000 g. Se eliminó el sobrenadante y se lavó el pellet de ARN con 1 ml de etanol al 75%. Tras centrifugar a 7500 g durante 5 minutos se eliminó el sobrenadante y se dejó secar el pellet antes de resuspenderlo en 20 μl de H2O-DEPC. Tras la cuantificación del ARN total, se partió de 3 μg del mismo para sintetizar ADNc empleando la transcripta inversa M-MLV (Invitrogen) y oligo(dT) como cebador en un volumen final de 20 μl. El proceso de retrotranscripción se llevó a cabo a 37 ºC durante 50 minutos tras los cuales se inactivó por incubación a 70 ºC durante 15 minutos. Las reacciones de PCR se realizaron con 0.1-2 μl de ADNc, en función del gen a estudiar, en un volumen final de 50 μl empleado los siguientes pares de cebadores (el tamaño del fragmento amplificado viene dado entre paréntesis) a una concentración final de 0.2 μM: TRAIL (443 pb) sentido 5’- GATCAAGACCATAGTGACCAA-3’ y antisentido 5’TGGCATGATCTCACCACAC-3’ 78 Bid (588 pb) sentido 5’- ATGGACTGTGAGGTCAACAACGG –3’ y antisentido 5’CACGTAGGTGCGTAGGTTCTGGTT A –3’ TRAIL-R1 (506 pb), sentido 5’-CTGAGCAACGCAGACTCGCTGTCCAC-3’, y antisentido 5’- TCCAAGGACACGGCAGAGCCTGTGCCAT-3’ TRAIL-R2 (502 pb), sentido 5’- GCCTCATGGACAATGAGATAAAGGTGGCT-3’ y antisentido 5’- CCAAATCTCAAAGTACGCACAAACGG-3’ TRAIL-R3 (612 pb), sentido 5'-GAAGAATTTGGTGCCAATGCCACTG-3' y antisentido 5'-CTCTTGGACTTGGCTGGGAGATGTG-3'. TRAIL-R4 (453 pb) sentido 5'-CTTTTCCGGCGGCGTTCATGTCCTTC-3' y antisentido 5'-GTTTCTTCCAGGCTGCTTCCCTTTGTAG-3' FLIPL (227 pb), sentido 5’-AATTCAAGGCTCAGAAGCGA-3’, y antisentido 5’GGCAGAAACTCTGCTGTTCC-3’ FLIPS (100 pb), sentido 5’-AATGTTCTCCAAGCAGCAATCC-3’, y antisentido 5’CCAAGAATTTTCAGATCAGGACAAT-3’ Bcl-2 (367 pb), sentido 5’-AGATGTCCAGCCAGCTGCACCTGAC-3’, y antisentido 5’-AGATAGGCACCAGGGTGAGCAAGCT-3’ Bcl-x (257 pb), sentido 5’- CATGGCAGCAGTAAAGCAAGC-3’, y antisentido 5’CTGCGATCCGACTCACCAATAC-3’ Mcl-1 (211 pb), sentido 5’- CTTAGTTGATATTTTGGGCTTGGG-3’, y antisentido 5’AGAAGTCAAAAAGTAGTCACTGGG-3’ XIAP ( 487 pb), sentido 5’-GGCCATCTGAGACACATGCAG-3’, y antisentido 5’GCATTCACTAGATCTGCAACC-3’ c-IAP1 (171 pb), sentido 5’- CCAGTTCTTTTCTACACTATAAT-3’, y antisentido 5’CTTAATCTGTTTATTTACAAGGG-3’ c-IAP2 (150 pb), sentido 5’- CAACATGGAGATTCGAAATCC-3’, y antisentido 5’CACATCACTCTTCTGTGAAGGG-3’ β-actina (661 pb), sentido 5’-TGACGGGGTCACCCACACTGTGCCCATCTA-3’ y antisentido 5’-CTAGAAGCATTTGCGGTGGACGATGGAGGG-3’ 79 Las condiciones de los ciclos para las PCRs fueron los siguientes: TRAIL, TRAIL-R1, TRAIL-R2 y TRAIL-R3: 1 min a 95 °C, 1 min a 55 °C, y 1 min a 72 °C. TRAIL-R4, Bcl-2 y β-actina: 1 min a 95 °C, 1 min a 60 °C, y 1 min a 72 °C FLIPS y FLIPL: 40 seg a 95 °C, 40 seg a 60 °C, y 40 seg a 72 °C. Bcl-x, Mcl-1, XIAP, cIAP1 y cIAP2 : 1 min a 95 ºC, 1 min a 57 °C y 1 min a 72 °C. El número de ciclos osciló entre los 30-35 en función del gen. Los productos de PCR migraron en geles de agarosa (Ecogen) al 1% en tampón TAE y 0,5 μg/ml de bromuro de etidio. La reacción de PCR a tiempo real se llevó a cabo en el termociclador iCycler iQ detection system (Bio-Rad) empleando los reactivos iQ SYBR Green Supermix (BioRad), de acuerdo con las instrucciones facilitadas por el fabricante: 12.5 μl de reactivo Supermix 2x, 0.5 μM de cada uno de los cebadores y 1 μl de ADNc en un volumen final de 25 μl. Cada reacción se llevó a cabo por triplicado. El agente intercalante SYBR Green se une al ADN de doble cadena inmediatamente después de su síntesis y emite fluorescencia tras su estimulación (absorción a 497 nm y emisión a 520 nm). La emisión es recogida por el sistema detector del equipo en cada uno de los ciclos de PCR, siendo la fluorescencia proporcional a la cantidad de ADN formado. El ciclo de la PCR en que se comienza a detectar el incremento de la señal de denomina ciclo umbral o Ct (threshold cycle) y es inversamente proporcional a la concentración inicial de ADN de la muestra. Los valores de Ct de cada muestra se normalizaron con respecto a un gen de referencia, en concreto el ARNr 18S. La cuantificación relativa de la expresión génica se determinó usando el método comparativo descrito (Wang et al., 2002a) donde el número de veces de inducción es igual a 2-ΔΔCt, siendo ΔΔCt la diferencia entre el ΔCt de la muestra problema y el ΔCt de la muestra control (normalizados según la expresión del gen ARNr 18S). Para las reacciones de PCR a tiempo real se utilizaron los siguientes cebadores: FLIPS (100 pb), sentido 5’-AATGTTCTCCAAGCAGCAATCC-3’, y antisentido 5’CCAAGAATTTTCAGATCAGGACAAT-3’ FLIPL (102 pb), sentido 5’-AATTCAAGGCTCAGAAGCGA-3’, y antisentido 5’GGCAGAAACTCTGCTGTTCC-3’ 80 Caspasa-3 (73 pb) forward 5’-TTGAATTGATGCGTGATGTT-3’ y reverse 5’TGGCTCAGAAGCACACAAAC-3’ Caspasa-9 (64 pb) forward 5’-GCTTCTCCTCGCTGCATTT-3’ y reverse 5’AGCACCATTTTCTTGGCAGT-3’ 18s (98 pb) sentido 5’- GATATGCTCATGTGGTGTTG-3’ y antisentido 5’AATCTTCTTCAGTCGCTCCA-3’ En todos los casos se llevaron a cabo 40 ciclos de PCR en las siguientes condiciones: 30 seg a 95 °C, 30 seg a la Tª de hibridación y 45 seg a 72 °C. Las temperaturas de hibridación fueron 60 ºC para FLIPs, 56 ºC para FLIPL y 63 ºC para caspasa-3, caspasa-9. 8.- Microscopía de fluorescencia La translocación al núcleo de la subunidad p65 del factor de transcripción NF-κB se observó por microscopía de fluorescencia en linfocitos T activados procedentes de donantes sanos. Para ello se esterilizaron cubreobjetos con etanol 70% y, una vez secos, se dispusieron en placas de cultivo. Se añadió 2 ml de poli-L-lisina (Sigma) y se incubó 1 hora a 37 ºC. Una vez transcurrido este tiempo, se recogió el exceso de poli-L-lisina y se lavó la placa con PBS. Se añadieron 5x106 células en 1 ml de medio completo y se mantuvieron toda la noche para su adhesión al cubreobjetos. Tras el tratamiento en presencia o en ausencia del inhibidor de NF-κB durante 1 hora, se retiró el medio y se lavó la placa con PBS. Las células se fijaron en paraformaldehido al 4% durante 10 minutos a temperatura ambiente, retirando el exceso mediante lavado con PBS-Tween 0.1%. El marcaje de la proteína p65 se realizó incubando con una solución del anticuerpo monoclonal 1:100 en PBS-BSA 1% durante toda la noche a 4 ºC y manteniendo la placa húmeda (envuelta en papel empapado en agua). Posteriormente se lavó la placa con PBS-Tween 0.1% tres veces durante 5 minutos antes de añadir el anticuerpo secundario marcado con FITC a una concentración de 1:1000 durante 1 hora a temperatura ambiente. Por último, los núcleos se marcaron con diclorhidrato 4,6Diamino-2-fenil indol (DAPI, Sigma) a una concentración de 2 μg/ml incubando 10 minutos a temperatura ambiente en oscuridad. El exceso se retiró por lavado con PBS- 81 Tween 0.1%. Las células teñidas fueron analizadas en un microscopio de fluorescencia Leica TCS NT (Leica Microsystems, Alemania). 9.- Obtención y clonaje en el vector reportero de luciferasa (pXP2) de los fragmentos del promotor de TRAIL Los distintos fragmentos del promotor del gen de TRAIL, amablemente cedidos por el Dr. Abelardo López Rivas (CABIMER, Sevilla), se obtuvieron por PCR en base a la secuencia del promotor del gen humano de TRAIL publicada con número de acceso AF178756 del Genbank. Los cebadores utilizados, así como el protocolo seguido para llevar a cabo el clonaje de dichos fragmentes en el vector reportero de luciferasa pXP2, aparecen descritos en la tesis de la Dr. Carmen Ruiz de Almodóvar Egea (Granada, 2004). 10.-Transfecciones transitorias Las transfecciones transitorias se llevaron a cabo en células Jurkat empleando el reactivo Lipofectamina (Invitrogen). Las distintas construcciones del promotor de TRAIL incluidas en el vector pXP2 se mezclaron con Lipofectamina en una proporción de 3 μl del reactivo por 0.5 μg de plásmido, diluidos ambos previamente en 50 μl de medio OPTIMEM (GIBKO). Las diluciones del vector llevaban también 0.5 μg de un plásmido de expresión de β-galactosidasa, que se cotransfecta para normalizar la eficiencia de transfección. La mezcla de plásmidos y Lipofectamina se incubó durante 20 minutos a temperatura ambiente. Acto seguido, los 100 μl de OPTIMEM conteniendo el plásmido asociado a Lipofectamina se añadieron a cada pocillo conteniendo 5x105 células en 500 μl de medio RPMI y se llevó a cabo la incubación de dichas células durante 24 horas a 37 ºC. Pasado este tiempo las células fueron tratadas o no con los estímulos correspondientes durante 6 horas, se lisaron con 200 μl de tampón de lisis suministrado en el kit de luciferasa (Luciferase Assay System, Promega) y se midió la cantidad de proteínas de cada muestra mediante el método de Bradford. Finalmente, la actividad luciferasa se midió en un luminómetro siguiendo el protocolo 82 indicado en dicho kit. La eficiencia de transfección se normalizó mediante la medida de actividad β-galactosidasa. Para medir dicha actividad, se tomó la misma cantidad de lisado total que se utilizó para medir actividad luciferasa, se mezcló con una solución de fosfato sódico 0,1M con 0,88 mg de ONPG (Sigma) y 0,1 mM de MgCl2, y se incubó a 37 ºC hasta que la densidad óptica a 420 nm de la muestra se encontraba en el rango de 0.2 a 0.8. Se midió la densidad óptica y con el valor obtenido se normalizó la actividad luciferasa previamente determinada. De la misma manera se llevaron a cabo las transfecciones con el vector reportero de NF-kB, pKBF-Luc cedido amablemente por el Dr. Juan Miguel Redondo (CNIC, Madrid), así como la determinación de la actividad luciferasa en las células transfectadas con el mismo. 83 84 IV. RESULTADOS 85 86 1.REGULACIÓN DE LA EXPRESIÓN DE TRAIL EN CÉLULAS T ACTIVADAS. 1.1 Activación del promotor de TRAIL en células T en respuesta al tratamiento simultáneo con ionomicina y PMA Con el objeto de estudiar la regulación de la expresión del ligando de muerte TRAIL en linfocitos T en diferentes estados de activación, en primer lugar se analizó la expresión de dicho ligando, a nivel de ARNm, en células T de sangre periférica en reposo y a los 6 días de activación, como se describe en materiales y métodos. Los resultados de la figura 1 demuestran que la expresión del ARNm de TRAIL en linfocitos T activados es considerablemente superior a la observada en células T en reposo. R A TRAIL β-actina Figura 1. Expresión del ARNm de TRAIL en células T activadas. La expresión del ARNm de TRAIL se analizó mediante RT-PCR en linfocitos T en reposo (R) y a los 6 días de activación (A). Como control de carga se muestra la expresión de β-actina. Mediante el uso de programas informáticos, como TRANSFAC, se han identificado en el promotor de TRAIL distintos sitios de unión a factores de transcripción, como GATA, C/EBP, GAS, ISRE, NF-κB o NFAT. Como ya se ha mencionado en la introducción, se ha descrito la implicación de NF-κB en la regulación de la expresión de TRAIL en células T en respuesta a la estimulación con agentes que imitan la activación del TCR, posiblemente por unión de este factor a dos secuencias consenso localizadas entre las posiciones -384 a -375 (sitio κB2) y -256 a –265 pb (sitio κB1) del promotor de TRAIL, con respecto al sitio de inicio de transcripción (Baetu et al., 2001). Sin embargo, no se sabe nada acerca de la implicación de otros factores de transcripción que, como NF-κB, se activan en respuesta a la estimulación de la célula T. Para conocer mejor los factores que regulan la expresión de TRAIL en células T activadas, utilizamos distintas construcciones del promotor de este ligando que habían sido obtenidas previamente por la doctora Ruiz de Almodóvar (datos no publicados) mediante clonaje de diferentes fragmentos de 1295, 787 y 165 pb en el vector pXP2 que carece de promotor mínimo y que contiene el gen de luciferasa, tal como se describe en el apartado de materiales y métodos (figura 2A). 87 +1 A p1295Luc κB2 SP-1 κB2 p787Luc Actividad luciferasa (RLUsx105) Actividad luciferasa (RLUsx106) C +1 κB1 SP-1 p165Luc B Luc κB1 Luc +1 Luc SP-1 6 5 2.7x 4 3 2 1.4x 1.5x PMA Io 1 0 Control PMA+Io 2.5 4.2x 2 4.2x 4x 1.5 1 0.5 0 pXP2 + - + p165Luc - + p787Luc - + PMA+Io p1295Luc Figura 2. El tratamiento simultáneo con ionomicina y PMA induce la actividad transcripcional del promotor de TRAIL en células Jurkat. A) Esquema de los fragmentos de 165, 787 y 1295 pb del promotor de TRAIL que tras ser obtenidos por PCR fueron clonados en el vector pXP2 reportero de luciferasa. Se indican los sitios de unión a NF-κB descritos (κB1 y κB2) y los posibles sitios de unión a NFAT (el fragmento de 1295 pb presenta 12 sitios para NFAT, el de 787 pb presenta 10 sitios y el de 165 pb 3 sitios) B) Células Jurkat se transfectaron con la construcción p787Luc del promotor de TRAIL y la actividad luciferasa de la misma se analizó a las 6 horas de tratamiento con PMA (100 nM) +/- ionomicina (1 μg/ml). C) La actividad luciferasa de los fragmentos p1295Luc, p787Luc y p165Luc y del vector vacio pXP2 se analizó en células Jurkat transfectadas y tratadas con PMA + ionomicina. Para normalizar la eficiencia de transfección se contransfectó en todos los casos un vector de expresión de β-gal. Los resultados mostrados son representativos de al menos 3 experimentos independientes. 88 Se transfectaron transitoriamente células Jurkat con la construcción de 787 pb, que contiene los dos sitios de unión a NF-kB mencionados así como posibles sitios de unión al factor NFAT entre otros, y 24 horas después se activaron mediante tratamiento con ionomicina y/o PMA durante 6 horas. La actividad transcripcional del fragmento del promotor se cuantificó midiendo actividad luciferasa en lisados de las células transfectadas. Como se observa en la figura 2B, dicho fragmento tiene actividad transcripcional inducible en respuesta a la activación simultánea con ionomicina y el éster de forbol, pero no con dichos agentes por separado, indicando la importancia de los factores de transcripción regulados por ambos, NFAT y NF-kB respectivamente, en la regulación del promotor de TRAIL. A continuación se comparó la actividad transcripcional de los tres fragmentos, de 1295, 787 y 165 pb, basal y en respuesta al tratamiento con ionomicina y PMA. Como control de la actividad luciferasa basal se analizó también la actividad del vector vacio pXP2. Curiosamente, y a pesar de lo descrito en la literatura sobre la localización de los sitios de unión a NF-κB, no se observaron diferencias ni en la actividad basal ni en la inducible por el tratamiento con ionomicina y PMA entre los diferentes fragmentos (figura 2C), lo que sugiere que el fragmento más pequeño de 165 pb contiene no solo los elementos mínimos necesarios para controlar la expresión basal de este gen sino posiblemente también los sitios consenso de unión y respuesta a los factores de transcripción NFAT y NF-κB. 1.2 Regulación de la expresión y función de TRAIL por ciclosporina A en células T activadas La activación de la calcineurina, fosfatasa dependiente de Ca2+/calmodulina, es necesaria para la activación del factor de transcripción NFAT tras la estimulación de la célula T con agentes que imitan la activación del TCR. Para confirmar la implicación de este factor de transcripción en la regulación de la expresión de TRAIL en células T en respuesta a la activación utilizamos la droga inhibidora de calcineurina, ciclosporina A (Cs A) (Handschumacher et al., 1984). El tratamiento de células T activadas con ciclosporina A durante 8 horas produjo una disminución en los niveles de ARNm de TRAIL (figura 3A). 89 A Control CsA TRAIL β-actina B % Células apoptóticas 100 HeLa BT474 80 SKBr3 60 40 20 0 Control TRAIL 1:2 1:5 + T activadas C % Células apoptóticas 100 80 60 40 20 0 C TRAIL - + +T reposo - + CsA +T activadas Figura 3. El tratamiento con ciclosporina A reduce la expresión de TRAIL y la actividad citotóxica de células T activadas. A) Células T activadas fueron incubadas durante 8 horas en presencia o en ausencia de 500ng/ml CsA y los niveles de ARNm de TRAIL se determinaron mediante RT-PCR. Como control de carga se analizó la expresión de β-actina. B) Células Hela, SKBr3 y BT474 se co-cultivaron durante 24 horas con o sin células T activadas a las proporciones indicadas (célula tumoral:célula T) y se analizó el porcentaje de células tumorales en el pico sub-G1 del ciclo celular. C) Células T en reposo y activadas, pretratadas durante 24 horas con o sin 500ng/ml CsA, se co-incubaron con células Hela en una proporción 1:5 (Hela: células T). Tras 24 horas de co-cultivo se determinó el porcentaje de células Hela en apoptosis. Como control positivo de inducción de apoptosis, las células tumorales en B) y C) se trataron con TRAIL (250ng/ml). Los resultados son representativos de dos experimentos independientes. 90 La expresión de TRAIL en linfocitos T activados puede facilitar o potenciar la acción citotóxica de los mismos sobre células tumorales. Con objeto de conocer la posible actividad citotóxica de nuestro modelo de células T activadas, realizamos cocultivos de dichas células T con diferentes tipos de células tumorales sensibles y resistentes a TRAIL, a diferentes proporciones, y analizamos la inducción de apoptosis en las células tumorales. Los resultados de la figura 3B indican que la inducción de apoptosis en células Hela, sensibles a TRAIL, tras su co-cultivo con células T activadas en una proporción 1:5 (Hela: células T) es similar a la observada en respuesta al tratamiento con TRAIL recombinante. Sin embargo, las células T activadas no fueron capaces de inducir apoptosis en dos líneas de cáncer de mama, SKBr3 y BT474, resistentes a TRAIL. Sólo se observó un débil efecto en células SKBr3, similar con las dos proporciones de célula diana: célula efectora y comparable al efecto detectado en Hela a la proporción 1:2, por lo que pensamos que podría tratarse de un efecto tóxico inespecífico derivado de la co-incubación. El pretratamiento de los linfocitos T activados con ciclosporina A redujo parcialmente su capacidad de inducción de apoptosis en células Hela (figura 3C) sugiriendo la importancia de NFAT en la expresión del ligando de muerte TRAIL funcional en dichos linfocitos activados. Observamos además que, a diferencia de ellos, los linfocitos T en reposo no eran capaces de inducir apoptosis en células Hela (figura 3C). 2. MECANISMOS QUE REGULAN LA RESISTENCIA A TRAIL EN LINFOCITOS T PRIMARIOS HUMANOS 2.1 Resistencia de linfocitos T primarios en reposo y activados a la inducción de apoptosis por TRAIL Diversos grupos han descrito la resistencia a la apoptosis mediada por TRAIL en células normales y en diferentes subpoblaciones de células sanguíneas (Hasegawa et al., 2004; Mirandola et al., 2004). Para una mejor caracterización de los efectos de TRAIL sobre células T primarias, analizamos la inducción de apoptosis en linfocitos T activados y en reposo, procedentes de 10 donantes sanos diferentes, en respuesta al tratamiento con una dosis elevada de TRAIL recombinante (250 ng/ml). En todos loscasos las células primarias demostraron ser resistentes a la apoptosis mediada por 91 TRAIL (figura 4A). Para asegurarnos de la resistencia de este tipo celular repetimos los experimentos con tratamientos prolongados durante 48 horas y empleando dosis aún más elevadas del ligando de muerte (500 ng/ml), obteniéndose idénticos resultados (datos no mostrados). Como control positivo en todos estos experimentos se utilizaron células T leucémicas Jurkat que son sensibles a TRAIL. % Células apoptóticas A 100 100 80 80 REPOSO-CONTROL REPOSO-TRAIL 60 60 ACTIVADOS-CONTROL ACTIVADOS-TRAIL 40 40 JURKAT-CONTROL JURKAT-TRAIL 20 20 00 Reposo 0 B Reposo - + Activados Activados - + 2 Jurkat Jurkat - + TRAIL Caspasa-8 41-43 kDa Figura 4. Los linfocitos T en reposo y activados son resistentes a la apoptosis mediada por TRAIL. A) Linfocitos T en reposo o activados, procedentes de 10 donantes sanos, se trataron durante 24 horas con 250 ng/ml TRAIL. El porcentaje de células apoptóticas se determinó mediante análisis del pico sub-G1 por citometría de flujo. B) La activación de caspasa-8 se analizó mediante Western Blot en linfocitos T en reposo y activados tras 15 horas de tratamiento con 250 ng/ml TRAIL. En ambos casos, como control positivo de células sensibles a la inducción de apoptosis se utilizaron células Jurkat tratadas con TRAIL (100 ng/ml). Estudiamos también la activación de la caspasa-8 apical, como primer acontecimiento bioquímico que tiene lugar tras la unión de TRAIL a sus receptores en la superficie celular, mediante el análisis del procesamiento de las dos isoformas de procaspasa-8 (a y b) en los fragmentos proteolíticos intermedios de 43-41 kDa. Como se observa en la figura 4B, encontramos un bloqueo de la ruta de inducción de apoptosis por TRAIL a este nivel tan temprano, tanto en células T primarias en reposo. 92 2.2 Expresión de proteínas involucradas en la ruta de señalización de TRAIL en linfocitos T en reposo y activados Puesto que la apoptosis mediada por TRAIL requiere en primer lugar de la unión del ligando a sus receptores de muerte TRAIL-R1 y -R2, y dado que se ha descrito la implicación de los receptores señuelo TRAIL-R3 y –R4 en la resistencia de células normales a TRAIL, analizamos el patrón de expresión de los receptores de TRAIL en la membrana de linfocitos T en reposo y activados. Así, observamos que los niveles de TRAIL-R3 y -R1 eran prácticamente nulos y los de TRAIL-R2 muy bajos en todas las muestras de linfocitos analizadas, tanto en reposo como activados, encontrando mínimas diferencias entre donantes. A TRAIL-R1 TRAIL-R2 TRAIL-R3 TRAIL-R4 Reposo Activadas Intensidad de Fluorescencia Figura 5. Expresión de los receptores de TRAIL en células T en reposo y activadas. La expresión de los receptores de TRAIL en membrana se determinó por citometría de flujo en células T en reposo y activadas utilizando los anticuerpos apropiados (picos sombreados). La línea sólida corresponde a la fluorescencia basal del anticuerpo secundario. Los resultados mostrados son representativos de al menos 3 donantes diferentes. Por el contrario, en el caso de TRAIL-R4 observamos una alta expresión y, curiosamente, detectamos dos subpoblaciones celulares caracterizadas por la diferente intensidad de fluorescencia de los picos mostrados en el análisis por citometría de flujo (figura 5). El pico de alta intensidad de fluorescencia parece corresponderse con la subpoblación CD8+ (Hasegawa et al., 2004, Mirandola, 2004 #252) mientras el pico de baja intensidad de fluorescencia, que presenta una cierta variabilidad entre donantes en 93 cuanto a intensidad se refiere, corresponde a la expresión de TRAIL-R4 en las células CD4+. Encontramos además un cierto incremento en la intensidad de fluorescencia de las dos subpoblaciones celulares en linfocitos T activados con respecto a los linfocitos en reposo, así como un aumento del porcentaje de células con mayor expresión de TRAIL-R4 (figura 5), posiblemente debido al incremento en la relación CD8+/CD4+ que conlleva la activación linfocitaria. Analizamos a continuación los niveles de expresión de otras proteínas que, junto con los receptores de muerte, están implicadas en la formación del DISC y por tanto en la activación de la caspasa-8. Como se observa en la figura 6, tanto las células T activadas como en reposo expresaban la proteína adaptadora FADD siendo los niveles de esta proteína similares a los de células Jurkat sensibles a TRAIL. Sin embargo, en el caso de la proteína inhibidora c-FLIP encontramos que los niveles de la isoforma larga (cFLIPL) eran mucho mayores en células T primarias, en reposo y activadas, que en células Jurkat. También los niveles de la isoforma corta (c-FLIPS), aunque bajos en células T primarias, eran superiores a los encontrados en esta línea de células T sensibles a TRAIL (figura 6). Estos datos sobre la expresión de FLIP, junto con los obtenidos tras el análisis de expresión de los receptores de TRAIL en membrana, pueden explicar la resistencia de células T primarias a la apoptosis mediada por este ligando de muerte. Jurkat Reposo Activados FADD FLIPL FLIPS α-Tubulina Figura 6. Expresión de proteínas implicadas en la formación del DISC en linfocitos T en reposo y activados. Los niveles de FADD, FLIPL y FLIPs se analizaron en células T en reposo y activadas y en células Jurkat mediante Western-blot. Como control de carga se analizó la expresión de α-tubulina. Los resultados mostrados son representativos de al menos 3 donantes diferentes. Con excepción de TRAIL-R4, en los factores hasta aquí analizados no encontramos diferencias significativas entre los niveles de expresión entre células T en reposo y activadas, a pesar de que varios autores han propuesto una mayor sensibilidad a la muerte por apoptosis en células T activadas (Green et al., 2003; Krammer et al., 2007). Analizamos también la expresión de algunos miembros de la familia de Bcl-2, implicados en la ruta mitocondrial de señalización de TRAIL, en células T en los 94 diferentes estadios de activación y observamos que los niveles de la propia proteína anti-apoptótica Bcl-2 eran similares en células en reposo y activadas, mientras que otras proteínas pro-apoptóticas pertenecientes a la misma familia variaban su expresión con la activación (figura 7A). A Reposo B Activadas Bax Bid Bid Bcl-2 β-actina XIAP Reposo Activadas α-Tubulina Figura 7. Expresión de factores intracelulares implicados en la ruta de señalización de TRAIL en linfocitos T en reposo y activados. A) La expresión de Bax, Bid, Bcl-2 y XIAP se determinó por Western Blot en células T en reposo y activadas. Como control de carga se analizó la expresión de α-tubulina. Los resultados mostrados son representativos de al menos tres donantes diferentes. B) Los niveles de ARNm de Bid se determinaron en células T en reposo y activadas mediante PCR semi-cuantitativa, utilizando diferentes cantidades de ADNc (2, 1 y 0,5 μl). Como control de cantidad de ARNm se muestra la expresión de β-actina. En concreto, encontramos una reducción reproducible y muy significativa en la expresión de Bid en células T activadas, y un cierto incremento en el contenido intracelular de Bax en las células T activadas respecto al estado de reposo en la mayoría de los casos analizados. En cuanto a los niveles de la proteína inhibidora de apoptosis XIAP, no encontramos diferencias en la expresión en respuesta a la activación. Curiosamente, cuando analizamos los niveles de expresión del ARNm de Bid mediante PCR semi-cuantitativa observamos que eran similares en células T en reposo y activadas, lo que sugiere la existencia de mecanismos post-transduccionales responsables de la regulación de esta proteína en ambos tipos de células T (figura 7B). 95 2.3 Efecto de diferentes moduladores de la sensibilidad a TRAIL en la resistencia de linfocitos T primarios humanos Se han descrito numerosos modelos de células tumorales resistentes a TRAIL en los cuales dicha resistencia es debida, entre otras causas, a la baja expresión de receptores pro-apoptóticos y a los elevados niveles de proteínas anti-apoptóticas intracelulares. Sin embargo, la mayoría de estos tumores pueden sensibilizarse a TRAIL mediante el empleo de diferentes estrategias terapéuticas como son las drogas quimioterapéuticas, inhibidores de histona deacetilasa, inhibidores del proteasoma o inhibidores de rutas de supervivencia (Inoue et al., 2004; Kandasamy and Srivastava, 2002; Sayers and Murphy, 2006; Shankar and Srivastava, 2004; Singh et al., 2003). Decidimos estudiar el efecto de algunos agentes, pertenecientes a los mencionados grupos farmacológicos, sobre la resistencia de células T primarias a la apoptosis mediada por TRAIL. Concretamente, analizamos el efecto del agente genotóxico doxorubicina, del inhibidor de la polimerización de los microtúbulos de actina nocodazol, del inhibidor de la PKC bisindolilmaleimida (BIS), del inhibidor de PI3K LY294002, del inhibidor de histona deacetilasa ácido valproico (VPA) y del inhibidor del proteasoma velcade (bortezomib), utilizando concentraciones de dichos agentes establecidas en base a publicaciones previas y a su capacidad para sensibilizar a las líneas tumorales CEM-6, línea de células leucémicas T que presentan una moderada sensibilidad a TRAIL, y SKBr3, línea de células de cáncer de mama muy resistentes a este ligando de muerte. En la figura 8 se observa cómo doxorubicina, LY294002 y VPA eran capaces de potenciar la acción de TRAIL en células CEM-6 mientras que nocodazol y bortezomib sensibilizaban claramente a las células SKBr3, a las concentraciones establecidas. Sin embargo en ninguno de los casos encontramos sensibilización de linfocitos T primarios, ni en reposo ni activados, a la apoptosis mediada por TRAIL. Es importante señalar que tanto doxorubicina, como nocodazol, y en especial bortezomib presentaron una alta toxicidad para los linfocitos T primarios activados. También el inhibidor de PKC, BIS, presentó cierto efecto sobre estas células. Y el bortezomib resultó ser además ligeramente tóxico para los linfocitos T en reposo (figura 8A, D, E, F). Por el contrario, los inhibidores LY294002 y VPA mostraron una ausencia total de toxicidad para los linfocitos T primarios, tanto en reposo como activados, lo que sugiere su posible utilización como agentes antitumorales en combinación con TRAIL. 96 % Células Apoptóticas A 100 100 80 80 Control TRAIL DXR 60 60 DXR + TRAIL 40 40 20 20 0 0 1 Reposo B 100 2 Activados 3 CEM % Células Apoptóticas 100 80 80 60 60 Control TRAIL LY * LY + TRAIL 40 40 20 20 0 0 Reposo 1 C % Células Apoptóticas 100 100 100 80 80 80 60 60 60 Activados 2 CEM 3 Control Control TRAIL TRAIL VPA VPA VPA + TRAIL VPA + TRAIL 40 40 40 20 20 20 0 00 1 Reposo D 2 Activados 3 CEM % Células Apoptóticas 100 100 80 80 60 60 Control TRAIL NZ NZ + TRAIL 40 40 *** 20 20 00 Reposo 1 Activados 2 SKBr3 3 97 E % Células Apoptóticas 100 100 80 80 60 60 Control TRAIL BZ ** BZ + TRAIL 40 40 20 20 0 0 1 Reposo F 2 Activados 3 SKBr3 % Células Apoptóticas 100100 80 80 Control 60 60 TRAIL BIS-I 40 40 BIS-I + TRAIL 20 20 00 Reposo Activados Figura 8. Resistencia de linfocitos T primarios a TRAIL tras el tratamiento con distintos agentes sensibilizadores. Linfocitos T en reposo y activados fueron preincubados con (A) doxorubicina (DXR, 100 ng/ml ), (B) LY 294002 (10 μM), (E) bortezomib (BZ, 50 nM ) o (F) BIS (6 μM) durante 1 h; o con (C) VPA (1 mM ) o (D) nocodazol (NZ, 400 ng/ml) durante 7h. Tras la preincubación, las células fueron tratadas con 250 ng/ml de TRAIL durante 24 h. Células CEM (A-C) y SKBr3 (D, E) fueron preincubadas en las mismas condiciones antes de ser tratadas con 50 ng/ml o 250 ng/ml de TRAIL, respectivamente, durante 20 horas. El porcentaje de células apoptóticas se determinó por análisis del pico sub-G1 mediante citometría de flujo. Las barras de error muestran la SD de tres experimentos independientes. La significación estadística se corresponde con *p < 0.05; **p < 0.01; ***p < 0.001, según el test de Student. 2.4 La inhibición de NF-κB sensibiliza a linfocitos T activados a la apoptosis mediada por TRAIL Junto a los agentes descritos en el apartado anterior analizamos también el efecto del inhibidor de NF-κB, BAY 11-7085, sobre linfocitos T primarios y su resistencia a TRAIL. En el caso de las células T en reposo, encontramos una gran variabilidad en la respuesta a este inhibidor entre los diferentes donantes estudiados (figura 9A). En 9 de las 10 muestras analizadas el inhibidor del factor de transcripción por sí solo presentaba efectos citotóxicos de intensidad variable en función del donante. En cuanto al efecto regulador de la apoptosis mediada por TRAIL, si bien en 4 de los 10 casos se apreciaba una sensibilización significativa de los linfocitos T en reposo a este ligando de muerte, 98 en otros 5 casos el porcentaje de células apoptóticas era inferior en respuesta al tratamiento combinado con BAY 11-7085 y TRAIL que tras el tratamiento solo con el inhibidor. Como se observa en la figura 9B, el inhibidor de NF-κB también presentó una toxicidad variable, en función del donante, para los linfocitos T activados. Sin embargo, en este caso encontramos una sensibilización significativa en todas las muestras analizadas, con excepción de una de ellas en el que el inhibidor solo provocó un efecto tóxico muy elevado (93% de células en apoptosis). AA % Células apoptóticas 100 100 80 80 60 60 40 40 20 20 00 BB Control TRAIL BAY BAY+TRAIL BAY BAY+TRAIL % Células apoptóticas 100 100 80 80 60 60 40 40 20 20 00 Control TRAIL Figura 9. Sensibilización de linfocitos T primarios a TRAIL por inhibición de NF-κB. Linfocitos T en reposo (A) o activados (B) de 10 donantes diferentes fueron preincubados durante 1 hora con BAY 11-7085 (2.5 μM) y a continuación tratados con TRAIL (250 ng/ml) durante 24 horas. El porcentaje de células apoptóticas se determinó mediante análisis del pico sub-G1 del ciclo celular. Se llevó a cabo un estudio de dosis-respuesta con el inhibidor BAY 11-7085, tratando de encontrar la dosis que con menor efecto tóxico indujera sensibilización a TRAIL en células T activadas. Concentraciones inferiores a 2.5 μM, aunque 99 presentaban baja toxicidad, no sensibilizaban a TRAIL de forma estadísticamente significativa por lo que dicha dosis se estableció como la óptima para continuar el estudio del efecto de este inhibidor en linfocitos T activados (figura 10). Estos resultados coinciden con los publicados por otros autores en los que se utilizan dosis similares del inhibidor de NF-κB para inducir la sensibilización a TRAIL de distintas líneas de células tumorales (Huerta-Yepez et al., 2004). % Células Apoptóticas 100 ** ** CONTROL 80 TRAIL-250 60 40 20 0 Control 1 2,5 BAY 11-7085 (μM) Figura 10. Efecto de distintas dosis de BAY 11-7085 en la sensibilización de linfocitos T activados a TRAIL. Linfocitos T activados de distintos donantes sanos fueron preincubados con distintas dosis de BAY 11-7085 durante 1 hora antes del tratamiento con TRAIL (250 ng/ml) durante 24 horas. El porcentaje de células apoptóticas se determinó por citometría de flujo. Las barras de error muestran la SD de tres experimentos independientes. Significación estadística **p < 0.01, según el test de Student. Simultáneamente al estudio de sensibilización a TRAIL en linfocitos T comprobamos que el inhibidor BAY 11-7085 efectivamente bloqueaba la activación de NF-κB. Se ha descrito que este compuesto actúa inhibiendo a la quinasa IKK y así la fosforilación de IκB, necesaria para la activación y translocación al núcleo de NF-κB (Bremner and Heinrich, 2002). Analizamos por microscopía de fluorescencia la localización nuclear de la subunidad p65 de NF-κB en linfocitos T tratados o no con BAY 11-7085 (figura 11A). Aún presentando estas células una morfología esférica constituida por núcleo en su práctica totalidad, el bloqueo de la entrada de la subunidad p65 es claro en células tratadas, donde la mayor parte de dicha subunidad se localiza en las regiones perinucleares. Analizamos también la capacidad de BAY 11-7085 para inhibir la función de NF-κB como factor de transcripción. Para ello, se transfectaron células Jurkat con el vector pKBF-Luc, que contiene tres copias de la secuencia de unión a NF-κB del promotor de H-2Kb placed upstream of a herpes simplex virus thymidine kinase minimal promoter driving the luciferase reporter gene, y se trataron con ionomicina y PMA para inducir la activación de NF-κB. Como se muestra en la 100 figura 11B, el pretratamiento con distintas concentraciones de BAY 11-7085 provocó una inhibición, casi completa en el caso de la dosis más alta, de la actividad luciferasa inducida en respuesta a la activación con ionomicina y PMA. A CONTROL B Actividad Luciferasa (RLUs) 2400000 2000000 + BAY (1H.) Control Control PIo PMA+Io Pio+BAY-1 PMA+Io+BAY-1mM Pio+BAY2,5 PMA+Io+BAY-2,5mM 1600000 1200000 800000 400000 0 Figura 11. BAY 11-7085 inhibe la activación de NF-κB. A) LinfocitosT activados de donantes sanos se incubaron 1 hora con o sin BAY 11-7085 (2.5 μM) para determinar, mediante microscopía de fluorescencia, la localización de la subunidad p65 del factor de transcripción NF-κB (fluorescencia verde). La localización nuclear se estableció por tinción con DAPI (azul). B) Se transfectaron células Juktar con el vector NFκB-Luc. A las 24 horas se preincubaron con diferentes dosis de BAY 11-7085 (1 y 2.5 μM) durante 1 hora y a continuación se trataron con PMA (100 nM) + Ionomicina (1μg/ml) durante 4 horas para finalmente analizar la actividad luciferasa. Los datos mostrados son representativos de tres experimentos independientes. 101 Una vez analizada la capacidad de BAY 11-7085 para inhibir la activación de NFκB, se examinaron los efectos de otros inhibidores de este factor de transcripción para confirmar la implicación del mismo en el mecanismo de sensibilización de linfocitos T a TRAIL. En concreto, utilizamos sulfosalazina, partenolide y el éster feniletílico del ácido caféico (CAPE). Mientras que la sulfosalazina permitió la sensibilización de los linfocitos T activados a TRAIL, los otros dos agentes, aún habiéndose descrito también como inhibidores específicos de NF-κB (Bork et al., 1997; Natarajan et al., 1996; Wahl et al., 1998), presentaron un efecto muy débil (partenolide) o ningún efecto (CAPE) sobre la resistencia a TRAIL (figura 12A). Para comprobar que las concentraciones de los agentes empleados en el estudio inhibían la activación de NF-κB estudiamos la presencia de las subunidades p65 y p50 en el núcleo de células T activadas y tratadas con los inhibidores, esta vez mediante Western-blot. Así observamos que, tanto BAY 11-7085 como sulfosalazina y parthenolide reducían significativamente los niveles de expresión de ambas subunidades en el núcleo, en comparación con los niveles en células control no tratadas. Sin embargo no encontramos ningún efecto del CAPE sobre la expresión de NF-κB en el núcleo lo que explicaría la ausencia de sensibilización a TRAIL en respuesta al tratamiento con dicho agente (figura 12B). A B % Células apoptóticas 100 80 C BAY SS PT CAPE p65 60 CONTROL * TRAIL 40 p50 PARP 20 * 0 Control SS PT CAPE Figura 12. Efecto de diferentes inhibidores de NF-κB sobre la resistencia de linfocitos T activados a TRAIL. (A) Células T activadas fueron preincubadas 1 hora con 1.5 mM de sulfosalazina (SS), 2.5 μM de partenolinde (PT) o 50 μg/ml de CAPE antes de ser tratadas 24 horas con TRAIL (250 ng/ml). El porcentaje de células apoptóticas se determinó mediante análisis del pico sub-G1 por citometría de flujo. Las barras de error muestran la SD de tres experimentos independientes.* p<0.05 según el test T de Student. (B) La expresión de las subunidades de NF-κB p50 y p65 se determinó por Western Blot en los extractos nucleares procedentes de células T activadas y tratadas durante 4 horas con los inhibidores indicados en (A), o con BAY 11-7085 (2.5 μM). Como control de carga se utilizó la proteína nuclear PARP. 102 Por otro lado, comprobamos que la inducción de apoptosis por TRAIL en células T sensibilizadas por inhibición de NF-κB era un proceso dependiente de la activación de caspasas. Se preincubaron células T activadas con el inhibidor general de caspasas ZVAD antes del tratamiento con BAY 11-7085 y TRAIL, y así observamos una inhibición no solo del efecto sensibilizador del BAY 11-7085 sino también de su efecto citotóxico (figura 13A). De la misma manera se encontró un bloqueo de los efectos del inhibidor de NF-κB en células T en reposo (figura 13B). Solamente en aquellos casos en que la toxicidad del BAY 11-7085 era extrema, el pretratamiento con ZVAD no logró inhibir este efecto lo que sugiere que el inhibidor de NF-κB, por si solo, es capaz de activar vías de señalización de muerte celular dependientes e independientes de caspasas en linfocitos T (datos no mostrados). Observamos también la activación de caspasa-8 en respuesta a TRAIL en células T activadas preincubadas con BAY 11-7085, lo que confirma la activación de la ruta de señalización de TRAIL desde su extremo más apical en linfocitos T sensibilizados por el inhibidor de NF-κB (figura 13C). % Células Apoptóticas AA BB 100 100 8080 100 100 100 100 - 80 80 80 80 Z-VAD 6060 60 60 60 60 4040 40 40 40 40 20 20 20 20 20 20 0 0 1 Control 2 TRAIL CC 3 BAY - 000 0 4 BAY+TRAIL + - + + + -Z-VAD Z-VAD Z-VAD Control 1 TRAIL 2 BAY 3 BAY+TRAIL 4 TRAIL BAY 11-7085 Caspasa-8 41-43 kDa α-Tubulina Figura 13. TRAIL induce la activación de caspasas en linfocitos T primarios sensibilizados por inhibición de NF-κB. Linfocitos T activados (A) o en reposo (B) fueron preincubados en presencia o en ausencia de Z-VAD (50 μM) y/o BAY 11-7085 (2.5μM) durante 1 hora y a continuación tratados con o sin TRAIL (250 ng/ml) durante 24 horas. El porcentaje de células apoptóticas se determinó por citometría de flujo. C) La activación de caspasa-8 se analizó mediante Western Blot en linfocitos T activados tratados con TRAIL (250 ng/ml) durante 20 horas tras pretratamiento de 1 hora con BAY 11-7085 (2.5μM). Como control de carga se analizó la expresión de α-tubulina. Los datos mostrados son resultados representativos de tres experimentos independientes con diferentes donantes. 103 2.5 BAY 11-7085 regula la expresión de receptores de TRAIL y de genes antiapoptóticos en linfocitos T activados El papel regulador del factor de transcripción NF-κB sobre la expresión de receptores y proteínas de la ruta de señalización de TRAIL se encuentra ampliamente descrito (Bernard et al., 2001; Micheau et al., 2001; Ravi et al., 2001). A fin de comprender el mecanismo por el que la inhibición de este factor de transcripción induce sensibilización a TRAIL en linfocitos T primarios, estudiamos la posible regulación de diversos factores implicados en la apoptosis mediada por este ligando y utilizamos para ello células T activadas como modelo celular, dado que la sensibilización en dichas células era mucho más consistente y reproducible que en el caso de los linfocitos T en reposo. Analizamos en primer lugar la expresión de los receptores TRAIL-R1, TRAILR2 y TRAIL-R4 en la superficie de células T activadas tras el tratamiento con BAY 117085. Como se observa en la figura 14A, no encontramos efecto alguno sobre la expresión de los receptores pro-apoptóticos en membrana mientras que los niveles del receptor anti-apoptótico TRAIL-R4 se veían reducidos sensiblemente tras el tratamiento con el inhibidor en todos los casos estudiados, afectando dicha reducción de forma similar a las subpoblaciones CD4+ y CD8+. A nivel de ARNm observamos una pérdida en la expresión de todos los receptores, dependiente del tiempo de tratamiento con BAY 11-7085 (figura 14B). B A TRAIL-R1 Tiempo (horas) TRAIL-R1 TRAIL-R2 TRAIL-R2 TRAIL-R4 β-Actina TRAIL-R4 104 0 6 9 C % Células Apoptóticas C 100 80 60 Control TRAIL BAY BAY + TRAIL 40 20 0 Cels T Totales CD4+ CD8+ Figura 14. Expresión de receptores de TRAIL en linfocitos T activados en respuesta a BAY 11-7085. A) Linfocitos T activados fueron incubados en presencia (picos no sombreados) o ausencia (picos sombreados) de BAY 11-7085 (2.5 μM) durante 15 horas y la expresión de los receptores TRAIL-R1, R2 y -R4 fue analizada por citometría de flujo. La línea discontinua muestra la florescencia basal del anticuerpo secundario. B) Los niveles de ARNm de los receptores fueron analizados por RT-PCR en linfocitos T activados tras el tratamiento con BAY 11-7085 (2.5 μM) a los tiempos indicados. Como control de la cantidad de ARNm se analizó la expresión de β-actina. C) La población total de linfocitos T activados de donantes sanos y las subpoblaciones separadas CD4+ y CD8+ se pretrataron 1 hora con BAY 11-7085 (2.5 μM) antes de ser incubadas con TRAIL (250 ng/ml) durante 24 horas. El porcentaje de células apoptóticas se determinó por citometría de flujo. Los resultados mostrados son representativos de tres experimentos independientes utilizando células de distintos donantes. La diferencia en la expresión de TRAIL-R4 entre las subpoblaciones CD4+ y CD8+ parece acentuarse aún más tras el tratamiento con el inhibidor de NF-κB, dado que en este contexto el porcentaje de células CD4+ que expresan dicho receptor es muy bajo (pico no sombreado de menor fluorescencia en el panel inferior de la figura 14A) mientras que el 100% de las células CD8 siguen expresándolo, aunque con menor intensidad. Estudiamos por ello la inducción de apoptosis por TRAIL en ambas subpoblaciones linfocitarias tras el pretratamiento con BAY 11-7085 y observamos que no existían diferencias significativas en la sensibilización de las dos subpoblaciones con respecto a la población total (figura 14C). Esto sugiere que la pérdida de expresión de este receptor, si bien puede jugar algún papel en la sensibilización a TRAIL de los linfocitos T activados por inhibición de NF-κB, no debe ser la única causa de la misma. Estudiamos entonces la expresión de distintas proteínas anti-apoptóticas como cFLIP, Bcl-2, Bcl-x, Mcl-1, XIAP, c-IAP1 y c-IAP2 en linfocitos T activados en respuesta al tratamiento con BAY 11-7085, no observando cambios en los niveles de ARNm de c-FLIPL, Bcl-x y Mcl-1 tras 9 horas de tratamiento (figura 15A). Por el 105 contrario, el descenso en los niveles de ARNm de c-FLIPs, Bcl-2, XIAP, c-IAP1 y cIAP2 fue significativo y dependiente del tiempo, tras la exposición al inhibidor. A Tiempo (horas) 0 6 9 B B - + BAY / 24 horas c-FLIPL c-FLIPL c-FLIPS c-FLIPS Bcl-2 c-IAP2 Bcl-x XIAP Mcl-1 Bcl-2 XIAP α-Tubulina c-IAP1 - + c-IAP2 Bcl-2 Incremento expresión (tanto por 1) β-Actina C BAY / 30 horas α-Tubulina D 2 - + BAY / 20 horas Bid α-Tubulina 1 0.33 0 - + BAY / 6 horas Figura 15. Expresión de factores intracelulares implicados en la ruta de señalización de TRAIL en céluas T activadas en respuesta a BAY 11-7085. Linfocitos T activados fueron tratados con BAY 117085 (2.5 μM) durante los tiempos indicados. Los niveles de ARNm de los genes c-FLIP, Bcl-2, Bcl-x, Mcl-1, XIAP, c-IAP1 y c-IAP2 se determinaron por RT-PCR (A). Los niveles de proteína de los genes mencionados (B) y de Bid (D) se analizaron por Western-blot. Como control de cantidad de carga se analizó la expresión de β-actina (A) o α-tubulina (B, D). C) Los niveles de ARNm de Bcl-2 se determinaron mediante PCR a tiempo real, llevando a cabo los experimentos por triplicado y normalizando los resultados con respecto a la expresión del ARNr 18S, para cada una de las muestras. Las barras de error representan la SD del triplicado. En todas las figuras los datos mostrados son representativos de al menos 3 experimentos independientes con células de diferentes donantes. 106 Determinamos también si los cambios observados a nivel de ARNm se correspondían con un descenso de los niveles de proteína y encontramos una pérdida de todas las analizadas tras 24 horas de tratamiento con el inhibidor, con la excepción de Bcl-2 (figura 15B, panel superior). El hecho de que los niveles de Bcl-2 no varíen a este tiempo puede deberse a que la vida media de esta proteína es muy larga (Reed, 1996). Así, encontramos que efectivamente se producía una reducción en la expresión de Bcl-2 tras 30 horas de tratamiento con BAY 11-7085 (figura 15B, panel inferior). Confirmamos también, mediante PCR a tiempo real, que la proteína Bcl-2 se regulaba a nivel transcripcional por el inhibidor de NF-κB en células T activadas (figura 15C). Además de todos los factores anti-apoptóticos mencionados, estudiamos la posible regulación por BAY 11-7085 de la proteína pro-apoptótica Bid, dada la disminución de los niveles de expresión de la misma en células T activadas con respecto a las células T en reposo (figura 7). Sin embargo, como se observa en la figura 15D, no encontramos ningún cambio en la expresión de Bid en las células T activadas tratadas con el inhibidor de NF-κB. En conjunto, todos estos datos indican que la regulación de diversas proteínas anti-apoptóticas, incluyendo entre ellas el receptor TRAIL-R4, puede ser el mecanismo por el cual la inhibición de NF-κB sensibiliza a las células T activadas a la apoptosis mediada por TRAIL. 3. REGULACIÓN DE LA SENSIBILIDAD A TRAIL EN CÉLULAS T LEUCÉMICAS. 3.1 Efecto de diferentes agentes moduladores de la sensibilidad a TRAIL en células T leucémicas Como ya se ha mencionado en el apartado anterior, numerosos agentes con diferentes mecanismos de acción se están ensayando en estrategias de terapia antitumoral combinada con TRAIL gracias a la capacidad de los mismos para romper la resistencia a este ligando o para potenciar su acción a dosis mínimas. Paralelamente al estudio descrito en el apartado anterior sobre la regulación de la resistencia a TRAIL de linfocitos T primarios de donantes sanos, analizamos la posible sensibilización a TRAIL en líneas de células T leucémicas, en concreto en las líneas celulares Jurkat, CEM-6 y MOLT-4. 107 % Células Apoptoticas A 100 80 Control Roscovitina TRAIL Roscovitina + TRAIL 60 40 20 0 B % Células Apoptoticas JURKAT 1 C 100 80 TRAIL Nocodazol + TRAIL % Células Apoptoticas CELs T ACTIVADAS 4 60 40 20 0 JURKAT 1 100 % Células Apoptoticas Nocodazol 80 CEM-6 2 MOLT-4 3 Control BAY TRAIL BAY + TRAIL CELs T ACTIVADAS 4 60 40 20 0 1 100 80 CEM-6 2 Control LY TRAIL LY + TRAIL MOLT-4 3 60 40 20 0 JURKAT 1 108 MOLT-4 3 Control JURKAT D CEM-6 2 CEM-6 2 MOLT-4 3 CELs T ACTIVADAS 4 E % Células Apoptóticas 100 80 Control VPA TRAIL VPA + TRAIL 60 40 20 0 1 JURKAT 2 CEM-6 3 MOLT-4 Figura 16. Inducción de apoptosis por agentes moduladores de la sensibilidad a TRAIL en células T leucémicas. Las líneas leucémicas T Jurkat, CEM-6 y MOLT-4 fueron pretratadas con (A) roscovitina 20 μM, (B) nocodazol 400 ng/ml o (E) VPA 1 mM durante 7 horas; o con (C) BAY 11-7085 2.5μM o (D) LY 294002 10μM durante 1 hora. Tras la preincubación, las células fueron tratadas con 100 ng/ml de TRAIL durante 20 h. El porcentaje de células apoptóticas se determinó por análisis del pico sub-G1 mediante citometría de flujo. Los datos son representativos de tres experimentos independientes. En A,B y C se muestra el resultado de un tratamiento idéntico, pero con 250 ng/ml TRAIL, en linfocitos T activados como control del efecto citotóxico de la droga (A,B) o de sensibilización a TRAIL (C). En la figura 16 se muestra la actividad de diferentes agentes, utilizados a concentraciones seleccionadas en base a publicaciones previas, sobre dichas líneas. Tanto la roscovitina, inhibidor de quinasas dependientes de ciclinas, como el nocodazol eran capaces de inducir muerte celular en las tres líneas, de forma similar a la observada en células T activadas (figura 16A, B). Y el tratamiento combinado con cualquiera de estos agentes y TRAIL solo suponía, a lo sumo, una suma de los efectos de ambos por separado. Curiosamente, cuando analizamos el efecto del inhibidor de NF-κB BAY 117085 encontramos que, a diferencia de la sensibilización que producía en linfocitos T primarios, en líneas leucémicas T no potenciaba la inducción de apoptosis por TRAIL, aunque sí presentaba un efecto citotóxico importante en las mismas (figura 16C). Estudiamos también el efecto de bortezomib, doxorubicina, BIS, PD 98059 (inhibidor de MEK) e IFN-α. De todos ellos, únicamente la doxorubicina presentaba un cierto efecto potenciador de la apoptosis en las tres líneas celulares, especialmente en células CEM-6 (figura 8A), mientras que el resto no producían ningún cambio en la viabilidad celular ni en la respuesta a TRAIL, a excepción del bortezomib que resultó ser ligeramente citotóxico (tabla I). 109 JURKAT CEM-6 MOLT-4 BAY 117085 (2.5 mM) +/- +/- +/- NOCODAZOL (400 ng/ml) +/- +/- + /- ROSCOVITINA (20 mM) +/- + /- + /- BORTEZOMIB (50 nM) (+) / - (+) / - (+) / - DOXO (100 ng/ml) - / (+) -/+ (+) /(+) BIS (6 mM) -/- -/- -/- PD 98059 (50 mM) -/- -/- -/- IFN-a (3 ng/ml) -/- -/- -/- LY 294002 (10 mM) (+) / + (+) / + (+) / + VPA (1 mM) -/+ -/+ -/+ Tabla I. Efecto de diferentes compuestos sobre la viabilidad y la sensibilización a TRAIL de células T leucémicas. Células Jurkat, CEM-6 y MOLT-4 se preincubaron con los agentes indicados durante 1 hora, excepto con nocodazol, roscovitina y VPA en que la preincubación fue de 7 horas y con IFN-α en que fue de 20 horas. Tras el pretratamiento se incubaron con o sin TRAIL (100 ng/ml) y se determinó la inducción de apoptosis por citometría de flujo. Se indica: citotoxicidad / sensibilización a TRAIL. Los resultados previos obtenidos con linfocitos T primarios sugerían la posible utilización del inhibidor de PI3K, LY294002, así como del inhibidor de HDAC, VPA, en el tratamiento de tumores dada la baja toxicidad de ambos compuestos para dichas células. Además habíamos visto que los dos potenciaban la apoptosis mediada por TRAIL en células CEM-6 (figura 8B, C). Los mismos resultados fueron obtenidos cuando analizamos su efecto en células Jurkat y MOLT-4. Especialmente llamativo fue el efecto del VPA, tanto por su escasa acción citotóxica como por su mayor capacidad para potenciar la acción de TRAIL en las tres líneas celulares estudiadas (figura 16D,E). Este efecto potenciador del VPA se observó incluso reduciendo el tiempo de preincubación con el mismo a 4 horas y utilizando dosis bajas de TRAIL del orden de 10 a 50 ng/ml (figura 17). 110 % Células Apoptoticas 100 80 JURKAT control 60 CEM control JURKAT + VPA CEM + VPA 40 MOLT control 20 MOLT + VPA 0 0 10 20 30 40 50 ng/ml TRAIL Figura 17. Efecto potenciador de VPA de la apoptosis mediada por TRAIL en células T leucémicas. Células leucémicas T Jurkat, CEM-6 y MOLT-4 fueron pretratadas o no durante 4 horas con VPA 1mM y a continuación incubadas con 10, 20 ó 50 ng/ml de TRAIL durante 20 horas. El porcentaje de células apoptóticas se determinó por análisis del pico sub-G1 mediante citometría de flujo. Los datos son representativos de tres experimentos independientes. 3.2 Regulación por VPA y LY 294002 de factores implicados en la ruta de señalización de TRAIL en líneas leucémicas T A fin de caracterizar el posible efecto de los inhibidores de HDAC y de PI3K sobre la expresión de proteínas intracelulares pertenecientes a la vía de señalización de TRAIL en células T leucémicas, estudiamos los niveles de ARNm de Bcl-2, Bcl-XL, cIAP1, c-IAP2 y XIAP tras 15 horas de tratamiento con dichos inhibidores (figura 18A). En ninguna de las tres líneas leucémicas T empleadas en el estudio encontramos diferencias entre las células tratadas y los controles no tratados. Sólo se muestran los resultados de células Jurkat como modelo representativo. En paralelo se analizó la expresión de dichas proteínas en células T activadas como modelo celular en el que el tratamiento con VPA y LY 294002 no regula la sensibilidad a TRAIL. A Jurkat C VPA T activadas LY C VPA LY Bcl-2 Bcl-x c-IAP1 c-IAP2 XIAP β-Actina 111 B JHM1 C VPA CEM LY C VPA T Activadas MOLT LY C C VPA LY VPA LY FLIPL FLIPS β -Actina CC T activadas Jurkat 2 2 1 1 0,31 0,35 VPA LY 0 0 C C VPA LY Figura 18. Expresión de factores anti-apoptóticos en líneas leucémicas T y células T activadas en respuesta al tratamiento con VPA y LY 294002. Células T activadas, células Jurkat (A, B, C), CEM-6 y MOLT-4 (B) fueron tratadas con LY 294002 10μM o VPA 1 mM durante 15 horas. Los niveles de ARNm de Bcl-2, Bcl-x, c-IAP1, c-IAP2, XIAP (A) y FLIP (B) se determinaron mediante RT-PCR, empleándose el producto de PCR de la β-actina como control de carga. En (C) los niveles de ARNm de FLIPs se analizaron mediante PCR a tiempo real llevando a cabo los experimentos por triplicado y normalizando los resultados con respecto a la expresión del ARNr 18S, para cada una de las muestras. Las barras de error representan la SD del triplicado. Los resultados mostrados en los 3 paneles son representativos de tres donantes diferentes en el caso de linfocitos T activados, y de tres experimentos independientes en el caso de las líneas celulares. Sin embargo, cuando analizamos los niveles de FLIP sí observamos un descenso claro en los niveles del ARNm de FLIPs en las tres líneas leucémicas, Jurkat, CEM-6 y MOLT-4, en respuesta al tratamiento con ambos inhibidores, pero no en las células T no transformadas (figura 18B). En los niveles de FLIPL, por el contrario, no se observó variación alguna ni en células T normales ni en leucémicas. La regulación selectiva de la expresión de FLIPs por VPA y LY 294002 en células T leucémicas se confirmó analizando los niveles de ARNm por PCR a tiempo real en células Jurkat y en células T activadas (figura 18C). Se ha descrito la regulación de la expresión del receptor TRAIL-R2 por inhibidores de HDAC en otros modelos tumorales (Insinga et al., 2005; VanOosten et al., 2005). 112 Por ello, analizamos también la capacidad del VPA para incrementar los niveles de expresión en membrana de este receptor en las diferentes líneas leucémicas T. Como se observa en la figura 19, el tratamiento con VPA produjo un cierto aumento en los niveles de TRAIL-R2 en células Jurkat y CEM-6, pero muy débil en células MOLT-4. Jurkat CEM-6 MOLT-4 Intensidad de Fluorescencia Figura 19. Expresión de TRAIL-R2 en membrana en células T leucémicas en respuesta al tratamiento con VPA. Células Jurkat, CEM-6 y MOLT-4 fueron incuabadas con (picos no sombreados) o sin (picos sombreados) VPA 1 mM durante 24 horas. La expresión del receptor TRAIL-R2 en membrana se analizó mediante citometría de flujo. La línea discontinua muestra la fluorescencia basal del anticuerpo secundario. 3.3 Potenciación por HDACi de la apoptosis mediada por TRAIL en líneas leucémicas T sensibles La ausencia de toxicidad del VPA en linfocitos T primarios, así como su gran capacidad para potenciar la inducción de apoptosis por TRAIL en líneas de células T leucémicas, sitúan a este compuesto, de entre todos los analizados en nuestro estudio, como el mejor candidato para ser utilizado en terapias combinadas con TRAIL. Decidimos entonces estudiar el efecto de distintos inhibidores de HDAC, pertenecientes a diferentes familias estructurales, en la sensibilidad a TRAIL de células T primarias y de células leucémicas T, para de este modo establecer posibles diferencias en los mecanismos de acción de los mismos y conocer cual de ellos es el más efectivo y selectivo en el tratamiento de leucemias de células T. En concreto, trabajamos con los siguientes HDACi: tricostatina A (TSA) y vorinostat (SAHA), pertenecientes a la familia de los hidroxamatos; VPA y butirato sódico (NaB), representativos del grupo de ácidos alifáticos; MS-275, perteneciente a la familia de las benzamidas; y apicidina como inhibidor del grupo de péptidos cíclicos. 113 En primer lugar tratamos células Jurkat y linfocitos T activados procedentes de donantes sanos con distintas concentraciones de cada uno de los inhibidores de HDAC, para comparar la posible acción citotóxica sobre ambos tipos celulares y establecer el límite de tolerancia de las células no transformadas. La selección de las concentraciones se realizó en base a las utilizadas por otros autores en diferentes modelos de células tumorales. A % Células apoptóticas 100 80 60 40 20 0 C 2 TSA TSA 1 3 VPA VPA 4 SAHA SAHA 5 MS-275 MS-275 6NaB NaB 7 Apicidina Apicidina 5 MS-275 6 NaB 7 Apicidina B % Células apoptóticas 100 80 60 40 20 0 C 1 2 TSA 3 VPA 4 SAHA Figura 20. Inducción de apoptosis por diferentes HDACi en células Jurkat y linfocitos T activados. Linfocitos T activados (A) y células Jurkat (B) fueron incubadas durante 24 horas en presencia de HDACi a distintas concentraciones: TSA 10, 30, 50, 75 y 100 ng/ml; VPA 0.5, 0.75, 1, 2.5 y 5 mM; SAHA 0.1, 0.3, 0.5, 1 y 2 μM; MS-275 0.5, 0.75, 1, 2.5 y 5 μM; NaB 0.1, 0.3, 0.5, 1 y 5 mM; y apicidina 10, 25, 50, 100 y 250 nM. El porcentaje de células apoptóticas se determinó por análisis del pico sub-G1 del ciclo celular. Los datos son representativos de al menos tres donantes independientes en el caso de linfocitos T y de 3 experimentos independientes en el caso de células Jurkat. 114 Las células T activadas resultaron ser resistentes a todas las dosis de los distintos HDACi, con excepción de los hidroxamatos (SAHA y especialmente TSA, figura 20A). El mismo patrón de resistencia se observó en células T no estimuladas (datos no mostrados). Por el contrario, las células Jurkat resultaron mucho más sensibles a la inducción de apoptosis por estos inhibidores (figura 20B). Las líneas T leucémicas CEM-6 y MOLT-4 mostraron una sensibilidad similar a la de células Jurkat (datos no mostrados). A partir de estos resultados seleccionamos una dosis de cada uno de los HDACi no tóxica para los linfocitos T primarios y además subletal para las líneas de células T leucémicas, de modo que se pudiese claramente diferenciar el posible efecto potenciador de la apoptosis mediada por TRAIL. Así establecimos las siguientes dosis: 10 ng/ml de TSA, 1 mM de VPA, 0.5 μM de SAHA, 1 μM de MS-275, 0.5 mM de NaB y 50 nM de apicidina. Para comprobar que las dosis elegidas de estos inhibidores efectivamente producían un efecto sobre la acetilación de histonas, estudiamos por citometría de flujo los niveles de la histona H4 acetilada. Observamos en células Jurkat un nivel máximo de acetilación a las 4 horas de tratamiento con los HDACi mientras que a las 24 horas el nivel de acetilación basal se recuperaba (figura 21). Resultados similares se obtuvieron en células CEM-6 por lo que se estableció el tiempo de 4 horas Intensidad de fluorescencia como el óptimo de pretratamiento con los HDACi. 60 Negativo Control TSA VPA 40 SAHA MS-275 30 NaB Apicidina 50 20 10 0 4 1 horas 24 2horas Figura 21. HDACi incrementan el nivel de acetilación de histonas en células Jurkat. Células Jurkat fueron tratadas con los distintos HDACi a concentraciones subletales (TSA 10 ng/ml, VPA 1mM, SAHA 0.5 μM , MS-275 1μM, NaB 0.5 mM y apicidina 50 nM) durante 4 y 24 horas. Los niveles de acetilación se analizaron por citometría de flujo como se describe en Materiales y Métodos. Los datos mostrados son representativos de tres experimentos independientes. 115 Una vez establecidas las dosis y tiempos de tratamiento con los inhibidores comenzamos a analizar el efecto sobre la apoptosis mediada por TRAIL en células Jurkat, CEM-6 y MOLT-4. Los HDACi de las distintas familias estructurales potenciaban de forma similar en las tres líneas celulares la inducción de apoptosis por TRAIL, con excepción de la apicidina que a la concentración empleada incrementaba la sensibilidad a TRAIL tan solo en células Jurkat, pero no en las líneas CEM-6 y MOLT4 (figura 22A, B, C). De forma paralela, se analizó la posible sensibilización a la apoptosis mediada por TRAIL en linfocitos T activados de donantes sanos, no encontrándose efecto alguno en ninguno de los casos analizados (figura 22D). % Células apoptóticas 100 Jurkat 80 + TRAIL 60 60 40 40 20 20 C TSA 100 SAHA MS-275 NaB Apicidina MOLT-4 - 80 VPA 60 0 + TRAIL C TSA 40 40 20 20 C TSA VPA SAHA MS-275 NaB Apicidina 0 SAHA MS-275 NaB Apicidina Linfocitos T activados + TRAIL 60 0 VPA 100 80 + TRAIL CEM-6 - 80 0 % Células apoptóticas 100 C TSA VPA SAHA MS-275 NaB Apicidina Figura 22. HDACi potencian la apoptosis mediada por TRAIL en líneas leucémicas T sensibles, pero no sensibilizan linfocitos T activados. Células Jurkat, CEM-6, MOLT-4 y linfocitos T activados fueron pretratadas durante 4 horas con los distintos HDACi a las siguientes concentraciones: TSA 10 ng/ml, VPA 1mM, SAHA 0.5 mM , MS-275 1mM, NaB 0.5 mM y apicidina 50 nM y posteriormente se incubaron con TRAIL (100 ng/ml en líneas y 250 ng/ml en primarias) durante 20 horas. El porcentaje de células apoptóticas se determinó por análisis del pico sub-G1 mediante citometría de flujo. Las barras de error representan la SD de al menos 3 experimentos independientes. 116 Estudiamos también el efecto de HDACi a las concentraciones establecidas sobre otra línea de células T leucémicas, HPB-ALL, que se caracteriza por presentar una alta resistencia a TRAIL, a diferencia de las tres líneas anteriores que se consideran sensibles a este ligando. En este caso, a pesar de utilizar una dosis más alta de TRAIL, no encontramos ningún efecto de sensibilización (figura 23A). Tampoco observamos sensibilización a la apoptosis mediada por TRAIL en linfocitos primarios de sangre periférica de pacientes con linfoma T cutáneo, leucemia T aguda, leucemia T precursora y leucemia T crónica (figura 23B-E). Estos resultados sugieren que los HDACi son capaces de potenciar o facilitar la inducción de apoptosis en células T leucémcias sensibles a TRAIL pero no en aquellas que son resistentes a este ligando. % Células apoptóticas % Células apoptóticas 100 80 - 60 + TRAIL - 60 + TRAIL 40 20 20 0 100 80 C TSA VPA 0 SAHA MS-275 NaB Apicidina 60 60 40 40 20 20 C TSA VPA NaB Apicidina HPB-ALL - 80 MS-275 + TRAIL 60 40 20 0 C TSA VPA SAHA TSA MS-275 NaB Apicidina 0 VPA SAHA MS-275 NaB Apicidina Leucemia T Crónica - 80 + TRAIL 0 C 100 Leucemia T Precursora - Leucemia T Aguda 80 40 100 % Células apoptóticas 100 Linfoma T Cutáneo + TRAIL C TSA VPA SAHA MS-275 NaB Apicidina Figura 23. HDACi no facilitan la apoptosis mediada por TRAIL en distintos modelos de leucemias T resistentes. La línea celular HPB-ALL y las células primarias de pacientes con linfoma T cutáneo, leucemia T aguda, leucemia T precursora y leucemia T crónica fueron pretratadas durante 4 horas con los distintos HDACi (TSA 10 ng/ml, VPA 1mM, SAHA 0.5 μM , MS-275 1μM, NaB 0.5 mM y apicidina 50 nM) y posteriormente se incubaron con TRAIL (250 ng/ml) durante 24 horas. El porcentaje de células apoptóticas se determinó por análisis del pico sub-G1 mediante citometría de flujo. 117 3.4 HDACi potencian la activación de caspasas y las señales mitocondriales mediadas por TRAIL en líneas leucémicas T Una vez demostrada la potenciación del proceso apoptótico mediante la combinación de los HDACi y TRAIL en células Jurkat, CEM-6 y MOLT-4, analizamos a qué nivel de la ruta de señalización de TRAIL tiene lugar dicha potenciación. En primer lugar determinamos la activación de caspasas-8, -9 y –3 en respuesta a TRAIL en células Jurkat preincubadas o no con los diferentes HDACi. Como se demuestra en la figura 24, todos los inhibidores, en mayor o menor medida, aumentaron la activación de todas las caspasas. Resultados similares se obtuvieron en células CEM y MOLT-4, excepto con la apicidina (datos no mostrados). HDACi TRAIL TSA VPA SAHA MS _____ _____ _____ _____ - + - + - + - + - + NaB Apicidina ______ ______ - + - + - + Caspasa-8 43/41 kDa Caspasa-9 37-35 kDa Caspasa-3 20/17 kDa Figura 24. HDACi potencian la activación de caspasas -8, -9 y -3 en respuesta a TRAIL en células Jurkat. Células Jurkat fueron pretratadas durante 4 horas con los distintos HDACi a las concentraciones subletales anteriormente descritas y a continuación fueron tratadas con TRAIL (100 ng/ml) durante 20 horas. La fragmentación de caspasas se determinó por Western Blot. Para determinar si el efecto fundamental de los HDACi sobre la ruta de TRAIL tiene lugar en las etapas iniciales en las que se activa la caspasa-8 o en una fase posterior, a nivel de las señales mitocondriales y la activación de caspasa-9, se comparó el efecto de inhibidores específicos de ambas caspasas sobre la inducción de apoptosis por HDACi 118 y TRAIL con el del inhibidor general de caspasas Z-VAD. Encontramos que tanto el inhibidor de caspasa-8, Z-IETD, como el de caspasa-9, Z-LEHD, producían un bloqueo similar al del Z-VAD de la apoptosis mediada por TRAIL en células Jurkat pretratadas con HDACi (figura 25). Estos datos sugieren que ambas caspasas son necesarias para la potenciación de la apoptosis por HDACi. % Células apoptóticas 100 80 60 HDACi - HDACi+TRAIL TRAIL ZVAD50uM +HDACi Z-VAD ZVAD50uM +HDACi+T Z-VAD + TRAIL Z-IETD inhCASP8-50uM +HDACi Z-IETD + TRAIL inhCASP8-50uM +HDACi+T Ac-LEHD inhCASP9-50uM +HDACi Ac-LEHD + TRAIL inhCASP9-50uM +HDACi+T 40 20 0 Control TSA VPA SAHA MS-275 NaB Apicidina Figura 25. La inhibición de caspasas bloquea la apoptosis inducida por el tratamiento combinado de HDACi y TRAIL. Células Jurkat se pretrataron durante 4 horas con las concentraciones subletales de los distintos HDACi en presencia o ausencia de los inhibidores de caspasas Z-VAD, Z-IETD o ZLEHD (20 μM). Posteriormente se trataron con TRAIL (100 ng/ml) durante 20 horas y el porcentaje de células apoptóticas se determinó por análisis del pico sub-G1 mediante citometría de flujo. Analizamos entonces el efecto de los inhibidores de caspasas en la activación de las mismas en células Jurkat preincubadas con un HDACi, en este caso MS-275, y tratadas con TRAIL (figura 26). Observamos, como era de esperar, que el inhibidor de la caspasa-8 no solo bloqueaba la activación de esta caspasa sino también la de la –9 y la 3 y la degradación de un sustrato de esta última, PARP. Pero también el inhibidor de caspasa-9 evitaba la activación de caspasa-8, de caspasa-3 y la degradación de PARP tanto en células tratadas con TRAIL como en los tratamientos combinados de HDACi y TRAIL. Los mismos resultados se obtuvieron con otros inhibidores de HDACi. Estos datos parecen indicar que el incremento en la activación de la caspasa-8 podría deberse en gran parte a una potenciación en la activación de la caspasa-9 que, vía caspasa-3 por 119 retroalimentación, activaría a la caspasa-8. La dosis de inhibidores de caspasas utilizada en estos experimentos (20 μM) es la mínima concentración descrita con capacidad para inhibir de forma específica la activación de caspasas. A pesar de ello, no podemos desechar la posibilidad de una acción inespecífica generalizada de los inhibidores empleados. MS-275 TRAIL Z-IETD-FMK Ac-LEHD-CMK - + - + - + + - + + - + + + - + + + + + Caspasa 8 43/41 kDa β-Actina Caspasa 9 37-35 kDa PARP 85 kDa β-Actina Caspasa 3 20/17 kDa β-Actina Figura 26. Efecto de los inhibidores de caspasas -8 y -9 en la activación de caspasas en respuesta al tratamiento combinado con HDACi y TRAIL. Células Jurkat fueron pretratadas durante 4 horas con MS-275 (1 μM) en presencia o ausencia de Z-IETD o Z-LEHD (20 μM) y posteriormente se trataron con TRAIL (100 ng/ml) durante 20 horas. La fragmentación de caspasas se determinó por Western Blot. Estudiamos también la posible potenciación por HDACi de señales mitocondriales descritas como necesarias para la inducción de apoptosis por TRAIL en la gran mayoría de las células tumorales. En concreto, analizamos la translocación de Bax a la mitocondria así como la salida de citocromo c al citosol, eventos que tienen lugar como consecuencia de la activación de la caspasa-8 apical. En la figura 27 podemos observar 120 que el pretratamiento de células Jurkat con VPA (como modelo de HDACi) potenciaba la translocación de Bax y especialmente la liberación de citocromo c en respuesta a TRAIL. Estos datos confirman la acción de los inhibidores de HDAC sobre las señales iniciales de la ruta de inducción de apoptosis por este ligando. VPA - - + + - - + + TRAIL - + - + - + - + Bax Bax Citocromo C Citocromo c Bcl-2 β-Actina PELLET CITOSOL Figura 27. VPA potencia la salida de citocromo c y el reclutamiento de Bax a la mitocondria en respuesta a TRAIL. Células Jurkat fueron pretratadas con VPA 1mM durante 4 horas y a continuación se incubaron con TRAIL 100 ng/ml durante 20 horas. Se prepararon extractos citosólicos y membranosos (pellet) y se detectaron las proteínas citocromo c y Bax por Western Blot. La expresión de β-actina y Bcl-2 se analizó como control de carga de los extractos citosólicos y de membrana, respectivamente. 3.5 Regulación por HDACi de proteínas involucradas en la ruta de señalización de TRAIL en células T leucémicas Los estudios preliminares con VPA habían demostrado la regulación de la expresión del receptor TRAIL-R2 en líneas de células T leucémicas, especialmente en células Jurkat y CEM-6 (figura 19). Analizamos si este fenómeno se reproducía en respuesta al tratamiento con todos los HDACi a las concentraciones previamente seleccionadas como subletales que potencian la apoptosis mediada por TRAIL. En la figura 28A se observa que, con excepción del TSA, efectivamente todos ellos inducían un cierto incremento en la expresión de este receptor pro-apoptótico en células Jurkat, siendo dicho incremento de menor magnitud en el caso de la apicidina. En células CEM-6 también se observó la regulación de la expresión de TRAIL-R2, aunque de manera más débil que en Jurkat, con todos los HDACi excepto con TSA y apicidina (figura 28B). Estudiamos además si estos inhibidores afectaban a la expresión del receptor TRAIL-R1, pero en este caso no encontramos ningún tipo de regulación consistente de manera que la expresión de este receptor en la membrana de células 121 Jurkat y CEM-6 es prácticamente nula en todos los casos figura, tanto en células control como tratadas (28C, D). A TSA MS-275 BIntensidad de Fluorescencia TSA MS-275 Intensidad de Fluorescencia C TSA MS-275 Intensidad de Fluorescencia 122 VPA NaB VPA NaB VPA NaB SAHA Apicidina SAHA Apicidina SAHA Apicidina D TSA MS-275 VPA NaB SAHA Apicidina Intensidad de Fluorescencia Figura 28. HDACi incrementan la expresión de TRAIL-R2 pero no de TRAIL-R1 en células T leucémicas. Las líneas leucémicas Jurkat (A y C) y CEM-6 (B y D) fueron tratadas durante 20 horas con las concentraciones subletales de los distintos HDACi. La expresión de los receptores TRAIL-R2 (A, B) y –R1 (C, D) se determinó por citometría de flujo. Los picos sombreados corresponden a la expresión del receptor en las células sin tratar mientras que los no sombreados corresponden a la expresión en células tratadas. La fluorescencia basal del anticuerpo secundario se representa con la línea discontinua. Los resultados son representativos de dos experimentos independientes. Al igual que ocurre con el receptor TRAIL-R2, la literatura describe el incremento de TRAIL tras el tratamiento con HDACi en otros modelos tumorales (Insinga et al., 2005; Nebbioso et al., 2005), siendo ésta una de las posibles causas del efecto citotóxico de los inhibidores por sí mismos. Las concentraciones subletales de HDACi utilizadas para potenciar la inducción de apoptosis por TRAIL en nuestro modelo celular son similares a las usadas por dichos autores. Analizamos por tanto si a dichas concentraciones tenía lugar algún tipo de regulación en la expresión de este ligando de muerte, obteniendo un resultado negativo con todos los HDACi (figura 29A). Sin embargo encontramos que, concentraciones más elevadas de algunos de estos inhibidores, que sí resultaban tóxicas para nuestras células (figura 20B), eran capaces de inducir la expresión de TRAIL (figura 29B). En concreto observamos una clara regulación de la expresión de este ligando en respuesta al tratamiento con TSA, VPA y NaB, que podría explicar en parte la inducción de apoptosis por los mismos a dichas concentraciones. 123 A TSA NaB VPA MS-275 Apicidina B TSA NaB VPA MS-275 Apicidina Intensidad de Fluorescencia Figura 29. Regulación de la expresión de TRAIL por HDACi en células T leucémicas. Células Jurkat fueron tratadas durante 20 horas con (A) concentraciones subletales de los distintos HDACi o (B) concentraciones elevadas de los mismos (TSA 50ng/ml, VPA 2.5mM, MS-275 2.5μM, NaB 5mM y apicidina 0.25 μM) y la expresión de TRAIL se determinó por citometría de flujo. El pico discontinuo corresponde a la fluorescencia basal del anticuerpo secundario, el pico sombreado refleja la expresión de TRAIL en las células sin tratar mientras que le pico no sombreado corresponde a la expresión del ligando tras el tratamiento con los distintos HDACi. Los resultados son representativos de dos experimentos independientes y similares a los obtenidos en la línea CEM-6. Siguiendo la misma línea de trabajo basada en la comparación de la actividad de los distintos HDACi tratamos de caracterizar el efecto de los mismos, incluyendo el VPA ya estudiado previamente, sobre proteínas intracelulares pertenecientes a la vía de señalización de TRAIL. Los efectos del NaB en algunos casos no fueron analizados por 124 ser un inhibidor de la misma familia estructural que el VPA, ácidos alifáticos, y haber demostrado las mismas acciones que éste en todas las determinaciones hasta aquí realizadas. Esto no ocurre sin embargo con TSA y SAHA, pertenecientes ambos a la familia de los ácidos hidroxámicos, por lo que continuamos el análisis de los dos inhibidores. Estudiamos los niveles de expresión del ARNm de FLIP, Bcl-XL, Mcl-1, cIAP1 y c-IAP2 en células Jurkat y en CEM-6 en respuesta al tratamiento con los diferentes inhibidores de HDAC a las concentraciones subletales. Tanto en células CEM-6 como en Jurkat, de las que se muestran los resultados obtenidos como representación de las líneas leucémicas, no encontramos diferencias entre células tratadas y controles en los niveles de ARNm de ninguno de los factores analizados, con excepción de FLIPs cuyos niveles se reducían especialmente tras el tratamiento con VPA, SAHA y MS-275, y ligeramente en respuesta al TSA (figura 30A). Estos datos coinciden con los ya obtenidos al analizar los efectos del VPA de manera individual (figura 18). Comprobamos los posibles cambios a nivel de proteína de FLIP y de otros factores como Bcl-2 y XIAP en células Jurkat. En el caso de estos dos últimos no encontramos regulación en respuesta a ninguno de los HDACi (figura 30B). Sin embargo, sí observamos una reducción más o menos pronunciada en los niveles de FLIPL, lo que sugiere una posible regulación postransduccional de esta proteína No pudimos analizar la expresión de FLIPs a nivel de proteína, posiblemente por no alcanzar dicha expresión los umbrales de detección del método y/o el anticuerpo empleado. Analizamos en paralelo los niveles de estas proteínas en linfocitos T activados de donantes sanos, como ya hicimos previamente con los de ARNm en respuesta a VPA (figura 18), para establecer una relación entre las posibles variaciones de las mismas y el efecto potenciador de la apoptosis mediada por TRAIL. Como era de esperar, no observamos ningún cambio en los niveles de las proteínas estudiadas, ni siquiera en los de FLIPL (figura 30B). Tanto en linfocitos T activados como en células Jurkat comprobamos que los tratamientos con HDACi habían resultado efectivos mediante determinación de la acetilación de la histona H4 a las 4 horas de tratamiento (figura 30B). Resultados similares a los de células Jurkat, incluyendo la regulación postransduccional de FLIPL, se obtuvieron en células CEM-6 y MOLT-4 (figura 30C y datos no mostrados). 125 A FLIPL FLIPs β-Actina Bcl-x Mcl-1 c-IAP1 c-IAP2 β-Actina B Jurkat C TSA VPA SAHA Linfocitos T activados MS C TSA VPA SAHA MS H4-Ac FLIPL XIAP Bcl-2 β-Actina MOLT-4 CEM-6 C C TSA VPA SAHA MS C TSA VPA SAHA MS FLIPL β-Actina Figura 30. Expresión de factores antiapoptóticos en líneas leucémicas T en respuesta al tratamiento con HDACi. A) Células Jurkat fueron tratadas con concentraciones subletales de los diferentes HDACi durante 15 horas. Los niveles de ARNm se determinaron mediante RT-PCR. B) Células Jurkat y linfocitos T activados se incubaron durante 4 horas (para detectar acetilación de histona H4) o 24 horas (para analizar el resto de proteínas) con concentraciones subletales de diferentes HDACi. C) Células CEM-6 y MOLT-4 se incubaron 24 horas como en (B). Los niveles de las proteínas indicadas en B y C se analizaron mediante Western-blot. En todos los casos se muestra la expresión de β-actina como control de carga. Los resultados son representativos de tres experimentos independientes. 126 Dado que no podíamos observar los niveles de FLIPs a nivel de proteína, confirmamos la regulación de la expresión de este factor anti-apoptótico a nivel de ARNm mediante PCR a tiempo real. Como se observa en la figura 31 encontramos una reducción significativa en la expresión de este gen en células Jurkat en respuesta al tratamiento con VPA, SAHA, MS-275 y NaB (datos no mostrados) pero no con TSA y apicidina. Analizamos también por esta técnica los niveles de ARNm de FLIPL pero en este caso, aunque se apreciaban ciertas variaciones, no eran significativas lo que corroboraba los resultados previamente obtenidos mediante PCR no cuantitativa. FLIPs Incremento expresión (tanto por 1) FLIPL 4 2 1,74 3 0,87 1,41 0,87 1 2 0,38 0,41 0,81 0,71 1,15 0,29 1 0 C TSA VPA SAHA MS Apicidina 0 C TSA VPA SAHA MS Apicidina Figura 31. El tratamiento con determinados HDACi reduce la expresión del ARNm de FLIPs en células T leucémicas. Células Jurkat fueron tratadas con concentraciones subletales de los diferentes HDACi durante 15 horas. Los niveles de ARNm de FLIPs y FLIPL se determinaron mediante PCR a tiempo real, llevando a cabo los experimentos por triplicado y normalizando los resultados con respecto a la expresión del ARNr 18S, para cada una de las muestras. Las barras de error representan la SD del triplicado. En los estudiados realizados sobre la activación de caspasas, especialmente en células Jurkat y CEM-6, en ocasiones encontrábamos un cierto incremento en la expresión de caspasa-3 y de caspasa-9 en respuesta al tratamiento con algunos de los inhibidores de HDAC. Otros autores han descrito la regulación de caspasas por HDACi en diferentes modelos tumorales. Analizamos más en detalle esta posible regulación de la expresión de caspasa-3 y -9 en células Jurkat, ya que también podría contribuir al efecto potenciador de la inducción de apoptosis por TRAIL. Los resultados de la figura 127 32 indican que, efectivamente, a nivel de proteína los HDACi parecen regular la expresión de ambas caspasas. Sin embargo dicha regulación no es significativa cuando se analizan los niveles de ARNm mediante PCR a tiempo real, con excepción de la inducción de caspasa-3 por VPA. Es posible que en este caso esté ocurriendo también algún tipo de regulación postransduccional, como con FLIPL. A C TSA VPA SAHA MS Caspasa-3 Caspasa-9 β-Actina Incremento expresión (tanto por 1) B Caspasa-3 8 Caspasa-9 4 1,74 4,29 1,41 3 6 4 0,93 2 2,14 0,71 0,71 1,52 1,32 2 1 0,71 0 0 C TSA VPA SAHA MS Apicidina C TSA VPA SAHA MS Apicidina Figura 32. Regulación de la expresión de caspasas por HDACi en células Jurkat. Células Jurkat fueron tratadas con dosis subletales de HDACi durante 24 (A) o 15 (B) horas. En (A) los niveles de caspasa-3 y -9 se analizaron a nivel de proteína por Western Blot determinándose la expresión de βactina como control de carga. En (B) se analizaron los niveles de ARNm de ambas caspasas por PCR a tiempo real, llevando a cabo los experimentos por triplicado y normalizando los resultados con respecto a la expresión del ARNr 18S, para cada una de las muestras. Las barras de error representan la SD del triplicado. Los resultados mostrados son representativos de al menos dos experimentos independientes. En resumen, todos estos datos sugieren que, al menos en el caso del VPA, SAHA, NaB y MS-275, los HDACi pueden potenciar la apoptosis mediada por TRAIL en células T leucémicas desde sus estadios iniciales, de forma que el incremento previamente observado en la activación de la caspasa -8 se podría correlacionar con el 128 aumento en la expresión del receptor TRAIL-R2 y la pérdida de FLIP. Pero no podemos descartar la posible regulación de otros factores anti- o pro-apoptóticos, como las caspasas, que contribuyan a potenciar las señales mitocondriales y la activación de caspasa-9. 129 130 V. DISCUSIÓN 131 132 1. REGULACIÓN DEL PROMOTOR DE TRAIL La importancia de TRAIL como agente terapéutico frente al cáncer radica en su conocida implicación inmunológica frente al desarrollo de tumores, además de en su acción selectiva sobre células tumorales y no sobre células normales. La expresión de TRAIL en superficie se incrementa en células T y en otras células del sistema inmune en respuesta a señales de activación, mediando en gran parte la actividad citotóxica de estas células (Kemp et al., 2005; Takeda et al., 2002). La descripción de los elementos reguladores de la expresión de TRAIL es fundamental para poder controlar su expresión en dichas células, de manera que TRAIL puede ser una herramienta terapéutica muy útil en el tratamiento del cáncer no solo administrándolo de manera exógena sino también potenciando su expresión endógena en células con capacidad citotóxica. Los trabajos iniciales sobre el promotor de TRAIL describieron la presencia de numerosos sitios de unión a factores de trascripción, entre ellos NFAT, y demostraron su regulación por interferón (Gong and Almasan, 2000; Wang et al., 2000a). En células T se ha descrito la implicación de NF-κB en la regulación de la expresión de TRAIL tras el tratamiento con agentes que imitan la estimulación del TCR - PMA/ionomicina, PHA, anticuerpos frente a CD3 y CD28 – y se ha caracterizado un sitio de unión a este factor de transcripción en la región -265 a -256 del promotor de TRAIL (Baetu et al., 2001). Sin embargo, el mayor incremento en la expresión de TRAIL en respuesta a tratamientos combinados con PMA/ionomicina que en tratamientos sólo con PMA parece indicar una cooperación de NF-κB con NFAT en la regulación del promotor. Nuestro trabajo ha tratado de establecer el posible papel de NFAT en la regulación de TRAIL en respuesta a la activación de la célula T, al igual que se ha descrito su implicación en la regulación de la expresión de otros ligandos de muerte como son TNF-α y CD95L (McCaffrey et al., 1994; Rengarajan et al., 2000). En nuestro modelo de células Jurkat hemos demostrado que efectivamente el tratamiento con PMA solo no es suficiente para inducir la expresión del promotor de TRAIL, como tampoco lo es el tratamiento solo con ionomicina, lo que confirma la importancia tanto de NF-κB como de NFAT en la regulación de TRAIL en células T en respuesta a la activación. Por otro lado, el descenso observado en los niveles de expresión de TRAIL en células T activadas en respuesta al tratamiento con el inhibidor de NFAT, ciclosporina A, también confirma la implicación de este factor de transcripción en la regulación del ligando de 133 muerte y coincide con los resultados obtenidos previamente en células T de ratón, en las que se observó una reducción en los niveles de TRAIL en membrana tras el tratamiento con dicho inhibidor (Mariani and Krammer, 1998b). Además, por primera vez, nuestros resultados demuestran que el tratamiento con ciclosporina A determina no sólo el descenso en los niveles de ARNm de TRAIL en células T activadas, sino también el descenso en la acción citotóxica de las mismas sobre células tumorales sensibles a TRAIL, indicando la importancia de NFAT en la regulación de la expresión del ligando de muerte con capacidad para funcionar como tal en la membrana de la célula T. Es importante mencionar que las células Hela, utilizadas como modelo de línea tumoral en estos experimentos, son resistentes a CD95L (Ref. Muñoz-Pinedo, JBC, 2003) por lo que podemos descartar que el efecto citotóxico regulable por ciclosporina observado sea debido a la expresión de este ligando en los linfocitos T activados. En resultados preliminares no mostrados hemos encontrado que dicho efecto citotóxico es similar en linfocitos T CD4+ y T CD8+ activados purificados, e incluso ligeramente superior en los primeros, lo que está de acuerdo con datos previamente publicados describiendo la actividad antitumoral de células T CD4+ citotóxicas mediada por TRAIL (Dorothee et al., 2002). Los resultados obtenidos tras el estudio de la actividad de los distintos fragmentos del promotor de TRAIL demuestran que el tratamiento simultáneo con PMA/ionomicina determina la activación del promotor en una magnitud similar utilizando los fragmentos de 165, 787 y 1295 pb, de lo que se deduce la importancia de la región de 165 pb más próxima al sitio de inicio de transcripción en su regulación, aunque esta región no contiene el sitio para NF-κB descrito por Baetu y colaboradores. En este sentido, nuestros resultados coinciden los de un trabajo reciente sobre la inhibición de NF-κB mediante el tratamiento con prostaglandinas y su implicación en la expresión de TRAIL en células T (Fionda et al., 2007), donde observan una inducción con PMA/ionomicina similar en fragmentos de 300 y 165 pb del promotor. De hecho, se ha descrito recientemente un sitio de unión para NF-κB entre los nucleótidos -75 a -65 que no había sido señalado previamente debido a que este sitio se solapa con el sitio SP1 encontrado en el análisis inicial de la secuencia del promotor (Matsuda et al., 2005). La unión de NF-kB a este sitio podría explicar la estimulación por PMA/ionomicina en nuestro fragmento de 165 pb del promotor de TRAIL. Por otro lado, podemos decir que el sitio de unión a NFAT también se encuentra en esta región. 134 Según el estudio realizado con programas informáticos, en este fragmento de 165 pb existen 3 secuencias consenso para NFAT. Sería interesante caracterizar en un futuro cual o cuales de ellos son los responsables de la regulación de la expresión de TRAIL por este factor de transcripción. 2. REGULACIÓN DE LA RESISTENCIA DE LINFOCITOS T PRIMARIOS A TRAIL La ausencia de toxicidad de TRAIL para la mayoría de células no transformadas es el hecho más importante a tener en cuenta a la hora de considerarlo como un agente antitumoral, si bien el mecanismo implicado en esta resistencia no está completamente caracterizado. De ahí que en el presente trabajo hayamos tratado de analizar la respuesta de linfocitos T en reposo y activados a TRAIL. Hemos encontrado que la ruta de señalización de apoptosis se ve inhibida en el nivel más apical, esto es, no llega a activarse la caspasa-8 en células T en ninguno de los dos estadios analizados: reposo y activación. El análisis de la expresión de las proteínas más importantes que participan en la formación del DISC indica que: - La activación de células T no regula la expresión de los niveles de FADD, caspasa-8, c-FLIP ni de los receptores de TRAIL, con la excepción de TRAIL-R4. - Comparando con la línea de células T leucémicas sensibles a TRAIL, Jurkat, la expresión de FADD y caspasa-8 en células T normales es similar mientras que los niveles de c-FLIP son mucho mayores que en la línea celular. Recientemente se ha descrito esta proteína inhibidora de la apoptosis como una de las claves de la resistencia de células NK a la muerte por TRAIL (Mirandola et al., 2004). Es posible que en linfocitos T juegue un papel similar. - Los niveles de TRAIL-R1, -R2 y -R3 en membrana son muy bajos, mientras que en el caso del receptor anti-apoptótico TRAIL-R4 podemos distinguir dos picos con baja y alta intensidad de fluorescencia, respectivamente. Este patrón de expresión de receptores pro- y anti-apoptóticos podría estar íntimamente ligado con la ausencia de activación de caspasa-8 tras tratamiento con TRAIL y, por tanto, con la resistencia de linfocitos T a la muerte mediada por TRAIL. 135 En relación con la expresión de los receptores de TRAIL, encontramos ciertas discrepancias con los trabajos publicados por otros autores. Mientras que estudios previos aseguran que células T en reposo no presentan una significativa expresión de receptores con excepción de TRAIL-R4 en la subpoblación de células CD8+ (Mirandola et al., 2004), nuestros datos obtenidos mediante análisis citofluorimétrico revelan la existencia de un pico de baja intensidad de fluorescencia de TRAIL-R4, que se presenta con una cierta variabilidad entre células T de distintos donantes y que debe corresponderse con la subpoblación de linfocitos T CD4+. Por otra parte, dichos trabajos afirman que la activación de la subpoblación CD8+ incrementa la expresión de los receptores TRAIL-R1 y -R2 (Hasegawa et al., 2004) o de TRAIL-R2, -R3 y -R4 (Mirandola et al., 2004), mientras que en la subpoblación CD4+ la activación no provoca cambio alguno en la expresión de receptores (Hasegawa et al., 2004). En contraste con los estudios de Mirandola, realizados con poblaciones purificadas CD4+ y CD8+, nosotros trabajamos con la población global de células T CD3+, es decir, en condiciones similares a las fisiológicas donde la coexistencia de ambas subpoblaciones durante el cultivo y la activación celular puede influir en su comportamiento y por tanto en el patrón de expresión de receptores de las mismas. Las discrepancias encontradas pueden deberse también a la forma de activación empleada. La inhibición de NF-κB se ha propuesto recientemente como un mecanismo de sensibilización de algunos tumores resistentes a TRAIL ya que este factor de transcripción se encuentra activado de forma constitutiva modulando la expresión de ciertas proteínas antiapoptóticas en algunos modelos tumorales (Huerta-Yepez et al., 2004). En este trabajo se muestra como los inhibidores de NF-κB pueden igualmente sensibilizar a los linfocitos T activados a la muerte por TRAIL. Este efecto se ha observado especialmente con los inhibidores BAY 11-7085 y sulfosalazina. En el caso de partenolide el efecto es demasiado bajo para ser significativo, a pesar de que inhibe claramente la translocación de las subunidades p50 y p65 de NF-κB al núcleo, pudiendo este hecho deberse a que este compuesto presenta otros efectos colaterales como es la activación de la vía de JNK y la inhibición de STAT3 (Nakshatri et al., 2004; Sobota et al., 2000) que pueden interferir con las señales derivadas de la inhibición de NF-κB. Si bien es cierto que la expresión de los receptores pro-apoptóticos TRAIL-R1 y -R2 en membrana es demasiado baja en células T activadas como para ser detectada mediante análisis citofluorimétrico, y que el tratamiento con BAY 11-7085 induce una 136 reducción a nivel de ARNm de dichos receptores, hemos encontrado una clara activación de caspasa-8 en respuesta a TRAIL en células pretratadas con el inhibidor de NF-κB. Por tanto, los niveles de ambos receptores o de cualquiera de ellos en la membrana de linfocitos T activados deben ser suficientes para transmitir la señal de muerte tras la interacción con su ligando TRAIL. Además de la regulación de estos dos receptores hemos demostrado un descenso claro en la expresión de TRAIL-R4, tanto a nivel de ARNm como de proteína en membrana. Esta reducción debería tener más relevancia en el caso de linfocitos T CD4+ ya que supone que el porcentaje de células que expresan dicho receptor tras el pretramiento con BAY 11-7085 es muy bajo, mientras que el 100% de células T CD8+ siguen siendo positivas para el mismo. A pesar de ello, los resultados obtenidos con poblaciones de células T CD4+ y CD8+ aisladas demuestran que la sensibilización a TRAIL por el inhibidor de NF-κB es similar en ambas subpoblaciones. Estos resultados pueden explicarse si tenemos en cuenta el papel regulador propuesto recientemente para TRAIL-R4 (Clancy et al., 2005). Estos autores demuestran que TRAIL-R4 puede asociarse con TRAIL-R2 formando un complejo inhibidor de muerte, de manera independiente de la presencia del ligando, y que dicho complejo preformado parece controlar la sensibilidad de linfocitos T CD8+ a la apoptosis mediada por TRAIL. De este modo, la reducción de los niveles de expresión de TRAIL-R4 por inhibición de NF-κB en la población CD8+ puede contribuir de manera significativa en su sensibilización a TRAIL. En cualquier caso, no podemos descartar la existencia de otros mecanismos implicados en dicha sensibilización. Además de los niveles de TRAIL-R4, el tratamiento con BAY 11-7085 disminuye la expresión de proteínas anti-apoptóticas previamente descritas como regulables por NF-κB, tales como FLIPs, XIAP y c-IAP2. Aunque se desconoce la contribución de cada uno de estos factores en la resistencia a la muerte mediada por TRAIL de los linfocitos T activados, en conjunto, la regulación todos ellos puede explicar la sensibilización a TRAIL de estas células en respuesta a la inhibición de NFκB. En el caso de Bcl-2, aunque se produce una reducción temprana en los niveles de ARNm, nuestros datos sugieren que este factor anti-apoptótico no debe estar implicado en el efecto del inhibidor de NF-κB ya que en los tiempos en que se detecta sensibilización a TRAIL (24 horas) todavía no se observan cambios en los niveles de proteína. 137 En relación con los linfocitos T en reposo, el comportamiento observado es distinto al de las células T activadas de modo que la respuesta de estas células al tratamiento con el inhibidor de NF-κB varía entre diferentes donantes. En estado de reposo, el nivel de activación de este factor de transcripción es muy bajo, incrementándose posteriormente en respuesta a señales activadoras. A este hecho debe unirse la presencia de complejos homodiméricos de p50 en células en reposo que actúan como represores de la transcripción, en contraposición con los heterodímeros de p50/p65 que se localizan en células T activadas y que son activadores de la transcripción (Algarte et al., 1995). Estas características del factor de transcripción NF-κB en células T en reposo, junto con las propias particularidades de cada donante, pueden influir sobre los efectos del inhibidor BAY 11-7085 en dichas células. Además, debemos tener en cuenta la capacidad de TRAIL para activar NF-κB cuando se une a los receptores pro-apoptóticos y a TRAILR4 en determinados tipos celulares (Franco et al., 2001). Es posible que esto pueda estar ocurriendo en las células T en reposo, lo que explicaría que TRAIL pueda contrarrestar los efectos citotóxicos del propio inhibidor de NF-κB en estas células. Por otro lado está descrito que, en general, los linfocitos T en reposo presentan una mayor resistencia a la muerte por apoptosis que los linfocitos T activados (Wesselborg et al., 1993), por lo que deben existir mecanismos diferentes en ambos estadios de activación que regulan la inducción de apoptosis y que por tanto pueden afectar al umbral de sensibilización a TRAIL. Diferentes autores han descrito diversos cambios en la expresión de factores pro- y anti-apoptóticos como posible causa de las diferencias en la susceptibilidad a apoptosis entre células T en reposo y activadas (Bosque et al., 2005; Broome et al., 1995; Schmitz et al., 2003). Nuestro análisis comparativo entre ambos tipos de células muestra una menor expresión de Bax en linfocitos T en reposo que podría explicar su mayor resistencia a la muerte por apoptosis. Sin embargo, la expresión del receptor señuelo TRAIL-R4 también es menor en estas células y los niveles de la proteína Bid son significativamente mayores que en células T activadas, lo que en principio estaría relacionado con una mayor sensibilidad a TRAIL. Sería interesante realizar un análisis más exhaustivo de los mecanismos de resistencia a TRAIL y la posible activación de NF-κB en respuesta a este ligando en células T en reposo. En relación con los bajos niveles de Bid encontrados en linfocitos T activados, pensamos que deben ser suficientes para permitir la sensibilización a TRAIL dado que no se incrementan en respuesta al tratamiento con BAY 11-7085. 138 La activación de NF-κB es esencial en numerosos procesos fisiológicos y juega un papel crucial en el desarrollo de la respuesta inmunitaria, por ejemplo en la proliferación de linfocitos y en la producción de citoquinas, lo que sugiere que el uso de inhibidores de NF-κB puede dar lugar a una gran variedad de efectos colaterales. Nuestros resultados demuestran que dichos inhibidores no solo sensibilizan a linfocitos T activados a la apoptosis mediada por TRAIL sino que también presentan una toxicidad variable para las células T, tanto en reposo como activadas. Recientemente se ha descrito la implicación de NF-κB en la resistencia de células uroteliales normales humanas a TRAIL (Steele et al., 2006). En conjunto, estos datos sugieren que NF-κB puede jugar algún papel en la resistencia de algunas células primarias a TRAIL y cuestionan la posible utilización de inhibidores de NF-κB en terapia antitumoral. Aunque los inhibidores del proteasoma bloquean la activación de NF-κB, no hemos observado efectos en la resistencia de células T a la apoptosis mediada por TRAIL tras el pretratamiento con bortezomib. Esto es debido posiblemente a que dichos inhibidores presentan multitud de efectos biológicos de manera que el resultado neto de todos ellos puede no ser apropiado para inducir la sensibilización de los linfocitos T a TRAIL. Sin embargo, sí presentan una elevada toxicidad especialmente para los linfocitos T activados. De forma similar, otros moduladores de la sensibilidad de células tumorales a TRAIL, como la doxorubicina o el nocodazol, muestran una toxicidad moderada para linfocitos T activados. Estos resultados ponen en duda la posible utilización de dichos compuestos en terapias combinadas con TRAIL. Por el contrario, otros agentes como los inhibidores de histona deacetilasa (HDAC) se presentan como una prometedora estrategia en la sensibilización de células tumorales a la apoptosis mediada por TRAIL debido a su falta de toxicidad y a su incapacidad para sensibilizar a células T primarias. También los inhibidores de la vía PI3K/Akt podrían ser útiles en el tratamiento de tumores con una elevada actividad de esta ruta de supervivencia que produce un bloqueo en la señalización de apoptosis en respuesta a TRAIL (Nam et al., 2003; Nesterov et al., 2001). 139 3. POTENCIACIÓN POR INHIBIDORES DE HDAC DE LA APOPTOSIS MEDIADA POR TRAIL EN CÉLULAS T LEUCÉMICAS Como hemos indicado previamente, la importancia de TRAIL como agente terapéutico frente al cáncer radica en su falta de toxicidad para la mayoría de las células no transformadas. En linfocitos T de donantes sanos hemos observado que la ruta de señalización de apoptosis mediada por TRAIL se ve inhibida desde su etapa más apical, esto es, a nivel de la activación de la caspasa-8 iniciadora, posiblemente debido a que los niveles de la proteína inhibidora de apoptosis c-FLIP son elevados y a que la expresión de los receptores pro-apoptóticos es muy baja. En el presente trabajo demostramos que inhibidores de HDAC pertenecientes a diferentes categorías estructurales potencian la ruta de señalización de TRAIL en células T leucémicas sin que se llegue a romper la resistencia apical presentada por los linfocitos T primarios. Curiosamente la potenciación de la apoptosis por HDACi parece deberse en parte a la regulación selectiva de la expresión del receptor TRAIL-R2 y de la proteína antiapoptótica FLIP en células T leucémicas, pero no en células T normales. Entre los posibles agentes empleados en terapia antitumoral, los HDACi se han perfilado en los últimos años como una estrategia interesante en la lucha contra el cáncer en combinación con TRAIL. La mayor parte de los estudios se han centrado hasta ahora en leucemia linfática crónica B (B-CLL), donde se ha observado que la sensibilización a TRAIL por HDACi, como el TSA, está mediada por un aumento en la formación del DISC asociado al receptor TRAIL-R1 (MacFarlane et al., 2005); y en leucemia mieloide aguda (AML), donde se ha demostrado que el MS-275 incrementa la expresión de TRAIL (Nebbioso et al., 2005). Algunos de los HDACi frecuentemente utilizados, como el VPA o el MS-275 son específicos de HDAC tipo I (HDAC1, HDAC2, HDAC3 y HDAC8) mientras que los hidroxamatos (TSA y SAHA) actúan de forma no selectiva, tanto sobre las HDAC de tipo I como de tipo II (HDAC4, HDAC5, HDAC6 y HDAC7). Se ha demostrado que ciertos tumores solamente son susceptibles a la inducción de apoptosis por determinados HDACi, como es el caso de algunas leucemias linfáticas agudas B y T en las que el MS-275 no es capaz de provocar la muerte celular, mientras que sí lo hacen VPA y SAHA (Tsapis et al., 2007). También se ha descrito que la inhibición de HDAC de clase I, pero no de clase II, es fundamental para la sensibilización a TRAIL de células de B-CLL (Inoue et al., 2006a). Nuestros 140 resultados demuestran un comportamiento uniforme de distintos HDACi, con independencia del tipo de HDAC sobre el que ejercen su acción inhibidora, en el mantenimiento de la resistencia a TRAIL de linfocitos no transformados, así como en la sensibilización a TRAIL de las diferentes líneas de leucemias T sensibles a este ligando. La única excepción la constituye la apicidina, ya que en el caso de este HDACi observamos una menor potenciación de la inducción de apoptosis por TRAIL en células Jurkat, en comparación con el resto de los inhibidores, y prácticamente nula en el caso de las líneas CEM-6 y MOLT-4, a pesar de que a las concentraciones empleadas se detecta claramente el incremento en los niveles de acetilación de histonas en dichas células. En realidad la apicidina, comparada con el resto de HDACi utilizados en este trabajo, constituye una excepción en la mayoría de los aspectos analizados. Diversos estudios han descrito el incremento de los niveles de TRAIL-R2 en extractos citosólicos de diferentes tipos de células sensibles al ligando de muerte TRAIL en respuesta al tratamiento con HDACi (Inoue et al., 2004; Nakata et al., 2004). El estudio comparativo del efecto de HDACi pertenecientes a las distintas familias estructurales en líneas de células T leucémicas nos ha permitido demostrar que la mayoría de ellos inducen un incremento en los niveles del receptor TRAIL-R2 en membrana, con excepción de la apidicina y del TSA. Además de los cambios en la expresión de este receptor, se ha descrito la regulación de diversas proteínas pro- y antiapoptóticas por diferentes HDACi en diversos modelos de células tumorales (Rosato et al., 2003; Sanda et al., 2007). Tras el análisis de la expresión de numerosos genes implicados en la ruta de señalización de TRAIL, nuestros resultados han demostrado que la mayoría de los HDACi analizados, de nuevo con la excepción de TSA y apicidina, provocan un descenso en los niveles de ARNm de FLIPs en las distintas líneas leucémicas T sensibles a TRAIL. Aunque los niveles de esta proteína son bajos en las líneas leucémicas, con respecto a la expresión en linfocitos T de donantes sanos, es posible que juegue un papel importante en la sensibilización a TRAIL. Además hemos observado una pérdida en la expresión de la proteína FLIPL que parece tener lugar a nivel postransduccional, ya que no va a acompañada de una regulación de los niveles de ARNm. Por tanto, la regulación de FLIP, junto con el incremento en los niveles de TRAIL-R2, pueden explicar la mayor activación de la caspasa-8 iniciadora en los tratamientos combinados de HDACi y TRAIL. Es importante señalar que recientemente se ha publicado un trabajo donde se demuestra que la potenciación de la 141 ruta de TRAIL por HDACi no depende del incremento en la expresión de receptor TRAIL-R2 en diferentes modelos tumorales (Inoue et al., 2006b). En general, nuestros datos indican que no todos los HDACi parecen compartir un mismo mecanismo de acción ni presentar similares efectos sobre las líneas T leucémicas. La escasa o nula capacidad de la apicidina para potenciar la acción de TRAIL en las distintas líneas leucémicas T correlaciona con su baja o nula capacidad para regular los niveles tanto del receptor TRAIL-R2 como de la proteína FLIPs. Recientemente se ha descrito que la apicidina inhibe selectivamente a las HDAC2 y HDAC3 (Khan et al., 2008), lo que sugiere que ambas HDAC pueden no estar implicadas en la regulación de FLIPs y TRAIL-R2 en células T leucémicas y que solo en células Jurkat podrían jugar algún papel en la regulación de la sensibilidad a TRAIL, por un mecanismo independiente de la modulación de estos genes. El caso del TSA es más controvertido ya que, si bien hemos mostrado su incapacidad para modular la expresión del receptor TRAIL-R2 y de FLIPs a la concentración seleccionada, también es cierto que este HDACi sí potencia la inducción de apoptosis por TRAIL en las tres líneas leucémicas sensibles. Por el contrario el SAHA, perteneciente a la misma familia y con igual espectro de acción que el TSA (inhibición de HDAC tipos I y II), presenta ambos efectos de incremento del receptor y potenciación de la apoptosis. Es posible que la actividad del TSA sea menor que la del SAHA sobre determinadas HDAC cuya inhibición es necesaria para regular la expresión del receptor de TRAIL y de FLIPs. En cualquier caso, estos datos sugieren que la causa de la potenciación de la apoptosis mediada por TRAIL no debe ser la regulación de uno o dos genes solamente, hecho que además viene apoyado por el amplio espectro de la acción reguladora de los HDACi. Uno de los múltiples factores que pueden contribuir a la acción de HDACi es NF-κB. Algunos estudios han demostrado que TSA inhibe la activación de este factor de transcripción favoreciendo la entrada en apoptosis de la célula tumoral (Hu and Colburn, 2005; Yao et al., 2006) mientras que en otros se ha descrito la activación de NF-κB por distintos HDACi, como TSA, SAHA y apicidina (Ashburner et al., 2001; Domingo-Domenech et al., 2007; Kim et al., 2006). Si esta última hipótesis es correcta, la capacidad de activación de NF-κB por parte de los distintos HDACi en nuestro modelo de células T leucémicas puede ser diferente, de forma que el efecto proapoptótico de los inhibidores puede estar compensado en mayor o menor medida por la activación de la vía de supervivencia de NF-κB(Mayo et al., 2003). Además se sabe que 142 TRAIL-R2 y FLIP pueden ser regulados por NF-κB por lo que los niveles de estas proteínas en respuesta al tratamiento con HDACi se verían afectados de forma diferente dependiendo del grado de activación de este factor de transcripción. Por otra parte, aunque se ha descrito la inducción de apoptosis en linfomas T cutáneos por el tratamiento con elevadas dosis de SAHA (Duvic and Vu, 2007), a las dosis subletales utilizadas en nuestro trabajo de este y de otros HDACi no hemos encontrado sensibilización a TRAIL de células primarias procedentes de pacientes con linfoma T cutáneo o con diferentes tipos de leucemias T. La baja toxicidad de los HDACi y la ausencia de sensibilización a TRAIL en células T leucémicas primarias así como en células T normales también podrían venir determinadas por una mayor activación de NF-κB en estas células. La diferencia en el mecanismo de acción de los HDACi parece ir ligada no solo a su estructura y a su actividad sobre HDAC concretas, sino que también varía en función del modelo tumoral empleado. Así, estudios realizados por varios autores sobre el efecto del ácido hidroxámico SAHA indican que este inhibidor puede regular la expresión de genes diferentes en distintos tipos de células tumorales. Por ejemplo, en células de hepatoma induce un aumento en la expresión de Bim y de Bcl-xs, mientras que en células de cáncer de mama puede modular la expresión Bim, Bak, XIAP, survivina y Bcl-2, y en células de mieloma múltiple es capaz de inducir la expresión de otras proteínas pro-apoptóticas como Bax, Noxa y Puma (además de Bim y Bak) (Emanuele et al., 2007; Fandy et al., 2005). En nuestro modelo de células T leucémcias no hemos observado regulación de ninguna de las proteínas analizadas de la familia de Bcl-2 ni de los IAPs en respuesta al tratamiento con SAHA ni con ningún otro HDACi. En relación con esta ausencia de regulación de proteínas de la familia de Bcl-2, no debemos olvidar que las líneas tumorales analizadas necesitan activar la ruta mitocondrial a través de Bid para completar la señalización de muerte celular en respuesta a TRAIL. Un estudio reciente demuestra cómo la sobreexpresión de Bcl-2 o de Bcl-xL pueden bloquear la inducción de apoptosis por combinación de ciertos HDACi y TRAIL en células Jurkat, poniendo de manifiesto la relevancia de esta ruta mitocondrial (Shankar et al., 2005b). De hecho, según algunos trabajos, la mitocondria es la vía fundamental afectada por los HDACi en ciertos modelos tumorales como la línea de leucemia mieloide sensible a TRAIL U937, ya que la potenciación de la apoptosis no está asociada a un incremento de receptores de muerte ni a un descenso en 143 los niveles de FLIP (Rosato et al., 2003). En el trabajo de Shankar y colaboradores (Ref. Shankar) el tratamiento con HDACi a concentraciones ligeramente superiores a las de nuestro estudio provoca en células U937 el descenso de XIAP, c-IAP, Mcl-1, Bcl-2 y Bcl-xL. En nuestro caso, a pesar de no haber observado regulación de las proteínas analizadas relacionadas con la ruta mitocondrial no podemos descartar un posible efecto a nivel de esta ruta, por regulación de otros factores pro- o anti-apoptóticos distintos a los hasta ahora analizados, en la acción de los HDACi en células T leucémicas, especialmente en el caso del TSA que solo es capaz de regular los niveles de FLIPL pero no de TRAIL-R4 ni FLIPs. Ciertos estudios han demostrado también la regulación de la expresión de caspasas-3 y -9 en células tratadas con diversos inhibidores de HDAC (Shankar et al., 2005b; Wallace et al., 2006). Nuestro estudio comparativo de los niveles de ARNm de ambas caspasas por PCR a tiempo real ha demostrado que tan sólo se produce un incremento en los mismos tras el tratamiento con VPA, pero no con el resto de inhibidores, lo que vuelve a confirmar la acción diferencial sobre distintos genes de los distintos HDACi en nuestro modelo celular. Sin embargo a nivel de proteína sí hemos observado un cierto incremento en la expresión de ambas caspasas en respuesta al tratamiento con todos los HDACi analizados. Esta regulación podría jugar un papel importante en la potenciación de la apoptosis mediada por TRAIL y, sobretodo, en la inducida por TSA. La regulación a nivel de proteína, y no de ARNm, de caspasas puede explicarse debido a la capacidad de los HDAC para modificar distintas proteínas no histonas, algunas de las cuales presentan distintos efectos a nivel postraduccional. Uno de estos efectos está relacionado con la estabilidad de las proteínas. Se ha descrito que tanto la acetilación como la ubiquitinación pueden tener lugar en el mismo residuo de lisina, existiendo una regulación cruzada de ambas modificaciones (Caron et al., 2005). La competencia entre ambos procesos influye en la estabilidad de la proteína ya que las HDACs pueden disminuir la vida media mediante cambios conformacionales que provocan la exposición de los residuos de lisina para su ubiquitinación (Giandomenico et al., 2003). De esta forma los distintos HDACi, al permitir la acetilación de los residuos de lisina, pueden estar impidiendo la degradación de las caspasas-3 y -9 a nivel del proteasoma, provocando una mayor actividad de las mismas a pesar de no existir modulación a nivel del promotor. En el caso de FLIPL hemos observado una regulación contraria a la de caspasas, es decir, una pérdida de expresión a nivel de proteína, pero no 144 es de extrañar si tenemos en cuenta que las HDACs pueden tener muchos otros efectos por ejemplo en la interacción o en la localización de proteínas, cuyas consecuencias pueden ser muy variadas. Por otro lado, los resultados obtenidos con los inhibidores de caspasas parecen demostrar la importancia de la vía mitocondrial en la potenciación de la apoptosis. Dado que el inhibidor de caspasa-9 bloquea de manera muy significativa la activación de caspasa-8 en respuesta al tratamiento con HDACi y TRAIL, debemos entender que estos facilitan la activación de caspasa-9 de forma independiente de la activación de la caspasa-8 apical y que en gran medida esta última se activa como consecuencia de la activación de la caspasa-9 y de caspasas efectoras (ruta de amplificación de la señal de muerte celular) (Slee et al., 1999). Un estudio reciente muestra cómo la potenciación de la apoptosis por combinación de TSA y TRAIL en células de cáncer torácico se bloquea mediante la inhibición de la caspasa-9 (Reddy et al., 2007). Sin embargo, no debemos olvidar la posible acción inespecífica de los inhibidores de caspasas a las concentraciones utilizadas por lo que sería interesante realizar un estudio más exhaustivo de la acción de los HDACi en células sin caspasa-8. Por tanto, ensayos con ARNi de esta caspasa podrían ser muy útiles para conocer la verdadera implicación de la caspasas-8 y –9 en la potenciación de la apoptosis por dichos inhibidores. Además, el análisis de la posible potenciación de la formación del DISC podría ayudar a establecer la relevancia de dichas caspasas. Por otra parte, futuros estudios deberían incluir la regulación de la expresión y función por HDACi de otras proteínas que favorecen la activación de caspasa-9, como son Smac/DIABLO y Apaf-1, a la vista de los datos obtenidos por otros autores mediante ensayos de micro-array (Peart et al., 2005). También se ha descrito que, junto con inducción de apoptosis por regulación de la expresión de TRAIL en células leucémicas, los HDACi a concentraciones elevadas pueden provocar la muerte celular mediante producción de especies reactivas de oxígeno (ROS) (Ruefli et al., 2001). Sería interesante analizar si el tratamiento con HDACi a las concentraciones empleadas para la sensibilización de leucemias T a TRAIL provoca igualmente un incremento en la producción de ROS que favorece la acción de este ligando de muerte. 145 146 VI. CONCLUSIONES 147 148 1.- El factor de transcripción NFAT está implicado en la regulación de la expresión del ligando de muerte TRAIL funcional en células T activadas, encontrándose el sitio de unión a este factor en la región de 165 pb del promotor proximal al sitio de inicio de transcripción. 2.- Los linfocitos T en reposo y activados presentan una resistencia similar a la apoptosis mediada por TRAIL. Entre los posibles mecanismos que controlan dicha resistencia encontramos la expresión de las proteínas anti-apoptóticas TRAIL-R4 y FLIP así como la baja expresión en membrana de los receptores pro-apoptóticos TRAIL-R1 y –R2. 3.- Inhibidores de la activación del factor de transcripción NF-κB, como el compuesto BAY 11-7085, sensibilizan a los linfocitos T activados a la apoptosis mediada por TRAIL mediante la regulación de la expresión de diversos factores anti-apoptóticos implicados en la ruta de señalización de TRAIL. 4.- Inhibidores de HDAC pertenecientes a diferentes grupos estructurales, con excepción del péptido cíclico apicidina, potencian la apoptosis mediada por TRAIL en diferentes líneas leucémicas T sensibles a este ligando, pero no en células T leucémicas resistentes ni en linfocitos T primarios. 5.- Los inhibidores de HDAC potencian todas las señales implicadas en la ruta de apoptosis mediada por TRAIL en líneas de células T leucémicas: activación de caspasas-8 y –9, eventos mitocondriales y activación de caspasas efectoras como la caspasa-3. 6.- Los inhibidores de HDAC SAHA, VPA, MS-275 y butirato sódico, incrementan la expresión del receptor pro-apoptótico TRAIL-R2 y reducen la expresión de FLIP en células T leucémicas. 149 150 VII. BIBLIOGRAFÍA 151 152 Adams, J. (2004) The proteasome: a suitable antineoplastic target. Nat Rev Cancer, 4, 349-360. Algarte, M., Lecine, P., Costello, R., Plet, A., Olive, D. and Imbert, J. (1995) In vivo regulation of interleukin-2 receptor alpha gene transcription by the coordinated binding of constitutive and inducible factors in human primary T cells. Embo J, 14, 5060-5072. Alladina, S.J., Song, J.H., Davidge, S.T., Hao, C. and Easton, A.S. (2005) TRAILinduced apoptosis in human vascular endothelium is regulated by phosphatidylinositol 3-kinase/Akt through the short form of cellular FLIP and Bcl-2. J Vasc Res, 42, 337-347. Amran, D., Sanchez, Y., Fernandez, C., Ramos, A.M., de Blas, E., Breard, J., Calle, C. and Aller, P. (2007) Arsenic trioxide sensitizes promonocytic leukemia cells to TNFalpha-induced apoptosis via p38-MAPK-regulated activation of both receptor-mediated and mitochondrial pathways. Biochim Biophys Acta, 1773, 1653-1663. Anest, V., Hanson, J.L., Cogswell, P.C., Steinbrecher, K.A., Strahl, B.D. and Baldwin, A.S. (2003) A nucleosomal function for IkappaB kinase-alpha in NF-kappaBdependent gene expression. Nature, 423, 659-663. Ashburner, B.P., Westerheide, S.D. and Baldwin, A.S., Jr. (2001) The p65 (RelA) subunit of NF-kappaB interacts with the histone deacetylase (HDAC) corepressors HDAC1 and HDAC2 to negatively regulate gene expression. Mol Cell Biol, 21, 7065-7077. Ashkenazi, A., Pai, R.C., Fong, S., Leung, S., Lawrence, D.A., Marsters, S.A., Blackie, C., Chang, L., McMurtrey, A.E., Hebert, A., DeForge, L., Koumenis, I.L., Lewis, D., Harris, L., Bussiere, J., Koeppen, H., Shahrokh, Z. and Schwall, R.H. (1999) Safety and antitumor activity of recombinant soluble Apo2 ligand. J Clin Invest, 104, 155-162. Baetu, T.M., Kwon, H., Sharma, S., Grandvaux, N. and Hiscott, J. (2001) Disruption of NF-kappaB signaling reveals a novel role for NF-kappaB in the regulation of TNF-related apoptosis-inducing ligand expression. J Immunol, 167, 3164-3173. 153 Banner, D.W., D'Arcy, A., Janes, W., Gentz, R., Schoenfeld, H.J., Broger, C., Loetscher, H. and Lesslauer, W. (1993) Crystal structure of the soluble human 55 kd TNF receptor-human TNF beta complex: implications for TNF receptor activation. Cell, 73, 431-445. Basseres, D.S. and Baldwin, A.S. (2006) Nuclear factor-kappaB and inhibitor of kappaB kinase pathways in oncogenic initiation and progression. Oncogene, 25, 6817-6830. Baxter, A.G. and Cooke, A. (1995) The genetics of the NOD mouse. Diabetes Metab Rev, 11, 315-335. Baylin, S.B. and Ohm, J.E. (2006) Epigenetic gene silencing in cancer - a mechanism for early oncogenic pathway addiction? Nat Rev Cancer, 6, 107-116. Bernard, D., Quatannens, B., Vandenbunder, B. and Abbadie, C. (2001) Rel/NF-kappaB transcription factors protect against tumor necrosis factor (TNF)-related apoptosis-inducing ligand (TRAIL)-induced apoptosis by up-regulating the TRAIL decoy receptor DcR1. J Biol Chem, 276, 27322-27328. Bertin, J., Armstrong, R.C., Ottilie, S., Martin, D.A., Wang, Y., Banks, S., Wang, G.H., Senkevich, T.G., Alnemri, E.S., Moss, B., Lenardo, M.J., Tomaselli, K.J. and Cohen, J.I. (1997) Death effector domain-containing herpesvirus and poxvirus proteins inhibit both Fas- and TNFR1-induced apoptosis. Proc Natl Acad Sci U S A, 94, 1172-1176. Boatright, K.M., Renatus, M., Scott, F.L., Sperandio, S., Shin, H., Pedersen, I.M., Ricci, J.E., Edris, W.A., Sutherlin, D.P., Green, D.R. and Salvesen, G.S. (2003) A unified model for apical caspase activation. Mol Cell, 11, 529-541. Bodmer, J.L., Meier, P., Tschopp, J. and Schneider, P. (2000) Cysteine 230 is essential for the structure and activity of the cytotoxic ligand TRAIL. J Biol Chem, 275, 20632-20637. Bodmer, J.L., Schneider, P. and Tschopp, J. (2002) The molecular architecture of the TNF superfamily. Trends Biochem Sci, 27, 19-26. Bork, P.M., Schmitz, M.L., Kuhnt, M., Escher, C. and Heinrich, M. (1997) Sesquiterpene lactone containing Mexican Indian medicinal plants and pure 154 sesquiterpene lactones as potent inhibitors of transcription factor NF-kappaB. FEBS Lett, 402, 85-90. Borner, C. (2003) The Bcl-2 protein family: sensors and checkpoints for life-or-death decisions. Mol Immunol, 39, 615-647. Bosque, A., Pardo, J., Martinez-Lorenzo, M.J., Iturralde, M., Marzo, I., Pineiro, A., Alava, M.A., Naval, J. and Anel, A. (2005) Down-regulation of normal human T cell blast activation: roles of APO2L/TRAIL, FasL, and c- FLIP, Bim, or Bcl-x isoform expression. J Leukoc Biol, 77, 568-578. Bradbury, D.A., Simmons, T.D., Slater, K.J. and Crouch, S.P. (2000) Measurement of the ADP:ATP ratio in human leukaemic cell lines can be used as an indicator of cell viability, necrosis and apoptosis. J Immunol Methods, 240, 79-92. Bremner, P. and Heinrich, M. (2002) Natural products as targeted modulators of the nuclear factor-kappaB pathway. J Pharm Pharmacol, 54, 453-472. Brooks, A.D. and Sayers, T.J. (2005) Reduction of the antiapoptotic protein cFLIP enhances the susceptibility of human renal cancer cells to TRAIL apoptosis. Cancer Immunol Immunother, 54, 499-505. Broome, H.E., Dargan, C.M., Krajewski, S. and Reed, J.C. (1995) Expression of Bcl-2, Bcl-x, and Bax after T cell activation and IL-2 withdrawal. J Immunol, 155, 2311-2317. Buggy, J.J., Sideris, M.L., Mak, P., Lorimer, D.D., McIntosh, B. and Clark, J.M. (2000) Cloning and characterization of a novel human histone deacetylase, HDAC8. Biochem J, 350 Pt 1, 199-205. Carapeti, M., Aguiar, R.C., Watmore, A.E., Goldman, J.M. and Cross, N.C. (1999) Consistent fusion of MOZ and TIF2 in AML with inv(8)(p11q13). Cancer Genet Cytogenet, 113, 70-72. Caron, C., Boyault, C. and Khochbin, S. (2005) Regulatory cross-talk between lysine acetylation and ubiquitination: role in the control of protein stability. Bioessays, 27, 408-415. Carrozza, M.J., Utley, R.T., Workman, J.L. and Cote, J. (2003) The diverse functions of histone acetyltransferase complexes. Trends Genet, 19, 321-329. 155 Clancy, L., Mruk, K., Archer, K., Woelfel, M., Mongkolsapaya, J., Screaton, G., Lenardo, M.J. and Chan, F.K. (2005) Preligand assembly domain-mediated ligand-independent association between TRAIL receptor 4 (TR4) and TR2 regulates TRAIL-induced apoptosis. Proc Natl Acad Sci U S A, 102, 1809918104. Clarke, N., Jimenez-Lara, A.M., Voltz, E. and Gronemeyer, H. (2004) Tumor suppressor IRF-1 mediates retinoid and interferon anticancer signaling to death ligand TRAIL. Embo J, 23, 3051-3060. Cleary, M.L. and Sklar, J. (1987) Formation of a hybrid bcl-2/immunoglobulin transcript as a result of t(14;18) chromosomal translocation. Haematol Blood Transfus, 31, 314-319. Collins, I. and Garrett, M.D. (2005) Targeting the cell division cycle in cancer: CDK and cell cycle checkpoint kinase inhibitors. Curr Opin Pharmacol, 5, 366-373. Corazza, N., Jakob, S., Schaer, C., Frese, S., Keogh, A., Stroka, D., Kassahn, D., Torgler, R., Mueller, C., Schneider, P. and Brunner, T. (2006) TRAIL receptormediated JNK activation and Bim phosphorylation critically regulate Fasmediated liver damage and lethality. J Clin Invest, 116, 2493-2499. Cory, S. and Adams, J.M. (2002) The Bcl2 family: regulators of the cellular life-ordeath switch. Nat Rev Cancer, 2, 647-656. Crazzolara, R. and Bernhard, D. (2005) CXCR4 chemokine receptors, histone deacetylase inhibitors and acute lymphoblastic leukemia. Leuk Lymphoma, 46, 1545-1551. Cryns, V. and Yuan, J. (1998) Proteases to die for. Genes Dev, 12, 1551-1570. Cuconati, A., Mukherjee, C., Perez, D. and White, E. (2003) DNA damage response and MCL-1 destruction initiate apoptosis in adenovirus-infected cells. Genes Dev, 17, 2922-2932. Cha, S.S., Sung, B.J., Kim, Y.A., Song, Y.L., Kim, H.J., Kim, S., Lee, M.S. and Oh, B.H. (2000) Crystal structure of TRAIL-DR5 complex identifies a critical role of the unique frame insertion in conferring recognition specificity. J Biol Chem, 275, 31171-31177. 156 Chao, J.R., Wang, J.M., Lee, S.F., Peng, H.W., Lin, Y.H., Chou, C.H., Li, J.C., Huang, H.M., Chou, C.K., Kuo, M.L., Yen, J.J. and Yang-Yen, H.F. (1998) mcl-1 is an immediate-early gene activated by the granulocyte-macrophage colonystimulating factor (GM-CSF) signaling pathway and is one component of the GM-CSF viability response. Mol Cell Biol, 18, 4883-4898. Chaudhary, P.M., Eby, M., Jasmin, A., Bookwalter, A., Murray, J. and Hood, L. (1997) Death receptor 5, a new member of the TNFR family, and DR4 induce FADDdependent apoptosis and activate the NF-kappaB pathway. Immunity, 7, 821830. Chen, L., Willis, S.N., Wei, A., Smith, B.J., Fletcher, J.I., Hinds, M.G., Colman, P.M., Day, C.L., Adams, J.M. and Huang, D.C. (2005) Differential targeting of prosurvival Bcl-2 proteins by their BH3-only ligands allows complementary apoptotic function. Mol Cell, 17, 393-403. Chen, Y., Zhou, J., Yue, B., Xiang, Z., Song, X., Li, C. and Wang, B. (1998) Bcl-2 gene and its family genes Bax, Bcl-Xl as well as Fas/Apo-1 and their clinical significance in acute leukemia. Chin Med J (Engl), 111, 682-685. Dai, Y., Dent, P. and Grant, S. (2003) Tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) promotes mitochondrial dysfunction and apoptosis induced by 7-hydroxystaurosporine and mitogen-activated protein kinase kinase inhibitors in human leukemia cells that ectopically express Bcl-2 and Bcl-xL. Mol Pharmacol, 64, 1402-1409. Degli-Esposti, M. (1999) To die or not to die--the quest of the TRAIL receptors. J Leukoc Biol, 65, 535-542. Degli-Esposti, M.A., Dougall, W.C., Smolak, P.J., Waugh, J.Y., Smith, C.A. and Goodwin, R.G. (1997a) The novel receptor TRAIL-R4 induces NF-kappaB and protects against TRAIL-mediated apoptosis, yet retains an incomplete death domain. Immunity, 7, 813-820. Degli-Esposti, M.A., Smolak, P.J., Walczak, H., Waugh, J., Huang, C.P., DuBose, R.F., Goodwin, R.G. and Smith, C.A. (1997b) Cloning and characterization of TRAIL-R3, a novel member of the emerging TRAIL receptor family. J Exp Med, 186, 1165-1170. 157 Deveraux, Q.L., Takahashi, R., Salvesen, G.S. and Reed, J.C. (1997) X-linked IAP is a direct inhibitor of cell-death proteases. Nature, 388, 300-304. Domingo-Domenech, J., Pippa, R., Tapia, M., Gascon, P., Bachs, O. and Bosch, M. (2007) Inactivation of NF-kappaB by proteasome inhibition contributes to increased apoptosis induced by histone deacetylase inhibitors in human breast cancer cells. Breast Cancer Res Treat. Dorothee, G., Vergnon, I., Menez, J., Echchakir, H., Grunenwald, D., Kubin, M., Chouaib, S. and Mami-Chouaib, F. (2002) Tumor-infiltrating CD4+ T lymphocytes express APO2 ligand (APO2L)/TRAIL upon specific stimulation with autologous lung carcinoma cells: role of IFN-alpha on APO2L/TRAIL expression and -mediated cytotoxicity. J Immunol, 169, 809-817. Du, C., Fang, M., Li, Y., Li, L. and Wang, X. (2000) Smac, a mitochondrial protein that promotes cytochrome c-dependent caspase activation by eliminating IAP inhibition. Cell, 102, 33-42. Duan, H., Heckman, C.A. and Boxer, L.M. (2005) Histone deacetylase inhibitors downregulate bcl-2 expression and induce apoptosis in t(14;18) lymphomas. Mol Cell Biol, 25, 1608-1619. Duan, S., Hajek, P., Lin, C., Shin, S.K., Attardi, G. and Chomyn, A. (2003) Mitochondrial outer membrane permeability change and hypersensitivity to digitonin early in staurosporine-induced apoptosis. J Biol Chem, 278, 13461353. Duvic, M. and Vu, J. (2007) Vorinostat: a new oral histone deacetylase inhibitor approved for cutaneous T-cell lymphoma. Expert Opin Investig Drugs, 16, 1111-1120. Ehrhardt, H., Fulda, S., Schmid, I., Hiscott, J., Debatin, K.M. and Jeremias, I. (2003) TRAIL induced survival and proliferation in cancer cells resistant towards TRAIL-induced apoptosis mediated by NF-kappaB. Oncogene, 22, 3842-3852. Emanuele, S., Lauricella, M., Carlisi, D., Vassallo, B., D'Anneo, A., Di Fazio, P., Vento, R. and Tesoriere, G. (2007) SAHA induces apoptosis in hepatoma cells and synergistically interacts with the proteasome inhibitor Bortezomib. Apoptosis, 12, 1327-1338. 158 Eskes, R., Desagher, S., Antonsson, B. and Martinou, J.C. (2000) Bid induces the oligomerization and insertion of Bax into the outer mitochondrial membrane. Mol Cell Biol, 20, 929-935. Fandy, T.E., Shankar, S., Ross, D.D., Sausville, E. and Srivastava, R.K. (2005) Interactive effects of HDAC inhibitors and TRAIL on apoptosis are associated with changes in mitochondrial functions and expressions of cell cycle regulatory genes in multiple myeloma. Neoplasia, 7, 646-657. Finnin, M.S., Donigian, J.R., Cohen, A., Richon, V.M., Rifkind, R.A., Marks, P.A., Breslow, R. and Pavletich, N.P. (1999) Structures of a histone deacetylase homologue bound to the TSA and SAHA inhibitors. Nature, 401, 188-193. Fionda, C., Nappi, F., Piccoli, M., Frati, L., Santoni, A. and Cippitelli, M. (2007) Inhibition of trail gene expression by cyclopentenonic prostaglandin 15-deoxydelta12,14-prostaglandin J2 in T lymphocytes. Mol Pharmacol, 72, 1246-1257. Fischle, W., Dequiedt, F., Fillion, M., Hendzel, M.J., Voelter, W. and Verdin, E. (2001) Human HDAC7 histone deacetylase activity is associated with HDAC3 in vivo. J Biol Chem, 276, 35826-35835. Fischle, W., Emiliani, S., Hendzel, M.J., Nagase, T., Nomura, N., Voelter, W. and Verdin, E. (1999) A new family of human histone deacetylases related to Saccharomyces cerevisiae HDA1p. J Biol Chem, 274, 11713-11720. Follows, G.A., Tagoh, H., Lefevre, P., Hodge, D., Morgan, G.J. and Bonifer, C. (2003) Epigenetic consequences of AML1-ETO action at the human c-FMS locus. Embo J, 22, 2798-2809. Franco, A.V., Zhang, X.D., Van Berkel, E., Sanders, J.E., Zhang, X.Y., Thomas, W.D., Nguyen, T. and Hersey, P. (2001) The role of NF-kappa B in TNF-related apoptosis-inducing ligand (TRAIL)-induced apoptosis of melanoma cells. J Immunol, 166, 5337-5345. Frisch, S.M. and Screaton, R.A. (2001) Anoikis mechanisms. Curr Opin Cell Biol, 13, 555-562. Frye, R.A. (1999) Characterization of five human cDNAs with homology to the yeast SIR2 gene: Sir2-like proteins (sirtuins) metabolize NAD and may have protein ADP-ribosyltransferase activity. Biochem Biophys Res Commun, 260, 273-279. 159 Fuller, K., Wong, B., Fox, S., Choi, Y. and Chambers, T.J. (1998) TRANCE is necessary and sufficient for osteoblast-mediated activation of bone resorption in osteoclasts. J Exp Med, 188, 997-1001. Galm, O., Herman, J.G. and Baylin, S.B. (2006) The fundamental role of epigenetics in hematopoietic malignancies. Blood Rev, 20, 1-13. Gao, L., Cueto, M.A., Asselbergs, F. and Atadja, P. (2002) Cloning and functional characterization of HDAC11, a novel member of the human histone deacetylase family. J Biol Chem, 277, 25748-25755. Georgakis, G.V., Li, Y., Humphreys, R., Andreeff, M., O'Brien, S., Younes, M., Carbone, A., Albert, V. and Younes, A. (2005) Activity of selective fully human agonistic antibodies to the TRAIL death receptors TRAIL-R1 and TRAIL-R2 in primary and cultured lymphoma cells: induction of apoptosis and enhancement of doxorubicin- and bortezomib-induced cell death. Br J Haematol, 130, 501510. Gerber, M. and Shilatifard, A. (2003) Transcriptional elongation by RNA polymerase II and histone methylation. J Biol Chem, 278, 26303-26306. Giandomenico, V., Simonsson, M., Gronroos, E. and Ericsson, J. (2003) Coactivatordependent acetylation stabilizes members of the SREBP family of transcription factors. Mol Cell Biol, 23, 2587-2599. Giles, F., Verstovsek, S., Faderl, S., Vey, N., Karp, J., Roboz, G., Khan, K.D., Cooper, M., Bilgrami, S.F., Ferrant, A., Daenen, S., Karsten, V., Cahill, A., Albitar, M., Kantarjian, H., O'Brien, S. and Feldman, E. (2006) A phase II study of cloretazine (VNP40101M), a novel sulfonylhydrazine alkylating agent, in patients with very high risk relapsed acute myeloid leukemia. Leuk Res, 30, 1591-1595. Glaser, K.B., Staver, M.J., Waring, J.F., Stender, J., Ulrich, R.G. and Davidsen, S.K. (2003) Gene expression profiling of multiple histone deacetylase (HDAC) inhibitors: defining a common gene set produced by HDAC inhibition in T24 and MDA carcinoma cell lines. Mol Cancer Ther, 2, 151-163. Glozak, M.A., Sengupta, N., Zhang, X. and Seto, E. (2005) Acetylation and deacetylation of non-histone proteins. Gene, 363, 15-23. 160 Golay, J., Cuppini, L., Leoni, F., Mico, C., Barbui, V., Domenghini, M., Lombardi, L., Neri, A., Barbui, A.M., Salvi, A., Pozzi, P., Porro, G., Pagani, P., Fossati, G., Mascagni, P., Introna, M. and Rambaldi, A. (2007) The histone deacetylase inhibitor ITF2357 has anti-leukemic activity in vitro and in vivo and inhibits IL6 and VEGF production by stromal cells. Leukemia, 21, 1892-1900. Goltsev, Y.V., Kovalenko, A.V., Arnold, E., Varfolomeev, E.E., Brodianskii, V.M. and Wallach, D. (1997) CASH, a novel caspase homologue with death effector domains. J Biol Chem, 272, 19641-19644. Gomyo, Y., Sasaki, J., Branch, C., Roth, J.A. and Mukhopadhyay, T. (2004) 5-aza-2'deoxycytidine upregulates caspase-9 expression cooperating with p53-induced apoptosis in human lung cancer cells. Oncogene, 23, 6779-6787. Gong, B. and Almasan, A. (2000) Genomic organization and transcriptional regulation of human Apo2/TRAIL gene. Biochem Biophys Res Commun, 278, 747-752. Gray, S.G. and Ekstrom, T.J. (2001) The human histone deacetylase family. Exp Cell Res, 262, 75-83. Green, D.R., Droin, N. and Pinkoski, M. (2003) Activation-induced cell death in T cells. Immunol Rev, 193, 70-81. Green, D.R. and Kroemer, G. (2004) The pathophysiology of mitochondrial cell death. Science, 305, 626-629. Greil, R., Egle, A. and Villunger, A. (1998) On the role and significance of Fas (Apo1/CD95) ligand (FasL) expression in immune privileged tissues and cancer cells using multiple myeloma as a model. Leuk Lymphoma, 31, 477-490. Griffith, T.S. and Lynch, D.H. (1998) TRAIL: a molecule with multiple receptors and control mechanisms. Curr Opin Immunol, 10, 559-563. Griffith, T.S., Wiley, S.R., Kubin, M.Z., Sedger, L.M., Maliszewski, C.R. and Fanger, N.A. (1999) Monocyte-mediated tumoricidal activity via the tumor necrosis factor-related cytokine, TRAIL. J Exp Med, 189, 1343-1354. Griffiths, G.J., Dubrez, L., Morgan, C.P., Jones, N.A., Whitehouse, J., Corfe, B.M., Dive, C. and Hickman, J.A. (1999) Cell damage-induced conformational changes of the pro-apoptotic protein Bak in vivo precede the onset of apoptosis. J Cell Biol, 144, 903-914. 161 Grinberg, M., Sarig, R., Zaltsman, Y., Frumkin, D., Grammatikakis, N., Reuveny, E. and Gross, A. (2002) tBID Homooligomerizes in the mitochondrial membrane to induce apoptosis. J Biol Chem, 277, 12237-12245. Gruss, H.J. and Dower, S.K. (1995) Tumor necrosis factor ligand superfamily: involvement in the pathology of malignant lymphomas. Blood, 85, 3378-3404. Gu, W., Shi, X.L. and Roeder, R.G. (1997) Synergistic activation of transcription by CBP and p53. Nature, 387, 819-823. Guenther, M.G., Lane, W.S., Fischle, W., Verdin, E., Lazar, M.A. and Shiekhattar, R. (2000) A core SMRT corepressor complex containing HDAC3 and TBL1, a WD40-repeat protein linked to deafness. Genes Dev, 14, 1048-1057. Gurley, K.E., Vo, K. and Kemp, C.J. (1998) DNA double-strand breaks, p53, and apoptosis during lymphomagenesis in scid/scid mice. Cancer Res, 58, 31113115. Hagemann, C. and Blank, J.L. (2001) The ups and downs of MEK kinase interactions. Cell Signal, 13, 863-875. Hahntow, I.N., Schneller, F., Oelsner, M., Weick, K., Ringshausen, I., Fend, F., Peschel, C. and Decker, T. (2004) Cyclin-dependent kinase inhibitor Roscovitine induces apoptosis in chronic lymphocytic leukemia cells. Leukemia, 18, 747-755. Han, S., Choi, H., Ko, M.G., Choi, Y.I., Sohn, D.H., Kim, J.K., Shin, D., Chung, H., Lee, H.W., Kim, J.B., Park, S.D. and Seong, R.H. (2001) Peripheral T cells become sensitive to glucocorticoid- and stress-induced apoptosis in transgenic mice overexpressing SRG3. J Immunol, 167, 805-810. Handschumacher, R.E., Harding, M.W., Rice, J., Drugge, R.J. and Speicher, D.W. (1984) Cyclophilin: a specific cytosolic binding protein for cyclosporin A. Science, 226, 544-547. Hasegawa, H., Yamada, Y., Harasawa, H., Tsuji, T., Murata, K., Sugahara, K., Tsuruda, K., Masuda, M., Takasu, N. and Kamihira, S. (2004) Restricted expression of tumor necrosis factor-related apoptosis-inducing ligand receptor 4 in human peripheral blood lymphocytes. Cell Immunol, 231, 1-7. 162 Hazzalin, C.A., Cuenda, A., Cano, E., Cohen, P. and Mahadevan, L.C. (1997) Effects of the inhibition of p38/RK MAP kinase on induction of five fos and jun genes by diverse stimuli. Oncogene, 15, 2321-2331. Heinzel, T., Lavinsky, R.M., Mullen, T.M., Soderstrom, M., Laherty, C.D., Torchia, J., Yang, W.M., Brard, G., Ngo, S.D., Davie, J.R., Seto, E., Eisenman, R.N., Rose, D.W., Glass, C.K. and Rosenfeld, M.G. (1997) A complex containing N-CoR, mSin3 and histone deacetylase mediates transcriptional repression. Nature, 387, 43-48. Hengartner, M.O. (2000) The biochemistry of apoptosis. Nature, 407, 770-776. Hernandez, A., Thomas, R., Smith, F., Sandberg, J., Kim, S., Chung, D.H. and Evers, B.M. (2001) Butyrate sensitizes human colon cancer cells to TRAIL-mediated apoptosis. Surgery, 130, 265-272. Hiebert, S.W., Reed-Inderbitzin, E.F., Amann, J., Irvin, B., Durst, K. and Linggi, B. (2003) The t(8;21) fusion protein contacts co-repressors and histone deacetylases to repress the transcription of the p14ARF tumor suppressor. Blood Cells Mol Dis, 30, 177-183. Holen, I., Croucher, P.I., Hamdy, F.C. and Eaton, C.L. (2002) Osteoprotegerin (OPG) is a survival factor for human prostate cancer cells. Cancer Res, 62, 1619-1623. Hsu, S.S., Huang, C.J., Cheng, H.H., Chou, C.T., Lee, H.Y., Wang, J.L., Chen, I.S., Liu, S.I., Lu, Y.C., Chang, H.T., Huang, J.K., Chen, J.S. and Jan, C.R. (2007) Anandamide-induced Ca2+ elevation leading to p38 MAPK phosphorylation and subsequent cell death via apoptosis in human osteosarcoma cells. Toxicology, 231, 21-29. Hsu, S.Y., Kaipia, A., McGee, E., Lomeli, M. and Hsueh, A.J. (1997) Bok is a proapoptotic Bcl-2 protein with restricted expression in reproductive tissues and heterodimerizes with selective anti-apoptotic Bcl-2 family members. Proc Natl Acad Sci U S A, 94, 12401-12406. Hu, E., Dul, E., Sung, C.M., Chen, Z., Kirkpatrick, R., Zhang, G.F., Johanson, K., Liu, R., Lago, A., Hofmann, G., Macarron, R., de los Frailes, M., Perez, P., Krawiec, J., Winkler, J. and Jaye, M. (2003) Identification of novel isoform-selective 163 inhibitors within class I histone deacetylases. J Pharmacol Exp Ther, 307, 720728. Hu, J. and Colburn, N.H. (2005) Histone deacetylase inhibition down-regulates cyclin D1 transcription by inhibiting nuclear factor-kappaB/p65 DNA binding. Mol Cancer Res, 3, 100-109. Hu, S., Vincenz, C., Ni, J., Gentz, R. and Dixit, V.M. (1997) I-FLICE, a novel inhibitor of tumor necrosis factor receptor-1- and CD-95-induced apoptosis. J Biol Chem, 272, 17255-17257. Hu, Y., Benedict, M.A., Wu, D., Inohara, N. and Nunez, G. (1998) Bcl-XL interacts with Apaf-1 and inhibits Apaf-1-dependent caspase-9 activation. Proc Natl Acad Sci U S A, 95, 4386-4391. Huang, D.C. and Strasser, A. (2000) BH3-Only proteins-essential initiators of apoptotic cell death. Cell, 103, 839-842. Huang, T.T. and Miyamoto, S. (2001) Postrepression activation of NF-kappaB requires the amino-terminal nuclear export signal specific to IkappaBalpha. Mol Cell Biol, 21, 4737-4747. Hubbert, C., Guardiola, A., Shao, R., Kawaguchi, Y., Ito, A., Nixon, A., Yoshida, M., Wang, X.F. and Yao, T.P. (2002) HDAC6 is a microtubule-associated deacetylase. Nature, 417, 455-458. Huerta-Yepez, S., Vega, M., Jazirehi, A., Garban, H., Hongo, F., Cheng, G. and Bonavida, B. (2004) Nitric oxide sensitizes prostate carcinoma cell lines to TRAIL-mediated apoptosis via inactivation of NF-kappa B and inhibition of Bcl-xl expression. Oncogene, 23, 4993-5003. Huletsky, A., de Murcia, G., Muller, S., Hengartner, M., Menard, L., Lamarre, D. and Poirier, G.G. (1989) The effect of poly(ADP-ribosyl)ation on native and H1depleted chromatin. A role of poly(ADP-ribosyl)ation on core nucleosome structure. J Biol Chem, 264, 8878-8886. Inoue, S., MacFarlane, M., Harper, N., Wheat, L.M., Dyer, M.J. and Cohen, G.M. (2004) Histone deacetylase inhibitors potentiate TNF-related apoptosis-inducing ligand (TRAIL)-induced apoptosis in lymphoid malignancies. Cell Death Differ, 11 Suppl 2, S193-206. 164 Inoue, S., Mai, A., Dyer, M.J. and Cohen, G.M. (2006a) Inhibition of histone deacetylase class I but not class II is critical for the sensitization of leukemic cells to tumor necrosis factor-related apoptosis-inducing ligand-induced apoptosis. Cancer Res, 66, 6785-6792. Inoue, S., Twiddy, D., Dyer, M.J. and Cohen, G.M. (2006b) Upregulation of TRAIL-R2 is not involved in HDACi mediated sensitization to TRAIL-induced apoptosis. Cell Death Differ, 13, 2160-2162. Insinga, A., Monestiroli, S., Ronzoni, S., Gelmetti, V., Marchesi, F., Viale, A., Altucci, L., Nervi, C., Minucci, S. and Pelicci, P.G. (2005) Inhibitors of histone deacetylases induce tumor-selective apoptosis through activation of the death receptor pathway. Nat Med, 11, 71-76. Ionov, Y., Yamamoto, H., Krajewski, S., Reed, J.C. and Perucho, M. (2000) Mutational inactivation of the proapoptotic gene BAX confers selective advantage during tumor clonal evolution. Proc Natl Acad Sci U S A, 97, 10872-10877. Irmler, M., Thome, M., Hahne, M., Schneider, P., Hofmann, K., Steiner, V., Bodmer, J.L., Schroter, M., Burns, K., Mattmann, C., Rimoldi, D., French, L.E. and Tschopp, J. (1997) Inhibition of death receptor signals by cellular FLIP. Nature, 388, 190-195. Jeremias, I., Herr, I., Boehler, T. and Debatin, K.M. (1998) TRAIL/Apo-2-ligandinduced apoptosis in human T cells. Eur J Immunol, 28, 143-152. Jilka, R.L., Weinstein, R.S., Bellido, T., Roberson, P., Parfitt, A.M. and Manolagas, S.C. (1999) Increased bone formation by prevention of osteoblast apoptosis with parathyroid hormone. J Clin Invest, 104, 439-446. Johnson, C.A. and Turner, B.M. (1999) Histone deacetylases: complex transducers of nuclear signals. Semin Cell Dev Biol, 10, 179-188. Johnson, G.L. and Lapadat, R. (2002) Mitogen-activated protein kinase pathways mediated by ERK, JNK, and p38 protein kinases. Science, 298, 1911-1912. Johnstone, R.W. (2002) Histone-deacetylase inhibitors: novel drugs for the treatment of cancer. Nat Rev Drug Discov, 1, 287-299. Kandasamy, K. and Srivastava, R.K. (2002) Role of the phosphatidylinositol 3'kinase/PTEN/Akt kinase pathway in tumor necrosis factor-related apoptosis165 inducing ligand-induced apoptosis in non-small cell lung cancer cells. Cancer Res, 62, 4929-4937. Kang, J., Kisenge, R.R., Toyoda, H., Tanaka, S., Bu, J., Azuma, E. and Komada, Y. (2003) Chemical sensitization and regulation of TRAIL-induced apoptosis in a panel of B-lymphocytic leukaemia cell lines. Br J Haematol, 123, 921-932. Karin, M. and Delhase, M. (2000) The I kappa B kinase (IKK) and NF-kappa B: key elements of proinflammatory signalling. Semin Immunol, 12, 85-98. Karin, M. and Lin, A. (2002) NF-kappaB at the crossroads of life and death. Nat Immunol, 3, 221-227. Kasuga, C., Ebata, T., Kayagaki, N., Yagita, H., Hishii, M., Arai, H., Sato, K. and Okumura, K. (2004) Sensitization of human glioblastomas to tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) by NF-kappaB inhibitors. Cancer Sci, 95, 840-844. Kayagaki, N., Yamaguchi, N., Nakayama, M., Eto, H., Okumura, K. and Yagita, H. (1999) Type I interferons (IFNs) regulate tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) expression on human T cells: A novel mechanism for the antitumor effects of type I IFNs. J Exp Med, 189, 1451-1460. Kelley, S.K., Harris, L.A., Xie, D., Deforge, L., Totpal, K., Bussiere, J. and Fox, J.A. (2001) Preclinical studies to predict the disposition of Apo2L/tumor necrosis factor-related apoptosis-inducing ligand in humans: characterization of in vivo efficacy, pharmacokinetics, and safety. J Pharmacol Exp Ther, 299, 31-38. Kelly, M.M., Hoel, B.D. and Voelkel-Johnson, C. (2002) Doxorubicin pretreatment sensitizes prostate cancer cell lines to TRAIL induced apoptosis which correlates with the loss of c-FLIP expression. Cancer Biol Ther, 1, 520-527. Kemp, T.J., Ludwig, A.T., Earel, J.K., Moore, J.M., Vanoosten, R.L., Moses, B., Leidal, K., Nauseef, W.M. and Griffith, T.S. (2005) Neutrophil stimulation with Mycobacterium bovis bacillus Calmette-Guerin (BCG) results in the release of functional soluble TRAIL/Apo-2L. Blood, 106, 3474-3482. Kerr, J.F., Wyllie, A.H. and Currie, A.R. (1972) Apoptosis: a basic biological phenomenon with wide-ranging implications in tissue kinetics. Br J Cancer, 26, 239-257. 166 Khan, N., Jeffers, M., Kumar, S., Hackett, C., Boldog, F., Khramtsov, N., Qian, X., Mills, E., Berghs, S.C., Carey, N., Finn, P.W., Collins, L.S., Tumber, A., Ritchie, J.W., Jensen, P.B., Lichenstein, H.S. and Sehested, M. (2008) Determination of the class and isoform selectivity of small-molecule histone deacetylase inhibitors. Biochem J, 409, 581-589. Khorasanizadeh, S. (2004) The nucleosome: from genomic organization to genomic regulation. Cell, 116, 259-272. Kiernan, R., Bres, V., Ng, R.W., Coudart, M.P., El Messaoudi, S., Sardet, C., Jin, D.Y., Emiliani, S. and Benkirane, M. (2003) Post-activation turn-off of NF-kappa Bdependent transcription is regulated by acetylation of p65. J Biol Chem, 278, 2758-2766. Kim, E.H., Kim, S.U., Shin, D.Y. and Choi, K.S. (2004) Roscovitine sensitizes glioma cells to TRAIL-mediated apoptosis by downregulation of survivin and XIAP. Oncogene, 23, 446-456. Kim, K., Fisher, M.J., Xu, S.Q. and el-Deiry, W.S. (2000) Molecular determinants of response to TRAIL in killing of normal and cancer cells. Clin Cancer Res, 6, 335-346. Kim, M.S., Kwon, H.J., Lee, Y.M., Baek, J.H., Jang, J.E., Lee, S.W., Moon, E.J., Kim, H.S., Lee, S.K., Chung, H.Y., Kim, C.W. and Kim, K.W. (2001) Histone deacetylases induce angiogenesis by negative regulation of tumor suppressor genes. Nat Med, 7, 437-443. Kim, Y.K., Lee, E.K., Kang, J.K., Kim, J.A., You, J.S., Park, J.H., Seo, D.W., Hwang, J.W., Kim, S.N., Lee, H.Y., Lee, H.W. and Han, J.W. (2006) Activation of NFkappaB by HDAC inhibitor apicidin through Sp1-dependent de novo protein synthesis: its implication for resistance to apoptosis. Cell Death Differ, 13, 2033-2041. Kischkel, F.C., Lawrence, D.A., Chuntharapai, A., Schow, P., Kim, K.J. and Ashkenazi, A. (2000) Apo2L/TRAIL-dependent recruitment of endogenous FADD and caspase-8 to death receptors 4 and 5. Immunity, 12, 611-620. Klose, R.J. and Bird, A.P. (2006) Genomic DNA methylation: the mark and its mediators. Trends Biochem Sci, 31, 89-97. 167 Koenig, U., Eckhart, L. and Tschachler, E. (2001) Evidence that caspase-13 is not a human but a bovine gene. Biochem Biophys Res Commun, 285, 1150-1154. Kouzarides, T. (2002) Histone methylation in transcriptional control. Curr Opin Genet Dev, 12, 198-209. Krajewski, S., Krajewska, M. and Reed, J.C. (1996) Immunohistochemical analysis of in vivo patterns of Bak expression, a proapoptotic member of the Bcl-2 protein family. Cancer Res, 56, 2849-2855. Kramer, O.H., Zhu, P., Ostendorff, H.P., Golebiewski, M., Tiefenbach, J., Peters, M.A., Brill, B., Groner, B., Bach, I., Heinzel, T. and Gottlicher, M. (2003) The histone deacetylase inhibitor valproic acid selectively induces proteasomal degradation of HDAC2. Embo J, 22, 3411-3420. Krammer, P.H., Arnold, R. and Lavrik, I.N. (2007) Life and death in peripheral T cells. Nat Rev Immunol, 7, 532-542. Kraus, J., Borner, C. and Hollt, V. (2003) Distinct palindromic extensions of the 5'TTC...GAA-3' motif allow STAT6 binding in vivo. Faseb J, 17, 304-306. Krogan, N.J., Keogh, M.C., Datta, N., Sawa, C., Ryan, O.W., Ding, H., Haw, R.A., Pootoolal, J., Tong, A., Canadien, V., Richards, D.P., Wu, X., Emili, A., Hughes, T.R., Buratowski, S. and Greenblatt, J.F. (2003) A Snf2 family ATPase complex required for recruitment of the histone H2A variant Htz1. Mol Cell, 12, 1565-1576. Krueger, A., Schmitz, I., Baumann, S., Krammer, P.H. and Kirchhoff, S. (2001) Cellular FLICE-inhibitory protein splice variants inhibit different steps of caspase-8 activation at the CD95 death-inducing signaling complex. J Biol Chem, 276, 20633-20640. Kuendgen, A., Strupp, C., Aivado, M., Bernhardt, A., Hildebrandt, B., Haas, R., Germing, U. and Gattermann, N. (2004) Treatment of myelodysplastic syndromes with valproic acid alone or in combination with all-trans retinoic acid. Blood, 104, 1266-1269. Kyritsis, A.P., Tachmazoglou, F., Rao, J.S. and Puduvalli, V.K. (2007) Bortezomib sensitizes human astrocytoma cells to tumor necrosis factor related apoptosis- 168 inducing ligand induced apoptosis. Clin Cancer Res, 13, 6540-6541; author reply 6541-6542. Lacey, D.L., Timms, E., Tan, H.L., Kelley, M.J., Dunstan, C.R., Burgess, T., Elliott, R., Colombero, A., Elliott, G., Scully, S., Hsu, H., Sullivan, J., Hawkins, N., Davy, E., Capparelli, C., Eli, A., Qian, Y.X., Kaufman, S., Sarosi, I., Shalhoub, V., Senaldi, G., Guo, J., Delaney, J. and Boyle, W.J. (1998) Osteoprotegerin ligand is a cytokine that regulates osteoclast differentiation and activation. Cell, 93, 165-176. Lamhamedi-Cherradi, S.E., Zheng, S.J., Maguschak, K.A., Peschon, J. and Chen, Y.H. (2003) Defective thymocyte apoptosis and accelerated autoimmune diseases in TRAIL-/- mice. Nat Immunol, 4, 255-260. Lamkanfi, M., Declercq, W., Kalai, M., Saelens, X. and Vandenabeele, P. (2002) Alice in caspase land. A phylogenetic analysis of caspases from worm to man. Cell Death Differ, 9, 358-361. Landry, J., Sutton, A., Tafrov, S.T., Heller, R.C., Stebbins, J., Pillus, L. and Sternglanz, R. (2000) The silencing protein SIR2 and its homologs are NAD-dependent protein deacetylases. Proc Natl Acad Sci U S A, 97, 5807-5811. Lane, D., Robert, V., Grondin, R., Rancourt, C. and Piche, A. (2007) Malignant ascites protect against TRAIL-induced apoptosis by activating the PI3K/Akt pathway in human ovarian carcinoma cells. Int J Cancer, 121, 1227-1237. Le Bras, M., Clement, M.V., Pervaiz, S. and Brenner, C. (2005) Reactive oxygen species and the mitochondrial signaling pathway of cell death. Histol Histopathol, 20, 205-219. Lee, H.J., Wang, C.J., Kuo, H.C., Chou, F.P., Jean, L.F. and Tseng, T.H. (2005) Induction apoptosis of luteolin in human hepatoma HepG2 cells involving mitochondria translocation of Bax/Bak and activation of JNK. Toxicol Appl Pharmacol, 203, 124-131. Lee, J.T., Jr. and McCubrey, J.A. (2002) The Raf/MEK/ERK signal transduction cascade as a target for chemotherapeutic intervention in leukemia. Leukemia, 16, 486-507. 169 Li, H., Zhu, H., Xu, C.J. and Yuan, J. (1998) Cleavage of BID by caspase 8 mediates the mitochondrial damage in the Fas pathway of apoptosis. Cell, 94, 491-501. Li, J., Yen, C., Liaw, D., Podsypanina, K., Bose, S., Wang, S.I., Puc, J., Miliaresis, C., Rodgers, L., McCombie, R., Bigner, S.H., Giovanella, B.C., Ittmann, M., Tycko, B., Hibshoosh, H., Wigler, M.H. and Parsons, R. (1997) PTEN, a putative protein tyrosine phosphatase gene mutated in human brain, breast, and prostate cancer. Science, 275, 1943-1947. Li, L.Y., Luo, X. and Wang, X. (2001) Endonuclease G is an apoptotic DNase when released from mitochondria. Nature, 412, 95-99. Li, W., Zhang, X. and Olumi, A.F. (2007) MG-132 sensitizes TRAIL-resistant prostate cancer cells by activating c-Fos/c-Jun heterodimers and repressing c-FLIP(L). Cancer Res, 67, 2247-2255. Lim, S.J., Lee, Y.J. and Lee, E. (2006) p38MAPK inhibitor SB203580 sensitizes human SNU-C4 colon cancer cells to exisulind-induced apoptosis. Oncol Rep, 16, 1131-1135. Lin, Y., Devin, A., Cook, A., Keane, M.M., Kelliher, M., Lipkowitz, S. and Liu, Z.G. (2000) The death domain kinase RIP is essential for TRAIL (Apo2L)-induced activation of IkappaB kinase and c-Jun N-terminal kinase. Mol Cell Biol, 20, 6638-6645. Lindemann, R.K., Gabrielli, B. and Johnstone, R.W. (2004) Histone-deacetylase inhibitors for the treatment of cancer. Cell Cycle, 3, 779-788. Lindsten, T., Ross, A.J., King, A., Zong, W.X., Rathmell, J.C., Shiels, H.A., Ulrich, E., Waymire, K.G., Mahar, P., Frauwirth, K., Chen, Y., Wei, M., Eng, V.M., Adelman, D.M., Simon, M.C., Ma, A., Golden, J.A., Evan, G., Korsmeyer, S.J., MacGregor, G.R. and Thompson, C.B. (2000) The combined functions of proapoptotic Bcl-2 family members bak and bax are essential for normal development of multiple tissues. Mol Cell, 6, 1389-1399. Liston, P., Fong, W.G. and Korneluk, R.G. (2003) The inhibitors of apoptosis: there is more to life than Bcl2. Oncogene, 22, 8568-8580. 170 Liu, L., Scolnick, D.M., Trievel, R.C., Zhang, H.B., Marmorstein, R., Halazonetis, T.D. and Berger, S.L. (1999) p53 sites acetylated in vitro by PCAF and p300 are acetylated in vivo in response to DNA damage. Mol Cell Biol, 19, 1202-1209. Liu, Q.Y. and Stein, C.A. (1997) Taxol and estramustine-induced modulation of human prostate cancer cell apoptosis via alteration in bcl-xL and bak expression. Clin Cancer Res, 3, 2039-2046. Liu, S., Yu, Y., Zhang, M., Wang, W. and Cao, X. (2001) The involvement of TNFalpha-related apoptosis-inducing ligand in the enhanced cytotoxicity of IFNbeta-stimulated human dendritic cells to tumor cells. J Immunol, 166, 54075415. Liu, Y., Borchert, G.L., Surazynski, A., Hu, C.A. and Phang, J.M. (2006) Proline oxidase activates both intrinsic and extrinsic pathways for apoptosis: the role of ROS/superoxides, NFAT and MEK/ERK signaling. Oncogene, 25, 5640-5647. Luger, K., Rechsteiner, T.J., Flaus, A.J., Waye, M.M. and Richmond, T.J. (1997) Characterization of nucleosome core particles containing histone proteins made in bacteria. J Mol Biol, 272, 301-311. Lum, J.J., Bren, G., McClure, R. and Badley, A.D. (2005) Elimination of senescent neutrophils by TNF-related apoptosis-inducing [corrected] ligand. J Immunol, 175, 1232-1238. Luo, X., Budihardjo, I., Zou, H., Slaughter, C. and Wang, X. (1998) Bid, a Bcl2 interacting protein, mediates cytochrome c release from mitochondria in response to activation of cell surface death receptors. Cell, 94, 481-490. Lutter, M., Perkins, G.A. and Wang, X. (2001) The pro-apoptotic Bcl-2 family member tBid localizes to mitochondrial contact sites. BMC Cell Biol, 2, 22. MacFarlane, M., Ahmad, M., Srinivasula, S.M., Fernandes-Alnemri, T., Cohen, G.M. and Alnemri, E.S. (1997) Identification and molecular cloning of two novel receptors for the cytotoxic ligand TRAIL. J Biol Chem, 272, 25417-25420. MacFarlane, M., Kohlhaas, S.L., Sutcliffe, M.J., Dyer, M.J. and Cohen, G.M. (2005) TRAIL receptor-selective mutants signal to apoptosis via TRAIL-R1 in primary lymphoid malignancies. Cancer Res, 65, 11265-11270. 171 Maison, C., Bailly, D., Peters, A.H., Quivy, J.P., Roche, D., Taddei, A., Lachner, M., Jenuwein, T. and Almouzni, G. (2002) Higher-order structure in pericentric heterochromatin involves a distinct pattern of histone modification and an RNA component. Nat Genet, 30, 329-334. Mariani, S.M. and Krammer, P.H. (1998a) Differential regulation of TRAIL and CD95 ligand in transformed cells of the T and B lymphocyte lineage. Eur J Immunol, 28, 973-982. Mariani, S.M. and Krammer, P.H. (1998b) Surface expression of TRAIL/Apo-2 ligand in activated mouse T and B cells. Eur J Immunol, 28, 1492-1498. Marks, P.A., Richon, V.M., Breslow, R. and Rifkind, R.A. (2001) Histone deacetylase inhibitors as new cancer drugs. Curr Opin Oncol, 13, 477-483. Marmorstein, R. and Roth, S.Y. (2001) Histone acetyltransferases: function, structure, and catalysis. Curr Opin Genet Dev, 11, 155-161. Marson, C.M., Mahadevan, T., Dines, J., Sengmany, S., Morrell, J.M., Alao, J.P., Joel, S.P., Vigushin, D.M. and Charles Coombes, R. (2007) Structure-activity relationships of aryloxyalkanoic acid hydroxyamides as potent inhibitors of histone deacetylase. Bioorg Med Chem Lett, 17, 136-141. Marzo, I., Brenner, C., Zamzami, N., Jurgensmeier, J.M., Susin, S.A., Vieira, H.L., Prevost, M.C., Xie, Z., Matsuyama, S., Reed, J.C. and Kroemer, G. (1998) Bax and adenine nucleotide translocator cooperate in the mitochondrial control of apoptosis. Science, 281, 2027-2031. Matsuda, T., Almasan, A., Tomita, M., Uchihara, J.N., Masuda, M., Ohshiro, K., Takasu, N., Yagita, H., Ohta, T. and Mori, N. (2005) Resistance to Apo2 ligand (Apo2L)/tumor necrosis factor-related apoptosis-inducing ligand (TRAIL)mediated apoptosis and constitutive expression of Apo2L/TRAIL in human Tcell leukemia virus type 1-infected T-cell lines. J Virol, 79, 1367-1378. Mattson, M.P. and Kroemer, G. (2003) Mitochondria in cell death: novel targets for neuroprotection and cardioprotection. Trends Mol Med, 9, 196-205. Mayo, M.W., Denlinger, C.E., Broad, R.M., Yeung, F., Reilly, E.T., Shi, Y. and Jones, D.R. (2003) Ineffectiveness of histone deacetylase inhibitors to induce apoptosis 172 involves the transcriptional activation of NF-kappa B through the Akt pathway. J Biol Chem, 278, 18980-18989. McCaffrey, P.G., Kim, P.K., Valge-Archer, V.E., Sen, R. and Rao, A. (1994) Cyclosporin A sensitivity of the NF-kappa B site of the IL2R alpha promoter in untransformed murine T cells. Nucleic Acids Res, 22, 2134-2142. McKinsey, T.A., Zhang, C.L. and Olson, E.N. (2000) Activation of the myocyte enhancer factor-2 transcription factor by calcium/calmodulin-dependent protein kinase-stimulated binding of 14-3-3 to histone deacetylase 5. Proc Natl Acad Sci U S A, 97, 14400-14405. Mercurio, F., Zhu, H., Murray, B.W., Shevchenko, A., Bennett, B.L., Li, J., Young, D.B., Barbosa, M., Mann, M., Manning, A. and Rao, A. (1997) IKK-1 and IKK2: cytokine-activated IkappaB kinases essential for NF-kappaB activation. Science, 278, 860-866. Micheau, O., Lens, S., Gaide, O., Alevizopoulos, K. and Tschopp, J. (2001) NF-kappaB signals induce the expression of c-FLIP. Mol Cell Biol, 21, 5299-5305. Mirandola, P., Ponti, C., Gobbi, G., Sponzilli, I., Vaccarezza, M., Cocco, L., Zauli, G., Secchiero, P., Manzoli, F.A. and Vitale, M. (2004) Activated human NK and CD8+ T cells express both TNF-related apoptosis-inducing ligand (TRAIL) and TRAIL receptors but are resistant to TRAIL-mediated cytotoxicity. Blood, 104, 2418-2424. Monleon, I., Martinez-Lorenzo, M.J., Monteagudo, L., Lasierra, P., Taules, M., Iturralde, M., Pineiro, A., Larrad, L., Alava, M.A., Naval, J. and Anel, A. (2001) Differential secretion of Fas ligand- or APO2 ligand/TNF-related apoptosisinducing ligand-carrying microvesicles during activation-induced death of human T cells. J Immunol, 167, 6736-6744. Munk, M., Memon, A.A., Nexo, E. and Sorensen, B.S. (2007) Inhibition of the epidermal growth factor receptor in bladder cancer cells treated with the DNAdamaging drug etoposide markedly increases apoptosis. BJU Int, 99, 196-201. Munoz-Pinedo, C., Ruiz de Almodovar, C. and Ruiz-Ruiz, C. (2002) Death on the beach: a rosy forecast for the 21st century. Cell Death Differ, 9, 1026-1029. 173 Nakata, S., Yoshida, T., Horinaka, M., Shiraishi, T., Wakada, M. and Sakai, T. (2004) Histone deacetylase inhibitors upregulate death receptor 5/TRAIL-R2 and sensitize apoptosis induced by TRAIL/APO2-L in human malignant tumor cells. Oncogene, 23, 6261-6271. Nakshatri, H., Rice, S.E. and Bhat-Nakshatri, P. (2004) Antitumor agent parthenolide reverses resistance of breast cancer cells to tumor necrosis factor-related apoptosis-inducing ligand through sustained activation of c-Jun N-terminal kinase. Oncogene, 23, 7330-7344. Nam, S.Y., Jung, G.A., Hur, G.C., Chung, H.Y., Kim, W.H., Seol, D.W. and Lee, B.L. (2003) Upregulation of FLIP(S) by Akt, a possible inhibition mechanism of TRAIL-induced apoptosis in human gastric cancers. Cancer Sci, 94, 1066-1073. Natarajan, K., Singh, S., Burke, T.R., Jr., Grunberger, D. and Aggarwal, B.B. (1996) Caffeic acid phenethyl ester is a potent and specific inhibitor of activation of nuclear transcription factor NF-kappa B. Proc Natl Acad Sci U S A, 93, 90909095. Nebbioso, A., Clarke, N., Voltz, E., Germain, E., Ambrosino, C., Bontempo, P., Alvarez, R., Schiavone, E.M., Ferrara, F., Bresciani, F., Weisz, A., de Lera, A.R., Gronemeyer, H. and Altucci, L. (2005) Tumor-selective action of HDAC inhibitors involves TRAIL induction in acute myeloid leukemia cells. Nat Med, 11, 77-84. Nechushtan, A., Smith, C.L., Hsu, Y.T. and Youle, R.J. (1999) Conformation of the Bax C-terminus regulates subcellular location and cell death. Embo J, 18, 23302341. Nesterov, A., Lu, X., Johnson, M., Miller, G.J., Ivashchenko, Y. and Kraft, A.S. (2001) Elevated AKT activity protects the prostate cancer cell line LNCaP from TRAIL-induced apoptosis. J Biol Chem, 276, 10767-10774. Nijhawan, D., Fang, M., Traer, E., Zhong, Q., Gao, W., Du, F. and Wang, X. (2003) Elimination of Mcl-1 is required for the initiation of apoptosis following ultraviolet irradiation. Genes Dev, 17, 1475-1486. Nimmanapalli, R., Perkins, C.L., Orlando, M., O'Bryan, E., Nguyen, D. and Bhalla, K.N. (2001) Pretreatment with paclitaxel enhances apo-2 ligand/tumor necrosis 174 factor-related apoptosis-inducing ligand-induced apoptosis of prostate cancer cells by inducing death receptors 4 and 5 protein levels. Cancer Res, 61, 759763. Ocker, M. and Schneider-Stock, R. (2007) Histone deacetylase inhibitors: signalling towards p21cip1/waf1. Int J Biochem Cell Biol, 39, 1367-1374. Oh, K.J., Barbuto, S., Meyer, N., Kim, R.S., Collier, R.J. and Korsmeyer, S.J. (2005) Conformational changes in BID, a pro-apoptotic BCL-2 family member, upon membrane binding. A site-directed spin labeling study. J Biol Chem, 280, 753767. Ohtsuka, T. and Zhou, T. (2002) Bisindolylmaleimide VIII enhances DR5-mediated apoptosis through the MKK4/JNK/p38 kinase and the mitochondrial pathways. J Biol Chem, 277, 29294-29303. Oka, N., Tanimoto, S., Taue, R., Nakatsuji, H., Kishimoto, T., Izaki, H., Fukumori, T., Takahashi, M., Nishitani, M. and Kanayama, H.O. (2006) Role of phosphatidylinositol-3 kinase/Akt pathway in bladder cancer cell apoptosis induced by tumor necrosis factor-related apoptosis-inducing ligand. Cancer Sci, 97, 1093-1098. Olsson, A., Diaz, T., Aguilar-Santelises, M., Osterborg, A., Celsing, F., Jondal, M. and Osorio, L.M. (2001) Sensitization to TRAIL-induced apoptosis and modulation of FLICE-inhibitory protein in B chronic lymphocytic leukemia by actinomycin D. Leukemia, 15, 1868-1877. Osley, M.A. (2004) H2B ubiquitylation: the end is in sight. Biochim Biophys Acta, 1677, 74-78. Ottilie, S., Diaz, J.L., Horne, W., Chang, J., Wang, Y., Wilson, G., Chang, S., Weeks, S., Fritz, L.C. and Oltersdorf, T. (1997) Dimerization properties of human BAD. Identification of a BH-3 domain and analysis of its binding to mutant BCL-2 and BCL-XL proteins. J Biol Chem, 272, 30866-30872. Palladino, F., Laroche, T., Gilson, E., Pillus, L. and Gasser, S.M. (1993) The positioning of yeast telomeres depends on SIR3, SIR4, and the integrity of the nuclear membrane. Cold Spring Harb Symp Quant Biol, 58, 733-746. 175 Pan, G., Ni, J., Wei, Y.F., Yu, G., Gentz, R. and Dixit, V.M. (1997a) An antagonist decoy receptor and a death domain-containing receptor for TRAIL. Science, 277, 815-818. Pan, G., O'Rourke, K., Chinnaiyan, A.M., Gentz, R., Ebner, R., Ni, J. and Dixit, V.M. (1997b) The receptor for the cytotoxic ligand TRAIL. Science, 276, 111-113. Park, S.J., Wu, C.H., Gordon, J.D., Zhong, X., Emami, A. and Safa, A.R. (2004) Taxol induces caspase-10-dependent apoptosis. J Biol Chem, 279, 51057-51067. Peart, M.J., Smyth, G.K., van Laar, R.K., Bowtell, D.D., Richon, V.M., Marks, P.A., Holloway, A.J. and Johnstone, R.W. (2005) Identification and functional significance of genes regulated by structurally different histone deacetylase inhibitors. Proc Natl Acad Sci U S A, 102, 3697-3702. Peinado, H., Ballestar, E., Esteller, M. and Cano, A. (2004) Snail mediates E-cadherin repression by the recruitment of the Sin3A/histone deacetylase 1 (HDAC1)/HDAC2 complex. Mol Cell Biol, 24, 306-319. Perlman, H., Pagliari, L.J., Nguyen, N., Bradley, K., Liu, H. and Pope, R.M. (2001) The Fas-FasL death receptor and PI3K pathways independently regulate monocyte homeostasis. Eur J Immunol, 31, 2421-2430. Petros, A.M., Medek, A., Nettesheim, D.G., Kim, D.H., Yoon, H.S., Swift, K., Matayoshi, E.D., Oltersdorf, T. and Fesik, S.W. (2001) Solution structure of the antiapoptotic protein bcl-2. Proc Natl Acad Sci U S A, 98, 3012-3017. Piekarz, R.L., Sackett, D.L. and Bates, S.E. (2007) Histone deacetylase inhibitors and demethylating agents: clinical development of histone deacetylase inhibitors for cancer therapy. Cancer J, 13, 30-39. Pistritto, G., Jost, M., Srinivasula, S.M., Baffa, R., Poyet, J.L., Kari, C., Lazebnik, Y., Rodeck, U. and Alnemri, E.S. (2002) Expression and transcriptional regulation of caspase-14 in simple and complex epithelia. Cell Death Differ, 9, 995-1006. Poulaki, V., Mitsiades, C.S., Kotoula, V., Tseleni-Balafouta, S., Ashkenazi, A., Koutras, D.A. and Mitsiades, N. (2002) Regulation of Apo2L/tumor necrosis factorrelated apoptosis-inducing ligand-induced apoptosis in thyroid carcinoma cells. Am J Pathol, 161, 643-654. 176 Prigent, C. and Dimitrov, S. (2003) Phosphorylation of serine 10 in histone H3, what for? J Cell Sci, 116, 3677-3685. Ravi, R., Bedi, G.C., Engstrom, L.W., Zeng, Q., Mookerjee, B., Gelinas, C., Fuchs, E.J. and Bedi, A. (2001) Regulation of death receptor expression and TRAIL/Apo2L-induced apoptosis by NF-kappaB. Nat Cell Biol, 3, 409-416. Reddy, R.M., Yeow, W.S., Chua, A., Nguyen, D.M., Baras, A., Ziauddin, M.F., Shamimi-Noori, S.M., Maxhimer, J.B., Schrump, D.S. and Nguyen, D.M. (2007) Rapid and profound potentiation of Apo2L/TRAIL-mediated cytotoxicity and apoptosis in thoracic cancer cells by the histone deacetylase inhibitor Trichostatin A: the essential role of the mitochondria-mediated caspase activation cascade. Apoptosis, 12, 55-71. Reed, J.C. (1996) A day in the life of the Bcl-2 protein: does the turnover rate of Bcl-2 serve as a biological clock for cellular lifespan regulation? Leuk Res, 20, 109111. Reed, J.C. (1999) Dysregulation of apoptosis in cancer. J Clin Oncol, 17, 2941-2953. Rengarajan, J., Mittelstadt, P.R., Mages, H.W., Gerth, A.J., Kroczek, R.A., Ashwell, J.D. and Glimcher, L.H. (2000) Sequential involvement of NFAT and Egr transcription factors in FasL regulation. Immunity, 12, 293-300. Renshaw, S.A., Parmar, J.S., Singleton, V., Rowe, S.J., Dockrell, D.H., Dower, S.K., Bingle, C.D., Chilvers, E.R. and Whyte, M.K. (2003) Acceleration of human neutrophil apoptosis by TRAIL. J Immunol, 170, 1027-1033. Rice, J.C. and Allis, C.D. (2001) Histone methylation versus histone acetylation: new insights into epigenetic regulation. Curr Opin Cell Biol, 13, 263-273. Richon, V.M., Sandhoff, T.W., Rifkind, R.A. and Marks, P.A. (2000) Histone deacetylase inhibitor selectively induces p21WAF1 expression and geneassociated histone acetylation. Proc Natl Acad Sci U S A, 97, 10014-10019. Rodriguez-Viciana, P., Marte, B.M., Warne, P.H. and Downward, J. (1996) Phosphatidylinositol 3' kinase: one of the effectors of Ras. Philos Trans R Soc Lond B Biol Sci, 351, 225-231; discussion 231-222. Ronzoni, S., Faretta, M., Ballarini, M., Pelicci, P. and Minucci, S. (2005) New method to detect histone acetylation levels by flow cytometry. Cytometry A, 66, 52-61. 177 Rosato, R.R., Almenara, J.A., Dai, Y. and Grant, S. (2003) Simultaneous activation of the intrinsic and extrinsic pathways by histone deacetylase (HDAC) inhibitors and tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) synergistically induces mitochondrial damage and apoptosis in human leukemia cells. Mol Cancer Ther, 2, 1273-1284. Roth, S.Y., Denu, J.M. and Allis, C.D. (2001) Histone acetyltransferases. Annu Rev Biochem, 70, 81-120. Ruefli, A.A., Ausserlechner, M.J., Bernhard, D., Sutton, V.R., Tainton, K.M., Kofler, R., Smyth, M.J. and Johnstone, R.W. (2001) The histone deacetylase inhibitor and chemotherapeutic agent suberoylanilide hydroxamic acid (SAHA) induces a cell-death pathway characterized by cleavage of Bid and production of reactive oxygen species. Proc Natl Acad Sci U S A, 98, 10833-10838. Sah, N.K., Munshi, A., Kurland, J.F., McDonnell, T.J., Su, B. and Meyn, R.E. (2003) Translation inhibitors sensitize prostate cancer cells to apoptosis induced by tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) by activating c-Jun N-terminal kinase. J Biol Chem, 278, 20593-20602. Sakaguchi, K., Herrera, J.E., Saito, S., Miki, T., Bustin, M., Vassilev, A., Anderson, C.W. and Appella, E. (1998) DNA damage activates p53 through a phosphorylation-acetylation cascade. Genes Dev, 12, 2831-2841. Salvador, L.M., Park, Y., Cottom, J., Maizels, E.T., Jones, J.C., Schillace, R.V., Carr, D.W., Cheung, P., Allis, C.D., Jameson, J.L. and Hunzicker-Dunn, M. (2001) Follicle-stimulating hormone stimulates protein kinase A-mediated histone H3 phosphorylation and acetylation leading to select gene activation in ovarian granulosa cells. J Biol Chem, 276, 40146-40155. Salvesen, G.S. (1999) Programmed cell death and the caspases. Apmis, 107, 73-79. Sanda, T., Okamoto, T., Uchida, Y., Nakagawa, H., Iida, S., Kayukawa, S., Suzuki, T., Oshizawa, T., Suzuki, T., Miyata, N. and Ueda, R. (2007) Proteome analyses of the growth inhibitory effects of NCH-51, a novel histone deacetylase inhibitor, on lymphoid malignant cells. Leukemia, 21, 2344-2353. 178 Santini, V., Gozzini, A. and Ferrari, G. (2007) Histone deacetylase inhibitors: molecular and biological activity as a premise to clinical application. Curr Drug Metab, 8, 383-393. Sarker, M., Ruiz-Ruiz, C. and Lopez-Rivas, A. (2001) Activation of protein kinase C inhibits TRAIL-induced caspases activation, mitochondrial events and apoptosis in a human leukemic T cell line. Cell Death Differ, 8, 172-181. Sattler, M., Liang, H., Nettesheim, D., Meadows, R.P., Harlan, J.E., Eberstadt, M., Yoon, H.S., Shuker, S.B., Chang, B.S., Minn, A.J., Thompson, C.B. and Fesik, S.W. (1997) Structure of Bcl-xL-Bak peptide complex: recognition between regulators of apoptosis. Science, 275, 983-986. Sayers, T.J. and Murphy, W.J. (2006) Combining proteasome inhibition with TNFrelated apoptosis-inducing ligand (Apo2L/TRAIL) for cancer therapy. Cancer Immunol Immunother, 55, 76-84. Scaffidi, C., Fulda, S., Srinivasan, A., Friesen, C., Li, F., Tomaselli, K.J., Debatin, K.M., Krammer, P.H. and Peter, M.E. (1998) Two CD95 (APO-1/Fas) signaling pathways. Embo J, 17, 1675-1687. Scaffidi, C., Schmitz, I., Krammer, P.H. and Peter, M.E. (1999) The role of c-FLIP in modulation of CD95-induced apoptosis. J Biol Chem, 274, 1541-1548. Scorrano, L., Oakes, S.A., Opferman, J.T., Cheng, E.H., Sorcinelli, M.D., Pozzan, T. and Korsmeyer, S.J. (2003) BAX and BAK regulation of endoplasmic reticulum Ca2+: a control point for apoptosis. Science, 300, 135-139. Schmitz, I., Krueger, A., Baumann, S., Schulze-Bergkamen, H., Krammer, P.H. and Kirchhoff, S. (2003) An IL-2-dependent switch between CD95 signaling pathways sensitizes primary human T cells toward CD95-mediated activationinduced cell death. J Immunol, 171, 2930-2936. Sebolt-Leopold, J.S. (2000) Development of anticancer drugs targeting the MAP kinase pathway. Oncogene, 19, 6594-6599. Secchiero, P., Melloni, E., Heikinheimo, M., Mannisto, S., Di Pietro, R., Iacone, A. and Zauli, G. (2004) TRAIL regulates normal erythroid maturation through an ERKdependent pathway. Blood, 103, 517-522. 179 Shankar, S., Chen, X. and Srivastava, R.K. (2005a) Effects of sequential treatments with chemotherapeutic drugs followed by TRAIL on prostate cancer in vitro and in vivo. Prostate, 62, 165-186. Shankar, S., Singh, T.R., Fandy, T.E., Luetrakul, T., Ross, D.D. and Srivastava, R.K. (2005b) Interactive effects of histone deacetylase inhibitors and TRAIL on apoptosis in human leukemia cells: involvement of both death receptor and mitochondrial pathways. Int J Mol Med, 16, 1125-1138. Shankar, S. and Srivastava, R.K. (2004) Enhancement of therapeutic potential of TRAIL by cancer chemotherapy and irradiation: mechanisms and clinical implications. Drug Resist Updat, 7, 139-156. Shimada, K., Nakamura, M., Ishida, E., Kishi, M. and Konishi, N. (2003) Roles of p38and c-jun NH2-terminal kinase-mediated pathways in 2-methoxyestradiolinduced p53 induction and apoptosis. Carcinogenesis, 24, 1067-1075. Shimizu, S., Narita, M. and Tsujimoto, Y. (1999) Bcl-2 family proteins regulate the release of apoptogenic cytochrome c by the mitochondrial channel VDAC. Nature, 399, 483-487. Shiozaki, E.N. and Shi, Y. (2004) Caspases, IAPs and Smac/DIABLO: mechanisms from structural biology. Trends Biochem Sci, 29, 486-494. Siegmund, D., Hausser, A., Peters, N., Scheurich, P. and Wajant, H. (2001) Tumor necrosis factor (TNF) and phorbol ester induce TNF-related apoptosis-inducing ligand (TRAIL) under critical involvement of NF-kappa B essential modulator (NEMO)/IKKgamma. J Biol Chem, 276, 43708-43712. Singh, R.K., Varney, M.L., Buyukberber, S., Ino, K., Ageitos, A.G., Reed, E., Tarantolo, S. and Talmadge, J.E. (1999) Fas-FasL-mediated CD4+ T-cell apoptosis following stem cell transplantation. Cancer Res, 59, 3107-3111. Singh, T.R., Shankar, S., Chen, X., Asim, M. and Srivastava, R.K. (2003) Synergistic interactions of chemotherapeutic drugs and tumor necrosis factor-related apoptosis-inducing ligand/Apo-2 ligand on apoptosis and on regression of breast carcinoma in vivo. Cancer Res, 63, 5390-5400. Slee, E.A., Harte, M.T., Kluck, R.M., Wolf, B.B., Casiano, C.A., Newmeyer, D.D., Wang, H.G., Reed, J.C., Nicholson, D.W., Alnemri, E.S., Green, D.R. and 180 Martin, S.J. (1999) Ordering the cytochrome c-initiated caspase cascade: hierarchical activation of caspases-2, -3, -6, -7, -8, and -10 in a caspase-9dependent manner. J Cell Biol, 144, 281-292. Smyth, M.J., Cretney, E., Takeda, K., Wiltrout, R.H., Sedger, L.M., Kayagaki, N., Yagita, H. and Okumura, K. (2001) Tumor necrosis factor-related apoptosisinducing ligand (TRAIL) contributes to interferon gamma-dependent natural killer cell protection from tumor metastasis. J Exp Med, 193, 661-670. Sobota, R., Szwed, M., Kasza, A., Bugno, M. and Kordula, T. (2000) Parthenolide inhibits activation of signal transducers and activators of transcription (STATs) induced by cytokines of the IL-6 family. Biochem Biophys Res Commun, 267, 329-333. Spotswood, H.T. and Turner, B.M. (2002) An increasingly complex code. J Clin Invest, 110, 577-582. Steele, L.P., Georgopoulos, N.T., Southgate, J., Selby, P.J. and Trejdosiewicz, L.K. (2006) Differential susceptibility to TRAIL of normal versus malignant human urothelial cells. Cell Death Differ, 13, 1564-1576. Stehlik, C., de Martin, R., Kumabashiri, I., Schmid, J.A., Binder, B.R. and Lipp, J. (1998) Nuclear factor (NF)-kappaB-regulated X-chromosome-linked iap gene expression protects endothelial cells from tumor necrosis factor alpha-induced apoptosis. J Exp Med, 188, 211-216. Strahl, B.D. and Allis, C.D. (2000) The language of covalent histone modifications. Nature, 403, 41-45. Strater, J., Walczak, H., Pukrop, T., Von Muller, L., Hasel, C., Kornmann, M., Mertens, T. and Moller, P. (2002) TRAIL and its receptors in the colonic epithelium: a putative role in the defense of viral infections. Gastroenterology, 122, 659-666. Sutherland, K.D., Vaillant, F., Alexander, W.S., Wintermantel, T.M., Forrest, N.C., Holroyd, S.L., McManus, E.J., Schutz, G., Watson, C.J., Chodosh, L.A., Lindeman, G.J. and Visvader, J.E. (2006) c-myc as a mediator of accelerated apoptosis and involution in mammary glands lacking Socs3. Embo J, 25, 58055815. 181 Takeda, K., Smyth, M.J., Cretney, E., Hayakawa, Y., Kayagaki, N., Yagita, H. and Okumura, K. (2002) Critical role for tumor necrosis factor-related apoptosisinducing ligand in immune surveillance against tumor development. J Exp Med, 195, 161-169. Taplick, J., Kurtev, V., Kroboth, K., Posch, M., Lechner, T. and Seiser, C. (2001) Homo-oligomerisation and nuclear localisation of mouse histone deacetylase 1. J Mol Biol, 308, 27-38. Thome, M., Schneider, P., Hofmann, K., Fickenscher, H., Meinl, E., Neipel, F., Mattmann, C., Burns, K., Bodmer, J.L., Schroter, M., Scaffidi, C., Krammer, P.H., Peter, M.E. and Tschopp, J. (1997) Viral FLICE-inhibitory proteins (FLIPs) prevent apoptosis induced by death receptors. Nature, 386, 517-521. Thornberry, N.A. and Lazebnik, Y. (1998) Caspases: enemies within. Science, 281, 1312-1316. Truneh, A., Sharma, S., Silverman, C., Khandekar, S., Reddy, M.P., Deen, K.C., McLaughlin, M.M., Srinivasula, S.M., Livi, G.P., Marshall, L.A., Alnemri, E.S., Williams, W.V. and Doyle, M.L. (2000) Temperature-sensitive differential affinity of TRAIL for its receptors. DR5 is the highest affinity receptor. J Biol Chem, 275, 23319-23325. Tsapis, M., Lieb, M., Manzo, F., Shankaranarayanan, P., Herbrecht, R., Lutz, P. and Gronemeyer, H. (2007) HDAC inhibitors induce apoptosis in glucocorticoidresistant acute lymphatic leukemia cells despite a switch from the extrinsic to the intrinsic death pathway. Int J Biochem Cell Biol, 39, 1500-1509. Tschopp, J., Martinon, F. and Hofmann, K. (1999) Apoptosis: Silencing the death receptors. Curr Biol, 9, R381-384. Utley, R.T. and Cote, J. (2003) The MYST family of histone acetyltransferases. Curr Top Microbiol Immunol, 274, 203-236. van Gurp, M., Festjens, N., van Loo, G., Saelens, X. and Vandenabeele, P. (2003) Mitochondrial intermembrane proteins in cell death. Biochem Biophys Res Commun, 304, 487-497. VanOosten, R.L., Moore, J.M., Karacay, B. and Griffith, T.S. (2005) Histone deacetylase inhibitors modulate renal cell carcinoma sensitivity to TRAIL/Apo- 182 2L-induced apoptosis by enhancing TRAIL-R2 expression. Cancer Biol Ther, 4, 1104-1112. Varfolomeev, E.E., Boldin, M.P., Goncharov, T.M. and Wallach, D. (1996) A potential mechanism of "cross-talk" between the p55 tumor necrosis factor receptor and Fas/APO1: proteins binding to the death domains of the two receptors also bind to each other. J Exp Med, 183, 1271-1275. Verdin, E., Dequiedt, F. and Kasler, H.G. (2003) Class II histone deacetylases: versatile regulators. Trends Genet, 19, 286-293. Vulfovich, M. and Saba, N. (2005) Technology evaluation: mapatumumab, Human Genome Sciences/GlaxoSmithKline/Takeda. Curr Opin Mol Ther, 7, 502-510. Wade, P.A. (2001) Transcriptional control at regulatory checkpoints by histone deacetylases: molecular connections between cancer and chromatin. Hum Mol Genet, 10, 693-698. Wahl, C., Liptay, S., Adler, G. and Schmid, R.M. (1998) Sulfasalazine: a potent and specific inhibitor of nuclear factor kappa B. J Clin Invest, 101, 1163-1174. Walczak, H., Degli-Esposti, M.A., Johnson, R.S., Smolak, P.J., Waugh, J.Y., Boiani, N., Timour, M.S., Gerhart, M.J., Schooley, K.A., Smith, C.A., Goodwin, R.G. and Rauch, C.T. (1997) TRAIL-R2: a novel apoptosis-mediating receptor for TRAIL. Embo J, 16, 5386-5397. Wallace, D.M., Donovan, M. and Cotter, T.G. (2006) Histone deacetylase activity regulates apaf-1 and caspase 3 expression in the developing mouse retina. Invest Ophthalmol Vis Sci, 47, 2765-2772. Wang, C.Y., Mayo, M.W., Korneluk, R.G., Goeddel, D.V. and Baldwin, A.S., Jr. (1998a) NF-kappaB antiapoptosis: induction of TRAF1 and TRAF2 and c-IAP1 and c-IAP2 to suppress caspase-8 activation. Science, 281, 1680-1683. Wang, K., Gross, A., Waksman, G. and Korsmeyer, S.J. (1998b) Mutagenesis of the BH3 domain of BAX identifies residues critical for dimerization and killing. Mol Cell Biol, 18, 6083-6089. Wang, K.K., Liu, N., Radulovich, N., Wigle, D.A., Johnston, M.R., Shepherd, F.A., Minden, M.D. and Tsao, M.S. (2002a) Novel candidate tumor marker genes for lung adenocarcinoma. Oncogene, 21, 7598-7604. 183 Wang, M.J., Liu, S., Liu, Y. and Zheng, D. (2007) Actinomycin D enhances TRAILinduced caspase-dependent and -independent apoptosis in SH-SY5Y neuroblastoma cells. Neurosci Res, 59, 40-46. Wang, Q., Ji, Y., Wang, X. and Evers, B.M. (2000a) Isolation and molecular characterization of the 5'-upstream region of the human TRAIL gene. Biochem Biophys Res Commun, 276, 466-471. Wang, T.H., Wang, H.S. and Soong, Y.K. (2000b) Paclitaxel-induced cell death: where the cell cycle and apoptosis come together. Cancer, 88, 2619-2628. Wang, X., Yang, C., Chai, J., Shi, Y. and Xue, D. (2002b) Mechanisms of AIFmediated apoptotic DNA degradation in Caenorhabditis elegans. Science, 298, 1587-1592. Weber, M., Davies, J.J., Wittig, D., Oakeley, E.J., Haase, M., Lam, W.L. and Schubeler, D. (2005) Chromosome-wide and promoter-specific analyses identify sites of differential DNA methylation in normal and transformed human cells. Nat Genet, 37, 853-862. Wei, M.C., Lindsten, T., Mootha, V.K., Weiler, S., Gross, A., Ashiya, M., Thompson, C.B. and Korsmeyer, S.J. (2000) tBID, a membrane-targeted death ligand, oligomerizes BAK to release cytochrome c. Genes Dev, 14, 2060-2071. Wesselborg, S., Janssen, O. and Kabelitz, D. (1993) Induction of activation-driven death (apoptosis) in activated but not resting peripheral blood T cells. J Immunol, 150, 4338-4345. Widschwendter, M. and Jones, P.A. (2002) The potential prognostic, predictive, and therapeutic values of DNA methylation in cancer. Commentary re: J. Kwong et al., Promoter hypermethylation of multiple genes in nasopharyngeal carcinoma. Clin. Cancer Res., 8: 131-137, 2002, and H-Z. Zou et al., Detection of aberrant p16 methylation in the serum of colorectal cancer patients. Clin. Cancer Res., 8: 188-191, 2002. Clin Cancer Res, 8, 17-21. Wiley, S.R., Schooley, K., Smolak, P.J., Din, W.S., Huang, C.P., Nicholl, J.K., Sutherland, G.R., Smith, T.D., Rauch, C., Smith, C.A. and et al. (1995) Identification and characterization of a new member of the TNF family that induces apoptosis. Immunity, 3, 673-682. 184 Wolf, B.B. and Green, D.R. (1999) Suicidal tendencies: apoptotic cell death by caspase family proteinases. J Biol Chem, 274, 20049-20052. Wright, C.W. and Duckett, C.S. (2005) Reawakening the cellular death program in neoplasia through the therapeutic blockade of IAP function. J Clin Invest, 115, 2673-2678. Wyllie, A.H., Kerr, J.F. and Currie, A.R. (1980) Cell death: the significance of apoptosis. Int Rev Cytol, 68, 251-306. Xiao, C., Yang, B.F., Song, J.H., Schulman, H., Li, L. and Hao, C. (2005) Inhibition of CaMKII-mediated c-FLIP expression sensitizes malignant melanoma cells to TRAIL-induced apoptosis. Exp Cell Res, 304, 244-255. Yang, J., Liu, X., Bhalla, K., Kim, C.N., Ibrado, A.M., Cai, J., Peng, T.I., Jones, D.P. and Wang, X. (1997) Prevention of apoptosis by Bcl-2: release of cytochrome c from mitochondria blocked. Science, 275, 1129-1132. Yang, R., Xu, D., Zhang, A. and Gruber, A. (2001) Immature dendritic cells kill ovarian carcinoma cells by a FAS/FASL pathway, enabling them to sensitize tumorspecific CTLs. Int J Cancer, 94, 407-413. Yang, S., Thor, A.D., Edgerton, S. and Yang, X. (2006) Caspase-3 mediated feedback activation of apical caspases in doxorubicin and TNF-alpha induced apoptosis. Apoptosis, 11, 1987-1997. Yang, W.M., Tsai, S.C., Wen, Y.D., Fejer, G. and Seto, E. (2002) Functional domains of histone deacetylase-3. J Biol Chem, 277, 9447-9454. Yang, Y.L. and Li, X.M. (2000) The IAP family: endogenous caspase inhibitors with multiple biological activities. Cell Res, 10, 169-177. Yao, J., Duan, L., Fan, M. and Wu, X. (2006) NF-kappaB signaling pathway is involved in growth inhibition, G2/M arrest and apoptosis induced by Trichostatin A in human tongue carcinoma cells. Pharmacol Res, 54, 406-413. Yoshida, M., Kijima, M., Akita, M. and Beppu, T. (1990) Potent and specific inhibition of mammalian histone deacetylase both in vivo and in vitro by trichostatin A. J Biol Chem, 265, 17174-17179. 185 Zauli, G., Rimondi, E., Nicolin, V., Melloni, E., Celeghini, C. and Secchiero, P. (2004) TNF-related apoptosis-inducing ligand (TRAIL) blocks osteoclastic differentiation induced by RANKL plus M-CSF. Blood, 104, 2044-2050. Zauli, G. and Secchiero, P. (2006) The role of the TRAIL/TRAIL receptors system in hematopoiesis and endothelial cell biology. Cytokine Growth Factor Rev, 17, 245-257. Zerafa, N., Westwood, J.A., Cretney, E., Mitchell, S., Waring, P., Iezzi, M. and Smyth, M.J. (2005) Cutting edge: TRAIL deficiency accelerates hematological malignancies. J Immunol, 175, 5586-5590. Zhang, J.Q., Okumura, C., McCarty, T., Shin, M.S., Mukhopadhyay, P., Hori, M., Torrey, T.A., Naghashfar, Z., Zhou, J.X., Lee, C.H., Roopenian, D.C., Morse, H.C., 3rd and Davidson, W.F. (2004) Evidence for selective transformation of autoreactive immature plasma cells in mice deficient in Fasl. J Exp Med, 200, 1467-1478. Zhang, X.D., Franco, A.V., Nguyen, T., Gray, C.P. and Hersey, P. (2000) Differential localization and regulation of death and decoy receptors for TNF-related apoptosis-inducing ligand (TRAIL) in human melanoma cells. J Immunol, 164, 3961-3970. Zhang, X.D., Gillespie, S.K., Borrow, J.M. and Hersey, P. (2003) The histone deacetylase inhibitor suberic bishydroxamate: a potential sensitizer of melanoma to TNF-related apoptosis-inducing ligand (TRAIL) induced apoptosis. Biochem Pharmacol, 66, 1537-1545. Zhang, Y., Ng, H.H., Erdjument-Bromage, H., Tempst, P., Bird, A. and Reinberg, D. (1999) Analysis of the NuRD subunits reveals a histone deacetylase core complex and a connection with DNA methylation. Genes Dev, 13, 1924-1935. Zhou, L.L., Zhou, L.Y., Luo, K.Q. and Chang, D.C. (2005) Smac/DIABLO and cytochrome c are released from mitochondria through a similar mechanism during UV-induced apoptosis. Apoptosis, 10, 289-299. Zinkel, S., Gross, A. and Yang, E. (2006) BCL2 family in DNA damage and cell cycle control. Cell Death Differ, 13, 1351-1359. 186 Zong, W.X., Lindsten, T., Ross, A.J., MacGregor, G.R. and Thompson, C.B. (2001) BH3-only proteins that bind pro-survival Bcl-2 family members fail to induce apoptosis in the absence of Bax and Bak. Genes Dev, 15, 1481-1486. Zou, H., Li, Y., Liu, X. and Wang, X. (1999) An APAF-1.cytochrome c multimeric complex is a functional apoptosome that activates procaspase-9. J Biol Chem, 274, 11549-11556. 187 188 VIII. PUBLICACIONES 189 190 Molecular Immunology 44 (2007) 2587–2597 Regulation of the resistance to TRAIL-induced apoptosis in human primary T lymphocytes: Role of NF-B inhibition Jorge Carlos Morales, Marı́a José Ruiz-Magaña, Carmen Ruiz-Ruiz ∗ Departamento de Bioquı́mica y Biologı́a Molecular 3 e Inmunologı́a, Facultad de Medicina, Universidad de Granada, Avda. de Madrid 11, 18012 Granada, Spain Received 17 October 2006; received in revised form 13 December 2006; accepted 14 December 2006 Available online 25 January 2007 Abstract Several combined strategies have been recently proposed to overcome the resistance to tumor necrosis factor (TNF)-related apoptosis-inducing ligand (TRAIL) showed by some tumor cells, thus improving the use of this death ligand in antitumor therapy. However, the molecular mechanisms of the tumor selective activity of TRAIL are not completely understood and hence the effects of the combined therapy on normal cells are unknown. Here, we have studied the resistance of primary T lymphocytes to TRAIL-mediated apoptosis. No significant differences were found in the expression of proteins involved in TRAIL-mediated apoptosis between resting and activated T cells. The low expression of death receptors TRAIL-R1/-R2 as well as the high levels of the antiapoptotic proteins TRAIL-R4 and cellular Fas-associated death domain-like IL-1-converting enzyme-inhibitory protein (c-FLIP) may explain the lack of caspase-8 activation observed upon TRAIL treatment in both cell types. We have also analyzed the effect of different sensitizing agents such as genotoxic drugs, phosphatidylinositol-3 kinase (PI3K) inhibitors, proteasome inhibitors, microtubule depolymerizing agents, histone deacetilase inhibitors (HDACi), and NF-B inhibitors. Although some of them induced T cell death, only NF-B inhibitors sensitized activated T cells to TRAIL-induced apoptosis, maybe through the regulation of the antiapoptotic proteins TRAILR4, c-FLIPS and members of the inhibitors of apoptosis proteins (IAP) family. These results question the safety of the combined treatments with TRAIL and NF-B inhibitors against tumors. © 2007 Elsevier Ltd. All rights reserved. Keywords: Apoptosis; TRAIL; T lymphocytes; Antitumor therapy 1. Introduction TNF-related apoptosis-inducing ligand (TRAIL/APO-2L) and CD95 ligand (CD95L or FasL/APO-1L) are type II transmembrane proteins that belong to the tumour necrosis factor (TNF) superfamily. They are potent inducers of apoptosis upon binding to their respective death domain-containing receptors, also known as death receptors (Suda et al., 1993; Wiley et al., 1995). Cytoplasmic death domains serve to recruit intracellular adapter molecules such as Fas-associated death domain protein (FADD) that in turn engage procaspase-8, thereby forming the death-inducing signalling complex (DISC). Caspase-8 is activated in the DISC allowing the initiation of a cascade of events that leads to apoptotic cell death (Chinnaiyan et al., 1995; Sprick et al., 2000). Such direct connection to the cell’s death machin- ∗ Corresponding author. Tel.: +34 958 24 35 22; fax: +34 958 24 90 15. E-mail address: mcarmenr@ugr.es (C. Ruiz-Ruiz). 0161-5890/$ – see front matter © 2007 Elsevier Ltd. All rights reserved. doi:10.1016/j.molimm.2006.12.015 ery suggests a therapeutic potential for CD95L and TRAIL in cancer treatment. Concerning CD95L, although it can efficiently induce apoptosis in a variety of tumour cells, its administration may not be a useful strategy as it is associated with severe liver toxicity (Ogasawara et al., 1993). In contrast, TRAIL has no systemic toxicity in preclinical studies with mice and non-human primates when administered at doses that inhibit the growth of breast and colon cancer xenografts (Walczak et al., 1999). TRAIL can bind to four specific type I membrane receptors. TRAIL-R1/DR4 and TRAIL-R2/DR5 are death receptors, as they contain the intracellular death domain essential for transmitting the apoptotic signals (MacFarlane et al., 1997; Pan et al., 1997b; Walczak et al., 1997). TRAIL-R3/DcR1 and TRAILR4/DcR2 are known as antiapoptotic or decoy receptors because they do not contain an intact death domain. They act as nonfunctional binding partners for TRAIL, thereby attenuating its apoptotic activity (Degli-Esposti et al., 1997; MacFarlane et al., 1997; Pan et al., 1997a). TRAIL receptors appear to be ubiquitously expressed with transcripts detected in most human 2588 J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 tissues as well as in tumor cell lines of different lineages. Moreover, unlike CD95L, TRAIL is expressed constitutively and it is widely distributed in normal organs and tissues (Wiley et al., 1995), which is in relation with its lack of toxicity to normal cells. Even though a recombinant form of human TRAIL has been shown to cause apoptosis in human hepatocytes (Jo et al., 2000), more recent data have demonstrated that different recombinant versions of TRAIL vary considerably in toxicity towards normal human cells, but all of them maintain their antitumor properties (Lawrence et al., 2001). To date, the mechanisms of TRAIL resistance in normal cells and some tumor cells are not completely understood. As regulation of TRAIL-mediated apoptosis is exerted at several stages along the signalling pathway, resistance can occur by different means. Initially, the degree of TRAIL sensitivity or resistance was proposed to be dependent on the levels of expression, localization and function of death and decoy receptors (Sheridan et al., 1997; Zhang et al., 2000). However, a correlation between cell survival and the expression of decoy or death receptors has not been conclusively demonstrated. On the other hand, several intracellular molecules can block the apoptotic effect of TRAIL. The cellular Fas-associated death domain-like IL-1converting enzyme-inhibitory protein (c-FLIP) modulates the apoptotic pathway right at the beginning because it competes with caspase-8 for binding to FADD (MacFarlane et al., 2002; Siegmund et al., 2002). Bcl-2 and Bcl-XL impede the activation of the mitochondrial pathway thereby blocking the release of cytochrome c from mitochondria to cytosol (Ruiz de Almodovar et al., 2001). The inhibitors of apoptotic proteins XIAP, c-IAP1 and c-IAP2 act by inhibiting active caspases (Cummins et al., 2004; Li et al., 2004). The antiapoptotic signals induced by NFB (Ravi et al., 2001), protein kinase B (PKB)/Akt (Whang et al., 2004), protein kinase C (PKC) or mitogen-activated protein kinase (MAPK) (Ortiz-Ferron et al., 2006; Sarker et al., 2001) have also been involved in the resistance to TRAIL-induced apoptosis of different types of cells. Despite the controversial results about the physiological role of TRAIL in vivo, several studies point to a role for TRAIL in the suppression of autoimmune diseases and the immune surveillance of developing and metastatic tumors (Cretney et al., 2006). In this respect, it has been reported that TRAIL is implicated in the antitumor cytotoxicity of dendritic cells, monocytes, NK cells and even B cells (Fanger et al., 1999; Griffith et al., 1999; Kemp et al., 2004; Takeda et al., 2001). Moreover, TRAIL is induced on the surface of human T lymphocytes upon activation through the T cell antigen receptor (TCR) or treatment with type I interferons, mediating an important part of the cytotoxic activity of T cells (Dorothee et al., 2002; Kayagaki et al., 1999). The resistance of immune cells to TRAIL-induced apoptosis is essential for them to act as mediators of antitumoral response. In this work, we have investigated the mechanisms of TRAIL resistance in resting and activated primary human T lymphocytes. We have analyzed the expression of several proteins involved in the TRAIL signalling pathway, such as TRAIL receptors and components of the DISC, in both cell types. Furthermore, we have studied their susceptibility to TRAIL upon combination with agents that regulate TRAIL sensitivity in tumor cells. Our results show that NF-B inhibitors are able to sensitize primary activated T lymphocytes to TRAIL-mediated apoptosis and regulate the expression of several antiapoptotic proteins in these cells. 2. Materials and methods 2.1. Reagents and antibodies Human recombinant TRAIL was prepared as described previously (MacFarlane et al., 1997). Phytohemagglutinin, doxorubicin, LY294002, nocodazole, valproic acid, BAY 117085, sulfasalazine and mouse anti-␣-tubulin mAb were from Sigma–Aldrich (St. Louis, MO). Velcade (bortezomib) was from Janssen-Cilag SA (Madrid, Spain). Z-VAD-FMK was provided by Bachem (Bubendorf, Switzerland). Parthenolide, CAPE, anti-cFLIP monoclonal antibody NF6 and mouse anti-human TRAIL receptor antibodies for flow cytometry studies were purchased from Alexis Biochemicals (San Diego, CA). Mouse anti-human CD28 and polyclonal anti-human NF-B p50 antibodies were from eBioscience (San Diego, CA). Anti-human caspase-8 monoclonal antibody was purchased from Cell Diagnostica (Münster, Germany). Mouse anti-FADD was obtained from Transduction Laboratories (Lexington, KY). Monoclonal antibodies against poly(ADP-ribose) polymerase (PARP), XIAP and c-IAP2 were obtained from BD Biosciences (San Jose, CA). Mouse anti-p65 was obtained from Santa Cruz Biotechnology Inc. (Santa Cruz, CA). Monoclonal antibody anti-Bcl-2 was from Dako (Glostrup, Denmark). 2.2. Cells and cell culture Blood samples were obtained from healthy donors by informed consent and collected into citrate tubes. Peripheral blood mononuclear cells (PBMC) were prepared by Ficoll– Histopaque density gradient centrifugation (Sigma–Aldrich) and adherent monocytes were depleted by culture on plastic dishes for 1 h at 37 ◦ C. Peripheral blood T lymphocytes were then isolated by negative selection with an indirect magnetic labelling system consisting of a cocktail of biotin-conjugated antibodies against CD14, CD16, CD19, CD36, CD56, CD123, and CD235a (Human Pan T Cells Isolation Kit II, Miltenyi Biotec, GmbH). Purity of T cells was >95% CD3+ as determined by flow cytometry. Purified resting T lymphocytes were resuspended in RPMI 1640 medium (BioWhittaker Inc.) containing 10% fetal bovine serum, 1 mM l-glutamine, 100 U/ml penicillin and 100 g/ml streptomycin and incubated as indicated at 37 ◦ C in a humidified 5% CO2 , 95% air incubator. For activation, resting T cells were cultured at 2 × 106 cells/ml with 5 g/ml PHA and 1 g/ml anti-CD28 for 20 h. After washing, cells were incubated in complete medium supplemented with 25 U/ml IL-2 for an additional 5 days. The human cell lines Jurkat, CEM and SKBR3 were all maintained in culture in RPMI 1640 medium with 10% fetal bovine serum, l-glutamine, penicillin and streptomycin at 37 ◦ C in a humidified 5% CO2 , 95% air incubator. J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 2.3. Determination of apoptotic cells Hypodiploid apoptotic cells were detected by flow cytometry according to published procedures (Gong et al., 1994). Briefly, cells were washed with phosphate-buffered saline (PBS), fixed in cold 70% ethanol, and then stained with propidium iodide while treating with RNase. Quantitative analysis of sub-G1 cells was carried out in a FACScan cytometer using the Cell Quest software (BD Biosciences). 2.4. Cytofluorometric analysis of TRAIL receptors To detect TRAIL receptors at the cell surface, control or treated cells were incubated with primary antibodies (5 g/ml) at 4 ◦ C for 30 min. After washing with PBS to remove unbound primary antibody, cells were incubated with goat anti-mouse fluorescein isothiocyanate-conjugated antibody (Caltag Laboratories) for 30 min at 4 ◦ C. Cells were then washed again, resuspended in PBS and analyzed in a FACScan flow cytometer. 2.5. Immunoblot detection of proteins For detection of cytosolic proteins, cells were lysed in icecold lysis buffer (150 mM NaCl, 50 mM Tris–Cl, 1% NP-40) for 30 min. Proteins of cytosolic supernatants were resolved on 10% SDS-PAGE gels and detected as reported previously (Ruiz-Ruiz and Lopez-Rivas, 1999). For nuclear proteins extractions, cells were lysed with 300 l of hypotonic buffer (10 mM HEPES, pH 7.6, 10 mM KCl, 0.1 mM EDTA, 0.1 mM EGTA, 1 mM DTT, 0.5 mM phenylmethylsulfonyl fluoride (PMSF), 10 mM Na2 MoO4 , and protease inhibitors) containing 0.6% of Nonidet P-40. Nuclei were then centrifuged and incubated in 50 l of high saltcontaining buffer (20 mM HEPES, pH 7.6, 0.4 M KCl, 1 mM EDTA, 1 mM EGTA, 1 mM DTT, 0.5 mM PMSF, 10 mM Na2 MoO4 , and protease inhibitors) for 30 min on a rocking platform at 4 ◦ C. Nuclei were centrifuged at 13,000 × g for 10 min, to get the supernatants containing the nuclear extracts. Proteins were then separated through 10% SDS-PAGE gel and detected as described (Ruiz-Ruiz and Lopez-Rivas, 1999). 2.6. Reverse transcription (RT)-PCR Total RNA was extracted from T lymphocytes with Trizol Reagent (Invitrogen) as recommended by the supplier. cDNAs were synthesized from 3 g of total RNA by using M-MLV reverse transcriptase (Invitrogen) and oligo(dT) primer in a total volume of 20 l. Reverse transcription was performed at 37◦ for 50 min followed by 15 min at 70◦ for inactivation. PCR reactions were performed with 0, 1–2 l cDNA (depending on the gene) in a 50-l volume and using the following primer pairs (fragments size indicated in brackets): TRAIL-R1 (506 bp), forward 5 -CTGAGCAACGCAGACTCGCTGTCCAC-3 and reverse 5 -TCCAAGGACACGGCAGAGCCTGTGCCAT-3 ; TRAIL-R2 (502 bp), forward 5 -GCCTCATGGACAATGAGATAAAGGTGGCT-3 and reverse 5 -CCAAATCTCAAAGTACGCACAAACGG-3 ; TRAIL-R3 (612 bp), forward 5 -GA- 2589 AGAATTTGGTGCCAATGCCACTG-3 and reverse 5 -CTCTTGGACTTGGCTGGGAGATGTG-3 ; TRAIL-R4 (453 bp), forward 5 -CTTTTCCGGCGGCGTTCATGTCCTTC-3 and reverse 5 -GTTTCTTCCAGGCTGCTTCCCTTTGTAG-3 ; c-FLIPL (227 bp), forward 5 -AATTCAAGGCTCAGAAGCGA-3 and reverse 5 -GGCAGAAACTCTGCTGTTCC-3 ; cFLIPS (100 bp), forward 5 -AATGTTCTCCAAGCAGCAATCC-3 and reverse 5 -CCAAGAATTTTCAGATCAGGACAAT3 ; Bcl-2 (367 bp), forward 5 -AGATGTCCAGCCAGCTGCACCTGAC-3 and reverse 5 -AGATAGGCACCAGGGTGAGCAAGCT-3 ; Bcl-x (257 bp), forward 5 -CATGGCAGCAGTAAAGCAAGC-3 and reverse 5 -CTGCGATCCGACTCACCAATAC-3 ; Mcl-1 (211 bp), forward 5 -CTTAGTTGATATTTTGGGCTTGGG-3 and reverse 5 -AGAAGTCAAAAAGTAGTCACTGGG-3 ; XIAP (858 bp), forward 5 -GGCCATCTGAGACACATGCAG-3 and reverse 5 -GCATTCACTAGATCTGCAACC-3 ; c-IAP1 (171 bp), forward 5 -CCAGTTCTTTTCTACACTATAAT-3 and reverse 5 -CTTAATCTGTTTATTTACAAGGG-3 ; c-IAP2 (150 bp), forward 5 -CAACATGGAGATTCGAAATCC-3 and reverse 5 -CACATCACTCTTCTGTGAAGGG-3 ; -actin (661 bp), forward 5 -TGACGGGGTCACCCACACTGTGCCCATCTA-3 and reverse 5 -CTAGAAGCATTTGCGGTGGACGATGGAGGG-3 . PCR cycle conditions were as follows: 1 min at 95 ◦ C, 1 min at 55 ◦ C and 1 min at 72 ◦ C for TRAIL-R1, TRAIL-R2 and TRAIL-R3; 1 min at 95 ◦ C, 1 min at 60 ◦ C, and 1 min at 72 ◦ C for TRAILR4, Bcl-2 and -actin; 40 seg at 95 ◦ C, 40 seg at 60 ◦ C, and 40 seg at 72 ◦ C for c-FLIPS and c-FLIPL ; 1 min at 95 ◦ C, 1 min at 57 ◦ C, and 1 min at 72 ◦ C for Bcl-x, Mcl-1, XIAP, c-IAP1 and c-IAP2. The number of cycles varied between 30 and 35, depending on the gene analyzed. The products were resolved on a 1% agarose gel and visualized with ethidium bromide. 3. Results 3.1. Resistance of resting and activated primary human T lymphocytes to TRAIL-mediated apoptosis The resistance of different peripheral blood cell subpopulations to TRAIL-mediated cytotoxicity has recently been reported (Hasegawa et al., 2004; Mirandola et al., 2004). To further characterize the effect of TRAIL on primary human T lymphocytes we first analyzed the induction of apoptosis on resting and activated T cells from 10 different healthy donors after treatment for 24 h with 250 ng/ml recombinant TRAIL. As shown in Fig. 1A, all of the resting and activated primary T cell samples were resistant to TRAIL-induced apoptosis as assessed by sub-G1 DNA content. Moreover, we did not observe any effect on primary T cell viability after incubation either for 2 days or with higher doses of TRAIL (data not shown). In this set of experiments, we used TRAIL-sensitive Jurkat cells as a positive control (Fig. 1). We also examined the activation of apical caspase-8 as it is the first biochemical event that occurs upon ligation of TRAIL receptors at the cell surface. To this end, we analyzed the processing of the procaspase into the 43–41 kDa intermediate proteolytic fragments corresponding to the cleavage of 2590 J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 Fig. 1. Primary T cells are resistant to TRAIL-induced apoptosis. (A) Resting and activated T cells from 10 different healthy donors were treated for 24 h with 250 ng/ml recombinant TRAIL. Apoptosis was assessed by analysis of the percentage of cells with sub-G1 content. (B) Resting and activated T lymphocytes were incubated for 15 h with 250 ng/ml TRAIL and caspase-8 activation was determined by Western blot as described in Section 2. As a positive control, apoptosis (A) and caspase-8 activation (B) were determined in Jurkat T cells after treatment with 100 ng/ml TRAIL. (C) Cell surface TRAIL receptors expression was analyzed by flow cytometry in resting and activated T lymphocytes (shaded peaks). Solid lines represent background fluorescence with secondary antibody alone. (D) Expression of FADD and c-FLIP was determined by Western blot in Jurkat, resting and activated T cells. ␣-Tubulin was used as a control of loaded protein. In B–D, results shown are representative of at least three different donors. procaspase-8a and -8b. Data presented in Fig. 1B shows that TRAIL was not able to induce activation of caspase-8 in either resting or activated primary T lymphocytes, which indicates an apical block of the TRAIL signalling pathway in both types of cells. These results led us to examine the expression of proteins known to participate in the formation of the DISC and thus in the activation of caspase-8. Firstly, we analyzed the surface expression of TRAIL receptors in resting and activated T lymphocytes. In agreement with previous data (Hasegawa et al., 2004), we observed that the expression of death receptors TRAIL-R1 and -R2, as well as decoy receptor TRAIL-R3, was barely detectable in all analyzed resting T cells, with very weak differences among donors. In contrast, when we examined the expression of TRAIL-R4, two subsets of cells with different fluorescence intensities were detected (Fig. 1C). The highly positive TRAIL-R4 peak seems to correspond to the CD8+ T cell subpopulation (Hasegawa et al., 2004; Mirandola et al., 2004) while the less fluorescent peak, which varies from very low to moderate intensity among different donors, corresponds to CD4+ T cells. T cell activation did not change significantly the expression of TRAIL receptors except for TRAIL-R4 (Fig. 1C). For this receptor, we found a slight enhancement in the fluorescence intensity of the two subpopulations, as well as an increase in the percentage of TRAIL-R4 highly positive cells, which is likely due to the observed increment of the CD8+ /CD4+ ratio within the population of activated T cells compared to the resting one (data not shown). Regarding the expression of intracellular proteins involved in the activation of caspase-8, we observed that the levels of FADD in resting and activated T cells were similar to that found in the TRAIL-sensitive Jurkat cell line (Fig. 1D). However, it is also known that human T lymphocytes express the inhibitory protein c-FLIP (Bosque et al., 2005; Schmitz et al., 2003). When we compared the expression of this protein between Jurkat and primary T cells we detected a much higher level of the long isoform, c-FLIPL , in both resting and activated T lymphocytes than in Jurkat cells. The expression of the c-FLIPS isoform was low in primary T cells but barely perceptible in Jurkat cells (Fig. 1D). Taken together, these results suggest that several factors may be responsible for the resistance of resting and activated T lymphocytes to TRAIL-induced apoptosis. 3.2. Effect of different modulators of TRAIL sensitivity on the resistance of human primary T lymphocytes Deregulated expression of TRAIL receptors and intracellular antiapoptotic proteins seem to cause resistance to TRAILinduced apoptosis in several types of tumor cells, which reduces the effectiveness of TRAIL in cancer therapy. However, most of these TRAIL-resistant cancer cells can be sensitized by several therapeutic approaches such as conventional chemotherapeutic drugs, histone deacetylase inhibitors (HDACi), inhibitors of survival pathways or proteasome inhibitors (Inoue et al., 2004; Kandasamy and Srivastava, 2002; Sayers and Murphy, 2006; Shankar and Srivastava, 2004; Singh et al., 2003). We analyzed the effect of some agents belonging to these functional groups on the resistance of primary T lymphocytes to TRAILmediated apoptosis. As shown in Fig. 2, we specifically tested the genotoxic drug doxorubicin, the PI3K inhibitor LY294002, the HDACi valproic acid (VPA), the microtubule depolymerizing agent nocodazole and the proteasome inhibitor Velcade (bortezomib). Working concentrations of these agents were chosen according to previous reports and on the basis of experiments with the T-lymphoblastic leukemic CEM cell line and the breast J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 2591 Fig. 2. Primary T lymphocytes remain resistant to TRAIL upon treatment with different sensitizing agents. Resting and activated T lymphocytes were preincubated with (A) 100 ng/ml doxorubicin (DXR), (B) 10 M LY 294002 or (E) 50 nM bortezomib (BZ) for 1 h, or with (C) 1mM valproic acid (VPA) or (D) 400 ng/ml nocodazole (NZ) for 7 h. After preincubation, cells were treated with or without 250 ng/ml recombinant TRAIL for 24 h. CEM (A–C) and SKBr3 (D and E) cells were preincubated in the same conditions before treatment with 50 and 250 ng/ml recombinant TRAIL, respectively, for 20 h. The percentage of sub-G1 apoptotic cells was determined by flow cytometry. Error bars show S.D. from three independent experiments. * p < 0.05; ** p < 0.01; *** p < 0.001 by unpaired Student’s t-test (two-tailed). cancer SKBr3 cell line, which show a moderate sensitivity and a high resistance to TRAIL, respectively. Pretreatment with either doxorubicin, LY294002 or VPA increased the susceptibility of CEM cells to TRAIL while bortezomib and nocodazole clearly sensitized TRAIL-resistant SKBr3 cells. However, we did not find TRAIL sensitization in either resting or activated T cells in response to pretreatment with any of the indicated agents. Interestingly, doxorubicin, nocodazole and bortezomib showed a remarkable toxicity against activated T cells, whereas only bortezomib was slightly toxic to resting ones (Fig. 2A, D and E). 3.3. NF-κB inhibitors sensitize human activated T lymphocytes to TRAIL-induced apoptosis In addition to the above agents, we also analyzed the effect of the NF-B inhibitor BAY 11-7085. Data presented in Fig. 3A indicates that the response of resting T lymphocytes to this inhibitor varies among different donors. BAY 11-7085 alone was toxic to varying degrees to 9/10 samples. Surprisingly, while it caused a significant sensitization to TRAIL-induced apoptosis in 4/10 samples, in five different ones the percentage of apoptotic cells decreased following treatment with both BAY 11-7085 and TRAIL compared to cells treated with the NFB inhibitor alone. In the case of activated T lymphocytes, a variable toxicity was also found in response to BAY 11-7085 alone (Fig. 3B). However, we observed that preincubation with the NF-B inhibitor induced a different but substantial sensitization to TRAIL-mediated apoptosis in all donors analyzed, except for one in which the inhibitor alone showed an extreme toxicity. To further establish that the death process promoted by the NF-B inhibitor was apoptosis, resting and activated T lymphocytes from donors previously proved to be sensitized to TRAIL (Fig. 3A and B, respectively) were preincubated with the general caspase inhibitor Z-VAD-FMK before treatment with BAY 11-7085 and TRAIL. Representative samples in Fig. 3C and D show that not only TRAIL-mediated cell death but also the cytotoxic effect of BAY 11-7085 were almost completely inhibited by Z-VAD. It is worth mentioning that Z-VAD was 2592 J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 Fig. 3. BAY 11-7085 sensitizes activated T lymphocytes to TRAIL-induced apoptosis. Resting (A and C) and activated (B and D) T lymphocytes from 10 different healthy donors (A and B) and one representative donor (C and D) were preincubated for 1 h with 2.5 M BAY 11-7085 and then treated with or without 250 ng/ml TRAIL for 24 h. In C and D, preincubation was carried out in the presence or in the absence of 50 M Z-VAD. Apoptosis was determined by flow cytometry. Results in C and D are representative of three different experiments. (E) Activation of caspase-8 was determined by Western blot in activated T lymphocytes incubated with or without 250 ng/ml TRAIL for 20 h after pretreatment for 1 h with 2.5 M BAY 11-7085. ␣-Tubulin was used as a control of loaded protein. Data shown are representative of three different experiments. not able to inhibit cell death in the samples in which BAY 11-7085 alone was extremely toxic (data not shown). This suggests that BAY 11-7085 may activate both caspase-dependent and caspase-independent cell death signalling pathways in T lymphocytes. On the other hand, we observed the processing of apical procaspase-8 in response to TRAIL in activated T lymphocytes preincubated with BAY 11-7085 (Fig. 3E), which confirm the induction of the TRAIL signalling pathway in T cells sensitized by treatment with the NF-B inhibitor. To determine the specific involvement of NF-B in the mechanism of BAY 11-7085-induced sensitization, we examined the effects of different NF-B inhibitors on the resistance of activated T cells to TRAIL-mediated cell death. Sulfasalazine, parthenolide and caffeic acid phenetyl ester (CAPE) have all been reported to be specific NF-B inhibitors (Bork et al., 1997; Natarajan et al., 1996; Wahl et al., 1998). We observed that activated T lymphocytes were significantly sensitized to TRAIL-induced apoptosis upon pretreatment with sulfasalazine. However, parthenolide and CAPE showed a very week and no effect, respectively, on the resistance to TRAIL (Fig. 4A). We next confirmed that all these compounds effectively inhibited NF-B at the concentrations used in this study. To this end, we analyzed the presence of the p65 (RelA) and the p50 (NF-B1) subunits of NF-B in nuclear extracts of activated T lymphocytes following treatment with the NF-B inhibitors. Western blot analysis showed that BAY 11-7085, sulfasalazine and parthenolide significantly reduced the levels of p65 and p50 in the nucleus of activated T cells, while CAPE only had a slight effect on the nuclear translocation of the NF-B subunits (Fig. 4B), which may explain the lack of sensitization to TRAIL-induced apoptosis showed by this agent. 3.4. BAY 11-7085 regulates the expression of TRAIL receptors and antiapoptotic genes in activated T lymphocytes The transcription factor NF-B has been reported to regulate the expression of some TRAIL receptors as well as that of J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 2593 may be due to the long half-life of the protein (Reed, 1996). Altogether, regulation of these antiapoptotic proteins and changes in the expression of TRAIL-R4, may account for the sensitization of T lymphocytes to TRAIL-mediated apoptosis observed in response to NF-B inhibition. 4. Discussion Fig. 4. Effect of different NF-B inhibitors on the resistance of activated T cells to TRAIL. (A) Activated T cells were preincubated for 1 h with 1.5 mM sulfasalazine (SS), 2.5 M parthenolide (PT) or 50 g/ml CAPE, before treatment with 250 ng/ml TRAIL for 24 h. Sub-G1 apoptotic cells were analyzed by flow cytometry. Error bars show S.D. from three independent experiments. * p < 0.05 by unpaired Student’s t-test (two-tailed). (B) Expression of the p50 and p65 subunits of NF-B was determined by Western blot in nuclear extracts of activated T cells after treatment for 4 h with the indicated inhibitors. PARP was used as control of loaded protein. Results shown are representative of three independent experiments. several antiapoptotic proteins involved in TRAIL signalling apoptotic pathway (Micheau et al., 2001; Ravi et al., 2001; Stehlik et al., 1998; Wang et al., 1998). In order to understand the mechanism for NF-B inhibition-induced sensitization of human primary T lymphocytes to TRAIL-mediated apoptosis, we first analyzed the expression of TRAIL-R1, -R2 and -R4 on the surface of activated T lymphocytes upon treatment with BAY 11-7085. As shown in Fig. 5A, we did not observe any change in the expression of death receptors, although the surface level of the antiapoptotic receptor TRAIL-R4 was reduced in response to the NF-B inhibitor in all samples analyzed. Moreover, when we determined the expression of TRAIL receptors at the mRNA level we observed a clear time-dependent down-regulation of all of them after treatment with BAY 11-7085 (Fig. 5B). TRAIL-R3 expression was undetectable at the mRNA level (data not shown). We then examined the effects of BAY 11-7085 on the expression of different antiapoptotic genes, namely c-FLIP, Bcl-2, Bcl-x, Mcl-1, XIAP, c-IAP1 and c-IAP2, in activated T lymphocytes. We found no marked changes in the mRNA levels of c-FLIPL , Bcl-x and Mcl-1 for up to 9 h following treatment with BAY 11-7085 (Fig. 5C). In contrast, a significant and timedependent decrease in the mRNA expression of c-FLIPS , Bcl-2, XIAP, c-IAP1 and c-IAP2 was observed in T cells exposed to the NF-B inhibitor (Fig. 5C). We further analyzed whether the decrease of these antiapoptotic genes was paralleled by changes in the levels of the corresponding proteins. Results in Fig. 5D indicate that all the proteins analyzed, with the exception of Bcl-2, were down-regulated after 24 h of treatment with BAY 11-7085. The absence of down-regulation of Bcl-2 at this time The significance of TRAIL as an anti-cancer therapeutic agent is supported by its known involvement in immune surveillance against tumors. Surface expression of TRAIL is upregulated in virtually all cell types of the immune system in response to activation signals, mediating to a great degree the cytotoxic activity of these cells (Fanger et al., 1999; Griffith et al., 1999; Kayagaki et al., 1999; Kemp et al., 2004, 2005; Takeda et al., 2001). The lack of toxicity to most normal cells is an important feature for TRAIL to act as an anti-tumor agent although the molecular mechanisms of TRAIL resistance in normal cells are not completely understood. Here, we have analyzed the response of resting and activated T lymphocytes to TRAIL. We have shown for the first time that the TRAIL signalling pathway is inhibited at the most apical level in both, resting and activated T cells, as no caspase-8 activation was observed in response to TRAIL. The analysis of the expression of the most important proteins involved in the formation of the DISC indicate that: (1) long-term T cell activation (day 6) does not modulate the expression levels of FADD, caspase-8, c-FLIP and TRAIL receptors, with the exception of TRAIL-R4; (2) when comparison with the TRAIL sensitive Jurkat cells, the expression of the antiapoptotic protein c-FLIP is much higher in T lymphocytes than in this cell line; (3) the levels of expression of TRAIL-R1, -R2 and -R3 receptors on the surface of T cells are barely detectable while the expression of TRAIL-R4 shows two peaks with low and high fluorescence intensity, respectively. From these data we can infer that several factors may be responsible for the lack of caspase-8 activation upon treatment with TRAIL and hence for the resistance of resting and activated T lymphocytes to TRAIL-induced apoptosis. It has been recently described that c-FLIP is involved the resistance of NK cells to TRAIL-induced apoptosis (Mirandola et al., 2004). Our results about the expression of c-FLIP, in agreement with previous reports (Bosque et al., 2005; Mirandola et al., 2004; Schmitz et al., 2003), suggest that this inhibitory protein could also play a role in the resistance of T lymphocytes. Regarding the expression of TRAIL receptors, there are some discrepancies between authors. Previous studies have reported that resting T cells do not show significant expression of TRAIL receptors, with the exception of TRAIL-R4 on the CD8+ subpopulation (Hasegawa et al., 2004; Mirandola et al., 2004). However, we have observed that the intensity of the TRAIL-R4 less fluorescence peak, which seems to correspond to the CD4+ cells, may vary among donors. On the other hand, those reports show that activation of CD8+ T cells up-regulate the expression of either TRAIL-R1 and -R2 (Hasegawa et al., 2004) or TRAILR2, -R3 and -R4 (Mirandola et al., 2004) while no changes are detected in the expression of TRAIL receptors upon activation of CD4+ T cells (Hasegawa et al., 2004). In contrast to Miran- 2594 J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 Fig. 5. Expression of TRAIL receptors and antiapoptotic genes in activated T lymphocytes in response to BAY 11-7085. (A) Activated T cells were incubated with (unshaded peaks) or without (shaded peaks) 2.5 M BAY for 15 h and cell surface TRAIL-R1, -R2 and -R4 receptors expression was assessed by flow cytometry. Dashed lines show background fluorescence with secondary antibody. mRNA levels of TRAIL-R1, -R2, -R4 (B), c-FLIPL , c-FLIPS , Bcl-2, Bcl-x, Mcl-1, XIAP, c-IAP1 and c-IAP2 (C) were determined in activated T cells after treatment with 2.5 M BAY for 6 and 9 h by RT-PCR as described under Section 2. -Actin was used as control of RNA input. (D) Expression of c-FLIPL , c-FLIPS , Bcl-2, XIAP and c-IAP2 was analyzed by Western blot in activated T lymphocytes treated with or without 2.5 M BAY for 24 h. ␣-Tubulin was used as control of loaded protein. Data shown are representative of at least three independent experiments with different donors. dola et al., we have worked with the whole population of CD3+ T lymphocytes as CD4+ and CD8+ T cells subpopulations coexist and may influence each other in physiological conditions. Moreover, not only CD8+ cytotoxic T cells but also CD4+ T cells seem to be involved in antitumor immune response via TRAIL (Dorothee et al., 2002; Kayagaki et al., 1999). Contra- dictory data concerning the expression of TRAIL receptors in activated T lymphocytes may also be due to differences in the way of T cell activation. Independently of the reported profile of TRAIL receptors expression, it is important to emphasize that all studies agree in the resistance of activated T lymphocytes to TRAIL-induced apoptosis. J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 NF-B inhibition has been recently proposed as a mechanism for sensitization of some tumor resistant cells to TRAIL-induced apoptosis since this transcription factor is constitutively active in certain tumors modulating the expression of several antiapoptotic proteins (Huerta-Yepez et al., 2004; Karacay et al., 2004; Kasuga et al., 2004). Our findings reveal that several inhibitors of NF-B are also able to sensitize activated T lymphocytes to TRAIL-mediated apoptosis. Specifically, we have observed a clear sensitization to TRAIL with the NF-B inhibitors BAY 11-7085 and sulfasalazine while the effect of parthenolide is too low to be significant. As all these agents were able to inhibit the translocation of p65 and p50 subunits to the nucleus, we speculate that the weak sensitization observed with parthenolide may be due to the additional activities that have been reported for this compound, such as JNK activation and STAT3 inhibition (Nakshatri et al., 2004; Sobota et al., 2000), which can interfere in some way with the signals derived from NF-B inhibition. Interestingly, BAY 11-7085 reduces the mRNA expression of TRAIL-R1 and TRAIL-R2 receptors in activated T lymphocytes, although we could not estimate this decrease at the level of surface protein expression. Despite that the levels of death receptors are too low to be detected by FACS analysis, we have clearly observed activation of the initiator caspase-8 in response to TRAIL upon pretreatment of T lymphocytes with BAY 11-7085. Thus, the above data suggest that TRAIL-R1, TRAIL-R2 or both receptors must be present on the surface of activated T lymphocytes and are able to transmit the apoptotic signals. In addition, we have demonstrated for the first time that NF-B inhibition not only down-regulates the expression of TRAIL-R4 at the mRNA level but also modestly reduces TRAIL-R4 expression at the cell surface. TRAIL-R4 has been recently proposed to be a regulatory rather than a decoy receptor as it associates with TRAILR2 forming a ligand-independent, death-inhibitory complex (Clancy et al., 2005). Moreover, authors demonstrate that this complex regulates the sensitivity of CD8+ T cells to TRAILinduced apoptosis. Therefore, down-regulation of TRAIL-R4 by NF-B inhibition may be involved in the sensitization of activated T lymphocytes to TRAIL. We have further observed that BAY 11-7085 down-regulates the expression levels of some antiapoptotic proteins known to be transcriptionally modulated by NF-B, such as c-FLIPS , XIAP and c-IAP2. Although the contribution of each factor in mediating TRAIL resistance of activated T lymphocytes is unknown, the overall results provide a mechanism for the sensitization of activated T lymphocytes to TRAIL-mediated apoptosis by NF-B inhibition. Regarding Bcl-2, even though there is a clear decrease of mRNA levels, our findings suggest that this antiapoptotic factor must not be involved in the effect of the NF-B inhibitor as the protein levels do not change at the time we observe sensitization of T lymphocytes. Our data indicate that the response of resting T lymphocytes to TRAIL-induced apoptosis upon NF-B inhibition vary among different donors. While NF-B is actively involved in T cell proliferation and activation, only low levels of constitutive NF-B binding activity are usually found in resting T cells. Moreover, homodimeric complexes of the NF-B p50 subunit, which generally function as transcriptional repressors, have been reported 2595 to bind to the DNA in the nuclei of resting T cells, in sharp contrast with the transcriptionally active p65/p50 heterodimers found in activated T lymphocytes (Algarte et al., 1995; Kang et al., 1992). These special features of NF-B activity in resting T lymphocytes, together with individual singularities, may influence the effect of BAY 11-7085. Furthermore, TRAIL has been reported to activate NF-B through TRAIL-R1 and TRAILR2 in many cell types (Franco et al., 2001; Hao et al., 2003; Schneider et al., 1997). Our striking findings about the blockade of cell death observed in some resting T cells upon treatment with both, BAY 11-7085 and TRAIL, might be due to the activation of NF-B by TRAIL, which could counteract the cytotoxic effect of the NF-B inhibitor. It would be interesting to determine whether NF-B activation is the predominant effect of TRAIL receptors engagement in resting T lymphocytes. On the other hand, although we have detected similar levels of pro- and antiapoptotic genes in resting and activated T lymphocytes (Fig. 1 and data not shown) it is known that only the last ones are able to undergo activation-induced cell death (AICD) (Wesselborg et al., 1993) indicating that different mechanisms of apoptosis resistance work in both types of cells, which may also influence the sensitization to TRAIL. Although proteasome inhibitors block NF-B activation, we have not observed any effect of either bortezomib or MG132 on the resistance of T lymphocytes to TRAIL-mediated apoptosis (Fig. 2E and data not shown). This is not surprising as the biological effects of proteasome inhibitors are multiple and the final result of all of them may be not suitable for the sensitization of T cells to TRAIL. In spite of that, our results indicate that bortezomib is highly toxic especially for activated T lymphocytes. Similarly, other modulators of TRAIL sensitivity in tumor cells, such as doxorubicin and nocodazole, show a moderate toxicity against activated T cells. In contrast, and in agreement with previous reports, our findings suggest that HDAC inhibitors may be a valuable therapeutic strategy to sensitize tumor cells to TRAIL-induced apoptosis as they seem to be non-toxic and they do not regulate TRAIL resistance in primary T cells (Inoue et al., 2004). PI3K/Akt inhibitors may also be useful in the treatment of tumors with high level of activity of this survival pathway that seems to block the apoptotic signals derived from TRAIL receptors engagement (Nam et al., 2003; Nesterov et al., 2001). NF-B activation is essential to numerous physiological processes and plays a crucial role in the immune response, e.g. in lymphocyte proliferation and cytokine production, which suggests that general NF-B inhibitors will probably produce a myriad of side-effects. In the present study, we have demonstrated that NF-B inhibitors not only sensitize primary activated T lymphocytes to TRAIL-induced apoptosis but also show a variable toxicity to resting and activated T cells. It is worth mentioning a recent report describing that the mechanism of TRAIL resistance in normal human urothelial cells is mainly dependent on the NF-B pathway (Steele et al., 2006). Together with these data, our results suggest a likely role for NF-B in the resistance of some normal cells types to TRAIL-induced apoptosis and challenge the use of NF-B inhibitors as a useful approach in antitumor therapy. 2596 J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 Acknowledgements This work was supported by a grant from the Ministerio de Educación y Ciencia SAF2003-02486 (to C.R.-R.). J.C.M. was supported by a fellowship from the Ministerio de Educación y Ciencia. We would like to thank Dr. Abelardo López-Rivas and Dr. F. Javier Oliver for invaluable advice and for the generous donation of reagents. References Algarte, M., Lecine, P., Costello, R., Plet, A., Olive, D., Imbert, J., 1995. In vivo regulation of interleukin-2 receptor alpha gene transcription by the coordinated binding of constitutive and inducible factors in human primary T cells. EMBO J. 14, 5060–5072. Bork, P.M., Schmitz, M.L., Kuhnt, M., Escher, C., Heinrich, M., 1997. Sesquiterpene lactone containing Mexican Indian medicinal plants and pure sesquiterpene lactones as potent inhibitors of transcription factor NFkappaB. FEBS Lett. 402, 85–90. Bosque, A., Pardo, J., Martinez-Lorenzo, M.J., Iturralde, M., Marzo, I., Pineiro, A., Alava, M.A., Naval, J., Anel, A., 2005. Down-regulation of normal human T cell blast activation: roles of APO-2L/TRAIL, FasL, and c- FLIP, Bim, or Bcl-x isoform expression. J. Leukoc. Biol. 77, 568–578. Clancy, L., Mruk, K., Archer, K., Woelfel, M., Mongkolsapaya, J., Screaton, G., Lenardo, M.J., Chan, F.K., 2005. Preligand assembly domain-mediated ligand-independent association between TRAIL receptor 4 (TR4) and TR2 regulates TRAIL-induced apoptosis. Proc. Natl. Acad. Sci. U.S.A. 102, 18099–18104. Cretney, E., Shanker, A., Yagita, H., Smyth, M.J., Sayers, T.J., 2006. TNFrelated apoptosis-inducing ligand as a therapeutic agent in autoimmunity and cancer. Immunol. Cell Biol. 84, 87–98. Cummins, J.M., Kohli, M., Rago, C., Kinzler, K.W., Vogelstein, B., Bunz, F., 2004. X-linked inhibitor of apoptosis protein (XIAP) is a nonredundant modulator of tumor necrosis factor-related apoptosis-inducing ligand (TRAIL)-mediated apoptosis in human cancer cells. Cancer Res. 64, 3006–3008. Chinnaiyan, A.M., O’Rourke, K., Tewari, M., Dixit, V.M., 1995. FADD, a novel death domain-containing protein, interacts with the death domain of Fas and initiates apoptosis. Cell 81, 505–512. Degli-Esposti, M.A., Dougall, W.C., Smolak, P.J., Waugh, J.Y., Smith, C.A., Goodwin, R.G., 1997. The novel receptor TRAIL-R4 induces NF-kappaB and protects against TRAIL-mediated apoptosis, yet retains an incomplete death domain. Immunity 7, 813–820. Dorothee, G., Vergnon, I., Menez, J., Echchakir, H., Grunenwald, D., Kubin, M., Chouaib, S., Mami-Chouaib, F., 2002. Tumor-infiltrating CD4+ T lymphocytes express APO2 ligand (APO-2L)/TRAIL upon specific stimulation with autologous lung carcinoma cells: role of IFN-alpha on APO-2L/TRAIL expression and -mediated cytotoxicity. J. Immunol. 169, 809–817. Fanger, N.A., Maliszewski, C.R., Schooley, K., Griffith, T.S., 1999. Human dendritic cells mediate cellular apoptosis via tumor necrosis factor-related apoptosis-inducing ligand (TRAIL). J. Exp. Med. 190, 1155–1164. Franco, A.V., Zhang, X.D., Van Berkel, E., Sanders, J.E., Zhang, X.Y., Thomas, W.D., Nguyen, T., Hersey, P., 2001. The role of NF-kappaB in TNF-related apoptosis-inducing ligand (TRAIL)-induced apoptosis of melanoma cells. J. Immunol. 166, 5337–5345. Gong, J.P., Traganos, F., Darzynkiewicz, Z., 1994. A selective procedure for DNA extraction from apoptotic cells applicable for gel electrophoresis and flow cytometry. Anal. Biochem. 218, 314–319. Griffith, T.S., Wiley, S.R., Kubin, M.Z., Sedger, L.M., Maliszewski, C.R., Fanger, N.A., 1999. Monocyte-mediated tumoricidal activity via the tumor necrosis factor-related cytokine, TRAIL. J. Exp. Med. 189, 1343–1354. Hao, X.S., Hao, J.H., Liu, F.T., Newland, A.C., Jia, L., 2003. Potential mechanisms of leukemia cell resistance to TRAIL-induced apoptosis. Apoptosis 8, 601–607. Hasegawa, H., Yamada, Y., Harasawa, H., Tsuji, T., Murata, K., Sugahara, K., Tsuruda, K., Masuda, M., Takasu, N., Kamihira, S., 2004. Restricted expres- sion of tumor necrosis factor-related apoptosis-inducing ligand receptor 4 in human peripheral blood lymphocytes. Cell. Immunol. 231, 1–7. Huerta-Yepez, S., Vega, M., Jazirehi, A., Garban, H., Hongo, F., Cheng, G., Bonavida, B., 2004. Nitric oxide sensitizes prostate carcinoma cell lines to TRAIL-mediated apoptosis via inactivation of NF-kappaB and inhibition of Bcl-xl expression. Oncogene 23, 4993–5003. Inoue, S., MacFarlane, M., Harper, N., Wheat, L.M., Dyer, M.J., Cohen, G.M., 2004. Histone deacetylase inhibitors potentiate TNF-related apoptosisinducing ligand (TRAIL)-induced apoptosis in lymphoid malignancies. Cell Death Differ. 11 (Suppl. 2), S193–S206. Jo, M., Kim, T.H., Seol, D.W., Esplen, J.E., Dorko, K., Billiar, T.R., Strom, S.C., 2000. Apoptosis induced in normal human hepatocytes by tumor necrosis factor-related apoptosis-inducing ligand. Nat. Med. 6, 564–567. Kandasamy, K., Srivastava, R.K., 2002. Role of the phosphatidylinositol 3 -kinase/PTEN/Akt kinase pathway in tumor necrosis factor-related apoptosis-inducing ligand-induced apoptosis in non-small cell lung cancer cells. Cancer Res. 62, 4929–4937. Kang, S.M., Tran, A.C., Grilli, M., Lenardo, M.J., 1992. NF-kappa B subunit regulation in non-transformed CD4+ T lymphocytes. Science 256, 1452–1456. Karacay, B., Sanlioglu, S., Griffith, T.S., Sandler, A., Bonthius, D.J., 2004. Inhibition of the NF-kappaB pathway enhances TRAIL-mediated apoptosis in neuroblastoma cells. Cancer Gene Ther. 11, 681–690. Kasuga, C., Ebata, T., Kayagaki, N., Yagita, H., Hishii, M., Aral, H., Sato, K., Okumura, K., 2004. Sensitization of human glioblastomas to tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) by NF-kappaB inhibitors. Cancer Sci. 95, 840–844. Kayagaki, N., Yamaguchi, N., Nakayama, M., Eto, H., Okumura, K., Yagita, H., 1999. Type I interferons (IFNs) regulate tumor necrosis factor-related apoptosis-inducing ligand (TRAIL) expression on human T cells: a novel mechanism for the antitumor effects of type I IFNs. J. Exp. Med. 189, 1451–1460. Kemp, T.J., Ludwig, A.T., Earel, J.K., Moore, J.M., VanOosten, R.L., Moses, B., Leidal, K., Nauseef, W.M., Griffith, T.S., 2005. Neutrophil stimulation with Mycobacterium bovis bacillus calmette-guerin (BCG) results in the release of functional soluble TRAIL/APO-2L. Blood 106, 3474–3482. Kemp, T.J., Moore, J.M., Griffith, T.S., 2004. Human B cells express functional TRAIL/Apo-2 ligand after CpG-containing oligodeoxynucleotide stimulation. J. Immunol. 173, 892–899. Lawrence, D., Shahrokh, Z., Marsters, S., Achilles, K., Shih, D., Mounho, B., Hillan, K., Totpal, K., DeForge, L., Schow, P., Hooley, J., Sherwood, S., Pai, R., Leung, S., Khan, L., Gliniak, B., Bussiere, J., Smith, C.A., Strom, S.S., Kelley, S., Fox, J.A., Thomas, D., Ashkenazi, A., 2001. Differential hepatocyte toxicity of recombinant APO-2L/TRAIL versions. Nat. Med. 7, 383–385. Li, L., Thomas, R.M., Suzuki, H., De Brabander, J.K., Wang, X., Harran, P.G., 2004. A small molecule Smac mimic potentiates TRAIL-and TNFalphamediated cell death. Science 305, 1471–1474. MacFarlane, M., Ahmad, M., Srinivasula, S.M., Fernandes-Alnemri, T., Cohen, G.M., Alnemri, E.S., 1997. Identification and molecular cloning of two novel receptors for the cytotoxic ligand TRAIL. J. Biol. Chem. 272, 25417–25420. MacFarlane, M., Harper, N., Snowden, R.T., Dyer, M.J., Barnett, G.A., Pringle, J.H., Cohen, G.M., 2002. Mechanisms of resistance to TRAIL-induced apoptosis in primary B cell chronic lymphocytic leukaemia. Oncogene 21, 6809–6818. Micheau, O., Lens, S., Gaide, O., Alevizopoulos, K., Tschopp, J., 2001. NFkappaB signals induce the expression of c-FLIP. Mol. Cell. Biol. 21, 5299–5305. Mirandola, P., Ponti, C., Gobbi, G., Sponzilli, I., Vaccarezza, M., Cocco, L., Zauli, G., Secchiero, P., Manzoli, F.A., Vitale, M., 2004. Activated human NK and CD8+ T cells express both TNF-related apoptosis-inducing ligand (TRAIL) and TRAIL receptors but are resistant to TRAIL-mediated cytotoxicity. Blood 104, 2418–2424. Nakshatri, H., Rice, S.E., Bhat-Nakshatri, P., 2004. Antitumor agent parthenolide reverses resistance of breast cancer cells to tumor necrosis factor-related apoptosis-inducing ligand through sustained activation of c-Jun N-terminal kinase. Oncogene 23, 7330–7344. Nam, S.Y., Jung, G.A., Hur, G.C., Chung, H.Y., Kim, W.H., Seol, D.W., Lee, B.L., 2003. Upregulation of FLIP(S) by Akt, a possible inhibition mecha- J.C. Morales et al. / Molecular Immunology 44 (2007) 2587–2597 nism of TRAIL-induced apoptosis in human gastric cancers. Cancer Sci. 94, 1066–1073. Natarajan, K., Singh, S., Burke Jr., T.R., Grunberger, D., Aggarwal, B.B., 1996. Caffeic acid phenethyl ester is a potent and specific inhibitor of activation of nuclear transcription factor NF-kappaB. Proc. Natl. Acad. Sci. U.S.A. 93, 9090–9095. Nesterov, A., Lu, X., Johnson, M., Miller, G.J., Ivashchenko, Y., Kraft, A.S., 2001. Elevated AKT activity protects the prostate cancer cell line LNCaP from TRAIL-induced apoptosis. J. Biol. Chem. 276, 10767–10774. Ogasawara, J., Watanabe-Fukunaga, R., Adachi, M., Matsuzawa, A., Kasugai, T., Kitamura, Y., Itoh, N., Suda, T., Nagata, S., 1993. Lethal effect of the anti-Fas antibody in mice. Nature 364, 806–809. Ortiz-Ferron, G., Tait, S.W., Robledo, G., de Vries, E., Borst, J., LopezRivas, A., 2006. The mitogen-activated protein kinase pathway can inhibit TRAIL-induced apoptosis by prohibiting association of truncated Bid with mitochondria. Cell Death Differ. 13, 1857–1865. Pan, G., Ni, J., Wei, Y.F., Yu, G., Gentz, R., Dixit, V.M., 1997a. An antagonist decoy receptor and a death domain-containing receptor for TRAIL. Science 277, 815–818. Pan, G., O’Rourke, K., Chinnaiyan, A.M., Gentz, R., Ebner, R., Ni, J., Dixit, V.M., 1997b. The receptor for the cytotoxic ligand TRAIL. Science 276, 111–113. Ravi, R., Bedi, G.C., Engstrom, L.W., Zeng, Q., Mookerjee, B., Gelinas, C., Fuchs, E.J., Bedi, A., 2001. Regulation of death receptor expression and TRAIL/APO-2L-induced apoptosis by NF-kappaB. Nat. Cell Biol. 3, 409–416. Reed, J.C., 1996. A day in the life of the Bcl-2 protein: does the turnover rate of Bcl-2 serve as a biological clock for cellular lifespan regulation? Leuk. Res. 20, 109–111. Ruiz-Ruiz, M.C., Lopez-Rivas, A., 1999. p53-Mediated up-regulation of CD95 is not involved in genotoxic drug-induced apoptosis of human breast tumor cells. Cell Death Differ. 6, 271–280. Ruiz de Almodovar, C., Ruiz-Ruiz, C., Munoz-Pinedo, C., Robledo, G., LopezRivas, A., 2001. The differential sensitivity of Bc1-2-overexpressing human breast tumor cells to TRAIL or doxorubicin-induced apoptosis is dependent on Bc1-2 protein levels. Oncogene 20, 7128–7133. Sarker, M., Ruiz-Ruiz, C., Lopez-Rivas, A., 2001. Activation of protein kinase C inhibits TRAIL-induced caspases activation, mitochondrial events and apoptosis in a human leukemic T cell line. Cell Death Differ. 8, 172–181. Sayers, T.J., Murphy, W.J., 2006. Combining proteasome inhibition with TNFrelated apoptosis-inducing ligand (APO-2L/TRAIL) for cancer therapy. Cancer Immunol. Immunother. 55, 76–84. Schmitz, I., Krueger, A., Baumann, S., Schulze-Bergkamen, H., Krammer, P.H., Kirchhoff, S., 2003. An IL-2-dependent switch between CD95 signaling pathways sensitizes primary human T cells toward CD95-mediated activation-induced cell death. J. Immunol. 171, 2930–2936. Schneider, P., Thome, M., Burns, K., Bodmer, J.L., Hofmann, K., Kataoka, T., Holler, N., Tschopp, J., 1997. TRAIL receptors 1 (DR4) and 2 (DR5) signal FADD-dependent apoptosis and activate NF-kappaB. Immunity 7, 831–836. Shankar, S., Srivastava, R.K., 2004. Enhancement of therapeutic potential of TRAIL by cancer chemotherapy and irradiation: mechanisms and clinical implications. Drug Resist. Update 7, 139–156. Sheridan, J.P., Marsters, S.A., Pitti, R.M., Gurney, A., Skubatch, M., Baldwin, D., Ramakrishnan, L., Gray, C.L., Baker, K., Wood, W.I., Goddard, A.D., Godowski, P., Ashkenazi, A., 1997. Control of TRAIL-induced apoptosis by a family of signaling and decoy receptors. Science 277, 818–821. Siegmund, D., Hadwiger, P., Pfizenmaier, K., Vornlocher, H.P., Wajant, H., 2002. Selective inhibition of FLICE-like inhibitory protein expression with small 2597 interfering RNA oligonucleotides is sufficient to sensitize tumor cells for TRAIL-induced apoptosis. Mol. Med. 8, 725–732. Singh, T.R., Shankar, S., Chen, X., Asim, M., Srivastava, R.K., 2003. Synergistic interactions of chemotherapeutic drugs and tumor necrosis factor-related apoptosis-inducing ligand/Apo-2 ligand on apoptosis and on regression of breast carcinoma in vivo. Cancer Res. 63, 5390–5400. Sobota, R., Szwed, M., Kasza, A., Bugno, M., Kordula, T., 2000. Parthenolide inhibits activation of signal transducers and activators of transcription (STATs) induced by cytokines of the IL-6 family. Biochem. Biophys. Res. Commun. 267, 329–333. Sprick, M.R., Weigand, M.A., Rieser, E., Rauch, C.T., Juo, P., Blenis, J., Krammer, P.H., Walczak, H., 2000. FADD/MORT1 and caspase-8 are recruited to TRAIL receptors 1 and 2 and are essential for apoptosis mediated by TRAIL receptor 2. Immunity 12, 599–609. Steele, L.P., Georgopoulos, N.T., Southgate, J., Selby, P.J., Trejdosiewicz, L.K., 2006. Differential susceptibility to TRAIL of normal versus malignant human urothelial cells. Cell Death Differ. 13, 1564–1576. Stehlik, C., de Martin, R., Kumabashiri, I., Schmid, J.A., Binder, B.R., Lipp, J., 1998. Nuclear factor (NF)-kappa B-regulated X-chromosome-linked IAP gene expression protects endothelial cells from tumor necrosis factor alphainduced apoptosis. J. Exp. Med. 188, 211–216. Suda, T., Takahashi, T., Golstein, P., Nagata, S., 1993. Molecular cloning and expression of the Fas ligand, a novel member of the tumor necrosis factor family. Cell 75, 1169–1178. Takeda, K., Hayakawa, Y., Smyth, M.J., Kayagaki, N., Yamaguchi, N., Kakuta, S., Iwakura, Y., Yagita, H., Okumura, K., 2001. Involvement of tumor necrosis factor-related apoptosis-inducing ligand in surveillance of tumor metastasis by liver natural killer cells. Nat. Med. 7, 94–100. Wahl, C., Liptay, S., Adler, G., Schmid, R.M., 1998. Sulfasalazine: a potent and specific inhibitor of nuclear factor kappa B. J. Clin. Invest. 101, 1163– 1174. Walczak, H., Degli-Esposti, M.A., Johnson, R.S., Smolak, P.J., Waugh, J.Y., Boiani, N., Timour, M.S., Gerhart, M.J., Schooley, K.A., Smith, C.A., Goodwin, R.G., Rauch, C.T., 1997. TRAIL-R2: a novel apoptosis-mediating receptor for TRAIL. EMBO J. 16, 5386–5397. Walczak, H., Miller, R.E., Ariail, K., Gliniak, B., Griffith, T.S., Kubin, M., Chin, W., Jones, J., Woodward, A., Le, T., Smith, C., Smolak, P., Goodwin, R.G., Rauch, C.T., Schuh, J.C., Lynch, D.H., 1999. Tumoricidal activity of tumor necrosis factor-related apoptosis-inducing ligand in vivo. Nat. Med. 5, 157–163. Wang, C.Y., Mayo, M.W., Korneluk, R.G., Goeddel, D.V., Baldwin Jr., A.S., 1998. NF-kappaB antiapoptosis: induction of TRAF1 and TRAF2 and cIAP1 and c-IAP2 to suppress caspase-8 activation. Science 281, 1680– 1683. Wesselborg, S., Janssen, O., Kabelitz, D., 1993. Induction of activation-driven death (apoptosis) in activated but not resting peripheral blood T cells. J. Immunol. 150, 4338–4345. Whang, Y.E., Yuan, X.J., Liu, Y., Majumder, S., Lewis, T.D., 2004. Regulation of sensitivity to TRAIL by the PTEN tumor suppressor. Vitam. Horm. 67, 409–426. Wiley, S.R., Schooley, K., Smolak, P.J., Din, W.S., Huang, C.P., Nicholl, J.K., Sutherland, G.R., Smith, T.D., Rauch, C., Smith, C.A., Goodwin, R.G., 1995. Identification and characterization of a new member of the TNF family that induces apoptosis. Immunity 3, 673–682. Zhang, X.D., Franco, A.V., Nguyen, T., Gray, C.P., Hersey, P., 2000. Differential localization and regulation of death and decoy receptors for TNF-related apoptosis-inducing ligand (TRAIL) in human melanoma cells. J. Immunol. 164, 3961–3970.