Parámetros genéticos y ambientales para peso ajustado a 210 días
Anuncio

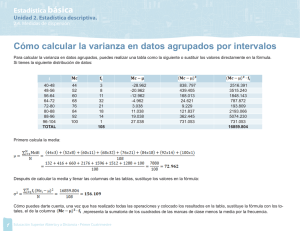
Resumen: V-007 UNIVERSIDAD NACIONAL DEL NORDEST E Comunicaciones Científicas y Tecnológicas 2005 Parámetros genéticos y ambientales para peso ajustado a 210 días en bovinos de raza Hereford 1 2 3 López, María V. - Arias Mañotti, Adolfo A. - Slobodzian, Ana 1- Facultad de Cs. Exactas y Naturales y Agrimensura (UNNE). 9 de Julio 1449.C.P.: 3400. Corrientes. Argentina. TE: (03783) 423126 - Fax (03783) 423968 2- Estación Experimental Agropecuaria Corrientes (INTA). C.C. 57. C.P.: 3400. Corrientes. Argentina. TE: (08783) 421786. 3-Estación Experimental Agropecuaria Las Breñas (INTA). C.C. 38. C.P.: 3722 . Las Breñas. Chaco. Argentina. TE: (03731) 460033. E-mail: mvlopez@exa.unne.edu.ar, ariasm@corrientes.inta.gov.ar, direbrenas@correo.inta.gov.ar Antecedentes Los bovinos de carne Hereford tuvieron su origen a comienzos del siglo XVIII en el condado de Herefordshire. Desde ese condado del sur de Inglaterra, no tardó en extenderse por todo el mundo. A nuestro país llegó en 1858, pero la primera importación de ganado Hereford de Pedigree fue realizada por el Sr. Leonardo Pereyra en 1862, cuando adquirió a Niágara 2131, instalándose primero en el centro del país y en la zona de la Pampa Húmeda desde donde se difundió rápidamente a otras zonas más difíciles, ocupando en poco tiempo el norte y el sur argentino. En 1917 ingresaron los primeros Polled Hereford, traídos por el Sr. Rafael Herrera Vegas, teniendo gran aceptación. Pruebas realizadas en nuestro país han demostrado la excelente capacidad de producción que esta raza tiene en nuestros sistemas pastoriles. Los Hereford son reconocidos por su alto poder de adaptación al medio ambiente. La docilidad es otra característica muy apreciada por razones de seguridad y facilidad de manejo, todo lo cual se traduce en mayor ahorro de dinero. Estas características son las que determinan que la raza Hereford se encuentre en permanente expansión en los rodeos de carne de nuestro país. (Sitio web del SENASA). Por otra parte, poseen una gran habilidad combinatoria general, lo cual los hace muy apropiados para cruzamientos en el subtrópico y otras regiones. Tienen un buen tamaño, crecimiento y masa muscular, fertilidad, precocidad y docilidad, lo que determina que esta raza sea utilizada en los cruzamientos con cebú, ya sea para programas de cruzamientos comerciales o en la formación de nuevas razas. En nuestro país, es requerida para ambos propósitos. El INTA EEA Corrientes ha desarrollado un esquema de selección para mejorar estas características en rodeos adaptados a las condiciones ambientales del subtrópico. La pregunta que un criador se plantea es dónde se ubica su rodeo respecto a los demás. Esta pregunta se responde conociendo la distribución del carácter que interesa en la población. La necesidad de comparar rodeos ha avanzado en el mundo. Esto ha llevado a cambios en la información que se está recibiendo. Por ejemplo, las Asociaciones de Hereford y Polled de EEUU y Canadá publicaron utilizando los recursos de la Universidad de Georgia, la primera evaluación internacional de la raza. Eso proporciona una gran simplificación para los criadores norteamericanos: tienen la misma base, los mismos criterios en la utilización de la precisión o accuracy y la misma tendencia genética, con lo cual pueden llegar a comparar DEPs (Diferencias Esperadas de la Progenie) de cualquiera de las cabañas que están siendo utilizadas en ese momento y que participan en el programa de toma de datos (Musi, 1995). En nuestro país, la Asociación Argentina Criadores de Hereford es la entidad responsable del PEG (Programa de Evaluación Genética). El mismo estudia la performance de manera exhaustiva y completa, a través de la elaboración de índices en algunos casos y DEPs en otros, surgidos de la Base de Datos de la Raza. Además ofrece una multitud de informes sobre los padres, madres e individuos evaluados. El análisis de los archivos se realiza conjuntamente con la Sociedad Rural Argentina, procesando la información para la elaboración de las DEPs en la universidad de Georgia, EEUU. El Puro Registrado A.A.C.H., es un plan de selección de animales machos y hembras Hereford y Polled Hereford con intención de reproducir hacienda de características fenotípicas superiores a la media de producción de la raza, protegiendo su pureza racial y privilegiando a individuos con características deseables en los diferentes órdenes de la producción animal de carne. Se trata de un registro abierto, regido por un reglamento y controlado por Inspectores de la Asociación (Sitio web del SENASA). Todo Programa de evaluación genética apunta a contar con estimaciones del mérito genético de cada reproductor en las variables de interés económico. Actualmente, la metodología más utilizada es la denominada Modelo Animal, por medio de la cual se obtienen las Diferencias Esperadas de la Progenie (DEPs). Se lo denomina Modelo Animal porque todos los animales que forman parte del archivo de análisis obtienen una evaluación genética individual para cada carácter considerado. El Modelo Animal contempla de manera simultánea factores genéticos y ambientales. Esta metodología predice el verdadero valor genético de cada reproductor, basándose para su cálculo en el propio comportamiento de cada animal, como así también en el desempeño (performance) de sus crías y la de todos sus parientes o pedigree (A.A.C.H., 1996). Los procedimientos de evaluación genética utilizan la metodología del Modelo Animal para incorporar toda la información disponible en la predicción del DEP de un individuo. La información generalmente disponible para un individuo incluye: a) la progenie o descendencia; b) el padre y la madre; c) los abuelos; d) el propio registro del individuo. Un aspecto importante de este procedimiento de análisis es que se tienen en cuenta la superioridad o inferioridad de los parientes del individuo, cuando la progenie está disponible. Esto reduce, si no elimina totalmente, Resumen: V-007 UNIVERSIDAD NACIONAL DEL NORDEST E Comunicaciones Científicas y Tecnológicas 2005 cualquier desvío potencial asociado con uniones específicas de padres y madres. Un segundo aspecto importante es la incorporación de las relaciones genéticas entre los animales en el procedimiento de análisis. Esto incorpora información sobre todos los antepasados y parientes colaterales en la predicción del DEP de un animal y también cuenta para la tendencia genética. El ajuste de la tendencia genética permite realizar comparaciones adecuadas entre animales jóvenes y más viejos (Bertrand et al.). El objetivo de pesar al destete es evaluar el crecimiento de las crías hasta el destete junto a la aptitud materna (producción de leche de los vientres). El peso al destete posee efectos directos (peso al destete propiamente dicho) y efectos indirectos (aptitud materna). Esto significa que el peso se ve afectado por los genes del ternero en cuanto a crecimiento, más el potencial genético de la madre en aptitud lechera, además de los factores ambientales que lo influencian. Para obtener las mejores estimaciones del valor genético para peso al destete, es necesario ajustar los registros individuales de los terneros a una base patrón de 210 días (A.A.C.H., 1996). El propósito de este trabajo fue estimar los parámetros genéticos y ambientales para peso ajustado a los 210 días de edad (I_PESOAJ_210) mediante la implementación del Modelo Animal. Preparación del archivo de datos para analizar el carácter peso ajustado a los 210 días. Se procesó una base de datos de 4264 registros de producción y genealogía de machos y hembras de la raza Hereford, correspondiente al período 1974-2002 y disponible en la EEA INTA Corrientes. La base de datos tiene la siguiente estructura: • I_NOM: Código del individuo. • T_NOM: Código del padre del individuo que produce el dato. • V_NOM: Código de la madre del individuo que produce el dato. • GC: Código de grupo contemporáneo. Estos grupos fueron definidos como animales nacidos en una misma cabaña, en el mismo año (carimbo) y del mismo sexo (GC = I_COD_CABA + I_CAR + I_SEXO), con un total de 110 grupos. • I_MESNAC: Mes de nacimiento del individuo. Se consideran los meses de marzo a noviembre. • V_EDAD: Edad de la madre. Está expresada en años y está tomada al momento del parto. Tiene 11 niveles, se inicia con la edad de 2 años y finaliza con la edad de 12 años. • V_LAC_PREV: Código de lactancia previa o anterior. Indica si la madre viene de amamantar su cría el año anterior y tiene 2 niveles (1=sí, 2=no). • I_EDAD_DES: Edad del animal en días al destete. Corresponde a la edad real del individuo al momento en que se registra el peso de los 6-7 meses. Se usa como covariable para analizar el peso ajustado a los 210 días. • I_PESOAJ_210: Peso del individuo ajustado a 210 días (kg). Es el peso vivo del animal tomado lo más cercano a los 6-7 meses y ajustado luego a los 210 días de edad. La fórmula de cálculo es: PD − PN I _ PESOAJ _ 210 = * 210 + PN I _ EDAD _ DES donde: PD = Peso al destete; PN = Peso al nacer; I_EDAD_DES = Edad en días al destete. El archivo de datos de pedigree y producción fue formateado adecuadamente para su procesamiento con el programa MTDFREML. Fue necesario recodificar los campos I_NOM, T_NOM y V_NOM, para cumplir con las restricciones de este programa: los códigos de los animales deben ser numéricos, los padres desconocidos deben ser codificados como 0, cada animal debe tener un código mayor que el de sus padres, el archivo debe estar en formato ASCII, entre otras. Luego se eliminaron los registros que no poseían información en el campo I_PESOAJ_210, quedando el archivo final con 4122 registros. Modelo para el análisis del carácter peso ajustado a los 210 días En este estudio, las estimaciones de los componentes de varianza, heredabilidad y los valores genéticos (DEPs) fueron obtenidas por el método DFREML, por medio del Modelo Animal univariado, utilizando el software MTDFREML (Boldman & Van Vleck, 1991; Boldman et al., 1993), con ecuaciones del Modelo Mixto (MME- Mixed Model Equations). Este software permite estimar los componentes de variancia y realizar la predicción de los valores genéticos de los animales. Implementa el método de máxima verosimilitud restricta (REML) (Patterson y Thompson, 1971), usando un algoritmo no derivativo (DFREML) propuesto por Smith y Graser (1986) y Graser et al. (1987). El programa MTDFREML se ejecutó en una PC Intel Pentium IV 1,6 Ghz, con 256 Mb de memoria RAM. El carácter peso ajustado a los 210 días fue analizado mediante un modelo estadístico univariado, considerando: • Efectos fijos: grupo contemporáneo (cabaña + año de nacimiento + sexo), mes de nacimiento, edad de la madre, lactancia previa de la madre, covariable edad del animal en días al destete. • Efectos aleatorios de los valores genéticos aditivos directos y maternos. • Efectos aleatorios del ambiente permanente de las madres. Resumen: V-007 UNIVERSIDAD NACIONAL DEL NORDEST E Comunicaciones Científicas y Tecnológicas 2005 El modelo matemático propuesto para este estudio es: yijklm = m + GCi + MEj + EMk + Ll + am + mm + pem + b1 (Eijklm) +eijklm, donde: yijklm = peso ajustado a 210 días, m = media general, GCi = efecto fijo del i-ésimo grupo contemporáneo (niveles 1-110), MEj = efecto fijo del j-ésimo mes de nacimiento (niveles 1-9), EMk = efecto fijo de la k-ésima categoría de edad de madre (niveles 1-11), Ll = efecto fijo de la l-ésima categoría de lactancia anterior (niveles 1-2), am = efecto aleatorio del valor genético aditivo directo del m-ésimo animal, mm = efecto aleatorio del valor genético aditivo materno del mésimo animal, pem = efecto aleatorio del ambiente permanente del m-ésimo animal, b1 = coeficiente de regresión lineal del peso ajustado a los 210 días sobre la edad del animal, Eijklm = edad en días al destete del m-ésimo animal (covariable),eijklm = efecto residual del m-ésimo animal (aleatorio). Fueron estimados los siguientes componentes de varianza y parámetros genéticos y ambientales, mediante el software MTDFREML: s2a (varianza de los efectos genéticos aditivos directos), s2m (varianza de los efectos genéticos aditivos maternos), sam (covarianza entre los efectos genéticos aditivos directos y maternos), s2pe (varianza ambiental permanente), s2e (varianza residual o error), s2p (varianza fenotípica total), h2a (heredabilidad para efectos genéticos directos), h2m (heredabilidad para efectos genéticos maternos), ram (correlación genética entre los efectos directos y maternos), e2 (proporción de la varianza fenotípica total debida a la varianza residual) y c2 (proporción de la varianza fenotípica total debida a la varianza ambiental permanente). Para obtener las predicciones de los parámetros genéticos, el software MTDFREML necesita que sean proporcionados los componentes de varianza apropiados, estimados en análisis previos u obtenidos de literatura. En este caso, los valores iniciales de las varianzas y covarianzas empleados fueron obtenidos de Meyer (1993). Además se obtuvieron las estimaciones de los valores genéticos (Breeding Values - BVs) y de las respectivas Predicciones de la Varianza del Error (Prediction Error Variance - PEVs). Cada BV predicho es acompañado de la PEV y de una precisión (PREC). Luego se calcularon las diferencias esperadas de las progenies (DEPs) de los animales dividiendo por dos los BVs. Discusión de resultados Las estimaciones de los componentes de varianza y covarianza, heredabilidad para el efecto directo y para el efecto materno, y la correlación genética entre estos dos parámetros se presentan en el Cuadro 1. Además, se incluyen en este cuadro, en la categoría de parámetros ambientales, las estimaciones de la varianza de ambiente permanente de la madre (c2) y de la varianza residual (e2), como proporciones de la varianza fenotípica. Cuadro 1: Estimaciones de parámetros genéticos y ambientales para I_PESOAJ_210 en la raza Hereford. s 2a 74,17 h2a 0,17 2 sm 111,12 h2m 0,25 sam -10,35 ram -0,11 s2pe 58,34 c2 0,13 2 se 214,45 e2 0,48 s 2p 447,74 Las estimaciones de los componentes de varianza obtenidas son similares o menores, según el caso, que las citadas en la literatura para raza Hereford (Meyer, 1993): s2a = 127,69, s2m = 113,5, s2pe = 163,16, s2e = 212,82 y s2p = 709,4. El valor estimado de h2a es similar al encontrado en la literatura: Meyer (1993) obtuvo h2a = 0,18 para este carácter. En cuanto al valor estimado de h2m, es superior al encontrado por Meyer (1993): h2m = 0,16. Por otra parte, Bertrand et al. encontraron para este carácter el valor 0,23 para h2a, siendo éste superior al obtenido en este trabajo. Finalmente, las medias de los valores citados en la literatura para este carácter en ganado de carne son: h2a = 0,25, h2m = 0,19 y ram = 0,11, coincidiendo exactamente el valor de ram con el obtenido en este análisis. El valor de la heredabilidad para este carácter, indica la factibilidad de obtener progreso genético en caso de seleccionar por el mismo. El valor estimado de c2, es menor al encontrado en la literatura para raza Hereford: (Meyer (1993) obtuvo c2 = 0,23), pero resulta concordante con la media de los valores publicados de c2 en ganado de carne para este carácter (0,12). El valor estimado de e2 es más alto que el obtenido por Meyer (1993) para raza Hereford (0,30). Las diferencias esperadas de la progenie (DEPs) calculadas a partir de los valores genéticos predichos (BVs) fueron graficadas en función del número de observaciones, dando una idea general de su distribución en la población analizada. En las figuras 1 y 2 se muestra la representación gráfica de la distribución de los DEPs para los efectos directo y materno en el carácter peso ajustado a los 210 días. La observación de los gráficos muestra claramente que la distribución de los DEPs se aproxima a la normal. Figura 1 Resumen: V-007 UNIVERSIDAD NACIONAL DEL NORDEST E Comunicaciones Científicas y Tecnológicas 2005 Figura 2 Conclusiones Se ha procesado una base de datos con información de producción y genealogía de bovinos de raza Hereford disponible en la EEA Corrientes, mediante el software MTDFREML que implementa el Modelo Animal, obteniéndose estimaciones de los factores ambientales y genéticos que influencian el carácter peso ajustado a los 210 días. Además, la metodología del Modelo Animal permitió evaluar los animales, y obtener las Diferencias Esperadas de la Progenie (DEPs) del carácter mencionado. Este estudio permitió obtener un panorama confiable acerca de la población Hereford evaluada y de sus posibilidades de mejoramiento, además de simplificar y hacer más manejable la información proporcionada a los productores en lo referente al carácter peso ajustado a 210 días, posibilitando a los mismos orientar objetivamente sus programas de mejoramiento. Referencias • A.A.C.H. Asociación Argentina de Criadores de Hereford. 1996. Manual del Criador. Buenos Aires. Argentina. • Bertrand, Keith, Little, Del, Nelson, Andra, Benyshek, Larry. North American Hereford Cattle Evaluation. Universidad de Georgia. Estados Unidos. En: www.missouri.edu/~anscbeef/hereford. • Boldman, K.G., Van Vleck, L.D. 1991. Derivative-Free Restricted Maximum Likelihood Estimation in Animal Model with a Sparse Matrix Solver. J. Dairy Sci, V.74. • Boldman,K.G., Kriese, L.A., Van Vleck, L.D., Kachman, S.D. 1993. A Manual for Use of MTDFREML – A Set of Programs to Obtain Estimates of Variances and Covariances. USDA-ARS. • Graser, H.U., Smith, S.P., Tier, B. 1987. A derivative-free approach for estimating variance components en animal models by restricted maximum likehood. J. Anim. Sci., V.64. • Meyer, K. 1993. Covariance matrices for growth traits of Australian Polled Hereford cattle. Anim. Prod. 1993, 57: 37-45. British Society of Animal Production. Animal Genetics and Breeding Unit. University of New England. Armidale. Australia. • Musi, Daniel. 1995. La genética y el criador. Revista “Hereford. La raza sin fronteras”. Año LXI. Nº 605. Primera Convención Nacional Hereford. • Patterson, H.D, Thompson, R. 1971. Recovery of inter.-block information when block sizes are unequal. Biometrika, V.58. • Sitio web del SENASA. Servicio Nacional de Sanidad y Calidad Agroalimentaria Av. Paseo Colón 367. CP: C1063ACD. Buenos Aires. Argentina. http://www.senasa.gov.ar/Publicaciones/razas/razadet/herefo.php. • Smith, S.P., Graser, H.U. 1986. Estimating variance components in a class of mixed models by restricted maximum likelihood. J. Dairy Sci., V.69.