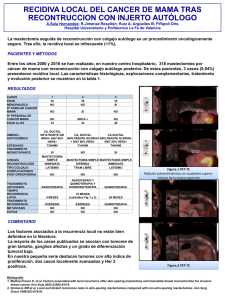
Tesis Arcos N. - Repositorio de la Universidad de Fuerzas Armadas
Anuncio

DEPARTAMENTO DE CIENCIAS DE LA VIDA Y DE LA AGRICULTURA CARRERA DE INGENIERÍA EN BIOTECNOLOGÍA TESIS PREVIO A LA OBTENCIÓN DEL TÍTULO DE INGENIERO EN BIOTECNOLOGÍA TEMA: ANÁLISIS DE LOS POLIMORFISMOS GENÉTICOS E17K, E17S Y L357P DEL GEN AKT1, Y SU ASOCIACIÓN CON CARACTERÍSTICAS HISTOPATOLÓGICAS E INMUNOHISTOQUÍMICAS EN LA POBLACIÓN MESTIZA ECUATORIANA AFECTA CON CÁNCER DE MAMA. AUTOR: ARCOS VILLACÍS, NATHALY ALEXANDRA DIRECTOR: DR. MARCELO GRIJALVA, M.D., Ph. D. CODIRECTOR: DRA. PATRICIA JIMÉNEZ., M.Sc. SANGOLQUÍ 2015 ii CERTIFICACIÓN Dr. Marcelo Grijalva, M.D., Ph. D. Dra. Patricia Jiménez., M.Sc. Certifican: Que el trabajo titulado “Análisis de los polimorfismos genéticos E17K, E17S y L357P del gen AKT1, y su asociación con características histopatológicas e inmunohistoquímicas en la población mestiza ecuatoriana afecta con cáncer de mama”, realizado por Nathaly Alexandra Arcos Villacís, ha sido guiado y revisado periódicamente y cumple normas estatutarias establecidas en el Reglamento de Estudiantes de la Universidad de las Fuerzas Armadas- ESPE. Sangolquí, Mayo 2015 Dr. Marcelo Grijalva, M.D., Ph. D. DIRECTOR Dra. Patricia Jiménez., M.Sc. CODIRECTOR iii DECLARACIÓN DE RESPONSABILIDAD Yo, NATHALY ALEXANDRA ARCOS VILLACÍS Declaro que: El proyecto de grado denominado “Análisis de los polimorfismos genéticos E17K, E17S y L357P del gen AKT1, y su asociación con características histopatológicas e inmunohistoquímicas en la población mestiza ecuatoriana afecta con cáncer de mama”, ha sido desarrollado con base a una investigación exhaustiva, respetando derechos intelectuales de terceros, conforme las citas que constan al pie de las páginas correspondientes, cuyas fuentes se incorporan en la bibliografía. Consecuentemente este trabajo es de mi autoría. En virtud de esta declaración, me responsabilizo del contenido, veracidad y alcance científico del proyecto de grado en mención. Sangolquí, Mayo 2015 Nathaly Alexandra Arcos Villacís iv AUTORIZACIÓN Yo, NATHALY ALEXANDRA ARCOS VILLACÍS Autorizo que: El proyecto de grado denominado “Análisis de los polimorfismos genéticos E17K, E17S y L357P del gen AKT1, y su asociación con características histopatológicas e inmunohistoquímicas en la población mestiza ecuatoriana afecta con cáncer de mama”; sea publicado en la biblioteca virtual de la Universidad de las Fuerzas Armadas – ESPE. Sangolquí, Mayo 2015 Nathaly Alexandra Arcos Villacís v DEDICATORIA Este trabajo lo dedico sobre todo a Dios, pues es Él quien ha sido mi principal guía, no sólo de esta investigación sino de mi vida. Él me ha fortalecido en todo tiempo y ha permitido que este trabajo llegue a buen fin. Dedico también este trabajo a mi Madre del Cielo y a mi familia: Pablo, María Alicia, mi madre y a mi padre, Antonio Arcos, que desde el cielo ha velado siempre por mí. vi AGRADECIMIENTO Agradezco a Dios por ser el motor de este y cada proyecto de mi vida, ya que juntos hemos caminado y avanzado cada etapa. Sin su misericordia, Su amor, Sus fuerzas, este trabajo no habría sido posible. Agradezco al Dr. Marcelo Grijalva, Director de este proyecto, y a la Dra. Patricia Jiménez, Codirectora, por su colaboración en esta investigación, su guía y su apoyo. Al Instituto de Investigaciones Biomédicas de la Universidad de las Américas (UDLA), que me permitió llevar a cabo esta investigación en su prestigioso laboratorio. Al Dr. César Paz-y-Miño, por dar acogida a este proyecto y permitirme adquirir experiencia laboral, de gran valor. Al Blgo. Andrés López, por su dirección en el proyecto, su paciencia y su guía en cada etapa del estudio. A cada uno de los profesionales y amigos que trabajan en el IIB, por compartir sus experiencias y permitirme adquirir nuevos conocimientos que serán de gran utilidad en mi futuro profesional. A mi familia, ya que sin ellos este sueño no tendría el mismo sentido, por su apoyo incondicional y su amor, que me empujan a no rendirme y dar mi mayor esfuerzo. A mis amigos, que han sido también un pilar de mi vida y me han animado en cada etapa a seguir adelante, confiando en Dios y en su respaldo. vii ÍNDICE DE CONTENIDOS CERTIFICACIÓN ............................................................................................ ii DECLARACIÓN DE RESPONSABILIDAD .................................................... iii AUTORIZACIÓN ............................................................................................ iv DEDICATORIA ............................................................................................... v AGRADECIMIENTO ...................................................................................... vi ÍNDICE DE CONTENIDOS ........................................................................... vii ÍNDICE DE CUADROS Y TABLAS ................................................................ xi ÍNDICE DE FIGURAS ................................................................................... xii ÍNDICE DE GRÁFICOS ............................................................................... xiii RESUMEN .................................................................................................... xv ABSTRACT .................................................................................................. xvi CAPÍTULO 1. INTRODUCCIÓN .....................................................................1 1.1 Formulación del problema ........................................................................ 1 1.2 Justificación del problema ........................................................................ 2 1.3 Objetivos de la investigación .................................................................... 3 1.3.1 Objetivo General ................................................................... 3 1.3.2 Objetivos Específicos ........................................................... 3 1.4 Marco Teórico........................................................................................... 5 1.4.1 Aspectos Generales: Cáncer ................................................ 5 1.4.1.1 Concepto y origen ................................................................................. 5 1.4.1.2 Incidencia mundial................................................................................. 6 1.4.1.3 Tumores .................................................................................................. 8 1.4.1.4 Genética del cáncer: oncogenes y genes supresores de tumores……. .............................................................................................................. 9 1.4.2. Cáncer de mama ................................................................ 10 1.4.2.1 Concepto............................................................................................... 10 viii 1.4.2.2 Epidemiología ...................................................................................... 10 1.4.2.3 Aspectos preliminares y factores de riesgo ................................... 12 1.4.2.3.1 Aspectos preliminares ..................................................................... 12 1.4.2.3.2 Factores de riesgo ........................................................................... 15 1.4.2.4 Clasificación del cáncer de mama .................................................... 16 1.4.2.4.1 Clasificación histológica .................................................................. 16 1.4.2.4.1.1 Carcinoma ductal.......................................................................... 17 1.4.2.4.1.2 Carcinoma lobulillar o lobular ..................................................... 20 1.4.2.4.1.3 Carcinoma inflamatorio ................................................................ 22 1.4.2.4.1.4 Tipos especiales de carcinoma invasivo del seno .................. 22 1.4.2.4.2 Clasificación molecular ................................................................... 24 1.4.2.4.2.1 Tipos luminal A, luminal B ........................................................... 24 1.4.2.4.2.2 Tipo HER2 ..................................................................................... 25 1.4.2.4.2.3 Tipo basal ...................................................................................... 25 1.4.2.5 Tratamiento .......................................................................................... 26 1.4.2.6 Genética del cáncer de mama .......................................................... 28 1.4.2.6.1 Mutaciones genéticas hereditarias................................................ 28 1.4.2.6.2 Mutaciones genéticas adquiridas .................................................. 29 1.4.2.7 Análisis del gen AKT1: implicaciones en el cáncer de mama………………………………………………………………………………..30 1.4.2.7.1 La vía de señalización PI3K/AKT .................................................. 30 1.4.2.7.2 AKT1 .................................................................................................. 31 1.4.2.7.2.1 Concepto y rol en el desarrollo del cáncer .............................. 31 1.4.2.7.2.2 Mutaciones en AKT1 .............................................................. 32 CAPÍTULO 2: MATERIALES Y MÉTODOS .................................................. 35 2.1 Revisión Bibliográfica ............................................................................. 35 2.2 Muestreo................................................................................................. 35 2.3 Diseño de Primers .................................................................................. 36 2.4 Análisis de historias clínicas ................................................................... 37 2.5 Análisis de laboratorio. Procesamiento de muestras .............................. 39 2.5.1 Extracción de ADN ............................................................. 39 2.5.1.1 Extracción de ADN a partir de sangre periférica ............................ 39 ix 2.5.1.2 Extracción de ADN a partir de tumores embebidos en parafina……………………………………………………………………………..40 2.5.2 Reacción en Cadena de la Polimerasa (PCR) .................... 42 2.5.2.1 Estandarización de PCR .................................................................... 42 2.5.3 Secuenciación .................................................................... 43 2.5.3.1 Purificación del producto PCR mediante columnas ....................... 43 2.5.3.2 Reacción de secuencia ...................................................................... 44 2.5.3.3 Purificación de la reacción de secuenciación ................................. 45 2.6 Análisis estadístico ................................................................................. 46 CAPÍTULO 3: RESULTADOS ....................................................................... 47 3.1 Recolección de muestras ....................................................................... 47 3.2 Clasificación de muestras tumorales ...................................................... 47 3.2.1 Tipo de carcinoma de mama .............................................. 47 3.2.2 Edad del paciente ............................................................... 48 3.2.3 Lateralidad .......................................................................... 49 3.2.4 Estadío tumoral................................................................... 50 3.2.5 Invasión a nódulo linfoide ................................................... 51 3.2.6 Grado tumoral/Histológico .................................................. 51 3.2.7 Receptores hormonales ...................................................... 52 3.2.8 Tipo de cáncer en base a receptores hormonales .............. 53 3.2.9 Márgenes quirúrgicos ......................................................... 54 3.3 Determinación de Genotipos .................................................................. 54 3.3.1 Extracción de ADN ............................................................. 54 3.3.2 Reacción en cadena de polimerasa (PCR) ......................... 55 3.3.2.1 Polimorfismos AKT1-E17 ................................................................... 56 3.3.2.2 Polimorfismo AKT1-L357P................................................................. 56 3.3.3 Secuenciación capilar ......................................................... 57 3.3.4 Análisis estadístico ............................................................. 60 x 3.3.4.1 Frecuencias genotípicas y alélicas ............................................. 60 3.3.4.2 Análisis de características clínicas en pacientes con cáncer de mama y asociación con el tipo de cáncer .................................................... 61 3.3.4.3 Asociación del polimorfismo E17K con características clínicas de pacientes con cáncer de mama....................................................................... 63 CAPÍTULO 4: DISCUSIÓN ........................................................................... 66 CAPÍTULO 5: CONCLUSIONES .................................................................. 72 CAPÍTULO 6: RECOMENDACIONES .......................................................... 73 CAPÍTULO 7. BIBLIOGRAFÍA ...................................................................... 74 CAPÍTULO 8. ANEXOS ................................................................................ 80 xi ÍNDICE DE CUADROS Y TABLAS Cuadro 1. Diferencias entre tumores benignos y malignos………………..…8 Cuadro 2. Subtipos de cáncer de mama determinados por perfiles de expresión génica. ......................................................................................... 26 Tabla 1. Incidencia y mortalidad por cáncer en población general ................ 6 Tabla 2. Criterios de selección empleados en el estudio............................. 35 Tabla 3. Descripción de las variables de estudio......................................... 37 Tabla 4. Condiciones estandarizadas para la PCR ..................................... 43 Tabla 5. Condiciones estandarizadas para la reacción de secuencia ......... 45 Tabla 6. Distribución de frecuencias genotípicas y alélicas del polimorfismo E17K ............................................................................................................ 60 Tabla 7. Distribución de características clínicas en pacientes con CM en función del tipo de cáncer (clasificación molecular) ..................................... 62 Tabla 8. Distribución de características clínicas, histopatológicas e inmunohistoquímicas en pacientes con CM en función del genotipo presentado, para la variante polimórfica E17K ............................................ 64 xii ÍNDICE DE FIGURAS Figura 1. Pictograma de la incidencia de cáncer de mama en el mundo. ... 11 Figura 2. Tasas estimadas de incidencia y mortalidad del cáncer de mama en el mundo. ................................................................................................ 12 Figura 3. Tejido mamario normal. ............................................................... 13 Figura 4. Ganglios linfáticos relacionados al seno. ..................................... 14 Figura 5. Tipos de tumores en cáncer de mama ......................................... 17 Figura 6. Carcinoma ductal in situ ............................................................... 18 Figura 7. Grados de carcinoma ductal hasta la generación de CDI ............ 19 Figura 8. Carcinoma lobulillar in situ. .......................................................... 20 Figura 9. Carcinoma lobulillar invasivo ........................................................ 21 Figura 10. Carcinoma inflamatorio. ............................................................. 22 Figura 11. Células tumorales en carcinoma mucinoso................................ 23 Figura 12. Células tumorales en carcinoma papilar .................................... 24 Figura 13. Esquema de la vía de señalización PI3K/AKT ........................... 31 Figura 14.Electroforesis de muestras de ADN extraído, de la población de estudio en geles de agarosa al 2%. ............................................................. 55 Figura 15. Electroforesis del fragmento de ADN de interés para el análisis del SNP E17, de 198 pb, en un gel de agarosa al 2% ................................. 56 Figura 16. Electroforesis del fragmento de ADN de interés para el análisis del SNP L357P, de 142 pb, en un gel de agarosa al 2% ............................. 57 xiii Figura 17. Identificación del polimorfismo L357P por secuenciación capilar y distribución en población total de estudio ................................................. 58 Figura 18. Identificación del polimorfismo E17K por secuenciación capilar y distribución en población total de estudio.. ............................................... 59 ÍNDICE DE GRÁFICOS Gráfico 1. Distribución de la población de estudio afecta con CM según el tipo de carcinoma......................................................................................... 48 Gráfico 2. Distribución de la población de estudio afecta con CM según la edad del paciente......................................................................................... 49 Gráfico 3. Distribución de la población de estudio afecta con CM en base a la lateralidad.............................................................................................. 49 Gráfico 4. Distribución de la población de estudio afecta con CM según el estadío tumoral ............................................................................................ 50 Gráfico 5. Distribución de la población de estudio afecta con CM en función de la invasión de células tumorales a nódulo linfoide...................... 51 Gráfico 6. Distribución de la población de estudio afecta con CM según el grado tumoral/histológico ............................................................................. 52 Gráfico 7. Distribución de la población de estudio afecta con CM en función de la presencia de receptores hormonales: Progesterona, Estrógeno, HER2. ........................................................................................ 53 Gráfico 8. Distribución de la población de estudio afecta con CM en función del tipo de cáncer en base a receptores hormonales. (TNBC: Triple Negative Breast Cancer, o Tipo Basal) .............................................. 53 xiv Gráfico 9. Distribución de la población de estudio afecta con CM en función de la presencia de células cancerígenas en bordes quirúrgicos ..... 54 xv RESUMEN En los últimos 8 años se han reportado estudios que indican una creciente evidencia que la quinasa AKT1 es un miembro central de una de las rutas más frecuentes de activación y proliferación del cáncer. Sin embargo, las mutaciones en el gen AKT1 no han sido ampliamente estudiadas. En esta investigación, el objetivo fue analizar los polimorfismos genéticos E17K, E17S y L357P del gen AKT1, y su asociación con características histopatológicas e inmunohistoquímicas en la población mestiza ecuatoriana afecta con cáncer de mama. Se llevó a cabo un estudio retrospectivo caso-control, en el que se recolectaron muestras de tejido mamario de 100 casos y muestras de sangre periférica de 140 controles. Se analizaron las historias clínicas de cada paciente y se registraron las características clínicas de cada uno de ellos. En el análisis de laboratorio, se realizó la extracción de ADN de cada muestra, la amplificación mediante PCR y finalmente secuenciación capilar. La variante polimórfica E17K fue detectada en (2/81) el 2.5% de la población; se encontraron dos genotipos homocigóticos en el caso del análisis de este polimorfismo: G/G y A/A (homocigoto raro). En cuanto a la variante L357P, sólo se encontró un genotipo: T/T. No se verificaron casos o controles con la variante E17S. A partir de estos datos, se generó un análisis de asociación de las distintas características histopatológicas e inmunohistoquímicas de los pacientes, con los genotipos encontrados, para el polimorfismo E17K; en ningún caso, se pudo afirmar la asociación del genotipo AA con alguna de las distintas características clínicas estudiadas (P>0.05). PALABRAS CLAVE: • AKT1 • CÁNCER • POLIMORFISMO GENÉTICO • CARACTERÍSTICAS HISTOPATOLÓGICAS • CARACTERÍSTICAS INMUNOHISTOQUÍMICAS xvi ABSTRACT In the last eight years, different studies have reported mounting evidence that kinase AKT1 is a central member of one of the most common pathways of cancer activation and proliferation. However, mutations in AKT1 gene have not been widely studied. The aim of this study was to analyze genetic polymorphisms E17K, E17S and L357P of AKT1 gene and their association with histopathological and immunohistochemical features in the Ecuadorian Mestizo population affected with breast cancer. For this purpose, a retrospective case-control study was conducted in which breast tissue samples from 100 breast cancer patients and peripheral blood samples from 140 controls were collected. The medical records of each patient were analyzed and clinical features were recorded. In laboratory analysis: DNA extraction, PCR and capillary sequencing were performed. The polymorphic variant E17K was detected in (2/81) 2.5% of affected population. In the analysis of this polymorphism, two homozygous genotypes G/G and A/A (rare homozygous) were found. Only one genotype (T/T) was found for the L357P variant. No cases or controls with E17S variant were found. From these data, an association analysis for the different histopathological and immunohistochemical features of patients and E17K genotypes, was performed. We found no association for AA genotype and clinical picture (P> 0.05). KEYWORDS: • AKT1 • CANCER • GENETIC POLYMORPHISM • HISTOPATHOLOGIC FEATURES • IMMUNOHISTOCHEMICAL FEATURES 1 CAPÍTULO 1. INTRODUCCIÓN 1.1 Formulación del problema En el Ecuador, el cáncer tiene una incidencia creciente y es considerado la segunda causa de mortalidad general después de las enfermedades cardiovasculares. En América Latina y el Caribe, el cáncer causa aproximadamente el 20% de fallecimientos por enfermedades crónicas no transmisibles. Se prevé que para el año 2020, existan alrededor de 833 mil defunciones en esta región como consecuencia de este grupo de enfermedades, lo que significa un incremento del 33% en relación a 1990 (MSP, Ministerio de Salud garantiza acceso a la salud de pacientes con cáncer, 2014). La caracterización del genoma, ha beneficiado a diversas áreas de la biomedicina como la oncología, ha contribuido tanto en la comprensión de los mecanismos básicos de los procesos de transformación neoplásica, como en el desarrollo de nuevos servicios para un mejor pronóstico y evaluación del riesgo en pacientes oncológicos (Hidalgo & Jiménez, 2009). Además, las investigaciones en líneas de cáncer se han multiplicado a nivel mundial, con el fin de encontrar posibles métodos de diagnóstico en fases tempranas del desarrollo de la enfermedad, y posibles dianas terapéuticas para su cura. Un método de investigación muy utilizado es el estudio de polimorfismos genéticos. A partir de los datos generados con estos estudios en pacientes con cáncer, se ha logrado entender parcialmente los mecanismos de susceptibilidad a esta enfermedad y han permitido la estimación de las frecuencias alélicas y análisis de asociación de polimorfismos con algunos tipos de cáncer, entre ellos el de mama (Checa, 2007). Sin embargo, aún deben realizarse diversos estudios de este tipo, que permitan entender el complejo mecanismo de desencadenamiento de la 2 enfermedad y cómo asociar estos conocimientos para el desarrollo de fármacos o metodologías terapéuticas aplicativas. 1.2 Justificación del problema El cáncer de mama es uno de los más frecuentes entre las mujeres de nuestro país, 30 de cada 100000 personas son diagnosticadas con esta enfermedad cada año y aproximadamente la mitad de ellas se detectan en etapas avanzadas de la enfermedad, lo cual disminuye las posibilidades de curación y supervivencia. Durante el año 2011, se realizaron 448.350 consultas de detección oportuna de cáncer de mama en el Ministerio de Salud Pública (MSP), sin embargo, esta enfermedad sigue siendo la tercera causa de muerte por cáncer en las mujeres del Ecuador (MSP, 2012). Ante esta creciente situación, las diferentes investigaciones a nivel oncológico se han incrementado; en el Ecuador a pesar de la urgencia de esta problemática, existen pocos estudios generados en líneas de cáncer de mama. En Ecuador, hasta el momento se han realizado estudios epidemiológicos multicéntricos para determinar marcadores moleculares de cáncer (SEP, 2013); otras investigaciones han buscado determinar la frecuencia de este cáncer en el País (Córdova & Coello, 2011) y otras, han empezado a centrarse en el estudio de polimorfismos relacionados con la enfermedad (Carrera, 2008). Estas investigaciones son el inicio de nuevos estudios que deberán llevarse a cabo, permitiendo la compresión de los mecanismos que generan susceptibilidad a la enfermedad y consecuentemente el desarrollo de metodologías de pronóstico y tratamiento oportunos. 3 Es fundamental iniciar con esta línea de investigación con el fin de conocer cómo las variantes polimórficas del gen AKT1 (E17S, E17 K y L357P) se presentan en la población mestiza ecuatoriana, comparando además sus resultados con poblaciones extranjeras estudiadas hasta el momento. 1.3 Objetivos de la investigación 1.3.1 Objetivo General • Analizar los polimorfismos genéticos E17K, E17S y L357P del gen AKT1, y su asociación con características histopatológicas e inmunohistoquímicas en la población mestiza ecuatoriana afecta con cáncer de mama. 1.3.2 Objetivos Específicos • Establecer frecuencias alélicas y genotípicas de la población mestiza ecuatoriana relacionadas con los polimorfismos genéticos E17K, E17S y L357P del gen AKT1. • Comparar las frecuencias alélicas y genotípicas obtenidas entre individuos afectos y controles. • Asociar las características histopatológicas e inmunohistoquímicas con las variantes genéticas estudiadas. • Relacionar la presencia de los polimorfismos genéticos estudiados con el riesgo a desarrollar cáncer de mama. 4 • Evaluar el potencial de los polimorfismos del gen AKT1, como biomarcadores moleculares para el diagnóstico preventivo de esta enfermedad. 5 1.4 Marco Teórico 1.4.1 1.4.1.1 Aspectos Generales: Cáncer Concepto y origen El cáncer se define como una enfermedad o el conjunto de ellas, caracterizado por una proliferación continua y descontrolada de células anormales. Puede originarse a partir de cualquier tipo de célula en cualquier tejido corporal, y se clasifica en función del tejido y de la célula de origen (National Cancer Institute [NCI], 2012). Existen distintas formas del cáncer, entre las que se destacan tres subtipos: los sarcomas, procedentes del tejido conectivo como huesos, cartílagos, nervios, vasos sanguíneos, músculos y tejido adiposo. Los carcinomas, que proceden de tejidos epiteliales como la piel o los epitelios que tapizan las cavidades y órganos corporales, y de los tejidos glandulares de la mama y de la próstata. Finalmente, en el tercer subtipo se encuentran las leucemias y los linfomas, que incluyen los cánceres de los tejidos formadores de las células sanguíneas, producen inflamación de los ganglios linfáticos, invasión del bazo y de la médula ósea, y sobreproducción de células blancas inmaduras (Instituto Nacional del Cáncer, 2011). En general, el cáncer se produce por alteraciones celulares que se han generado por una modificación en la clave cromosómica, causando que las células reciban mensajes erróneos, que provocan el descontrol de su propio desarrollo y que las células dejen de cumplir las funciones para las que fueron inicialmente programadas (Instituto Nacional de Cancerología, 2004). La enfermedad surge de una serie de cambios genéticos y epigenéticos, que le otorgan a la célula cancerosa su comportamiento maligno. En el nivel más simple, las células cancerosas se dividen en momentos inadecuados y no responden a las señales de alto y siga, que regulan el ciclo celular (mensajes químicos). 6 Posteriormente, cuando se transforman en células tumorales, adquieren una variedad de características que las ayudan a sobrevivir, proliferar, invadir y crecer, como: angiogénesis, cambios en el citoesqueleto, reducción de la adhesión celular, aumento de la movilidad y desarrollan acciones fisiológicas que bloquean los intentos del cuerpo de eliminar las células cancerosas mediante respuestas inmunológicas (NCI, 2012). 1.4.1.2 Incidencia mundial El cáncer puede ocurrir en aproximadamente uno de cada tres individuos. Las mutaciones del ADN normalmente suceden con una frecuencia de 1 en cada 20 millones por gen por división celular, por lo que sería lógico asumir que las poblaciones humanas a nivel mundial muestran esta misma frecuencia, sin embargo varios factores intervienen de manera influyente en el incremento de la incidencia de cáncer en algunas poblaciones, ya sean factores hereditarios o ambientales (Ion Baba, 2007). A continuación se muestran las tasas de incidencia y mortalidad por cáncer para el año 2012, en la Tabla 1 (World Health Organization [WHO], 2012). Tabla 1. Incidencia y mortalidad por cáncer en población general en el año 2012 Incidencia Cáncer Mortalidad Número de personas (%) Número de personas (%) Cavidad oral, labio 300373 2.1 145353 1.8 Nasofaringe 86691 0.6 50831 0.6 Otros cánceres ORL (no laringe) 142387 1.0 96105 1.2 Esófago 455784 3.2 400169 4.9 Estómago 951594 6.8 723073 8.8 7 Colorrectal 1360602 9.7 693933 8.5 Hígado 782451 5.6 745533 9.1 Vesícula biliar 178101 1.3 142823 1.7 Páncreas 337872 2.4 330391 4.0 Laringe 156877 1.1 83376 1.0 Pulmón 1824701 13.0 1589925 19.4 Melanoma cutáneo 232130 1.7 55488 0.7 Sarcoma de kaposi 44247 0.3 26974 0.3 Mama 1671149 11.9 521907 6.4 Cervix uterino 527624 3.8 265672 3.2 Cuerpo uterino 319605 2.3 76160 0.9 Ovario 238719 1.7 151917 1.9 Próstata 1094916 7.8 307481 3.7 Testículo 55266 0.4 10351 0.1 Riñón 337860 2.4 143406 1.7 Vejiga 429793 3.1 165084 2.0 Cerebro, sistema nervioso 256213 1.8 189382 2.3 Tiroides 298102 2.1 39771 0.5 Linfoma de Hodgkin 65950 0.5 25469 0.3 Linfoma no-Hodgkin 385741 2.7 199670 2.4 Mieloma múltiple 114251 0.8 80019 1.0 Leucemia 351965 2.5 265471 3.2 Todos los cánceres excluidos los cánceres de piel no melanoma 14067894 100.0 8201575 100.0 Fuente: (WHO, 2012) 8 1.4.1.3 Tumores El crecimiento anormal de las células puede formar, en la mayoría de los casos, masas de tejidos llamados tumores (Instituto Nacional de Cancerología, 2004). Es esencial diferenciar tumores benignos de malignos. En general, los tumores malignos tienen capacidad invasiva y proliferativa hacia otros tejidos, es decir poseen habilidad para desarrollar angiogénesis y metástasis, diferencia de los a benignos, cuya mayor complicación consiste en la posibilidad de ocasionar presión en tejidos y órganos sanos (American Cancer Society [ACS], 2014). En el Cuadro 1 se pueden observar las diferencias entre los tumores beningos y malignos (Agency for toxic substances and disease registry [ATSDR], 2005). Cuadro 1. Diferencias entre tumores benignos y malignos Características Tumores benignos Tasa de crecimiento Lenta; puede interrumpirse o retroceder Rápida, autónoma; generalmente no interrumpe o retrocede Se expande y desplaza Invade, destruye y reemplaza Crecen de manera ordenada Crecen desordenadamente Diferenciación Las células tumorales se asemejan a las células originales Las células tumorales tal vez no se asemejan a las células originales Metástasis No Sí Tipo de crecimiento Tumores malignos 9 Efectos en la salud Normalmente no producen efectos graves como la muerte. Pueden ocasionar la muerte en caso de diagnóstico y tratamiento tardíos. Fuente: (ATSDR, 2005) 1.4.1.4 Genética del cáncer: oncogenes y genes supresores de tumores Las más de 30.000 millones de células que constituyen el organismo del ser humano nacen, crecen, se dividen y mueren bajo la estricta vigilancia del ADN. La reproducción celular está supervisada por determinados sistemas de control, en los que participan una gran cantidad de moléculas como proteínas, enzimas, factores de crecimiento y genes que se activan y desactivan con gran precisión. El más mínimo error que se presente en uno de estos sistemas de control puede acarrear una tragedia celular (Ríos & Hernández, 2001). Las células de un tumor descienden de una ancestral común, que en algún momento, inició un programa de división indebido. La transformación maligna de una célula acontece por acumulación de mutaciones en unos genes específicos, los cuales son la clave molecular para entender las raíces del cáncer (Ríos & Hernández, 2001). En los últimos años se ha aceptado universalmente que los tumores surgen debido a la acumulación de mutaciones en genes encargados de controlar la proliferación, diferenciación o muerte celular. Estos genes pueden clasificarse en dos grandes grupos: 1) oncogenes y 2) genes supresores de tumores (Santos, 2013). 10 Los protooncogenes ejercen efectos de control positivo sobre la proliferación celular, mientras que los genes supresores de tumores cumplen un papel regulador negativo en tal proceso, lo que determina que su mutación en procesos tumorales les confiera un carácter recesivo (Alonso, 2013). De este modo, hoy en día, se considera que en el inicio del cáncer existe un desequilibrio de la balanza entre la proliferación celular (oncogenes = acelerador) y la represión de ésta (genes supresores de tumores = freno). Además de la activación de oncogenes e inactivación de genes supresores, las alteraciones genéticas que provocarían el cáncer pueden tener otros orígenes como: alteración en los genes de reparación del ADN, alteración de genes relacionados con la apoptosis, y otros mecanismos tales como la activación de telomerasa, genes interruptores, entre otros. (Alonso, 2013). 1.4.2. 1.4.2.1 Cáncer de mama Concepto El cáncer de mama se considera como la proliferación de células malignas en tejidos del seno (células epiteliales que revisten los conductos o lobulillos de la mama). Ocurre casi por completo en las mujeres, pero los hombres también la pueden padecer (ACS, 2014). 1.4.2.2 Epidemiología El cáncer de mama es el cáncer más frecuente en las mujeres, tanto en los países desarrollados como en los países en desarrollo. Los casos de cáncer de mama han aumentado en un 20% desde el 2008, convirtiéndolo en 11 el segundo tipo de cáncer más común en el mundo y el más frecuente entre mujeres (OMS, 2012). Del total de casos de cáncer diagnosticados en 2012, el 25 % fueron de mama, de los que 883.000 se registraron en países menos desarrollados y 794.000 en el mundo desarrollado, indicando grandes diferencias de atención entre ellos (El Universo, 2013). Esta enfermedad es una de las principales causas de muerte en los países menos desarrollados, lo que se debe en parte a un cambio en los estilos de vida que está ocasionando un incremento en la incidencia, y a que los avances clínicos para combatir la enfermedad no están alcanzando a las mujeres que viven en estas regiones (OMS, 2012). Figura 1. Pictograma de la incidencia de cáncer de mama en el mundo. Fuente: (WHO, 2012). 12 Figura 2. Tasas estimadas de incidencia y mortalidad del cáncer de mama en el mundo. Fuente: (WHO, 2012). La edad de máxima incidencia está por encima de los 50 años, pero aproximadamente un 6% se diagnostica en mujeres menores de 35 años (Sociedad Española de Oncología Médica [SEOM], 2013). 1.4.2.3 Aspectos preliminares y factores de riesgo 1.4.2.3.1 • Aspectos preliminares El seno Consiste principalmente en lobulillos (glándulas productoras de leche), conductos (llevan la leche desde los lobulillos al pezón) y estroma (el tejido 13 adiposo y el tejido conectivo que rodean los conductos y los lobulillos, los vasos sanguíneos y los vasos linfáticos) (Ver Figura 3) (ACS, 2014). Figura 3. Tejido mamario normal. Fuente: (ACS, 2014). La mayoría de los cánceres de seno comienza en las células que recubren los conductos. Algunos cánceres de seno se originan en las células que recubren los lobulillos, mientras que un pequeño número se origina en otros tejidos. • Sistema linfático del seno El sistema linfático forma parte del sistema de defensa del cuerpo y está compuesto de vasos linfáticos y de ganglios linfáticos. Los vasos linfáticos son tubos delgados que transportan glóbulos blancos y linfa. Los vasos linfáticos conducen a órganos pequeños en forma de frijol llamados ganglios linfáticos. Los ganglios linfáticos se encuentran cerca del seno, bajo el brazo, arriba de la clavícula, en el pecho y en otras partes del cuerpo (NCI, 2014). Los ganglios linfáticos filtran sustancias en la linfa para ayudar a combatir infecciones y enfermedades. Además, almacenan glóbulos blancos, llamados linfocitos, los cuales combaten enfermedades e infecciones (NCI, 2014). 14 Figura 4. Ganglios linfáticos relacionados al seno. Fuente: (ACS, 2014). Si las células cancerosas se han propagado a los ganglios linfáticos, existe una probabilidad mayor de que las células también hayan alcanzado el torrente sanguíneo y se hayan propagado (metástasis) a otros lugares del cuerpo y por tanto, a otros órganos (ACS, 2014). Debido a esto, si se detecta invasión de células cancerígenas en uno o más ganglios linfáticos, el pronóstico empeora y el plan de tratamiento es poco eficaz. Aun así, no todas las mujeres con células cancerosas en sus ganglios linfáticos presentan metástasis, y es posible también que algunas mujeres que no tengan células cancerosas en sus ganglios linfáticos, luego desarrollen metástasis (ACS, 2014). 15 1.4.2.3.2 Factores de riesgo Se han identificado algunos factores de riesgo para el desarrollo del cáncer de mama, como los que se mencionan a continuación (SEOM, 2013). • Edad: el riesgo se incrementa con la edad. La mayoría de casos ocurre en mujeres mayores de 60 años. Aproximadamente uno de ocho cánceres de seno se detecta en mujeres menores de 45 años de edad, mientras que aproximadamente dos de tres cánceres invasivos del seno se encuentran en mujeres de 55 años o más. • Raza: más frecuente en mujeres de raza blanca. En general, se sabe que las mujeres de raza blanca tienen una probabilidad ligeramente mayor de padecer cáncer de seno que las mujeres de raza negra, aunque éstas últimas tienen una mayor probabilidad de morir de este cáncer. Sin embargo, en las mujeres menores de 45 años de edad, el cáncer de seno es más común en las mujeres de raza negra. Las mujeres asiáticas, hispanas e indias americanas tienen un menor riesgo de padecer y morir de cáncer de mama. • Alcohol: su consumo excesivo aumenta el riesgo de padecer cáncer de seno. El riesgo aumenta con la cantidad de alcohol consumido. En comparación con las mujeres que no ingieren alcohol, las que consumen una bebida alcohólica diaria tienen un aumento muy ligero en el riesgo. • Aparición temprana de la primera regla y/o menopausia tardía: las mujeres que han tenido más ciclos menstruales debido a que comenzaron a menstruar temprano (antes de los 12 años) y/o experimentaron tarde la menopausia (después de los 55 años) tienen un riesgo ligeramente mayor de padecer cáncer de seno. Este aumento en el riesgo podría deberse a una exposición más prolongada a las hormonas estrógeno y progesterona durante la vida de la persona. 16 • Historia familiar: aumenta el riesgo si un familiar de primer grado (madre, hermana o hija) ha padecido la enfermedad. Si la enfermedad apareció en familiares más lejanos, el riesgo es menor. • Antecedentes personales: una mujer que ha tenido cáncer de mama, tiene más riesgo de padecer otro cáncer en la mama contralateral. • Enfermedades previas de la mama: algunas enfermedades de la mama como la hiperplasia atípica o el carcinoma lobulillar in situ pueden aumentar el riesgo. • Primer embarazo tardío o nuliparidad (ausencia de embarazo): Las mujeres que no han tenido hijos o aquellas que tuvieron su primer hijo después de los 30 años tienen en general un riesgo ligeramente mayor de padecer cáncer de seno. • Terapia hormonal sustitutiva prolongada: Durante el tratamiento hormonal sustitutivo existe un mayor riesgo de carcinoma de mama, si bien depende de los tipos de preparados, y vuelve al riesgo habitual una vez que cesa el tratamiento. 1.4.2.4 1.4.2.4.1 Clasificación del cáncer de mama Clasificación histológica Existen diversos tipos de cáncer de mama, como se muestra en la Figura 5. En algunos casos, un solo tumor del seno puede ser una combinación de estos tipos o ser una mezcla de cáncer in situ e invasivo (INC, 2014). 17 El tipo más común de cáncer de seno es el carcinoma ductal. Este cáncer empieza en las células que revisten los conductos del seno. Casi 7 de cada 10 mujeres con cáncer de seno tienen carcinoma ductal. El segundo tipo más común de cáncer de seno es el carcinoma lobulillar. Este cáncer empieza en un lobulillo del seno. Casi 1 de cada 10 mujeres con cáncer de seno tiene carcinoma lobulillar. Otras mujeres tienen una mezcla de los tipos de carcinoma ductal y lobulillar o tienen un tipo menos común de cáncer de seno (ACS, 2014). Figura 5. Tipos de tumores en cáncer de mama. Fuente: (Kumar, Aster, Fausto, & Abbas, 2010) 1.4.2.4.1.1 • Carcinoma ductal In situ El carcinoma ductal in situ (CDIS) es el tipo más común de cáncer de mama no invasivo. Ductal significa que el cáncer comienza dentro de los conductos lácteos, como se observa en la Figura 6, carcinoma se refiere a cualquier cáncer que se genera en la piel u otros tejidos (incluyendo tejido mamario) que cubren o revisten los órganos internos, e “in situ” significa en el lugar de origen (BreastCancer, 2014). 18 CDIS se considera un precáncer, ya que en algunos casos se puede convertir en un cáncer invasivo. Sin embargo, actualmente, no existe una metodología que permita conocer con certeza los casos que se convertirán en cánceres invasivos (ACS, 2014). Muy pocos casos de CDIS se presentan como una masa palpable; 80% se diagnostican a través de una mamografía. El CDIS abarca un grupo heterogéneo de lesiones histopatológicas que se clasifican en varios subtipos, principalmente de acuerdo con su perfil arquitectónico: micropapilar, papilar, sólido, cribiforme y comedón. Este último, se compone de células cuya citología parece maligna, con la presencia de núcleos de grado alto, pleomorfismo y abundante necrosis luminal central, parece ser un tumor más dinámico, con una probabilidad mayor de desembocar en carcinoma ductal invasivo (INC, 2014). Figura 6. Carcinoma ductal in situ. Fuente: (ACS, 2014). • Invasivo o infiltrante El carcinoma ductal invasivo (CDI), es el tipo más común de cáncer de mama. 19 Este cáncer comienza en un conducto lácteo del seno, penetra a través de la pared del conducto y crece en el tejido adiposo del seno (Ver Figura 7). En este punto puede tener la capacidad de propagarse hacia otras partes del cuerpo a través del sistema linfático y el torrente sanguíneo (ACS, 2014). Aunque este carcinoma puede afectar a mujeres de cualquier edad, resulta más común a medida que la mujer envejece. Según la Sociedad Americana del Cáncer, aproximadamente dos tercios de las mujeres que son diagnosticadas con cáncer de mama invasivo tienen aproximadamente 55 años de edad (BreastCancer, 2014). Figura 7. Grados de carcinoma ductal hasta la generación de CDI. Fuente: (BreastCancer, 2014). 20 1.4.2.4.1.2 • Carcinoma lobulillar o lobular In situ La expresión carcinoma lobulillar in situ (CLIS) no hace referencia a un verdadero cáncer. El término neoplasia lobulillar es más apropiado para esta lesión, ya que no se conoce como una lesión premaligna sino, más bien, como un marcador que identifica a mujeres con un riesgo mayor de presentar posteriormente de cáncer invasivo de mama (INC, 2014). En este carcinoma, las células anómalas comienzan a crecer en los lobulillos, es decir en las glándulas productoras de leche ubicadas en los extremos de los conductos mamarios (Ver Figura 8). El crecimiento anómalo permanece dentro del lobulillo y no se propaga hacia los tejidos circundantes. Las personas diagnosticadas con CLIS suelen tener más de un lobulillo afectado (BreastCancer, 2014). Figura 8. Carcinoma lobulillar in situ. Fuente: (ACS, 2014). • Invasivo o infiltrante El carcinoma lobulillar invasivo (CLI) es el segundo tipo de cáncer de mama más común después del carcinoma ductal invasivo. De acuerdo a la 21 Sociedad Americana del Cáncer, más de 180 000 mujeres en los Estados Unidos descubren anualmente que padecen un cáncer de mama invasivo. Alrededor del 10 % de estos casos de cáncer son carcinomas lobulares invasivos (BreastCancer, 2014) Este tipo de carcinoma comienza en las glándulas productoras de leche (lobulillos) y se propaga hacia los tejidos mamarios que lo rodean (Ver Figura 9). Con el paso del tiempo, un carcinoma lobular invasivo puede propagarse hacia los ganglios linfáticos y posiblemente otras zonas del cuerpo (ACS, 2014). Los casos de CLI tienden a aparecer en edades más avanzadas que los carcinomas ductales invasivos, alrededor de los 60 años. Algunas investigaciones sugieren que el uso de terapias de reemplazo hormonal durante y después de la menopausia puede incrementar el riesgo de desarrollar este carcinoma (BreastCancer, 2014). Figura 9. Carcinoma lobulillar invasivo. Fuente: (BreastCancer, 2014). 22 1.4.2.4.1.3 Carcinoma inflamatorio Es un cáncer poco común (1-3% de los carcinomas en seno). Las células tumorales infiltran los vasos linfáticos y la piel. La mama está globalmente enrojecida y caliente, como si estuviese inflamada, porque las células tumorales bloquean los vasos linfáticos de la piel (Ver Figura 10) (Asociación Española contra el Cáncer [AEEC], 2013). El seno afectado puede volverse más grande, firme, sensible, o puede presentar picazón. En sus etapas iniciales, se lo confunde con una infección en el seno (mastitis), y se trata como una infección administrando antibióticos. Si los síntomas son causados por cáncer, éstos no se aliviarán, y una biopsia encontrará células cancerosas. Este tipo de cáncer suele tener una mayor probabilidad de propagación y un peor pronóstico que el típico cáncer ductal invasivo o lobulillar invasivo (ACS, 2014). Figura 10. Carcinoma inflamatorio. Fuente: (ACS, 2014). 1.4.2.4.1.4 • Tipos especiales de carcinoma invasivo del seno Carcinoma mucinoso Es una forma poco frecuente de carcinoma ductal invasivo. Representa cerca del 2-3 % de todos los casos de cáncer de mama 23 Es un carcinoma ductal invasor de bajo grado de malignidad que se caracteriza por células tumorales inmersas en cúmulos de moco extracelular (Ver Figura 11). Comúnmente, esta mucosidad recubre la mayoría de las superficies internas del cuerpo humano, sin embargo, en el carcinoma mucinoso, la mucosidad se convierte en una parte principal del tumor y rodea las células del cáncer de mama (ACS, 2014). El carcinoma mucinoso tiene menos probabilidad de propagarse a los ganglios linfáticos que otros tipos de cáncer de mama, presentando un mejor pronóstico (BreastCancer, 2014). Figura 11. Células tumorales en carcinoma mucinoso. Fuente: (Moreno, 2010). • Carcinoma papilar Son carcinomas poco frecuentes y representan menos del 1-2 % de los casos de cáncer de mama invasivo. Generalmente tiene un borde bien definido y está formado por prolongaciones pequeñas en forma de dedos, como se muestra en la Figura 12. Con frecuencia, es de grado 2 (grado moderado) en una escala de 1 a 3. En la mayoría de los casos de carcinoma papilar invasivo, también está presente un carcinoma ductal in situ (CDIS) (BreastCancer, 2014). 24 Figura 12. Células tumorales en carcinoma papilar. Fuente: (Pérez, Mariángel, & Poblete, 2004). 1.4.2.4.2 Clasificación molecular La investigación sobre los patrones de expresión genética ha sugerido algunas nuevas formas de clasificar los cánceres de seno. Recientemente, se ha generado una clasificación basada en las características moleculares, que puede ser mejor al predecir el pronóstico y la respuesta de varios tipos de tratamiento contra el cáncer de seno. Se han identificado cuatro tipos básicos de cánceres de mama: Luminal A, Luminal B, Basal y Her2/neu (ACS, 2014). 1.4.2.4.2.1 Tipos luminal A, luminal B Los tipos luminales son receptor de estrógeno positivo, usualmente de bajo grado, que tiende a crecer lentamente. Los patrones de expresión genética de estos tipos son similares a las células normales que recubren los conductos y las glándulas del seno. Los cánceres luminal B, generalmente crecen con un poco más de rapidez que los luminal A (INC, 2014). Los tumores Luminal A tienden a ser RE+ y/o RP+ y HER2- y menos agresivos que otros subtipos. Normalmente, se asocian con un mejor pronóstico a corto plazo, ya que la expresión de los receptores hormonales predice una respuesta favorable al tratamiento hormonal. Sin embargo, la supervivencia a largo plazo es similar o incluso menor que en otros subtipos (American Cancer Society, 2013). 25 Los tumores Luminal B, son en su mayoría RE+ y/o RP+ y se distinguen de los demás subtipos, por la expresión de HER2. Del 10-20% de casos de cáncer de mama, son Luminal B (American Cancer Society, 2013). 1.4.2.4.2.2 Tipo HER2 Estos cánceres tienen copias adicionales del gen HER2, que codifica una proteína promotora del crecimiento, y de varios otros genes. Los tumores, tienden a crecer más rápidamente y a diseminarse con mayor agresividad que otros subtipos, por lo que se asocian con un pronóstico desfavorable a corto plazo, en comparación con tumores con RE+ (ACS, 2014). Sin embargo, el uso de terapias dirigidas, como el trastuzumab (Herceptin), han invertido gran parte del impacto provocado debido al pronóstico de sobre-expresión de HER2, permitiendo tratar exitosamente el cáncer en algunos casos (American Cancer Society, 2013). 1.4.2.4.2.3 Tipo basal La mayoría de estos cánceres son de los llamados tipo triple negativo, es decir, carecen de los receptores de estrógeno o progesterona y tienen cantidades normales de HER2. Los patrones de expresión genética de estos cánceres son similares a células en las capas basales más profundas de los conductos y las glándulas del seno. Este tipo es más común entre las mujeres con mutaciones genéticas BRCA1 (ACS, 2014). 26 Cuadro 2. Subtipos de cáncer de mama determinados por perfiles de expresión génica. Comportamiento Subtipo Inmunofenotipo Luminal A RE (+) y/o RP (+); HER2/neu (-) • Subtipo más común y menos agresivo. Buen pronóstico. • Bajo grado histológico. Respuesta hormonal. • Asociado a incremento de edad. Luminal B RE (+) y/o RP (-); HER2/neu (+) • Similar al Subtipo Luminal A. Peor resultado que el Subtipo Luminal A. • Más frecuentemente RE (+) / RP (-). Basal RE (-); RP (-); HER2/neu (-) CK 5/6 (+) y/o EGFR (+) • Subtipo agresivo. • Alto grado histológico e índice mitótico. • Riesgo en edades menores (<40 años). • Más frecuentemente en mujeres premenopáusicas afroamericancas. RE (-) y/o RP (-); HER2/neu (+) • Menos común. Subtipo altamente agresivo. • Alto grado histológico. • Riesgo en mujeres <40 años, mayor que el subtipo luminal. • La etnia afroamericana puede ser un factor de riesgo. • Resultado mejorado por HER2/neu (+). HER2/neu (+); RE(-) Modificada por: (Schnitt, 2009). 1.4.2.5 Tratamiento El tratamiento integral del cáncer de mama es multidisciplinario, los manejos más comunes son cirugía y radioterapia en cualquiera de sus modalidades (neoadyuvante, adyuvante y paliativa). El tratamiento sistémico, incluye quimioterapia, terapia endócrina y la terapia dirigida a blancos moleculares (Arce, 2011). Los tratamientos pueden ser clasificados en grupos amplios, basándose en su funcionamiento y al tipo de pacientes a que se dirigen (ACS, 2014). • Cirugía para el cáncer de seno Se emplea algún tipo de cirugía en la mayoría de las mujeres con cáncer de seno. Usualmente, se necesita cirugía para extirpar un tumor de seno. Las 27 opciones quirúrgicas son la cirugía conservadora del seno y la mastectomía. La cirugía también se hace para verificar si el cáncer se propagó a los ganglios linfáticos que están debajo del brazo. Las opciones para esto incluyen una biopsia del ganglio linfático centinela y una disección de los ganglios linfáticos axilares. • Terapia local y terapia sistémica La terapia local tiene como objetivo tratar un tumor en un sitio específico, sin afectar el resto del cuerpo. Algunos ejemplos de terapias locales son la cirugía y la radioterapia. La terapia sistémica se refiere a medicamentos que se administran por vía oral o directamente en el torrente sanguíneo para llegar a las células cancerosas en cualquier lugar del cuerpo. La quimioterapia, la terapia hormonal y la terapia dirigida son terapias sistémicas. • Terapia adyuvante Son tratamientos adicionales para ayudar a evitar que el cáncer regrese. A esto se le conoce como terapia adyuvante. Los médicos creen que incluso en las etapas tempranas de cáncer de seno, las células cancerosas se pueden desprender del tumor primario del seno y comenzar a propagarse. Estas células no se pueden detectar fácilmente, sin embargo pueden establecer tumores nuevos en los tejidos adyacentes, en otros órganos y huesos. El objetivo de la terapia adyuvante es destruir estas células ocultas. Tanto la terapia sistémica (como quimioterapia, terapia hormonal y terapia dirigida) como la radiación se pueden usar como terapia adyuvante. 28 1.4.2.6 Genética del cáncer de mama La mayoría de casos en cáncer de mama no tienen una causa identificable, pero aproximadamente del 5-10% son causados por mutaciones genéticas (Vidal, 2008). 1.4.2.6.1 Mutaciones genéticas hereditarias Ciertas mutaciones hereditarias en el ADN pueden aumentar dramáticamente el riesgo de padecer diversos tipos de cáncer. Los dos genes mejor conocidos vinculados al cáncer de mama son BRCA1 y BRCA2 (ACS, 2014). Juntas, las mutaciones en el BRCA1 y en el BRCA2 representan casi de 20 a 25% de los casos de cáncer de mama hereditarios y cerca de 5 a 10% de todos casos de éste cáncer. Además, las mutaciones en el BRCA1 y en el BRCA2 representan casi 15% de los casos de cáncer de ovario en general. Los cánceres de mama asociados con las mutaciones en el BRCA1 y en el BRCA2 tienden a presentarse a una edad más joven que los cánceres de mama esporádicos (INC, 2014). Hoy en día, las pruebas genéticas pueden identificar a algunas mujeres que han heredado las mutaciones en los genes supresores de tumores BRCA1 o BRCA2 (o con menos frecuencia en otros genes como PTEN o TP53). Estas mujeres pueden tomar medidas preventivas para reducir su riesgo de cáncer de mama y supervisarse continuamente a fin de detectar la enfermedad en una etapa tratable (ACS, 2014). Cambios en otros genes: otras mutaciones genéticas podrían también conducir a cáncer de mama, sin embargo éstas son menos frecuentes e indicadoras de la gravedad o riesgo de desarrollar la enfermedad. Algunos de los genes que pueden presentar mutaciones relacionadas al cáncer de mama, son los siguientes (ACS, 2014). 29 • ATM: el gen ATM ayuda normalmente a reparar el ADN dañado. Heredar dos copias anormales de este gen causa la enfermedad ataxia-telangiectasia. Por otro lado, heredar una copia mutada de este gen ha sido asociado con una alta tasa de cáncer de mama en algunas familias. • p53: las mutaciones hereditarias del gen p53 supresor de tumores causa el síndrome de Li-Fraumeni. Las personas con este síndrome tienen mayor riesgo de desarrollar cáncer de mama, al igual que otros cánceres. • PTEN: este gen ayuda normalmente a regular el crecimiento celular. Las mutaciones hereditarias en este gen causan el síndrome de Cowden, un trastorno poco común en el cual las personas están en un riesgo aumentado de tumores del seno, malignos y benignos. • CHEK2: mutaciones hereditarias en el gen CHEK2 pueden aumentar el riesgo de cáncer de mama alrededor del doble del porcentaje normal. • CDH1: las mutaciones hereditarias en este gen causan cáncer gástrico difuso hereditario, un síndrome en el cual las personas desarrollan un tipo poco común de cáncer de estómago a una edad temprana. Las mujeres con mutaciones en este gen también tienen mayor riesgo de cáncer de mama lobulillar invasivo. 1.4.2.6.2 Mutaciones genéticas adquiridas La mayoría de las mutaciones del ADN relacionadas con el cáncer de mama, se generan en el transcurso de la vida de una mujer, en lugar de ser heredadas (INC, 2014). Las mutaciones adquiridas de los oncogenes y/o de los genes supresores de tumores pueden ser el resultado de otros factores, tal como radiaciones u otros factores de riesgo, mencionados anteriormente. 30 Las pruebas para identificar cambios genéticos adquiridos pueden ayudar a predecir con más precisión el pronóstico de algunas mujeres con cáncer de mama, como las pruebas para identificación de células cancerosas con varias copias del oncogén HER2 (ACS, 2014). 1.4.2.7 Análisis del gen AKT1: implicaciones en el cáncer de mama 1.4.2.7.1 La vía de señalización PI3K/AKT La vía de señalización PI3K/AKT es activada por muchos estímulos, y controla aspectos claves como el crecimiento, proliferación, supervivencia, motilidad y metabolismo celular (Lema, 2012). La vía de la PI3K es estimulada fisiológicamente como consecuencia de la activación de receptores de membrana tirosin kinasa, los cuales autofosforilan y fosforilan el sustrato del receptor de insulina (IRS); este último fosforila la subunidad p85 de la PI3K. La fosforilación de la subunidad p85 conduce a un cambio conformacional de dicha proteína que conduce a la unión de la subunidad catalítica (p110). La PI3K activa, fosforila el fosfatidil inositol 3,4 difosfato (PIP2) convirtiéndolo en el segundo mensajero fosfatidil inositol 3,4,5 trifosfato (PIP3), el cual, corriente abajo, conduce a la activación de la proteína AKT (Ver Figura 13) (Pinzón, Serrano, & Sanabria, 2009). La AKT tiene múltiples blancos, responsables de los efectos de la activación de la vía. La activación anormal de esta vía conduce a una respuesta proliferativa y anti-apoptótica que se relaciona con el desarrollo de múltiples tipos de cáncer; por esto, su estudio es parte crucial para el entendimiento de los procesos de carcinogénesis (Pinzón, Serrano, & Sanabria, 2009). 31 Figura 13. Esquema de la vía de señalización PI3K/AKT. Fuente: Adaptado por (Pinzón, Serrano, & Sanabria, 2009), de (Hennessy, Smith, Ram, Lu, & Mills, 2005). 1.4.2.7.2 AKT1 1.4.2.7.2.1 Concepto y rol en el desarrollo del cáncer AKT1 (también conocida como proteína quinasa B) forma parte de una subfamilia de proteínas serina/treonina quinasas (Testa & Bellacosa, 2001). Es una enzima, codificada por el gen AKT1. Hay tres isoformas de AKT (AKT1, AKT2 y AKT3), y cada miembro de AKT contiene un dominio de homología pleckstrina N-terminal (PH), un 32 conector corto-α helicoidal y un dominio de quinasa C-terminal (Testa & Bellacosa, 2001) Todas las isoformas de AKT constituyen los principales blancos aguas abajo de los receptores de factores de crecimiento que se señalizan a través de fosfatidilinositol 3-quinasa (Testa & Bellacosa, 2001). Existe evidencia creciente de que la activación de las proteínas AKT es importante en el desarrollo del cáncer (Muise-Helmericks, et al., 1998; Dimmeler, et al., 1999; Ozes, et al.,1999; Mende, et al., 2001; Wei, et al., 2001). En la activación de la vía PI3K/AKT, se enfatiza la importancia de los dominios PH en AKT, que tienen gran afinidad por el PIP3 en la membrana, ya que normalmente, en su configuración inactiva, AKT está plegada sobre sí misma. Al unirse a PIP3 se estira y es activada por la fosforilación de la Tirosina 308 por la PDK1 y su Serina 473 por la mTORC2 (Lema, 2012). La fosfo-AKT, se encarga de la fosforilación de decenas de targets importantes para el metabolismo (FOXO, IRS1, GSK3 y las enzimas glucolíticas, entre otros), traducción (mTOR, TSC1/2, FOXO, PRAS40, S6K), proliferación (FOXO, p21, p27), supervivencia (BAD, Procaspasa 9, CREB, p21, p27, MDM2, GSK3) y angiogénesis (eNOS) y motilidad. Al final, tiene efectos metabólicos importantes en diabetes y enfermedades cardiovasculares, y mitogénicos importantes en cáncer; entre otros procesos celulares (Lema, 2012). 1.4.2.7.2.2 • Mutaciones en AKT1 Conceptos preliminares: Polimorfismos genéticos y asociación con características clínicas Un polimorfismo genético (single nucleotide polymorphism o SNP), se define como una variación genética, que hace referencia a la existencia en una población de múltiples alelos de un gen. Su estudio tiene diversas aplicaciones en el campo de la medicina así como en el desarrollo de 33 investigaciones biológicas (Checa, 2007). Numerosas publicaciones sobre el tema permiten vislumbrar su entrada en la práctica clínica. Se han reportado estudios que indican el uso de SNPs como marcadores moleculares en la genotipificación étnica, la expresión génica de enfermedades y como potenciales blancos farmacológicos (Spalvieri & Rotenberg, 2004). De este modo, se han desarrollado estudios en base al análisis de polimorfismos en enfermedades de índole genética, como el cáncer. Los principales blancos de estudio, han sido oncogenes, que podrían estar alterados, en rutas metabólicas de importancia en el desarrollo de la enfermedad. Las investigaciones sobre polimorfismos en el estudio del cáncer, se han enfocado en la búsqueda de relaciones entre los cambios en la secuencia de nucleótidos y el efecto sobre la función de las respectivas proteínas, con los distintos estadíos, tipos de cáncer y otras características clínicas (histológicas e inmunishistoquímicas) de importancia, con el fin de hallar blancos moleculares que puedan emplearse posteriormente como dianas terapéuticas (Cancer Research UK, 2013). • Polimorfismos en AKT1 Recientemente, varios grupos de investigación han descubierto mutaciones somáticas recurrentes del gen AKT1 en casos de cáncer de mama, cáncer colorectal y de ovario (Carpten, et al., 2007). Entre estas mutaciones, se destaca el polimorfismo E17K, el cual en un estudio se encontró en 5 de los 61 (8,2%) casos de cáncer de mama, 3 de cada 51 (5,9%) casos de cáncer colorrectal y 1 de cada 50 (2,0%) casos de cáncer de ovario. La mutación E17K era mutuamente excluyente con respecto a la mutación PIK3CA y a la pérdida de expresión de PTEN (Carpten, et al., 2007). En otros estudios, esta variante fue reportada en el 4.3% (Kim, Jeong, Yoo, & Lee, 2008) y 6% (Bleeker, et al., 2008) de las poblaciones de estudio respectivas, de pacientes con cáncer de mama. 34 E17K resulta en un cambio de aminoácido en la posición 17 en AKT1, de un ácido glutámico (E) por una lisina (K). Esta mutación se produce dentro del dominio de homología a pleckstrina (PHD) de AKT1 y resulta en la activación de la ruta fosfatidilinositol 3-quinasa (PI3K). Otras investigaciones realizadas han sugerido, mediante modelos computacionales, una variedad de SNP´s que podrían ser patogénicos y por tanto de gran utilidad en el diagnóstico molecular de enfermedades relacionadas con este gen, entre ellas, el cáncer de mama (Shanthi, Rajasekaran, & Ramanathan, 2014). Dentro de las variantes identificadas (E17K, E17S, E319G, L357P, y P388T) se encuentran las que han sido seleccionadas para este estudio: E17K, E17S y L357P, siendo E17K la única reportada en otros estudios. 35 CAPÍTULO 2: MATERIALES Y MÉTODOS 2.1 Revisión Bibliográfica Se realizó inicialmente la revisión bibliográfica del tema, en lo referente al cáncer de mama, su clasificación en base a características histopatológicas y moleculares. Además, se investigó sobre los polimorfismos del gen AKT1 que se han relacionado hasta el momento con una posible susceptibilidad a la enfermedad, específicamente los SNPs: L357P, E17S y E17K. 2.2 Muestreo Se recolectaron bloques de tejido mamario embebido en parafina, donados por la Unidad de Patología del Hospital de SOLCA y del Hospital de Especialidades Eugenio Espejo. En el caso de las muestras control, se utilizaron muestras de sangre periférica de pacientes del Hospital de Machachi, y del Banco de Muestras de ADN del Instituto de Investigaciones Biomédicas (UDLA), provenientes de mujeres sanas de entre 30 y 80 años. Los criterios empleados para la selección de casos y controles para este estudio, se muestran en la Tabla 2. Tabla 2. Criterios de selección empleados en el estudio Criterios de Selección Criterios de Inclusión CASOS • Mujeres diagnosticadas con cáncer de mama Criterios de Exclusión • Ausencia de diagnóstico positivo para cáncer de mama. 36 • Mujeres sanas: sin registro de carcinomas de mama. • Rango de edad de 30 a 80 años Etnia mestiza CONTROLES CASOS Y CONTROLES • • Mujeres diagnosticadas con cáncer de mama o que registren haberlo presentado. • Rango de edad menor a 30 años y mayor a 80 años. Etnia distinta a la mestiza. • Para la realización del estudio, se contó con la aprobación del Comité de Bioética de la Universidad de las Américas y se llevaron a cabo los respectivos consentimientos informados. 2.3 Diseño de Primers Se diseñaron diferentes pares de primers, luego de localizar las variantes: rs34409589, rs121434592, rs11555432 en el gen AKT1, mediante herramientas proporcionadas por el NCBI: dbSNP y Primer-BLAST, respectivamente (http://www.ncbi.nlm.nih.gov/snp/?term). Para las variantes: rs34409589, rs121434592, los primers seleccionados fueron: Primers Secuencia Tm Fw 5’-GGCCAAGGGGATACTTACGC- 3’ 60.53 Rv 5’-AGGGTCTGACGGGTAGAGTG- 3’ 60.35 Para el rs11555432: Primers Secuencia Tm Fw 5’-CCTTCTTGAGCAGCCCTGAA- 3’ 59.96 Rv 5’-TACGAGATGATGTGCGGTCG - 3’ 59.97 37 2.4 Análisis de historias clínicas En este estudio se seleccionaron pacientes que fueron diagnosticadas entre los años 2010 y 2013. Se analizaron las historias clínicas de las pacientes afectas con cáncer de mama, las mismas que contaban con la información necesaria respecto a las características histopatológicas e inmunohistoquímicas de los tumores de cada paciente. Estos datos, se emplearon como variables para el presente estudio (Tabla 3). Tabla 3. Descripción de las variables de estudio. Variables de estudio Edad Tipos de carcinoma de mama Definición Tiempo transcurrido desde el nacimiento hasta el momento del estudio. Indica la forma del carcinoma según su origen: los conductos, los lobulillos o, en algunos casos, el tejido intermedio. Dimensión Indicador • • <35 años 35-49 años • • • 50-80 años • • Ductal • • Lobulillar • Papilar • Mucinoso • • • Pre-menopausia Período de menopausia Post- menopausia El cáncer comienza dentro de los conductos lácteos. Las células anómalas comienzan a crecer en los lobulillos. Carcinoma con borde bien definido y formado por prolongaciones pequeñas en forma de dedos. Células tumorales inmersas en cúmulos de moco extracelular. 38 Receptores hormonales Invasión a ganglios linfáticos Lateralidad Grado Tumoral/Histológi co Estadío de la Enfermedad (Estadío Tumoral) Indica si las células del cáncer de mama tienen o no receptores para las hormonas estrógeno y progesterona, que son proteínas que reciben las señales que ordenan a las células multiplicarse. Indica si el cáncer se ha propagado o no hasta los ganglios linfáticos cercanos Indica la mama que es afectada por el desarrollo de un tumor maligno. Hace referencia al nivel diferenciación tumoral del paciente. Estado actual del tumor o de la enfermedad. • • Receptores Estrógeno Progesterona Receptor HER2/neu de y • • Presencia (RE+, RP+, Her2/neu +), o Ausencia (RE-, RP-, Her2/neu -) • Presencia de células cancerosas, o • Ausencia células cancerosas de • Presencia tumor, o Ausencia tumor del • Positivo • Negativo • • • Mama derecha. Mama Izquierda. Ambas mamas. • SBRI • SBRII • SBRIII • 0 • • I • • II • del • Grado tumoral I o bien diferenciado • Grado tumoral II o moderadamente diferenciado Grado tumoral III o pobremente diferenciado • • Carcinoma in situ, afección no invasiva. Cáncer formado, tumor no se disemina. El tumor mide más de 2 cm, pero no más de 5 cm, y el cáncer se encuentra entre 1 a 3 ganglios linfáticos; o el tumor mide más de 5 cm, pero no se ha diseminado hasta los ganglios linfáticos. 39 • III • El tumor supera los 5 cm. El cáncer se diseminó hasta más de 3 ganglios linfáticos axilares e incluso piel de la mama. Puede tornarse inoperable. 2.5 Análisis de laboratorio. Procesamiento de muestras 2.5.1 Extracción de ADN La extracción de ADN de las muestras (sangre periférica y tumores), se llevó a cabo mediante la utilización del kit PureLink Genomic DNA de Invitrogen, según el protocolo indicado en el manual, con las modificaciones mencionadas a continuación. 2.5.1.1 Extracción de ADN a partir de sangre periférica En el caso de las muestras de sangre periférica, inicialmente se precalentó el bloque térmico AccuBlockTM Digital Cry Block (Labnet, NJ, USA) a 55ºC. En un volumen de 200 µL de sangre (de cada muestra), se añadió 20 µL de Proteinasa K y 20 µL de RNasa A. Luego de homogenizar la mezcla, se incubó a temperatura ambiente por 2 minutos. A continuación, se añadió 200 µL del buffer de lisis celular y se incubó la mezcla a 55ºC para promover la digestión proteica. Luego de 10 minutos, se retiraron los tubos del bloque térmico, y se añadió 200 µL del etanol 100% al lisado. Con la finalidad de obtener una solución homogénea, se mezcló por vórtex y el volumen total generado, aproximadamente 640 µL, se traspasó a una columna de extracción provista por el kit. 40 Luego de centrifugar las columnas a 10.000 x g, por 1 minuto a temperatura ambiente, se descartaron los tubos de colección y se colocaron las columnas en nuevos tubos. Entonces, se adicionaron 500 µL del buffer de lavado 1, y se centrifugaron los tubos nuevamente, a 10.000 x g, por 1 minuto. Lo mismo se realizó para adicionar el buffer de lavado 2, pero se centrifugó a 13500 rpm por 3 minutos. Después de descartar los tubos de colección, se colocaron las columnas en tubos estériles de 1.5 mL y se añadió 50 µL del buffer de elución, en el centro de la membrana de las columnas. Cada muestra se incubo por 1 minuto a temperatura ambiente. Finalmente, se centrifugaron los tubos a 13500 rpm por 3 minutos y luego se desecharon las columnas. Los tubos con ADN, fueron rotulados y conservados -20ºC. El ADN obtenido se cuantificó con Nanodrop 2000 y se realizó una corrida electroforética en gel de agarosa al 2% para constatar la calidad de ADN obtenido. 2.5.1.2 Extracción de ADN a partir de tumores embebidos en parafina Previamente, se realizó el marcaje de la zona tumoral en los bloques de parafina, utilizando una aguja estéril. A continuación, se realizaron de 5 a 10 cortes longitudinales de 5 µm de espesor con el uso de un micrótomo (SLEE 6062, Mainz, Alemania). Los cortes se colocaron en tubos estériles de 1.5 mL, los que fueron rotulados y almacenados a temperatura ambiente, para formar un banco de muestras tumorales. Para realizar la extracción de ADN, se precalentó un bloque térmico (Labnet, NJ, USA) a 55ºC. Se tomaron 3 o 4 cortes de cada muestra y se colocaron en tubos estériles. A cada tubo se añadió 1 mL de xilol y se mezcló 41 por vórtex durante 2 minutos. Luego se centrifugaron los tubos a 1600 rpm y se desechó el sobrenadante. El lavado con xilol se repitió hasta este punto. Después, se añadió 1 mL de etanol 100%, se mezcló por vórtex durante 30 segundos y se centrifugó por 3 minutos a temperatura ambiente. Se retiró el sobrenadante y se conservó el pellet conteniendo el tejido tumoral. El lavado con etanol se realizó por duplicado. Luego, a fin de evaporar todo el alcohol, se colocaron las muestras con la tapa abierta durante 15 minutos, en la estufa a 37ºC. Concluido el tiempo, a cada tubo se añadió 180 μL de buffer de digestión y 40 μL de Proteinasa K (Quiagen, Hilden, Alemania). Se mezcló adecuadamente por vórtex y se colocó los tubos en el bloque térmico a 55ºC por un lapso de 12 a 24 horas, en dependencia del grado de digestión y lisado del tejido tumoral. Una vez transcurrido este tiempo, se centrifugaron las muestras a 13500 rpm, por 3 minutos a temperatura ambiente; se transfirió alrededor de 200 μL del sobrenadante a un microtubo estéril de 1,5 mL y se añadió 20 μL de RNasa. Luego de incubar las muestras por 2 minutos, se añadió 200 μL del buffer de lisis y de unión a la membrana y 200 μL de etanol 100%, y se mezcló por vórtex durante 5 segundos hasta formar una solución homogénea. Se obtuvo un volumen aproximado de 640 μL por muestra, el que se colocó en una columna de extracción provista por el kit. Las columnas se centrifugaron a 10.000 x g, por 1 minuto a temperatura ambiente. El tubo colector se descartó y se colocó la columna en un nuevo tubo de colección. Posteriormente se añadió 500 μL de buffer de lavado 1 a cada columna de extracción y se centrifugo a 10.000 x g, por 1 minuto a temperatura ambiente. Se descartó el tubo colector y se colocó la columna en un nuevo tubo recolector. Luego, se añadió 500 μL de buffer de lavado 2 y se centrifugaron los tubos a 13500 rpm por 3 minutos a temperatura ambiente. Se desechó el tubo colector y se colocó la columna a un tubo estéril de 1,5 mL para la elución final. 42 Finalmente, en cada columna se colocó 50 μL de buffer de elución, y se esperó 2 minutos como tiempo de incubación. Se centrifugaron los tubos a 13500 rpm por 2 minutos, y se desecharon las columnas de extracción. Los tubos con ADN fueron rotulados adecuadamente y almacenados a -20ºC. 2.5.2 Reacción en Cadena de la Polimerasa (PCR) Una vez obtenido el ADN de las muestras de casos y controles, se amplificaron los fragmentos de interés, para lo cual se estandarizó la Reacción de PCR (para cada par de primers, dirigidos a los SNPs de estudio) y se determinaron las condiciones necesarias de temperatura, concentración de ADN y MgCl2. 2.5.2.1 Estandarización de PCR Previamente a la estandarización de la PCR, los cebadores diseñados fueron verificados mediante un ensayo de PCR in sílico para confirmar la amplificación de la zona de interés y la generación de una sola zona de amplificación, para lo cual se empleó la herramienta proporcionada por el grupo de Bioinformática Genómica de la Universidad de California en Santa Cruz en base al genoma humano actualizado hasta diciembre del 2010 (https://genome.ucsc.edu). Luego de ello, se efectuó la estandarización de PCR, llegando a las siguientes condiciones y volúmenes: el volumen final de reacción se ajustó a 50 μL por cada muestra, la cual estuvo compuesta por 34 μL de H2O grado PCR, 5 μL de buffer (MgCl2)10X, 1.5 μL de MgCl2 (10mM), 1 μL de dNTPs (10 mM cada uno), 2 μL de cada primer (10 μM), 0.5 μL de Platinum® Taq DNA Polimerasa (5 U/μL, Invitrogen) y 4 μL de templado de ADN (200 ng). La reacción fue llevada a cabo en un termociclador SureCycler8800 (Agilent, Santa Clara, CA), bajo las siguientes condiciones, ver Tabla 4: 43 Tabla 4. Condiciones estandarizadas para la PCR Nº ciclos T (ºC) Tiempo (min) 1 95 5 94 1 35 1 E17: L357P: 60 56.4 1 72 1 72 5 4 ∞ Finalmente, se realizó una corrida electroforética de cada muestra, en gel de agarosa al 2% para constatar la calidad de producto PCR obtenido. 2.5.3 Secuenciación Con el producto de PCR previamente obtenido, se llevó a cabo la secuenciación de los fragmentos, para lo cual se realizó una purificación previa y posterior a la reacción de secuencia, como se describe a continuación. 2.5.3.1 Purificación del producto PCR mediante columnas Se realizó la purificación, con el fin de separar el producto de PCR de interés de subproductos contaminantes, primers y dNTPs. Para ello, se empleó el kit de Invitrogen PureLink® PCR Purification, según el cual, inicialmente, se colocaron 15 µL del producto de PCR en un tubo estéril de 0.2 mL, al que se añadió 60 µL de buffer de unión a la membrana (HC) y se homogenizó mediante vórtex. 44 A continuación, se transfirió la mezcla a una columna colocada en el tubo colector, y se centrifugó a 10.000 x g, por 1 minuto a temperatura ambiente. Luego, se añadió 130 µL de Buffer de lavado y se centrifugó la columna a 10.000 x g, por 1 minuto, y luego a 13500 rpm por 3 minutos, para garantizar la remoción total del buffer. Para la elución, se descartó el tubo colector y se colocó la columna en un tubo estéril de 1.5 mL, luego se añadió 20 µL de buffer de elución, que se dejó incubar por 2 minutos y finalmente, se centrifugó a 13500 rpm por 2 minutos. Los tubos con producto de PCR purificado se rotularon adecuadamente y conservaron a -20ºC. La calidad y cantidad de PCR, pudo verificarse empleando el instrumento Nanodrop 2000 y realizando una corrida electroforética en gel de agarosa al 2%, respectivamente. 2.5.3.2 Reacción de secuencia Se estandarizó la PCR de secuenciación, de modo que: el volumen final de reacción se ajustó a 4.4 μL por cada muestra, la cual estuvo compuesta por 1.4 μL de H2O grado PCR, 0.9 μL de buffer de secuenciación (BigDye Terminator) 5X, 0.5 μL de BigDye® Terminator v1.1, 1.6 μL del primer seleccionado (1 μM) y 1.5 μL del producto de PCR purificado. La reacción fue llevada a cabo en un termociclador SureCycler8800 (Agilent, Santa Clara, CA), bajo las siguientes condiciones, en 25 ciclos (Ver Tabla 5). 45 Tabla 5. Condiciones estandarizadas para la reacción de secuencia 2.5.3.3 T (ºC) Tiempo 96 3 min 96 10 s 50.5 5s 60 4 min 4 1 min 4 ∞ Purificación de la reacción de secuenciación Luego de la reacción de secuenciación, se realizó una nueva purificación, para remover principalmente los ddNTPs no incorporados. Se utilizó un kit de Applied Biosystems: kit de purificación BigDye® XTerminator™. Según el manual, se adicionaron 5 μL de Big Dye Xterminator Solution y 22.5 μL de SAM Solution a ≈5 μL de la PCR de secuenciación. Luego se mezcló por vórtex durante 30 min y se centrifugó, para emplear el sobrenadante en la secuenciación. A continuación se cargaron estas muestras en las placas del secuenciador ABI PRISM 3130 Genetic Analyzer (Applied Biosystems, Austin, TX) y finalmente se realizó el análisis de secuencias, a través del Sequencing Analysis Software 5.3.1 (Applied Biosystems, Austin, TX), para determinar los genotipos de cada una de las muestras. 46 2.6 Análisis estadístico Luego de determinar los genotipos de las muestras (casos y controles), se procedió a tabular los resultados, para asociarlos con las características histopatológicas e inmunohistoquímicas tumorales, registradas anteriormente para cada paciente. Adicionalmente, se analizaron las frecuencias alélicas y genotípicas del gen AKT1 (polimorfismo E17K), y se calculó el equilibrio Hardy-Weinberg utilizando una herramienta disponible en internet (http://www.genes.org.uk/software/hardy-weinberg.shtml). Mediante el programa estadístico IBM SPSS Statistics 22 (SPSS Inc, Chicago, IL), se aplicaron las pruebas estadísticas chi-square (χ2) y P value, para determinar la asociación entre la presencia de los SNPs estudiados, y las características histopatológicas e inmunohistoquímicas tumorales. Las características histopatológicas analizadas en los pacientes se dividieron en 3 rangos de edad (<35, 35-49, 50-80 años) y fueron las siguientes: tipo de carcinoma (Ductal, lobulillar, papilar, mucinoso), lateralidad (mama derecha, mama izquierda), grado tumoral/histológico (SBRI, SBRII, SBRIII), márgenes quirúrgicos (presencia vs ausencia), nódulo linfoide (presencia vs ausencia de células cancerosas) y estadío de la enfermedad (0, I, II, III). En cuanto a las características inmunohistoquímicas analizadas, fueron: receptores hormonales de estrógeno, progesterona y HER2neu (presencia vs ausencia). 47 CAPÍTULO 3: RESULTADOS 3.1 Recolección de muestras En total se recolectaron 100 bloques de tejido mamario embebidos en parafina (casos) de la Unidad de Patología del Hospital de SOLCA (86) y del Hospital de Especialidades Eugenio Espejo (14) y se utilizaron 140 muestras de sangre periférica de mujeres sanas (controles), recolectadas previamente de pacientes del Hospital de Machachi y del banco de muestras de ADN del Instituto de Investigaciones Biomédicas (UDLA). 3.2 Clasificación de muestras tumorales Con el acceso a las respectivas historias clínicas, se tabularon y analizaron los datos de los pacientes afectos con cáncer de mama, como: nombres y apellidos, lateralidad, grado de diferenciación tumoral, bordes quirúrgicos, receptores hormonales, entre otros (Ver Anexo 1). 3.2.1 Tipo de carcinoma de mama El principal tipo de carcinoma encontrado en la población de estudio, corresponde al Ductal invasor, con un porcentaje de 74.07%. Seguido, está el carcinoma de tipo ductal in situ, con 7.41%. A continuación, se encuentra el carcinoma ductal mixto, que corresponde a un tipo de carcinoma ductal con componente de otro tipo, con 6.17%. Y finalmente, se identificaron otros tipos de carcinoma, con menor frecuencia: lobulillar invasor (3.70%), mucinoso in situ (3.70%), papilar invasor (2.47%), mucinoso invasor (1.23%) y medular (1.23%) (Ver Gráfico 1). 48 Tipos de carcinomas Medular 1,23 Mucinoso invasor 1,23 Mucinoso in situ Papilar invasor Lobulillar invasor 3,70 2,47 3,70 Ca ductal mixto 6,17 Ductal invasor Ductal in situ 74,07 7,41 0,00 10,00 20,00 30,00 40,00 50,00 60,00 70,00 80,00 Casos (%) Gráfico 1. Distribución de la población de estudio afecta con CM según el tipo de carcinoma. 3.2.2 Edad del paciente Se clasificó a la población de estudio en tres grupos de edad, que buscaron abarcar los períodos de pre-menopausia, menopausia y postmenopausia, debido a la asociación de este factor con el desarrollo del cáncer de mama. Se encontró que la mayor parte de las mujeres afectas (56.79%) estaban en un rango de 50-80 años de edad, el 40.74% de ellas estuvieron entre los 35 y 49 años y el resto de ellas (2.47%) fueron menores a 35 años (Ver Gráfico 2). 49 Edad del paciente 2,47% 40,74% 56,79% <35 35-49 50-80 Gráfico 2. Distribución de la población de estudio afecta con CM según la edad del paciente 3.2.3 Lateralidad La mama afectada en la mayor parte de los casos de estudio fue la mama derecha, con un 58.02%, como se muestra en el Gráfico 3. De este modo, el 41.98% restante de casos mostró afectación en la mama izquierda. Además, no se presentó en ningún caso, afectación en ambas mamas. Lateralidad 70 % Casos 60 58,02 % 50 41,98 % 40 30 20 10 0 Derecha Izquierda Gráfico 3. Distribución de la población de estudio afecta con CM en base a la lateralidad 50 3.2.4 Estadío tumoral El estadío tumoral de los casos fue clasificado en cuatro grupos: To, T1T2, T3-T4 y No determinado. El análisis de esta variable, determinada por el Médico Patólogo, mostró que la mayoría de pacientes con cáncer de mama, diagnosticados entre los años 2010-2013, presentaron un estadío tumoral T1 o T2 (55%), como se observa en el Gráfico 4. En menor porcentaje, se encontraron casos en estadío tumoral T3-T4 (13%) y To (4%). El grupo de casos en la categoría “No determinado”, corresponde a aquellos en los que el patólogo no ha determinado el estadío tumoral en que se encuentran. Estadío tumoral 80 67,9 70 % Casos 60 50 40 30 16,05 20 10 11,11 4,94 0 To-Tx T1-T2 T3-T4 No determinado Gráfico 4. Distribución de la población de estudio afecta con CM según el estadío tumoral 51 3.2.5 Invasión a nódulo linfoide Esta variable fue clasificada en dos grupos: invasión presente (+) o ausente (-). Se encontró que el 48% de los casos presentaron invasión a nódulo linfoide y el 38% de ellos, ausencia. En el 14% de los casos, esta variable no fue determinada por el Patólogo (Ver Gráfico 5). Nódulo linfoide + - No determinado 14% 48% 38% Gráfico 5. Distribución de la población de estudio afecta con CM en función de la invasión de células tumorales a nódulo linfoide 3.2.6 Grado tumoral/Histológico El grado tumoral/histológico, que indica los niveles de diferenciación tumoral, se clasificó en tres grupos. La mayor parte de casos (40.74%) se asignaron al grado II o SBRII (moderadamente diferenciado), el 19.75% pertenecieron a la categoría SBRI (bien diferenciado) y 18.52%, al grado SBRIII (pobremente diferenciado). 52 Los casos sin determinación de grado tumoral representaron el 20.99% del total (Ver Gráfico 6). % Casos Grado tumoral/histológico 45 40 35 30 25 20 15 10 5 0 40,74 19,75 SBRI 18,52 SBRII SBRIII 20,99 No determinado Gráfico 6. Distribución de la población de estudio afecta con CM según el grado tumoral/histológico 3.2.7 Receptores hormonales El registro de la presencia o ausencia de receptores hormonales, de fundamental importancia en el diagnóstico y tratamiento de cáncer de mama, mostró los siguientes resultados: el 62.96% de los casos presentaron receptor de estrógeno positivo (o RE+), el 54.32% receptor de estrógeno positivo (o RP+) y sólo el 30.86% de los casos presentaron receptor 2 de factor de crecimiento epidérmico humano positivo (o HER2+). De este modo, se encontró ausencia de receptores de estrógeno, progesterona y HER2 en el 37.04%, 45.68% y 69.14% de los casos de estudio, respectivamente, como se muestra en el Gráfico 7. 53 % Casos Receptores hormonales 80 70 60 50 40 30 20 10 0 69,14 62,96 54,32 45,68 37,04 + Estrógeno - 30,86 + - Progesterona + Her2 neu Gráfico 7. Distribución de la población de estudio afecta con CM en función de la presencia de receptores hormonales: Progesterona, Estrógeno, HER2. 3.2.8 Tipo de cáncer en base a receptores hormonales Se encontró que la mayoría de casos pertenecían al tipo Luminal A con el 43.21% del total de pacientes con CM. A continuación, el 24.69% de los casos presentaron el tipo Basal o TNBC, el 19.75% se diagnosticó con cáncer de tipo Luminal B, y el 12.35% con el de tipo HER2 (neu) (Ver Gráfico 8). Clasificación molecular de pacientes con CM TNBC Her2 neu Luminal B luminal A 24,69% 12,35% 19,75% 43,21% Gráfico 8. Distribución de la población de estudio afecta con CM en función del tipo de cáncer en base a receptores hormonales. (TNBC: Triple Negative Breast Cancer, o Tipo Basal) 54 3.2.9 Márgenes quirúrgicos El análisis de márgenes quirúrgicos, realizado por el Patólogo a partir de las biopsias y registrado en las historias clínicas, mostró que en el 35% de los casos, el margen quirúrgico no contenía células cancerígenas (bordes libres), el 33% de ellos indicaron un resultado positivo y en el 32% de casos no se determinó esta variable, como se muestra en el Gráfico 9. Márgenes quirúrgicos + - No determinado 32% 33% 35% Gráfico 9. Distribución de la población de estudio afecta con CM en función de la presencia de células cancerígenas en bordes quirúrgicos 3.3 Determinación de Genotipos 3.3.1 Extracción de ADN Se realizó la extracción de ADN de casos y controles, utilizando el kit PureLink Genomic DNA de Invitrogen. 55 La calidad del ADN proveniente de tumores embebidos en parafina, se mostró inferior respecto al ADN obtenido de tejido fresco (sangre periférica), lo que sugiere degradación del material genético de los casos, debido a procesos propios del pre tratamiento y fijación del tejido tumoral (Ver Figura 14). A CM1 CM2 CM3 CM4 CM5 CM6 CM7 CM8 CM9 CM10 B TM1 TM2 T M3 TM4 TM5 TM6 TM7 TM8 TM9 TM10 Figura 14.Electroforesis de muestras de ADN extraído, de la población de estudio en geles de agarosa al 2%. A) Muestras de ADN proveniente de sangre periférica. B) Muestras de ADN proveniente de tumores embebidos en parafina. 3.3.2 Reacción en cadena de polimerasa (PCR) Se amplificaron los fragmentos de interés con los primers diseñados y las condiciones de estandarización adecuadamente establecidas. 56 3.3.2.1 Polimorfismos AKT1-E17 Luego de la PCR, se obtuvieron los fragmentos de interés, que contenían las regiones de ubicación de los polimorfismos: E17K y E17S. El fragmento obtenido fue de aproximadamente 198 pb, como se observa en la Figura 15. A 198 pb B 198 pb Figura 15. Electroforesis del fragmento de ADN de interés para el análisis del SNP E17, de 198 pb, en un gel de agarosa al 2%. A) Muestras control. B) Muestras de casos. 3.3.2.2 Polimorfismo AKT1-L357P Se amplificó correctamente un fragmento de 142pb de la zona del gen AKT1 que contiene el polimorfismo L357P, como se muestra la Figura 16. 57 A MM (-) CM1 CM2 CM3 CM4 CM5 CM6 CM7 CM8 CM9 142 pb B MM (-) TM1 TM2 TM3 TM4 TM5 TM6 TM7 TM8 142 pb Figura 16. Electroforesis del fragmento de ADN de interés para el análisis del SNP L357P, de 142 pb, en un gel de agarosa al 2%. A) Muestras control. B) Muestras de casos. 3.3.3 Secuenciación capilar Se realizó la secuenciación de los fragmentos obtenidos en PCR, para el análisis de los polimorfismos: E17K, E17S y L357P. En el caso de las variantes polimórficas E17K y E17S, estas se presentan cuando se ha producido el cambio de un Ac. Glutámico por Lisina o Serina, respectivamente, en la posición 17. De modo que, en los casos que presentaron la variante E17K, no se encontró la variante E17S. En cuanto al polimorfismo L357P, se presenta en aquellos casos en los que se ha producido un cambio de Leucina por Prolina, en la posición 357. 58 Como se muestra en la Figura 17, sólo se encontró el genotipo salvaje: T/T en el caso del análisis de la variante L357P. En cuanto al polimorfismo E17K, se encontraron dos genotipos homocigóticos: G/G y A/A (Figura 18). No se verificaron casos o controles con la variante E17S. Los resultados en cuanto al porcentaje de casos y controles para cada genotipo, se muestran a continuación. En la Figura 17, se muestra el genotipo T/T, representado por la curva señalada, correspondiente a la base A (adenina), es decir su base complementaria, lo que se debe al uso del primer reverse en la reacción de secuenciación. T/T 100 % 100 % Casos Controles 100 80 60 40 20 0 Figura 17. Identificación del polimorfismo L357P por secuenciación capilar y distribución en población total de estudio. A) Secuencia para homocigotos T/T. B) Cuadro de distribución porcentual del genotipo T/T. 59 En la sección A de la Figura 18, se muestra el genotipo G/G, representado por la curva señalada, correspondiente a la base C, es decir citosina. De igual manera, en la sección B, se observa el genotipo A/A, representado por la curva correspondiente a la base T (timina). A. B. C. D. G/G 100 85 70 55 40 25 10 97,5 % casos A/A 100 % 3 2,5 2 1,5 1 0,5 0 controles 2,5 % 0% casos controles Figura 18. Identificación del polimorfismo E17K por secuenciación capilar y distribución en población total de estudio. A) Secuencia para homocigotos G/G. B) Secuencia para homocigotos A/A. C) Cuadro de distribución porcentual del genotipo G/G en la población total de estudio. D) Cuadro de distribución porcentual del genotipo A/A en la población total de estudio. 60 3.3.4 Análisis estadístico 3.3.4.1 Frecuencias genotípicas y alélicas Los genotipos observados se ordenaron adecuadamente y se calcularon las frecuencias genotípicas y alélicas, como se muestra en la Tabla 6. Este análisis fue realizado únicamente en el caso del polimorfismo E17K, ya que solamente se halló un genotipo distinto al silvestre (homocigoto raro) en los casos analizados para esta variante. De este modo, a través de la prueba chi-cuadrado realizada, se obtuvo un valor de P mayor a 0.05, mostrando que ambos grupos (casos y controles) están en equilibrio Hardy-Weinberg (HWE) y que no existen diferencias significativas entre ellos. El HWE se calculó aproximado a 1, es decir que se consideró no significativo en relación a diferencias entre poblaciones, un dato fundamental en el análisis estadístico en modelos de estudio caso-control. Tabla 6. Distribución de frecuencias genotípicas y alélicas del polimorfismo E17K. Frecuencia Genotípica Gen/ SNP AKT1 / E17K Frecuencia Alélica Genotipo Casos Controles Todo Casos Controles Todo G/G 0,98 1,00 0,99 0,98 1,00 0,99 G/A 0,00 0,00 0,00 A/A 0,03 0,00 0,01 HWE, Chicuadr ado 1 0,03 0,00 0,01 Valor P 0,3 61 3.3.4.2 Análisis de características clínicas en pacientes con cáncer de mama y asociación con el tipo de cáncer La distribución de características clínicas en los pacientes con cáncer de mama, en función del tipo de cáncer (clasificación molecular), se muestra en la Tabla 7. En cuanto a la edad, se encontró que la mayoría de mujeres de entre 35 y 80 años (44.30%) padecían cáncer del tipo Luminal A y en menores porcentajes padecían los demás tipos: TNBC, Luminal B y HER2. En el análisis de la lateralidad, se encontró gran diferencia porcentual en el caso de los pacientes con cáncer de tipo Luminal A y Luminal B, que presentaron afectación principalmente en la mama derecha. En lo referente al estadío tumoral, es importante señalar que en todos los tipos de cáncer, predominó el grupo T1-T2, respecto a los demás. Adicionalmente, en los tipos de cáncer Luminal A y HER2, se encontró que la mayoría de los casos presentaron invasión a nódulo linfoide, un factor fundamental en el diagnóstico y pronóstico de la enfermedad. De manera similar, se encontró que la mayor parte de casos con cáncer de tipo Luminal A y TNBC, presentaron márgenes quirúrgicos con presencia de células cancerígenas. En el análisis del grado tumoral/histológico, se encontró que el grado II o SBRII predominó en los casos con cáncer de tipo Luminal A y Luminal B, no así con los de tipo HER2 y TNBC, en los que predominó el grado SBRIII. En cuanto a las variantes polimórficas encontradas, de genotipo AA, uno de los casos presentó cáncer de tipo Luminal A y el otro, el tipo TNBC. 62 Tabla 7. Distribución de características clínicas en pacientes con CM en función del tipo de cáncer (clasificación molecular). Luminal A Luminal B HER TNBC Total n % n % n % n % n % <35 0 0 0 0 1 10 1 5,00 2 2,47 35-49 14 40 6 37,5 5 50 8 40,00 33 40,74 50-80 21 60 10 62,5 4 40 11 55,00 46 56,79 Derecha 21 60,00 11 68,75 5 50,00 10 50,00 47 58,02 Izquierda 14 40,00 5 31,25 5 50,00 10 50,00 34 97,14 Estadío tumoral T1-T2 28 80,00 7 43,75 5 50,00 15 75,00 55 67,90 T3-T4 4 11,43 4 25,00 3 30,00 2 10,00 13 16,05 To-Tx No determinado Nódulo linfoide + 2 5,71 1 6,25 0 0,00 1 5,00 4 4,94 1 2,86 4 25,00 2 20,00 2 10,00 9 11,11 20 57,14 5 31,25 5 50,00 9 45,00 39 48,15 14 40,00 6 37,50 2 20,00 9 45,00 31 38,27 1 2,86 5 31,25 3 30,00 2 10,00 11 13,58 + 35 100,0 0 16 100,0 0 0 0,00 0 0,00 51 62,96 - 0 0,00 0 0,00 10 100,0 0 20 100,00 30 37,04 Edad Lateralidad No determinado Estrógeno Progesterona 6 37,50 1 10,00 2 10,00 44 54,32 0 100,0 0 0,00 10 62,50 9 90,00 18 90,00 37 45,68 + 3 8,57 10 62,50 10 2 10,00 25 30,86 - 32 91,43 6 37,50 0 18 90,00 56 69,14 + 35 HER2 100,0 0 0,00 63 Márgenes quirúrgicos + 15 42,86 2 12,50 2 20,00 8 40,00 27 33,33 No determinado 13 37,14 6 37,50 3 30,00 6 30,00 28 34,57 7 20,00 8 50,00 5 50,00 6 30,00 26 32,10 Grado tumoral hist. SBRI 13 37,14 1 6,25 1 10,00 1 5,00 16 19,75 SBRII 17 48,57 7 43,75 3 30,00 6 30,00 33 40,74 SBRIII No determinado 1 2,86 2 12,50 4 40,00 8 40,00 15 18,52 4 11,43 6 37,50 2 20,00 5 25,00 17 20,99 GG 34 97,14 16 100 10 100 19 95,00 79 97,53 AA 1 2,86 0 0,00 0 0,00 1 5,00 2 2,47 E17K 3.3.4.3 Asociación del polimorfismo E17K con características clínicas de pacientes con cáncer de mama Se tabularon los datos clínicos de los pacientes con cáncer de mama y se asociaron con los genotipos encontrados, para el polimorfismo E17K, como se muestra en la Tabla 8. En ningún caso, se puede afirmar la asociación del genotipo AA (Homocigoto raro) con alguna de las distintas características clínicas, debido al bajo de número de casos encontrados con este genotipo, que consecuentemente genera valores no significativos (P>0.05). 64 Tabla 8. Distribución de características clínicas, histopatológicas e inmunohistoquímicas en pacientes con CM en función del genotipo presentado, para la variante polimórfica E17K. AKT1 E17K rs121434592 GG AA n % n % Derecha 46 58,23 1 50,00 Izquierda 33 41,77 1 50,00 Lateralidad Xi2 0.054 Valor P 1 Estadío tumoral To 3 3,80 1 50 T1-T2 54 68,35 1 50 T3-T4 13 16,46 0 0 No determinado 9 11,39 0 0 Xi2 Grupos Xi2 y Valor P Valor P To / T1-T2 6.1 0.297 To / T3-T4 3.5 0.52 T1-T2 / T3-T4 0.24 1 Nódulo linfoide + 38 48,10 1 50,00 - 31 39,24 0 0,00 No determinado 10 12,66 1 50,00 Xi2 0.8 Valor P 1 Estrógeno + 50 63,29 1 50,00 - 29 36,71 1 50,00 Xi2 0.148 Valor P 1 Progesterona + 43 54,43 1 50,00 - 36 45,57 1 50,00 Xi2 0.015 Valor P 1 65 HER2 + 25 31,65 0 0,00 - 54 68,35 2 100,00 Xi2 0.915 Valor P 0.856 Márgenes quirúrgicos + 27 34,18 0 0,00 - 27 34,18 1 50,00 No determinado 25 31,65 1 50,00 Xi2 0.982 Valor P 1 66 CAPÍTULO 4: DISCUSIÓN La presente investigación, se llevó a cabo en base a la creciente necesidad de investigación en temas relacionados al cáncer de mama, para el desarrollo de metodologías de detección temprana aplicativas. Según Checa en el 2007, el estudio de polimorfismos comprende un método investigativo empleado para la comprensión parcial de los mecanismos de susceptibilidad a esta enfermedad, al asociar la presencia de una variante polimórfica con el riesgo a desarrollarla. El estudio del gen AKT1 es un blanco de gran interés, su importancia radica en los efectos metabólicos que se producen tras la activación de la vía PI3K/AKT, debido a la fosforilación de decenas de targets importantes para el metabolismo, traducción, proliferación, supervivencia, angiogénesis y motilidad (Lema, 2012). En base a esto, la presente investigación llevó a cabo el análisis de las variantes polimórficas: E17K, E17S y L357P del gen AKT1 y su asociación con características histopatológicas e inmunohistoquímicas en población mestiza ecuatoriana afecta con cáncer de mama, generando un estudio retrospectivo caso-control. En el análisis de la población estudiada, se encontró que el tipo de carcinoma más común, fue el Ductal invasivo (CDI) con un porcentaje total de 74.07%, información que resulta coherente con los datos estadísticos reportados a nivel mundial por la ACS (American Cancer Society) en el 2013, en los que estimaron que la mayoría de casos en ese año fueron diagnosticados con algún tipo de cáncer invasivo, principalmente CDI. Además, mencionan que por cada cuatro casos diagnosticados con CDI, uno es diagnosticado con cáncer ductal in situ (CDIS), lo que guarda similitud con los resultados de este estudio, en el que el CDIS se presentó en el 7.41% de la población afecta con CM. 67 Se determinó que el rango de edad más común entre las mujeres con cáncer de mama (56.79%), correspondía a la categoría de 50-80 años, lo que se debe a que la incidencia del CM y las tasas de mortalidad respectivas, se incrementan con la edad. De este modo, el 69% de nuevos casos y el 88% de las muertes por CM ocurren en mujeres de 50 años o más (American Cancer Society, 2013). En el análisis de la lateralidad, se verificó que la mayoría de pacientes presentaron afectación en la mama derecha (58.02%), en comparación a la izquierda (41.98%), lo que difiere en cuanto a estudios que han reportado que existen más mujeres diagnosticadas con cáncer en el seno izquierdo, que en el derecho (Senie, et al., 1980). Sin embargo, esta diferencia, se debe a que la población de estudio pertenecía a una etnia distinta a la de los estudios reportados (etnias: caucásica o afro-americana). Las mujeres con afectación en la mama izquierda, tienen mayor riesgo a desarrollar complicaciones cardiovasculares como oclusión de la arteria coronaria y trombosis cerebrovascular, como consecuencia de la aplicación de radio-terapia en el lado izquierdo del pecho (Amer, 2014). El principal grupo en el estadío tumoral, fue el grupo de los estadíos T1 y T2, que son casos que han superado la fase inicial del cáncer (To), o carcinoma in situ. Es decir que, en la mayor parte de los casos, el cáncer se ha formado y ha empezado a crecer y en algunos de ellos, a diseminarse (INC, 2014). Sin embargo, estos estadíos aun presentan altos porcentajes de supervivencia a 5 años y la posibilidad de aplicar distintas terapias curativas o cirugías para la remoción del cáncer de mama (American Cancer Society, 2013). Adicionalmente, el pronóstico de un paciente con cáncer de mama, depende de la invasión de células cancerígenas a nódulo linfoide, variable que en este estudio se encontró positiva para la mayoría de los casos (48%), lo que indica un riesgo mayor de propagación de la enfermedad o metástasis (Rubio, et al., 2009). 68 En el análisis del grado tumoral, se evidenció que en la mayoría de casos, los tumores estaban moderadamente diferenciados (SBRII), es decir que presentaban células cancerígenas con cierta semejanza a las células normales, pero con mayor velocidad de multiplicación y división que ellas (BreastCancer, 2013). Además, se realizó el análisis de los receptores hormonales: Progesterona, estrógeno y HER2, de gran importancia ya que a través de él, se conoce la posibilidad de aplicar terapias hormonales. Más del 50% de los casos, presentaron expresión de los receptores hormonales (RE y RP), lo que se correlaciona con tumores de bajo grado histológico que responden a tratamiento hormonal sobretodo en pacientes post-menopaúsicas; aproximadamente 77% de las pacientes con tumores RE y RP positivos responden a terapia hormonal, un 27% responden cuando son RE positivos y RP negativos, y 46% responden cuando son RE negativos y RP positivos (Mohammed, Lakatua, & Haus, 1986) . Por otro lado, la mayor parte de las pacientes, no presentaron expresión de HER-2, lo que es favorable, ya que cuando hay sobreexpresión, ésta puede ser predictiva de resistencia a terapia hormonal (Wright, Nicholson, & Angus, 1992). Adicionalmente, a partir del análisis de los receptores hormonales mencionados, el Médico Patólogo, estableció el tipo de Cáncer de mama, ya sea Luminal A, Luminal B, Basal o HER2. En este estudio, se determinó que el 43.21% de ellos pertenecían al tipo Luminal A, información coherente con las estadísticas actuales, que indican que alrededor del 40% de los casos corresponden a Luminal A, siendo el subtipo más común entre los afectos con cáncer de mama (Perou & Borresen-Dale, 2011). Del 10-20% de los casos son del tipo Luminal B, del 10-20% del tipo Basal y alrededor del 10% son de tipo HER2 (neu) (Voduc KD et al., 2010). Estos datos son similares a los encontrados en este estudio: 24.69% fueron casos de tipo Basal, 19.75% de tipo Luminal B, y 12.35% de tipo HER2 (neu). 69 En cuanto al análisis de los datos clínicos registrados, finalmente está el de márgenes quirúrgicos, el mismo que indicó que el 33% de la población de estudio presentaba células cancerígenas en los bordes quirúrgicos de las biopsias, de modo que en estos casos, se requerirá una cirugía adicional para extirpar las células cancerígenas restantes (BreastCancer, 2013). Para el análisis molecular de las muestras, inicialmente se llevó a cabo el proceso de extracción de ADN, en el que se observaron inconvenientes en cuanto a la baja calidad del ADN extraído a partir de tumores embebidos en parafina, lo que se debe al proceso de fijación que ha sufrido el tejido de interés. En general, la recuperación de los ácidos nucleicos (ADN y ARN), a partir de muestras que han sido fijadas y embebidas en parafina, se considera un reto, lo que se debe a una serie de motivos, como el uso de formaldehído, utilizado comúnmente en este proceso, que si bien no degrada físicamente los ácidos nucleicos, conduce a la generación de entrecruzamientos de los componentes proteicos del tejido, lo que dificulta los procesos de extracción. Además, los ácidos nucleicos se fragmentan en situaciones donde la solución que contiene el fijador, ha perdido gran cantidad de buffer, ya que el pH tiende a ser extremadamente bajo (Gilbert, et al., 2007). Debido a esto, se realizaron modificaciones en el proceso de extracción de ADN a partir de tumores, con el fin de optimizar el proceso y obtener mejores resultados en cuanto a la calidad y cantidad de ADN. De este modo, se incrementó el uso de Proteinasa K de 20 μL a 40 μL y se extendió el proceso de incubación del tejido con el buffer de digestión, hasta 24 h. Es importante señalar que algunas muestras de biopsias contenían poca cantidad de tejido, lo que se manifestó en las bajas concentraciones de ADN obtenidas. Según Gilbert y Cols en el 2007, los inconvenientes en la recuperación de ADN a partir de muestras embebidas en parafina, generan serios desafíos en 70 estudios genéticos desarrollados en torno a la reacción en cadena de la polimerasa (PCR), debido a la poca cantidad y calidad del ADN extraído. Esto se reflejó en la presente investigación, debido a que a partir de 100 muestras de tumores, sólo en 81 de ellas se obtuvieron amplicones-PCR óptimos para la secuenciación del fragmento. Luego del análisis de secuencias de los fragmentos amplificados, se encontró un sólo genotipo distinto al salvaje, para la variante E17K. Este polimorfismo, ha sido reportado en distintos estudios a nivel mundial, en los que se lo ha encontrado en el 4.3% (Kim, Jeong, Yoo, & Lee, 2008), 6% (Bleeker, et al., 2008) y 8% (Carpten, et al., 2007) de las poblaciones de estudio respectivas, en pacientes con cáncer de mama. En este estudio, la variante polimórfica E17K fue detectada sólo en (2/81), es decir el 2.5% de la población afecta, siendo ésta la primera investigación realizada en la población ecuatoriana en cuanto al análisis del gen AKT1. La diferencia respecto a las frecuencias anteriormente reportadas, en otras poblaciones, se relaciona estrechamente a las distintitas condiciones etnológicas entre ellas. Según las estadísticas actuales de la Sociedad Americana de Cáncer en el 2013, las tasas de incidencia de cáncer de mama son mayores en la población blanca (no hispánica), a la que corresponderían los estudios de Bleeker y Cols (2008) (Italia) y Carpten y Cols (2007) (USA), que en la población hispánica o latina, a la que corresponde el presente estudio. Es notable además, que la mutación E17K ha sido encontrada únicamente en casos con cáncer de mama ductal y lobular (Bleeker, et al., 2008), lo que se corrobora en esta investigación, debido a que los casos encontrados con el genotipo perteneciente al homocigoto raro A/A, padecían de cáncer ductal invasivo. En cuanto a los polimorfismos E17S y L3757P, no se encontraron casos o controles que presentaran un genotipo distinto al salvaje, por lo tanto no se 71 determinó ninguna de estas variantes. Esto contrasta con un estudio realizado por Shanthi, y Cols (2014), en el que a través de un modelo computacional se identificaron las variantes mencionadas como mutaciones potencialmente patogénicas del gen AKT1 y por tanto de posible utilidad en el diagnóstico molecular de enfermedades relacionadas; este hecho evidencia que si existe una diferencia al realizar un análisis a nivel computacional, en relación a uno a nivel de laboratorio. Hasta el día de hoy, todas las investigaciones realizadas, han verificado una sola mutación en el gen AKT1, que corresponde al polimorfismo E17K (Bleeker, et al., 2008). Posteriormente, en el análisis de las frecuencias genotípicas y alélicas, no se encontraron diferencias significativas entre grupos (p>0.05), ya que ambos estaban en equilibrio HWE. Esto se debe al bajo número de casos encontrados con un genotipo distinto al salvaje y a la ausencia de heterocigotos G/A en casos y controles. De este modo, en ningún caso, se pudo afirmar la asociación del genotipo AA (homocigoto raro) con alguna de las características clínicas, histopatológicas e inmunohistoquímicas de los pacientes, por el bajo de número de casos encontrados con este genotipo, que consecuentemente generó valores no significativos (p>0.05), en cada uno de los análisis de asociación. No se pudo llevar a cabo el análisis estadístico del Odds ratio, debido a la ausencia de controles que presentaran un genotipo distinto al salvaje, dato de suma importancia en el cálculo del riesgo relativo que representaría la presencia del polimorfismo E17K en cuanto al desarrollo de cáncer de mama. Así mismo, no se pudo establecer una asociación entre la variante y la incidencia de la enfermedad en la población mestiza ecuatoriana. 72 CAPÍTULO 5: CONCLUSIONES La presente investigación se llevó a cabo en base a la creciente evidencia que indica que las alteraciones en la vía de señalización del gen AKT, desempeñan un papel importante en el desarrollo del cáncer. En el análisis de los polimorfismos: E17K, E17S y L357P del gen AKT1, en la población mestiza ecuatoriana afecta con cáncer de mama, se verificó únicamente la presencia del SNP E17K. El polimorfismo E17K, se encontró presente en un 2.5% de la población afecta de estudio, bajo el genotipo A/A (homocigoto raro). Las variantes analizadas E17S y L357P, no han sido verificadas en otros estudios, pero han sido señaladas como posibles mutaciones patogénicas relacionadas al cáncer de mama. En esta investigación, la primera respecto al análisis del gen AKT1 en el Ecuador, no se pudo verificar la asociación de los polimorfismos mencionados, por su ausencia en la población de estudio. Estos resultados, que contrastan con el estudio de un modelo computacional que determinaba a las variantes E17S y L357P como marcadores potencialmente útiles en el cáncer de mama, muestran la importancia de la precisión en el uso de herramientas bioestadísticas predictivas, y la vulnerabilidad de tales estudios. El establecimiento de las frecuencias alélicas y genotípicas para E17K, evidenció que no habían diferencias significativas entre los individuos afectos y controles, encontrándose ambos grupos en equilibrio HWE. Debido al bajo número de casos encontrados con la mutación E17K, no se pudo establecer una asociación significativa con el riesgo a desarrollar cáncer de mama (Odds ratio) o con las distintas características histopatológicas e inmunohistoquímicas de los pacientes enfermos, por lo que no se puede corroborar el potencial de los polimorfismos del gen AKT1 en 73 estudio, como biomarcadores moleculares para el diagnóstico preventivo del cáncer de mama. CAPÍTULO 6: RECOMENDACIONES En los procesos de extracción de ADN a partir de muestras provenientes de tumores o biopsias embebidas en parafina, es recomendable la utilización de kits específicos o a su vez el uso de las modificaciones realizadas para el protocolo utilizado en este estudio. Además, en los procesos previos al análisis de la secuencia se sugiere purificar el producto de la PCR mediante columnas, y emplear un kit de purificación BigDye XTerminator para la purificación de la reacción de secuencia, como se realizó en esta investigación. Es necesario llevar a cabo nuevos estudios que analicen distintos polimorfismos del gen AKT1, debido a la importancia que tiene la ruta PI3K/AKT en el desarrollo del cáncer, donde se incluya un mayor número de casos y controles, de la población mestiza ecuatoriana. Es importante, llevar a cabo estudios de expresión génica con respecto al polimorfismo E17K y a nuevas variantes que puedan verse relacionadas con el desarrollo del cáncer de mama, con el fin de elucidar los resultados de este estudio o hallar posibles biomarcadores moleculares para la enfermedad. También, se recomienda estudiar la asociación del polimorfismo E17K con el tipo de cáncer Ductal, relacionado en todos los estudios anteriores y en esta investigación, con la presencia de la variante. 74 CAPÍTULO 7. BIBLIOGRAFÍA ACS. (2014). Cáncer de seno (mama). Recuperado el 15 de Octubre de 2014, de American Cancer Society: http://www.cancer.org/acs/groups/cid/documents/webcontent/002284pdf.pdf AEEC. (2013). Cáncer de mama. Recuperado el 15 de Octubre de 2014, de Asociación Española contra el Cáncer: https://www.aecc.es/sobreelcancer/cancerporlocalizacion/cancermam a/paginas/diagnosticoprecoz.aspx Alonso, Á. (2013). Mecanismos genéticos en cáncer hereditario. Recuperado el 14 de Octubre de 2014, de Instituto Roche: http://www.geneticaycancer.es/Area_Molecular/Mecanismos_genetico s_en_cancer_hereditario/Conceptos_basicos.html Amer, M. (2014). Genetic factors and breast cancer laterality. Cancer Management and Research, 191–203. American Cancer Society. (2013). Breast Cancer Facts & Figures 2013-2014. Atlanta. Arce, C. (2011). OncoGuía: Cáncer de mama. Recuperado el 17 de Octubre de 2014, de Instituto Nacional de Cancerología: http://www.incan.org.mx/revistaincan/elementos/documentosPortada/1 327324685.pdf ATSDR. (2005). ¿Qué es el cáncer? Recuperado el 14 de Octubre de 2014, de Agency for toxic substances and disease registry: http://www.atsdr.cdc.gov/es/general/cancer/es_cancer_fs.pdf Bleeker, F., Felicioni, L., Buttitta, F., Lamba, S., Cardone, L., Rodolfo, M., & Scarpa, A. (2008). AKT1E17K in human solid tumours. Oncogene, 5648–5650. BreastCancer. (2013). Diagnóstico. Obtenido de BreastCancer.org: http://www.breastcancer.org/es/sintomas/diagnostico BreastCancer. (2014). Tipos de cáncer de mama. Recuperado el 16 de Octubre de 2014, de BreastCancer.org: http://www.breastcancer.org/es/sintomas/tipos Cancer Research UK. (2013). Gene variations and cancer risk – more results, more answers and more questions. Obtenido de Cancer Research UK: 75 http://scienceblog.cancerresearchuk.org/2013/03/27/gene-variationsand-cancer-risk-more-results-more-answers-and-more-questions/ Carpten, J., Faber, A., Horn, C., Donoho, G., Briggs, S., Robbins, C., & Hostetter, G. (2007). A transforming mutation in the pleckstrin homology domain of AKT1 in cancer. Nature, 439 – 444. Carrera. (2008). Estudio del polimorfismo Ile 462 Val del gen CYP1A1 en población ecuatoriana con cáncer de mama. Recuperado el 13 de Octubre de 2014, de Repositorio de la PUCE: http://www.puce.edu.ec/portal/content/Tesis%3A%20Gen%C3%A9tica /519?link=oln30.redirect Checa, M. (2007). Polimorfismos genéticos: importancia y aplicaciones. Rev Inst Nal Enf Resp Mex, 213-221. Córdova, A., & Coello, P. (2011). Determinación de la frecuencia y tipos de cáncer de mama, SOLCA-Cuenca. Recuperado el 14 de Octubre de 2014, de Repositorio de la Universidad de Cuenca: http://dspace.ucuenca.edu.ec/bitstream/123456789/3500/1/MED65.pd f Dimmeler, S., Fleming, I., Fisslthaler, B., Hermann, C., Busse, R., & AM, Z. (1999). Activation of nitric oxide synthase in endothelial cells by Aktdependent phosphorylation. Nature, 601-605. El Universo. (12 de Diciembre de 2013). Cáncer de mama aumenta un 20% desde 2008, según la OMS. El Universo. Gilbert, M., Haselkorn, T., Bunce, M., Sanchez, J., & Lucas, S. (2007). The Isolation of Nucleic Acids from Fixed, Paraffin-Embedded Tissues– Which Methods Are Useful When? PLoS ONE, e537. Gutierrez, G. (2012). Detección de SNPs en genes asociados a obesidad y cáncer de mama en pacientes del noreste de México. Recuperado el 10 de Octubre de 2014, de http://eprints.uanl.mx/4445/1/1080256580.pdf Hennessy, B., Smith, D., Ram, P., Lu, Y., & Mills, G. (2005). Exploiting the PI3K/AKT pathway for cancer drug. Nature, 988-1004. Hidalgo, A., & Jiménez, G. (2009). Bases genómicas del cáncer de mama: avances hacia la medicina personalizada. Salud pública Méx, 00363634. 76 INC. (2014). Cáncer de seno (mama). Recuperado el 16 de Octubre de 2014, de Instituto Nacional del Cáncer: http://www.cancer.gov/espanol/pdq/tratamiento/seno/HealthProfession al/page4 Instituto Nacional de Cancerología. (2004). El Cáncer: aspectos básicos sobre su biología, clínica, prevención, diagnóstico y tratamiento. Recuperado el 15 de Octubre de 2014, de Instituto Nacional de Cancerología E.S.E: http://www.cancer.gov.co/~incancer/instituto//files/libros/archivos/f8fe1 8efc7008fbc9669068b86f9ae03_El%20cancer%20(%20aspectos%20 basicos%20)%20pacientes.pdf Instituto Nacional del Cáncer. (2011). Manual de Enfermería Oncológica. Argentina. Ion Baba, A. (2007). Comparative Oncology. Bucarest: Academiei Române. Kim, M., Jeong, E., Yoo, N., & Lee, S. (2008). Mutational analysis of oncogenic AKT E17K mutation in common solid cancers and acute leukaemias. British Journal of Cancer- Nature, 1533-1535. Kumar, V., Aster, J., Fausto, N., & Abbas, A. (2010). Patología estructural y funcional. España: Elsevier-Saunders Publications. Lema, M. (2012). RUTA PI3K / PTEN / AKT. Recuperado el 17 de Octubre de 2014, de CNIO Oncología Molecular: http://mauriciolema.webhost4life.com/Moloncol2012/files/MolOncol04_ PI3K_PTEN_AKT.pdf Mende, I., Malstrom, S., Tsichlis, P., Vogt, P., & Aoki, M. (2001). Oncogenic transformation induced by membrane-targeted Akt2 and Akt3. Oncogene, 4419 – 4423. Mohammed, R., Lakatua, A., & Haus, E. (1986). Estrogen and progesterone receptors in human breast cancer: correlation with histologic subtype and degree of differentiation. Cancer, 1076-1081. Moreno, A. (2010). SAC. Obtenido de Sociedad Argentina de Citología: http://sociedaddecitologia.org.ar/sac/imagen-fichas-carcinomamucinoso-de-mama/ MSP. (2012). El Ministerio de Salud comprometido en la lucha contra el cáncer de mama. Recuperado el 13 de Octubre de 2014, de Ministerio de Salud Pública: http://www.salud.gob.ec/el-ministerio-de-saludcomprometido-en-la-lucha-contra-el-cancer-de-mama/ 77 MSP. (2014). Ministerio de Salud garantiza acceso a la salud de pacientes con cáncer. Recuperado el 24 de Septiembre de 2014, de Ministerio de Salud Pública: http://www.salud.gob.ec/ministerio-de-salud-garantizaacceso-a-la-salud-de-pacientes-con-cancer-2/ Muise-Helmericks, R., Grimes, H., Bellacosa, A., Malstrom, S., Tsichlis, P., & Rosen, N. (1998). Cyclin D expression is controlled posttranscriptionally via a phosphatidylinositol 3-kinase/Akt-dependent pathway. J Biol Chem, 29864 – 29872. National Cancer Institute. (2014). NCI. Obtenido de Significado de los cambios en los senos: http://www.cancer.gov/espanol NCI. (2012). Cáncer: un cambio en la conversación. Recuperado el 15 de Octubre de 2014, de National Cancer Institute- NIH: http://www.cancer.gov/PublishedContent/Files/espanol/instituto/Bypas s_SPA_508Jul13v1.pdf OMS. (2012). Cáncer de mama: prevención y control. Recuperado el 15 de Octubre de 2014, de Organización Mundial de la Salud: http://www.who.int/cancer/detection/breastcancer/es/index1.html Ozes, O., Mayo, L., Gustin, J., Pfeffer, S., Pfeffer, L., & Donner, D. (1999). NFkappaB activation by tumour necrosis factor requires the Akt serinethreonine kinase. Nature, 82-85. Pérez, J., Mariángel, P., & Poblete, M. (2004). Carcinoma mamario papilar intraquístico. Cuad. Cir. , 70-75. Perou, C., & Borresen-Dale, A. (2011). Systems biology and genomics of breast. Cold Spring Harb Perspect Biol. Pinto, Y., Sánchez, W., Ibáñez, M., & Ramírez, S. (2006). Polimorfismos del Gen P53 en Cáncer de Mama Familiar en una Población Colombiana . SALUD UIS. Pinzón, C., Serrano, M., & Sanabria, M. (2009). Papel de la vía fosfatidilinositol 3 kinasa (PI3K/Akt) en humanos. Rev. Cienc. Salud. Bogotá (Colombia), 47-66. Ramírez, S., Escobar, S., & Rangel, N. (2009). Polimorfismos de los genes P53, CYP1A1, CYP1B1, GSTM1, GSTT1 Y GSTP1 en cáncer de seno familiar en una población colombiana. Recuperado el 13 de Octubre de 2014, de http://repository.urosario.edu.co/bitstream/handle/10336/1305/518662 51.pdf?sequence=7&isAllowed=y 78 Ríos, M., & Hernández, M. (2001). Los genes supresores de tumores y el cáncer . Rev Cubana Oncol, 65-71. Rubio, I., Roca, I., Sabadell, D., & Xercavins, J. (2009). Beneficio de la biopsia del ganglio linfático centinela en pacientes con carcinoma in situ de mama. CIR. ESP., 92– 95. Santos, E. (2013). Biología y genética molecular del cáncer. Ambiociencias, 3-6. Schnitt, S. (2009). Classification and prognosis of invasive breast cancer: From morphology to molecular taxonomy. Mod. Pathol. Recuperado el 17 de Octubre de 2014, de http://www.ncbi.nlm.nih.gov/pubmed/20436504 Senie, R., Rosen, P., Lesser, M., Snyder, R., Schottenfeld, D., & Duthie, K. (1980). Epidemiology of breast carcinoma II: factors related to the predominance of left-sided disease. Cancer, 1705–1713. SEOM. (2013). Las cifras del cáncer en España 2014. Recuperado el 15 de Octubre de 2014, de Sociedad Española de Oncología Médica: http://www.seom.org/seomcms/images/stories/recursos/Las_cifras_del _cancer_2014.pdf SEP. (2013). EPIMARC1 - Estudio Epidemiológico Multicéntrico de Marcadores Moleculares de Cáncer en Ecuador. Recuperado el 8 de Octubre de 2014, de Sociedad Ecuatoriana de Patología: http://www.sep-ec.com/index.php/epimarc1 Shanthi, V., Rajasekaran, R., & Ramanathan, K. (2014). Computational Identification of Significant Missense Mutations in AKT1 gene. Cell Biochem Biophys - Springer, 003-009. Sifuentes, A., Talamás, J., Reyes, F., & Reyes, M. (2012). Asociación del cáncer de mama con los polimorfismos T-66G y G-156GG del gen SPP1 y las concentraciones séricas de osteopontina. Ginecol Obstet Mex, 22-29. Spalvieri, M., & Rotenberg, R. (2004). Medicina genómica. Aplicaciones del polimorfismo de un nucleótido y micromatrices de ADN. Medicina (B. Aires), 1669-9106. Testa, J., & Bellacosa, A. (2001). AKT plays a central role in tumorigenesis. Proc Natl Acad Sci USA, 10983 – 10985. 79 Vidal, S. (2008). Cáncer de mama hereditario: identificación y elección de pacientes para estudio molecular de los genes BRCA. Cancerología, 51-61. Wei, L., Yang, Y., & Yu, Q. (2001). Tyrosine kinase-dependent, phosphatidylinositol 3'-kinase, and mitogen-activated protein kinaseindependent signaling pathways prevent lung adenocarcinoma cells from anoikis. Cancer Res, 2439 – 2444. WHO. (2012). GLOBOCAN 2012: Estimated Cancer Incidence, Mortality and Prevalence Worldwide in 2012. Recuperado el 16 de Octubre de 2014, de World Health Organization: International Agency for Research Cancer: http://globocan.iarc.fr/Pages/fact_sheets_cancer.aspx Wright, C., Nicholson, S., & Angus, B. (1992). Relationship between c-erbB— 2 protein product expression and response to endocrine therapy in advanced breast cancer. Br J Cancer, 118-121. 80 CAPÍTULO 8. ANEXOS Anexo 1 Resumen de historias clínicas de los pacientes afectos con cáncer de mama Información General del Paciente CARACTERISTICAS HISTOPATOLOGICAS-HISTOQUIMICAS Código IIB Nº Historial Clínico Edad TM 3 ADN 163585 37 Parafina Derecho TM 4 ADN 165358 62 Parafina Izquierdo TM 5 ADN 154216 46 Parafina Derecho TM 6 ADN 161868 61 Parafina Derecho Muestra Seno Afectad o Comenta Diagnost Bordes rio/ % ico Diferencia Quirurgi Tumor Patológi ción cos Células co Moderada Ca ductal mente A-90% profundo invasivo diferencia do SBR II Moderada mente diferencia do con Ca ductal libre A-80% invasivo diferencia cón neuroend ócrina Moderada mente Ca ductal libre A-30% invasivo diferencia do SBR II Ca ductal A-70% SBR II profundo invasivo Estadio Estróge Progest patológi no erona co HER2 NEU Clasificaci ón Molecular pT1c positivo No(sn) negativo negativo (3+) Mx Lo Vo HER2u pT2 N1a negativo negativo negativo Mx Lo V1 Basal pR1 N1 Mx positivo positivo positivo (2+) Luminal A pR1 pN1 positivo positivo negativo Luminal A pMx 81 TM 7 ADN 149045 38 Parafina Derecho A-70% TM 8 ADN 165239 52 Parafina Izquierdo A-70% TM 9 ADN 163492 37 Parafina Izquierdo A-30% TM 10 ADN 126200 43 Parafina Derecho A-60% TM 11 ADN 164771 81 Parafina Derecho A-60% TM 12 ADN 99856 76 Parafina Izquierdo A-30% TM 13 ADN 168972 36 Parafina Izquierdo TM 14 ADN 174230 47 Parafina Derecho 90% Bien Ca pR2 N1 positivo mucinos Diferencia profundo Mx do o bifocal Ca papilar grado nuclear pR2 N2 II con SBR II profundo positivo Mx compon ente ductal invasivo Ca Bajo a pR2 No intraduct libre positivo intermedio Mx al SBR II moderada Ca ductal pT1 pNx mente libre positivo invasivo pMx diferencia do Bien pT1a N1a Ca ductal diferencia profundo positivo invasivo Mx L1 Vo do SBR I Ca ductal microinv Bien pT1b No asor diferencia profundo positivo Mx Lo Vo (intraduc do SBR I tal) pT1b Ca dutal pN1c positivo invasivo pMx Moderada pT1b Ca ductal mente pN2a positivo invasivo diferencia pMx positivo negativo Luminal A positivo negativo Luminal A positivo negativo Luminal A positivo (2+) luminal B positivo negativo luminal A negativo positivo negativo Luminal A positivo negativo Luminal A positivo positivo (2+) Luminal A 82 TM 15 ADN 79576 45 Parafina Izquierdo A-70% TM 16 ADN 118745 46 Parafina Derecho A-50% 46 Parafina Derecho A-90% TM 17 ADN TM 19 ADN 161646 57 Parafina Derecho A-40% TM 20 ADN 165481 36 Parafina Izquierdo A-30% TM 22 ADN 57965 54 Parafina Derecho A-80% TM 26 ADN 165311 56 Parafina Derecho A-80% TM 27 ADN 164758 50 Parafina Izquierdo A-20% do SBR 7/9 pobrement e Ca ductal diferencia profundo invasivo do SBR 8/9 Ca ductal invasivo acompa ñado SBR G II profundo con Ca intraduct al Ca libre medular Moderada mente Ca ductal profundo invasivo diferencia do SBR II Bien Ca ductal diferencia libre invasor do SBR I Moderada Ca ductal mente profundo invasor difenrecia do SBR II bien Ca ductal diferencia libre invasor do SBR II Ca intraduct profundo al con necrosis pT1b pN2a pMx negativo negativo negativo Basal pT1b pNo negativo negativo negativo pMx Basal pT1b pNo negativo negativo negativo pMx Basal pT1C No negativo positivo positivo Mx Basal pT1c No(sn) positivo positivo negativo Luminal A Mx Lo Vo pT1c pN0/10 pMx pT1c pN1a pMx positivo positivo negativo Luminal A positivo positivo positivo pT1c pN2 negativo negativo negativo pMx Luminal B Basal 83 166700 43 Parafina Izquierdo A-90% Lobulillar infiltrant e pT1c pNo positivo positivo positivo pMx (1+) 165884 66 Parafina Izquierdo C-40% Ductal in situ pT1c pNo positivo positivo negativo Luminal A pMx TM 30 ADN 149604 59 Parafina Izquierdo TM 34 ADN 104281 59 Parafina Derecho TM 35 ADN 164443 39 Parafina Izquierdo TM 38 ADN 167234 37 Parafina Izquierdo TM 40 ADN 166944 84 Parafina Derecho 128516 52 Parafina Izquierdo TM 28 ADN TM 29 ADN TM 41 ADN Bien Ca ductal diferencia libre invasor do Moderada mente A-60%; 2 Ca ductal direfencia libre tumores invasor do SBR G II Moderada Ca ductal mente C-80% libre invasor diferencia do SBR II Moderada Ca ductal mente A-50% libre invasor diferencia do SBR II Ca A-30% lobulillar profundo invasor A-60% A-40% TM 43-1 ADN 164774 34 Parafina Derecho A-80% TM 44 ADN 98022 49 Parafina Derecho A-50% Ca ductal invasor SBR II Pobrement e Ca ductal diferencia invasor do SBR III 8/9 Ductal in situ Luminal B -pT1c, pN1, pMx positivo positivo negativo Luminal A pT2 N1 Mx positivo positivo negativo Luminal A pT2 No Mx positivo positivo negativo Luminal A pT2 No negativo negativo negativo Mx Lo Vo Basal pT2 No negativo negativo negativo Mx Lo Vo Basal profundo pT2 No negativo negativo negativo Mx Lo Vo Basal libre pT2 pN1 negativo negativo negativo pMx Basal pT2 pN1 Luminal A positivo positivo negativo pMx 84 TM 45 ADN 165158 49 Parafina Izquierdo A-5% Ca ductal invasor SBR I residual libre 168055 42 Parafina Izquierdo C-60% TM 47 ADN 167023 45 Parafina Derecho A-80% TM 48 ADN 164122 54 Parafina Derecho A-50% TM 51 ADN 166820 51 Parafina Derecho A-80% TM 52 ADN 97504 40 Parafina Derecho B-90% Parafina Izquierdo F-90%; M-90% (Primari oo Bien metásta Ca ductal diferencia profundo sis?); invasor do Grado I mama izq+ ovario der. 167262 77 Basal Moderada Ca ductal mente pT2 pN1a profundo positivo positivo negativo Luminal A invasor diferencia pMx do SBR II Moderada Ca ductal mente pT2 pN1a positivo libre negativo negativo HER2u invasor diferencia pMx (2+) do SBR II Moderada Ca ductal mente pT2 pN1c profundo positivo positivo negativo Luminal A invasor diferencia pMx do SBR II Moderada Ca ductal mente pT2 pN2a positivo libre positivo positivo Luminal B invasor diferencia pMx (3+) do SBR II Ca mixto pobrement (invasor e pT2 pNo profundo negativo negativo negativo Basal y papilar diferencia pMx invasor) do SBR III TM 46 ADN TM 53-1 ADN pT2 pN1 positivo negativo negativo pMx (1+) pT2 pNo positivo positivo negativo Luminal A pMx 85 TM 54-1 ADN 169252 44 Parafina Izquierd o A-40% TM 55 ADN 167244 59 Parafina Derecho C-30% Ca ductal invasor Ca papilar invasor TM 56 ADN 168806 82 Parafina Derecho A-40% Ca ductal invasor TM 57 ADN 168489 53 Parafina Derecho A-20% Ca ductal invasor 48 Parafina Derecho A-90% 60% TM 61 ADN TM 63 ADN 116310 67 Parafina Derecho TM 64 ADN 162485 52 Parafina Derecho TM 66 ADN 162204 55 Parafina Izquierd o 74% Izquierd células o tumorale s TM 69 ADN 169619 39 Parafina TM 72 ADN 169565 78 Parafina Derecho Ca ductal invasor Ca ductal invasor Ca ductal invasor 24% 27% células Ca ductal invasor Ca ductal invasor Ca ductal invasor SBR II profundo pT2 pNo negativ positivo positivo pMx o Luminal A libre pT2 pNo negativ negativ positivo pMx o o Luminal B Grado I bien pT2 pNo libre diferencia pMx do Grado I bien pT2 pNo libre diferencia pMx do pT2 Bien diferencia profundo pNo(c) do SBR I pMx Bien pT2(2) diferencia libre pN1 pMx do Pobremen pT2(m) te libre N3a Mx diferencia L1 V1 do SBR III Poco pT2, diferencia profundo pN1, do, Grado pMx 3 Poco diferencia pT2_pN1 libre do, Grado b_pMx 3 Moderada profundo mente positivo positivo negativ o Luminal A positivo positivo negativ o Luminal A negativ positivo positivo o (3+) positivo positivo negativ o HER2u Luminal A negativ o negativ positivo o (2+) HER2u negativ o negativ negativ o o Basal negativ o negativ positivo o (3+) HER2u pT3 pN1 negativ positivo positivo pMx o Luminal A 86 tumorale s TM 74 ADN 171371 49 Parafina TM 80 ADN 172194 63 Parafina Derecho Ca ductal invasor Izquierd o 80% TM 88 ADN 172418 71 Parafina Derecho E-30% TM 90 ADN 173526 57 Parafina Izquierd o No hay HQ TM 92 ADN 173046 37 Parafina Derecho TM 95 ADN 85056 51 Parafina Izquierd o A-80% TM 97 ADN 91910 56 Parafina Derecho A-80% TM 101 ADN 166598 50 Parafina Derecho A-40% 80% Ca ductal invasor diferencia do SBR III Poco diferencia do, Grado 3 Poco diferencia do, Grado 3 libre pT3_pN2 negativ _pMx o R2 Nx Mx negativ negativ o o Basal negativ negativ positivo o o Basal Ca moderada metaplá negativ mente sicoprofundo tRo No positivo positivo o diferencia mucinos do a Ca negativ negativ positivo ductal T1N1M0 o o (3+) invasor Bien Ca diferencia negativ ductal T3N1MO positivo positivo do SBR o invasor 5/9 bien Ca diferencia positivo ductal libre T3N0M0 positivo positivo do SBR (1+) invasor 4/3 Ca mucinos negativ o profundo T4N1M0 positivo positivo o intraduct al Moderada Ca mente negativ ductal profundo T2N0M0 positivo positivo diferencia o invasor do Luminal A HER2u Luminal A Luminal A Luminal A Luminal A 87 TM 102 ADN Parafina Izquierd o 174101 76 50% TM 104 ADN 168604 88 Parafina Izquierd o A-70% TM 107 ADN 73486 52 Parafina Derecho TM 114 ADN 173621 65 Parafina TM 117 ADN 174733 67 Parafina Derecho TM 118 ADN 59505 87 Parafina Izquierd o A-90% TM 119 ADN 182900 62 Parafina Derecho A-60% TM 122 ADN 173130 55 Parafina Izquierd o C-90% TM 130 ADN 129807 40 Parafina Derecho C TM 131 ADN 162235 41 Parafina Derecho A Izquierd o 20% A-80% 70% Ca ductal invasor grado III poco diferencia do TisN0M0 negativ o negativ negativ o o Ca negativ negativ papilar libre T2N2M0 positivo o o invasor Ca negativ positivo intraduct Alto grado T3N0M0 positivo o (3+) al moderada Ca mente negativ profundo T2N1M0 positivo positivo ductal y diferencia o lobulillar do Moderada Ca mente negativ positivo ductal profundo T3N2M0 positivo diferencia o (3+) invasor do SBR II Ca negativ negativ mucinos TisN0M0 positivo o o o Ca negativ negativ ductal T4N2M0 positivo o o invasor Bien Ca diferencia T0N1**M negativ ductal libre positivo positivo do SBR 0 o invasor 5/9 Pobremen Ca te negativ ductal libre T2N0M0 positivo positivo diferencia o invasor do SBR III Ca negativ negativ positivo ductal SBR II profundo T4N1M0 o o (3+) invasor Basal Luminal B Luminal B Luminal A Luminal B Luminal B Basal Luminal A Luminal A HER2u 88 *TM100 ADN SOLCA 41 Parafina Derecho *TM113 ADN SOLCA 46 Parafina TM 146 ADN Q13-1414 65 Parafina Derecho TM 140 ADN Q13-1369 46 Parafina TM 155 ADN 745479 35 Biopsia TM 153 ADN 779241 80 Parafina TM 154 ADN 801632 44 Parafina Izquierd o Izquierd o derecho 68% Ca ductal SBR II invasor Ca ductal SBR III invasor Ca ductal SBR II invasor Ca ductal invasor, SBR II lobulillar y linfático Ca ductal infiltrant e SRB 7, G moderad II amente diferenci ado Ca ductal infiltrant SRB, G I e bien diferenci ado Ca ductal infiltrant SBR 6, G e II moderad amente T2N1M0 positivo positivo libre negativ o Luminal A T3N0M0 negativ o negativ positivo o HER2u T2N1M0 negativ o negativ negativ o o Basal T3N0M0 positivo positivo positivo Luminal B positivo positivo negativ a Luminal A positivo positivo negativ o Luminal A positivo positivo positivo Luminal B pT2, pN2a pMx 89 TM 148 ADN 811520 30 Parafina TM 149 ADN 689662 50 Biopsia TM 150 ADN 17212751 37 46 Biopsia Izquierd o TM 163 ADN 810355 55 Tejido Derecho TM 156 ADN 800791 40 Parafina Izquierd o TN 157 ADN 516818 78 Biopsia diferenci ado Ca ductal infiltrant e Ca ductal infiltrant SBR 8, G e poco III diferenci ado Ca ductal infiltrant SBR P, G e poco III diferenci ado Ca ductal SBRA, G infiltrant III e Lobulilla r infiltrant e Ca ductal infiltrant e SBR, G II moderad (6/9) amente diferenci ado pt4 pt4 negativ o negativ positivo o (3+) HER2u negativ o negativ positivo o (3+) HER2u positivo negativ positivo o Luminal B negativ o negativ negativ o o Basal negativ negativ positivo o o Basal positivo negativ negativ o o Luminal B 90 TM 160 ADN 17069267 79 53 Biopsia Izquierd o TM 164 ADN 56014130 4 66 Biopsia Derecho TM 147 ADN 17096604 2 45 Biopsia TM 151 ADN 779241 80 Parafina Derecho 80% Ca ductal infiltrant e SBR 8, G pobreme III nte diferenci ado Ca ductal infiltrant e Fibroade noma intracani cular (invasor) Ca ductal infiltrant SBR, G I e bien diferenci ado profundo pT2 pN2a pMx negativ o negativ positivo o (3+) HER2u positivo negativ negativ o o Luminal B positivo negativ negativ o o Luminal B positivo positivo positivo Luminal B