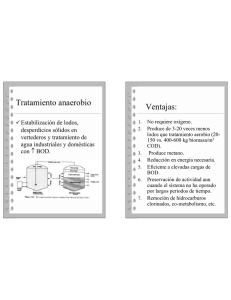
cuerpo memoria2 - Repositorio Académico
Anuncio

UNIVERSIDAD DE CHILE FACULTAD DE CIENCIAS FÍSICAS Y MATEMÁTICAS DEPARTAMENTO DE INGENIERÍA QUÍMICA Y BIOTECNOLOGÍA DINÁMICA DE LA BIODIVERSIDAD EN PROCESOS DE BIOLIXIVIACIÓN MEMORIA PARA OPTAR AL TÍTULO DE INGENIERO CIVIL QUÍMICO MEMORIA PARA OPTAR AL TÍTULO DE INGENIERO CIVIL EN BIOTECNOLOGÍA PAULINA BRUSADELLI ACUÑA PROFESOR GUÍA BLANCA ESCOBAR MIGUEL MIEMBROS DE LA COMISIÓN ORIANA SALAZAR AGUIRRE TOMÁS VARGAS VALERO SANTIAGO DE CHILE OCTUBRE 2007 RESUMEN DE LA MEMORIA PARA OPTAR AL TÍTULO DE INGENIERO CIVIL QUÍMICO E INGENIERO CIVIL EN BIOTECNOLOGÍA POR: PAULINA BRUSADELLI ACUÑA FECHA: 23/10/2007 PROF.GUÍA: Sra. BLANCA ESCOBAR M. “DINÁMICA DE LA BIODIVERSIDAD EN PROCESOS DE BIOLIXIVIACIÓN” El presente trabajo de título tiene por objetivo estudiar la dinámica de la biodiversidad en procesos de biolixiviación, es decir, busca establecer alguna relación entre la variación de los parámetros fisicoquímicos presentes en el ambiente en estudio y las bacterias identificadas para cada condición. Su importancia en estos procesos, radica en determinar los parámetros fisicoquímicos que estimulan o inhiben la actividad de los microorganismos involucrados, para optimizar la recuperación de cobre, importante en nuestro país. Para esto, se utilizó la técnica de determinación de los polimorfismos de los largos de los fragmentos terminales de restricción (T-RFLP) en soluciones obtenidas de una mini columna de biolixiviación, con lo que se logró identificar las poblaciones bacterianas presentes. Para corroborar la información obtenida con el protocolo propuesto por Gabriela Morales, se buscó una enzima de restricción adicional para el método de T-RFLP. La segunda enzima seleccionada correspondió a MspI, la cual mostró ser capaz de distinguir entre dos de las especies bacterianas encontradas en los procesos de biolixiviación, L.ferrooxidans y Sb.thermosulfidooxidans, pero no proporciona ningún aporte frente al otorgado por RsaI, que es capaz de distinguir entre A. ferrooxidans y A.thiooxidans. Se determinó que es válido considerar como bacterias presentes en la solución, aquellas cuyos TRFs se encuentren presentes en la muestra analizada al digerir con cada enzima de restricción por separado., sin poder descartar plenamente las que se encuentren sólo con una enzima por poder asociarse a errores del método de T-RFLP. En la mayoría de las muestras se encontraron α proteobacterias y A.ferrooxidans. Además, en las soluciones estudiadas se determinó la presencia de A.thiooxidans, Sb.thermosulfidooxidans, Sb.acidophilus Desulfosporosinus spp., Shewanella spp. Arthrobacter spp., o Ferrimicrobium acidiphilium, Acidomonas methanolica, Acidocella spp., Acidiphilum spp. o Acidimicrobium ferrooxidans. Al analizar lo parámetros fisicoquímicos de las muestras estudiadas, se destaca la disminución de la diversidad bacteriana a medida que aumentan los iones metálicos en solución y la mayor cantidad de bacterias generadas en las muestras con aireación. Los parámetros fisicoquímicos de la muestra 4 mostraron ser los más favorables para la diversidad bacteriana: Eh de 597 mV, pH inicial de 1,6, pH final de 1,4, concentración inicial de FeSO4 de 6 g/l y aire. A través del índice de diversidad de especies, no se logró observar, en ninguno de los casos, una correspondencia entre el aumento de las condiciones inhibitorias y la disminución del índice. En el caso del índice de Shannon-Wiener, se observó una cierta tendencia a disminuir en algunas muestras a medida que aumentaron los iones metálicos en solución, pese a que ambos índices pueden verse afectados por los errores asociados al T-RFLP. Se observó concordancia entre los dendrogramas elaborados a partir de la aparición de las bacterias en cada muestra, según los resultados de la digestión con RsaI o MspI, debido a que los TRFs asociados a las mismas bacterias fueron agrupados de igual forma. Además se observó que las agrupaciones realizadas eran congruentes con las características de las bacterias encontradas en el sistema estudiado. El análisis de componentes principales mostró no ser concluyente a la hora de agrupar los TRFs asociados a bacterias biolixiviantes según su aparición en las soluciones, principalmente debido a que su elaboración toma en cuenta las abundancias relativas de los fragmentos considerados. Finalmente, no se encontró una relación entre las soluciones, a partir de las bacterias encontradas en cada una, que fuera congruente con los parámetros fisicoquímicos analizados. A mis padres, Mario y Mª Alicia, mis hermanos, Cristóbal, Italo y Romina, y Marco. Agradecimientos En el momento en que termino una etapa más de mi vida, miro hacia atrás y doy las gracias a todos los que en algún momento influyeron en la persona en que me he convertido. En primer lugar, a mi familia le agradezco su apoyo incondicional. Agradezco a mi papá por darme la oportunidad de estudiar la carrera que quise, en donde quise. A mi mamá, por brindarme siempre su apoyo y por tratar de hacerme la vida más sencilla, para poder así enfocarme en mis estudios. A mis hermanos, Cristóbal e Italo simplemente por estar ahí sin importar nada, y a Romina por su alegría de niña que siempre me ha hecho recordar lo lindo que es jugar sin preocupaciones y por su cariño incondicional. No puedo dejar de agradecer a la “mamma”, mi abuelita Alicia, por su constante apoyo y sus rezos de ayuda en los momentos complicados, ni a la “nonna”, Sonia, por su preocupación constante. A Marco le agradezco su apoyo constante, su ayuda y su paciencia desde el primer momento, porque simplemente es mi amigo y compañero, con el que me siento feliz. Agradezco la preocupación y el apoyo incondicional de mi amiga de la vida, Andrea, además de la buena disposición a pasar un momento agradable siempre que fuera requerido. A Karla e Ivana, les agradezco su comprensión y apoyo en este último tiempo, y, por supuesto, su grata compañía. A Germán le agradezco su compañía, su alegría, sus locuras, sus tonteras y su amistad, por sacarme una sonrisa y hacerme olvidar las preocupaciones aunque fuera por un momento. A todos aquellos que me han acompañado, y, cómo olvidarlos, a los que compartieron conmigo en la “salita” les agradezco esos gratos momentos de estudio, ocio y divagaciones. Finalmente, agradezco a mi profesora guía, Sra. Blanca Escobar, por su apoyo y ayuda en el transcurso de la memoria. ..y a todos los que se me hayan quedado en el tintero, gracias… Amigos míos, nos vemos en la vida… ii Índice Capítulo 1. Introducción....................................................................................................................................2 1. 1. Aspectos Generales ..............................................................................................................................2 1. 2. Antecedentes Bibliográficos ................................................................................................................3 1. 2. 1. Microorganismos lixiviantes ........................................................................................................4 1. 2. 2. Factores que influyen en la rapidez de la biolixiviación ..........................................................6 1. 2. 3. Inhibición de la actividad enzimática de los microorganismos biolixiviantes......................7 1. 2. 4. Técnica de T-RFLP.......................................................................................................................7 1. 2. 5. Comparación de poblaciones microbianas a través de análisis estadísticos .........................8 1. 3. Descripción del Proyecto.....................................................................................................................9 1. 4. Objetivos..............................................................................................................................................10 1. 4. 1. Objetivo General........................................................................................................................ 10 1. 4. 2. Objetivos específicos................................................................................................................. 10 1. 5. Alcances................................................................................................................................................10 Capítulo 2. Metodología.................................................................................................................................. 11 2. 1. Materiales .............................................................................................................................................11 2. 2. Métodos................................................................................................................................................11 2. 2. 1. Selección de la segunda enzima a utilizar ............................................................................... 11 2. 2. 2. Obtención de muestras de bacterias desde una mini-columna de biolixiviación ............. 12 2. 2. 3. Extracción de DNA desde soluciones de procesos de biolixiviación ................................ 14 2. 2. 4. T-RFLP........................................................................................................................................ 15 2. 2. 4. 1. Amplificación de un fragmento del rDNA 16S............................................................ 15 2. 2. 4. 2. Purificación y cuantificación del DNA .......................................................................... 15 2. 2. 4. 3. Digestión de los productos de PCR y electroforesis capilar ....................................... 16 2. 2. 4. 4. Procesamiento de los resultados de la electroforesis capilar....................................... 16 2. 2. 5. Análisis utilizados para comparar las especies bacterianas encontradas ............................ 17 2. 2. 5. 1. Determinación de la abundancia relativa de los TRFs................................................. 17 2. 2. 5. 2. Índices de biodiversidad................................................................................................... 17 2. 2. 5. 3. Análisis de cluster.............................................................................................................. 18 2. 2. 5. 4. Análisis de componentes principales.............................................................................. 19 Capítulo 3. Resultados..................................................................................................................................... 20 3. 1. Selección de la segunda enzima a utilizar en los análisis de T-RFLP ..........................................20 3. 2. Variación de los parámetros fisicoquímicos en las muestras estudiadas.....................................21 3. 3. Obtención de microorganismos, DNA genómico y producto amplificado...............................25 3. 4. Determinación de las poblaciones bacterianas en solución y su comparación mediante diversos análisis............................................................................................................................................28 3. 4. 1. Determinación de la abundancia Relativa de los TRFs ........................................................ 28 3. 4. 2. Comparación de los índices de biodiversidad........................................................................ 32 3. 4. 3. Comparaciones de las muestras estudiadas ............................................................................ 33 3. 4. 3. 1. Comparación entre los TRF asociados a bacterias según su aparición en las muestras ............................................................................................................................................... 33 3. 4. 3. 2. Comparación entre las soluciones según la aparición de bacterias en solución....... 35 iii 3. 4. 3. 3. Comparación de los TRFs mediante el análisis de componentes principales .......... 37 Capítulo 4. Discusión ...................................................................................................................................... 39 4. 1. Selección de la segunda enzima de restricción a utilizar ...............................................................39 4. 2. Análisis de los parámetros fisicoquímicos de las muestras estudiadas........................................40 4. 3. Obtención de microorganismos, DNA genómico y fragmentos amplificados del rDNA 16S41 4. 4. Análisis de los TRFs encontrados y su relación con las condiciones de la mini-columna.......42 4. 5. Análisis de la diversidad biológica en la mini-columna .................................................................46 4. 5. 1. Análisis de los índices diversidad de especies y de Shannon-Wiener ................................. 46 4. 5. 2. Comparación entre las distintas muestras obtenidas de la mini-columna ......................... 47 4. 5. 2. 1. Comparación entre las bacterias encontradas en solución.......................................... 47 4. 5. 2. 2. Comparación entre las soluciones según las bacterias encontradas........................... 49 4. 6. Efectos de los errores asociados a la metodología de T-RFLP....................................................50 4. 6. 1. Asociados a la extracción de DNA.......................................................................................... 50 4. 6. 2. Asociados a la reacción de PCR............................................................................................... 50 4. 6. 3. Asociados a la digestión enzimática ........................................................................................ 50 Capítulo 5. Conclusiones ................................................................................................................................ 52 Bibliografía ........................................................................................................................................................ 54 Anexos............................................................................................................................................................... 57 Anexo A. Partidores utilizados..................................................................................................................57 Anexo B. Soluciones utilizadas..................................................................................................................57 Anexo C. Protocolos...................................................................................................................................59 (a) Protocolo de electroforesis en gel de agarosa al X%. .................................................................. 59 (b) Protocolo de determinación de Fe+2 y fierro total. ...................................................................... 59 Anexo D. Tablas de las enzimas de restricción, su sitio de restricción y el largo de los fragmentos que se forman...............................................................................................................................................60 (a) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Acidithiobacillus caldus. (776 pb) .............................................................................................................. 60 (b) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Acidithiobacillus ferooxidans. (776 pb) ...................................................................................................... 65 (c) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Acidithiobacillus thiooxidans. (776 pb) ...................................................................................................... 70 (d) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Leptospirillum ferrooxidans. (778 pb) ........................................................................................................ 74 (e) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Sulfobacillus thermosulfidooxidans. (766 pb) .............................................................................................. 78 Anexo E. Digestión virtual ........................................................................................................................82 iv Índice de Tablas Tabla 1. Máxima concentración de iones metálicos (en [g/l]) en la cual se observa crecimiento o actividad enzimática............................................................................................................................................7 Tabla 2. Condiciones y concentraciones iniciales de los iones de cada muestra en la columna de biolixiviación..................................................................................................................................................... 13 Tabla 3. Concentraciones necesarias de los reactivos para la reacción de PCR. .................................... 15 Tabla 4. Programa de PCR utilizado............................................................................................................. 15 Tabla 5. Concentración de los reactivos en la digestión de los productos de PCR ............................... 16 Tabla 6. Tamaño en pb de los TRF obtenidos con el programa Virtual Diegest a través de la digestión con las enzimas de restricción RsaI y MspI, de forma independiente y de forma combinada (RsaI+MspI). .................................................................................................................................................... 20 Tabla 7. Valores de los parámetros estudiados para cada muestra a la que fue sometida la minicolumna. ............................................................................................................................................................ 22 Tabla 8. Abundancias relativas de los TRFs más significativos obtenidos en todas las muestras digeridas con RsaI............................................................................................................................................ 28 Tabla 9. Abundancias relativas (en porcentaje) de los TRFs más significativos obtenidos en todas las muestras para la digestión con MspI............................................................................................................. 29 Tabla 10. Bacterias presentes en las soluciones a partir del análisis de T-RFLP con RsaI y MspI de forma independiente........................................................................................................................................ 30 Tabla 11. Índice de diversidad de especies (S) de cada muestra según la enzima de restricción utilizada (RsaI o MspI). ................................................................................................................................... 32 Tabla 12. Índice de Shannon-Wiener (H) de cada muestra según la enzima de restricción utilizada (RsaI o MspI).................................................................................................................................................... 32 Tabla 13. Composición de medio MC. ......................................................................................................... 57 Tabla 14. Composición del Buffer A2X. ........................................................................................................ 57 Tabla 15. Composición del Buffer de lavado................................................................................................. 57 Tabla 16. Composición del Buffer TE............................................................................................................ 58 Tabla 17.Composición del Buffer de carga 6X.............................................................................................. 58 Tabla 18. Composición del Buffer TAE 50X. ............................................................................................... 58 Tabla 19. Composición del Buffer de almacenamiento................................................................................ 58 Tabla 20. Composición del estándar de peso molecular, 1 Kb. ................................................................ 58 Tabla 21. Composición de la solución de la RNasa.................................................................................... 58 Tabla 22. Tamaños de los TRFs obtenidos de la digestión virtual de las bacterias asociadas a procesos de biolixiviación................................................................................................................................................ 82 v Índice de Figuras Figura 1. Esquema de los distintos tipos de lixiviación de un sulfuro según Tributsh (obtenida de Ballester 2005). ....................................................................................................................................................4 Figura 2. Esquema del método de t-RFLP......................................................................................................8 Figura 3. Esquema del proceso de selección de la segunda enzima a utilizar. ........................................ 12 Figura 4. Mini columna de biolixiviación. (a) Foto lateral de la mini columna de biolixiviación; (b) foto del mineral a procesar; (c) esquema de la mini columna de biolixiviación. ................................... 13 Figura 5. Evolución del potencial redox en las muestras 4, 5, 6, 7, 8 y 9 de la mini-columna de biolixiviación operada bajo diferentes condiciones (Tabla 7). La simbología utilizada para cada muestra se encuentra en la parte inferior del gráfico. ................................................................................. 21 Figura 6. Evolución de la cantidad de cobre lixiviado (en [Kg])............................................................... 23 Figura 7. Evolución de la cantidad de ión férrico en generado (en [Kg])................................................ 23 Figura 8. Evolución de la cantidad de sulfato generado (en [g])............................................................... 24 Figura 9. Evolución de la cantidad de células generadas a partir de la muestra 2. ................................. 25 Figura 10. Gel de agarosa al 0,8% del DNA genómico de las muestras 3 y 5 . Carril 1: muestra 5 ; carril 2: muestra 5 tratada con RNasa; carril 3: muestra 3; carril 4: muestra 3 tratada con RNasa; PM: Estándar de tamaño molecular 1Kb.............................................................................................................. 26 Figura 11. Gel de 1,4% de agarosa de los productos de PCR de la muestra 4 para distintas diluciones. Carriles 1y 2: amplificado de la muestra 4 sin diluir; carriles 3 y 4: amplificado de la dilución 1:10 de la muestra 4; carriles 5 y 6: amplificado de la dilución 1:100 de la muestra 4; PM: estándar de tamaño molecular 1Kb. ................................................................................................................................................. 26 Figura 12. Variación de la abundancia relativa de A.ferrooxidans según el análisis con la enzima RsaI. ............................................................................................................................................................................ 29 Figura 13. Relación de los TRFs significativos (a) obtenidos de la digestión con RsaI, (b) obtenidos de la digestión con MspI................................................................................................................................. 33 Figura 14. Relación de las bacterias encontradas en solución a partir de la Tabla 10............................ 34 Figura 15. Dendrogramas que muestran la relación entre las soluciones a partir de las poblaciones desarrolladas en ellas (a) según la digestión con RsaI, (b) según la digestión con MspI. ...................... 35 Figura 16. Dendrograma de la relación entre las soluciones a partir de las poblaciones desarrolladas en ellas, según los datos obtenidos en la Tabla 10. ..................................................................................... 36 Figura 17. Gráfico del análisis de componentes principales para los TRF obtenidos de la digestión con RsaI. Los números corresponden a los tamaños de los TRF. ........................................................... 37 Figura 18. Gráfico del análisis de componentes principales para los TRF obtenidos de la digestión con MspI. Los números corresponden a los tamaños de los TRF........................................................... 38 Figura 19. (a) Modelo esquemático de la formación de pseudo-TRF, plegamiento parcial de la hebra simple podría o no formar pseudo-TRF. (b) Ejemplo de las estructuras secundarias formadas. (Egert y Friedrich 2003) .............................................................................................................................................. 51 vi Capítulo 1. Introducción 1. 1. Aspectos Generales La importancia económica de la producción de cobre en Chile, impulsa el óptimo aprovechamiento de los recursos minerales explotados. El cobre se presenta fundamentalmente en dos formas: como minerales oxidados y como minerales sulfurados. Los primeros son fácilmente solubles por lo que son tratados por medio de lixiviación ácida, mientras que los minerales sulfurados, principalmente calcopirita (CuFeS2), calcosina (Cu2S) y covelina (CuS) en Chile, son insolubles incluso en ácidos concentrados. Es en este contexto que la biolixiviación o lixiviación bacteriana se presenta como una opción para recuperar metales de interés a través de su solubilización bajo la acción directa o indirecta de microorganismos desde, por ejemplo, botaderos y pilas. Dependiendo de la composición del mineral sulfurado, la biolixiviación es una alternativa ventajosa a las tecnologías pirometalúrgicas, si bien es más lenta en cuanto a la obtención del mineral, ya que demanda menores inversiones de capital y menores costos de operación. Además se presenta como una tecnología más limpia que las practicadas comúnmente, debido a que no se emite SO2 al ambiente en su periodo de operación (Ballester 2005). Es así, como la búsqueda de las condiciones óptimas de crecimiento y actividad de los microorganismos envueltos en estos procesos, es decir los parámetros que estimulan o inhiben su accionar, se vuelve de vital importancia para la industria. Para lograr esto, se deben identificar las poblaciones bacterianas envueltas en el proceso en estudio y cómo éstas se ven influenciadas por las características fisicoquímicas que las rodean, lo que contribuiría a mejorar el entendimiento de los procesos de biolixiviación. Sin embargo, dado que no todos los microorganismos presentes en estos procesos serán cultivables, las técnicas de cultivo tradicionales no logran detectar la realidad del ecosistema estudiado. Por esta razón, las técnicas moleculares independientes de cultivo se presentan como la alternativa indicada para determinar los microorganismos involucrados en un proceso en un tiempo dado. En este contexto, el presente trabajo pretende identificar las poblaciones bacterianas asociadas a un ambiente de lixiviación específico, a partir de una técnica molecular, y buscar alguna relación entre éstas y las variaciones de los parámetros fisicoquímicos relevantes del sistema. 2 1. 2. Antecedentes Bibliográficos Los microorganismos presentes en los procesos de biolixiviación tienen un rol catalítico en la oxidación del ión ferroso (Fe+2) y del azufre elemental producidos durante la lixiviación química, a ión férrico (Fe+3) e ión sulfato, respectivamente (Ecuación 1 y Ecuación 2). Los iones férricos corresponden a los agentes químicos oxidantes que disolverán el sulfuro metálico (Ballester 2005; Watling 2006). Ecuación 1. Oxidación biológica del ión ferroso a ión férrico (Watling H.R., 2006). 2 Fe +2 + 2 H + + 0,5O2 bacteria → 2 Fe +3 + H 2 O Ecuación 2. Oxidación biológica de azufre a sulfato (Watling H.R., 2006). 2 S + 3O2 + 2 H 2 O bacteria → 2 H 2 SO4 Los fundamentos bioquímicos de las reacciones de lixiviación han sido ampliamente estudiados en los últimos años, habiéndose propuesto 2 mecanismos para estos procesos. El primero consiste en la llamada “lixiviación indirecta” o “mecanismo indirecto”, en la que la oxidación del sulfuro metálico se realiza a través de los iones férricos sin mediar contacto entre los microorganismos y la superficie o alguna reacción enzimática, combinada con la oxidación biológica de los iones ferrosos en solución obtenidos de la reacción anterior, como se muestra en la Ecuación 1 (Figura 1) (Watling 2006). La “lixiviación por contacto” o “mecanismo por contacto” supone que la mayoría de las células se adhieren a la superficie de los minerales sulfurados, lo que se traduce en la disolución del mineral producto de los procesos electroquímicos que tienen lugar en la interfaz entre la pared del microorganismo y la superficie del sulfuro (Figura 1) (Watling 2006). Se añade a éstos mecanismos la “lixiviación cooperativa”, que no es más que la presencia simultánea de los mecanismos anteriores, lo que podría traducirse en un suministro óptimo de energía química a partir de una superficie limitada de sulfuros favoreciendo la supervivencia de las bacterias (Figura 1) (Ballester 2005). 3 Figura 1. Esquema de los distintos tipos de lixiviación de un sulfuro según Tributsh (obtenida de Ballester 2005). 1. 2. 1. Microorganismos lixiviantes La mayor parte de los microorganismos involucrados en los procesos de biolixiviación, se caracterizan por crecer quimiolitotróficamente en ambientes ácidos, con pH menor a 3, con alta concentración de iones metálicos y por ser capaces de utilizar el ión ferroso o los compuestos de azufre reducido como fuente de energía (Rawling 1998; Watling 2006). Sin embargo también se pueden encontrar microorganismos heterotróficos cuyo trabajo consiste en eliminar del ambiente los compuestos de carbono presentes producto de la muerte celular o de desechos metabólicos de los microorganismos quimiolitotrofos (Brierley 2000). Los microorganismos asociados a estos procesos se clasifican según la temperatura a la que se desarrollan de forma óptima, encontrándose los mesófilos (temperaturas menores a 40°C), termófilos moderados (temperaturas entre los 30°C y 55°C) e hipertermófilos (temperaturas entre los 50° y 85°C) (Brierley 2000). Entre las bacterias más estudiadas y presentes en la mayoría de los procesos de biolixiviación se destacan: (Rawlings 2002) • Acidithiobacillus ferrooxidans: filogenéticamente situadas entre el nodo que une las β y γ proteobacterias, son preferentemente aeróbicas, capaces de utilizar ión ferroso o compuestos de azufre reducidos como donadores de electrones, pero también pueden crecer utilizando el ión férrico como aceptor de electrones. Han sido encontradas como 4 • • • • • el microorganismo dominante en procesos de lixiviación de sulfuros de cobre, especialmente cuando existen altas concentraciones de ferroso. Son mesófilas y el rango de pH en el que se desarrollarían estaría entre 1,8 y 2,0. Acidithiobacillus thiooxidans: al pertenecer también al género de las Acidithiobacillus, se encuentran filogenéticamente situadas entre el nodo que une las β y γ proteobacterias, sin embargo están restringidas al uso de compuestos de azufre reducido como donador de electrones. Son mesófilas, probablemente encontrándose hasta los 35°C, y son más ácido tolerantes en comparación con A. ferrooxidans, con un rango de pH entre 0,5 y 5,5. Acidithiobacillus caldus: al pertenecer también al género de las Acidithiobacillus, poseen las características filogenéticas antes mencionadas. Sólo puede oxidar compuestos de azufre reducido y crecer a pH bajos. Son consideradas termófilas moderadas, siendo su temperatura óptima de crecimiento de 45°C aproximadamente. Leptospirillum: filogenéticamente perteneciente a la división de las Nitrospira, es una bacteria quimiolitoautotrófica ácido tolerante (pH óptimo entre 1,5 y 1,8), capaz de utilizar el ión ferroso como donador de electrones. Tienen una alta afinidad por el ión ferroso y alta tolerancia frente a la presencia de ión férrico. Entre ellas se encuentran las L. ferrooxidans, L. ferriphilium y L. thermoferrooxidans, las primeras mesófilas y la última termófila moderada. Sulfobacillus: perteneciente a la familia de las Alicyclobacillaceae (Taxonomy, NCBI), se destacan Sb. thermosulfidooxidans y Sb. acidophilus. Son termófilas moderadas, capaces de crecer autotróficamente utilizando ión ferroso o compuestos de azufre reducidos como donadores de electrones. También son capaces de crecer en ausencia de oxígeno utilizando el ión férrico como aceptor de electrones y compuestos de azufre como donadores de electrones. Se ha encontrado que su crecimiento se encontraría asociado al de Acidimicrobium ferrooxidans, bacteria heterotrófica, capaz de oxidar hierro. Acidiphilium: perteneciente a las α proteobacterias (Taxonomy, NCBI), son bacterias heterótrofas que son encontradas frecuentemente creciendo en las cercanías de las A. ferrooxidans, presumiblemente alimentándose de los compuestos orgánicos producidos por éstas durante su metabolismo. Entre ellas, Acidiphilium acidophilum es la única capaz de crecer autotróficamente usando compuestos de azufre reducidos, heterotróficamente usando distintas fuentes de carbono, o mixotróficamente usando carbón orgánico o inorgánico. 5 1. 2. 2. Factores que influyen en la rapidez de la biolixiviación Existen diversos factores asociados a los procesos de biolixiviación que condicionan el crecimiento de los microorganismos en estos ambientes, y éstos varían durante estos procesos provocando variaciones en las poblaciones microbianas (Brandl 2001). Entre los parámetros fisicoquímicos importantes en el proceso estudiado se encuentran el pH, potencial redox, acidez, concentración de hierro total, de ión ferroso y de cobre, temperatura contenido de oxígeno y viabilidad, y presencia de inhibidores entre otros. La concentración de hierro total, de ión ferroso y de cobre además del potencial redox, permiten monitorear la disolución del mineral mediada por férrico y observar el comportamiento de las bacterias hierrooxidantes. La presencia de inhibidores del crecimiento o la actividad de los microorganismos es importante a la hora de utilizar este proceso a escala industrial, debido a que estos influirán en el tiempo de obtención del mineral lixiviado. En cuanto a los parámetros a tener en cuenta relacionados con los microorganismos, se tienen la diversidad microbiana, la densidad de la población, la actividad microbiana, la tolerancia a los metales y las capacidades de adaptación de los microorganismos. Todos ellos estarán directamente relacionados con la rapidez de la lixiviación del mineral. Finalmente, las propiedades del mineral a lixiviar también son determinantes en estos procesos, ya que de ellas dependerá la factibilidad de la disolución del mineral tratado. Entre éstas se encuentran, por ejemplo, el tipo de mineral, su composición, el tamaño de partícula, el área superficial y la porosidad. 6 1. 2. 3. Inhibición de la actividad enzimática de los microorganismos biolixiviantes Los microorganismos requieren de la presencia de diversos metales que juegan un rol bioquímico esencial como catalizadores, co-factores de enzimas o estabilizadores de las estructuras de las proteínas (Dopson et al. 2003). No obstante, la acumulación de estos metales podría llegar a ser tóxica para los microorganismos, afectando los procesos en los que se encuentran envueltos, por lo cual es importante conocer cómo éstos los afectan. La Tabla 1 presenta algunos ejemplos de la resistencia de bacterias acidófilas a algunos metales encontrados en procesos de biolixiviación. Tabla 1. Máxima concentración de iones metálicos (en [g/l]) en la cual se observa crecimiento o actividad enzimática. Acidiphilium cryptum Acidiphilium angustum Acidocella aminolytica Acidocella facilis Acidithiobacillus ferrooxidans Leptospirrilllum ferrooxidans Sulfobacillus thermosulfidooxidans Cu(II) 0,95a 0,32a 1,91a 0,064a 15b –50,83a 0,32a 0,38a Fe(III) 16,76a < 20 a,b 27,93a - Al(III) 5,4c 5,4c 5,4c 6b - Zn(II) 8,17a 0,52a 32,69a 6,54 a 15b -70,02a 2,8a Datos obtenidos de las siguientes referencias: (a) Dopson et al. (2003); (b) Acevedo y Gentina (2005); (c) Wakao et al. (2002). 1. 2. 4. Técnica de T-RFLP La cantidad de bacterias, y microorganismos en general, conocidos en estos procesos, es consecuencia del uso de métodos de cultivo con medios enriquecidos y de aislamiento que provocan la selección de sólo algunas de las poblaciones existentes. Esto ha ido cambiando debido al desarrollo de técnicas moleculares que involucran la extracción y procesamiento del DNA sin que se utilicen etapas intermedias de cultivo (Watling 2006). Entre estas últimas se destaca la técnica de reconocimiento de los polimorfismos de los largos de los fragmentos terminales de restricción (T-RFLP), la cual permite obtener fragmentos terminales de restricción (TRF) de tamaño característico para determinadas especies, con lo cual podrían ser identificados. La técnica de T-RFLP se divide en diversos pasos consecutivos como se presenta a continuación (Figura 2): • • Extracción del DNA directamente desde la muestra en estudio; Amplificación del rDNA 16S bacteriano mediante partidores universales, que sean específicos para la mayor cantidad posible de especies presentes en la muestra, marcados en el extremo 5’ con un fluorósforo; 7 • • • Digestión mediante una enzima de restricción para generar fragmentos terminales de restricción (TRF) de distinto largo marcados en un extremo; Separación de los fragmentos mediante electroforesis capilar de alta resolución para poder determinar el largo de cada TRF mediante la excitación de los fluorósforos, además de obtener una intensidad asociada a cada largo de TRF; Obtención de los resultados en forma de un electroferograma y/o tablas. Extracción del DNA bacteriano Amplificación a través de PCR Purificación del producto amplificado Electroferograma obtenido de la electroforesis capilar Digestión del producto amplificado con una enzima de restricción Figura 2. Esquema del método de t-RFLP.1 1. 2. 5. Comparación de poblaciones microbianas a través de análisis estadísticos Finalmente, los análisis estadísticos permiten, entre otras cosas, obtener una estimación de la diversidad asociada al ambiente en estudio o agrupar los microorganismos de acuerdo a alguna característica. En particular, se usan índices de diversidad de especies, de Shannon-Wiener o de Simpson para calcular la diversidad de las especies presentes en las muestras en estudio. Los dos últimos dan cuenta del orden asociado al ambiente estudiado. Para agrupar especies según alguna característica se utilizan dendrogramas o análisis de componentes principales, que también darán cuenta del comportamiento de los microorganismos en las distintas muestras. 1 Imágenes obtenidas de la sección Books, de la página web de la NCBI (30 Mayo 2007). 8 1. 3. Descripción del Proyecto El proyecto consiste en el estudio de la dinámica de la biodiversidad en procesos de biolixiviación, es decir, se busca establecer alguna relación entre la variación de los parámetros fisicoquímicos presentes en el ambiente en estudio y las bacterias identificadas para cada condición. Para esto, se realizará el análisis mediante T-RFLP de las soluciones obtenidas de una minicolumna de biolixiviación montada en el laboratorio, para así identificar las poblaciones bacterianas presentes, debido a que, al ser una técnica independiente de cultivos, permite obtener una aproximación de los microorganismos presentes en solución. La técnica de T-RFLP consiste en realizar la concentración de las células presentes en la solución, la extracción del DNA de las células, su digestión con enzimas de restricción y la separación de los fragmentos terminales de restricción (TRF) obtenidos a través de electroforesis capilar. Para corroborar la información obtenida con el protocolo propuesto por Morales (2006), se busca una enzima de restricción adicional que sea capaz de distinguir entre dos de las especies bacterianas usualmente encontradas en los procesos de biolixiviación (A. ferrooxidans y A. thiooxidans), y que permita comparar las informaciones obtenidas en ambos electroferogramas para corroborar la presencia de un determinado microorganismo. La información obtenida de los electroferogramas se analiza por comparación y de forma estadística para buscar alguna relación entre las distintas bacterias identificadas y los parámetros fisicoquímicos medidos. Es importante destacar que sólo se estudiarán las bacterias presentes en las soluciones de biolixiviación, debido al uso de partidores universales específicos para bacterias, determinados por Morales (2006), y que éstas serán consideradas mesófilas, dado que no se le añadió ningún tipo de calefacción a la columna. Este trabajo se centra en la necesidad de determinar las condiciones óptimas de crecimiento y actividad de los microorganismos envueltos en los procesos de biolixiviación, es decir los parámetros que estimulan o inhiben su accionar, a la hora de su evaluación económica, debido a su incidencia directa en la rapidez y capacidad de lixiviación biológica de un determinado metal. 9 1. 4. Objetivos 1. 4. 1. Objetivo General A través del presente trabajo se busca identificar las poblaciones bacterianas presentes en soluciones de biolixiviación y relacionarlas con las características fisicoquímicas más relevantes de la solución en estudio en distintos tiempos. 1. 4. 2. Objetivos específicos Entre los objetivos específicos se encuentran: • • • • Seleccionar una segunda enzima de restricción para ser utilizada en la metodología de T-RFLP; Identificar mediante el método de T-RFLP las poblaciones bacterianas presentes en soluciones de biolixiviación; Relacionar las variaciones de las poblaciones bacterianas con las características fisicoquímicas más relevantes de la solución en un tiempo dado; Realizar un análisis estadístico de las poblaciones encontradas, correspondiente a la determinación de índices de diversidad, análisis de cluster y análisis de componentes principales. 1. 5. Alcances Este proyecto pretende establecer alguna relación entre la variación de algunos parámetros fisicoquímicos presentes en ambientes de biolixiviación y las bacterias identificadas para cada condición. Para esto, se identificarán mediante la técnica de T-RFLP, las poblaciones bacterianas presentes en 8 muestras obtenidas de una solución proveniente de una mini columna de biolixiviación de sulfuros de cobre montada en el laboratorio de Biohidrometalurgia. Mediante técnicas químicas se determinaran los parámetros fisicoquímicos de las soluciones, como pH, Eh y la concentración de los iones presentes: Cu(II), Fe(II), fierro total, Al(III), Zn(II) y sulfato. Paralelamente se buscará una enzima de restricción alternativa a la propuesta por Morales (2006), que permita distinguir entre dos de las especies bacterianas usualmente encontradas en los procesos de biolixiviación, y que además permita comparar las informaciones obtenidas en ambas enzimas por separado para corroborar la presencia de un determinado microorganismo. Cabe destacar que sólo se estudiarán las bacterias presentes en las soluciones de biolixiviación, debido al uso de partidores universales específicos para bacterias, determinados por Morales (2006), y que éstas serán consideradas mesófilas, dado que no se le añadió ningún tipo de calefacción a la columna. 10 Capítulo 2. Metodología En el presente capítulo se presentan los materiales y métodos utilizados en el estudio de la dinámica de las poblaciones bacterianas, producidas al variar las condiciones de una mini-columna de biolixiviación. 2. 1. Materiales Los materiales utilizados para el estudio de la dinámica de las poblaciones bacterianas, se encuentran enumerados en la metodología según corresponda. Asimismo, la composición de las soluciones requeridas en los métodos se encuentra en el Anexo B. 2. 2. Métodos A continuación se presentan los métodos utilizados para determinar la dinámica de las poblaciones bacterianas en ambientes de biolixiviación. En primer lugar, se determinó una segunda enzima de restricción a utilizar respecto de la metodología de T-RFLP implementada por Gabriela Morales (Morales 2006) en el laboratorio de Biohidrometalurgia de la Escuela de Ingeniería, Universidad de Chile, con el fin de poder corroborar los resultados obtenidos en la electroforesis capilar con la primera enzima (RsaI). Paralelamente, se puso en marcha una mini columna de biolixiviación en el laboratorio Biohidrometalurgia de la Escuela de Ingeniería, para así obtener bacterias asociadas a un proceso de biolixiviación de cobre realizado en distintas condiciones, como Eh, pH y concentración de iones en solución. Luego se extrajo el DNA de las bacterias obtenidas en solución, con el cual se realizó el método de T-RFLP, luego de lo cual fueron analizadas para conocer las especies bacterianas presentes y realizar pruebas estadísticas. 2. 2. 1. Selección de la segunda enzima a utilizar Para comenzar desde el sitio web del Nacional Center of Biotechnology Information (NCBI) se obtuvo la secuencia del rDNA 16S de las bacterias más conocidas y comúnmente encontradas en los procesos de biolixiviación: Acidithiobacillus ferrooxidans (locus: AF329205.1), Acidithiobacillus thiooxidans (locus: AY830898.1), Acidithiobacillus caldus (locus: Z29975.1), Leptospirillum ferrooxidans (locus: AF356834.1), Sulfobacillus thermosulfidooxidans (locus: AY140233.1). Luego se realizó la alineación virtual de los partidores universales (Anexo A) a utilizar en el método de T-RFLP experimental usando el programa de libre acceso Kalign (EBI). Para esto se utilizó el partidor 341f y la secuencia complementaria del partidor reverse 1100r. Con esto se obtiene la secuencia que se amplifica teóricamente en la reacción de PCR para una bacteria dada. El siguiente paso fue obtener el mapa de restricción de cada secuencia amplificada. Esto se logró utilizando el programa de libre acceso Webcutter 2.0, el cual permite ingresar la secuencia a analizar, lineal o circular, y elegir entre mostrar los sitios de restricción de una, algunas o todas las enzimas que 11 tiene asociadas el programa. El resultado del procesamiento de los datos se obtiene en forma de tablas, en las cuales aparece un listado de las enzimas de restricción que pueden cortar la secuencia, el sitio de restricción que dicha enzima reconoce, el número de cortes posibles y la posición en la cual se lleva a cabo dicho corte. Posteriormente se compararon las 5 tablas de enzimas de restricción y sitios de corte obtenidas, buscando una enzima que permita diferenciar las bacterias en estudio a través del TRF obtenido en teoría, al igual como lo hace la RsaI propuesta por Gabriela Morales (Morales 2006), o que permita diferenciar distintos géneros. Obtención de la secuencia de la bacteria (NCBI) Alineación de los partidores universales (Kalign, EBI|) Obtención de la secuencia amplificada Análisis de los mapas de restricción Figura 3. Esquema del proceso de selección de la segunda enzima a utilizar.2 Finalmente, se obtuvieron los tamaños virtuales de los TRFs asociados a bacterias biolixiviantes a partir del programa Virtual Diegest, de MICA, con lo que se pudo probar la segunda enzima seleccionada y determinar si era la más idónea para el análisis posterior. 2. 2. 2. Obtención de muestras de bacterias desde una mini-columna de biolixiviación Para la obtención de muestras frescas, se montó una mini columna de lixiviación en el Laboratorio de Biohidrometalurgia, rellenándola con mineral seco proveniente de la mina Quebrada Blanca (Figura 4 (a) y (b)). Para su puesta en marcha, se preparó medio MC pH 1,6 (Anexo B) con 3 [g/l] de Fe+2, el que se puso a circular en la columna como muestra el esquema (Figura 4 (c)) sin añadírsele aireación. Esta primera muestra fue retirada al cabo de 21 días, debido al lento aumento del Eh, y cambiada por una muestra fresca de MC pH 1,6 (Anexo B) con 6 [g/l] de Fe+2. 2 Imágenes obtenidas de la sección Books, de la página web de la NCBI (30 Mayo 2007). 12 Aireación Bomba Solución ácida Figura 4. Mini columna de biolixiviación. (a) Foto lateral de la mini columna de biolixiviación; (b) foto del mineral a procesar; (c) esquema de la mini columna de biolixiviación. En algunas de las condiciones estudiadas, se añadió aire al sistema a través de una bomba de pecera (Tabla 2) para considerar otro parámetro involucrado en el crecimiento de las bacterias. Las siguientes soluciones fueron cambiadas al alcanzar un Eh superior a 590 mV, excepto en los casos 2 y 3 que fueron retiradas a Eh menores para considerar estos casos en el análisis (Tabla 7). Un resumen de las condiciones iniciales se presenta en la Tabla 2. En las muestras el Fe+2 se agregó como FeSO4, en las muestras 6 y 7 se agregó sulfato como K2SO4, en las soluciones 8 y 9 se agregó aluminio en forma de Al2(SO4)3 y el zinc fue agregado en la muestra 9 como ZnSO4 (reactivos pro análisis, de Merck). El pH se ajustó con H2SO4 concentrado. El Eh y pH de la columna fue medido periódicamente, y según esta medición se ajustó el pH. Tabla 2. Condiciones y concentraciones iniciales de los iones de cada muestra en la columna de biolixiviación. Características Fe(II) Sulfato Al(III) Zn(II) pH Aireación Unidades g/l g/l g/l g/l - 1 3 1,6 no 2 6 1,6 no 3 6 1,6 si 13 Número de muestra 4 5 6 7 6 6 6 6 30 30 1,6 1,6 1,6 1,3 si no si si 8 6 60 8,43 1,6 si 9 6 71,75 8,43 8 1,6 si 2. 2. 3. Extracción de DNA desde soluciones de procesos de biolixiviación Antes de llevar a cabo la extracción del DNA, midió el pH y el Eh de la solución con bacterias mediante el uso de los electrodos correspondientes. Además se realizó el recuento directo de la cantidad de células por ml presentes en la solución. Se llevó a cabo la extracción del DNA desde una solución proveniente de un proceso de biolixiviación, debiendo usarse guantes y llevar a cabo las siguientes etapas: (Morales 2006) • Filtrar el volumen de la solución a tratar (aproximadamente 1 L) con una membrana de 0,22 µm. Una vez concluida la filtración, recuperar la membrana y depositarla al interior de un tubo de 40 ml para centrífuga. El líquido filtrado se utiliza para realizar los análisis de fierro total, ión ferroso, cobre, sulfato y, cuando corresponda, de aluminio y zinc en solución. • Lavar con 20 ml de PBS 1X pH 1,2 agitando en vortex. Centrifugar a 13.000 RPM por 15 minutos. Descartar sobrenadante. • Lavar con 10 ml de buffer de lavado agitando en vortex. Centrifugar a 13.000 RPM por 15 minutos. Descartar sobrenadante. Repetir el lavado. • Resuspender en 3 ml de buffer A2X. Adicionar lisozima a una concentración final de 3 mg/ml (243 µl de lisozima de concentración inicial de 40 mg/ml) e incubar por 1 hora a 37°C. • Agregar SDS a una concentración final de 4% p/v (3,706 ml de SDS 7,5% p/v). • Agregar 1,131 ml de NaCl 5M para llegar a una concentración final de 0,7M, y luego 898 µl de CTAB 10% p/v para tener una concentración final de 1% de CTAB. Incubar a 65°C por 1 hora. • Realizar 3 ciclos de congelamiento-descongelamiento de 3 minutos cada uno, a -20°C y 70°C respectivamente. • Centrifugar a 1.000 RPM por 2 minutos. Recuperar el sobrenadante repartiéndolo en tubos eppendorf de 1,6 ml con 0,7 ml aproximadamente. • Agregar un volumen de la fase inferior de fenol-cloroformo-alcohol isoamílico (25:24:1). Vortex hasta homogeneizar. Centrifugar a 12.000 RPM durante 5 minutos. Recuperar la fase superior y traspasar a tubos eppendorf de 1,6 ml limpios, colocando 0,7 ml por tubo aproximadamente. • Agregar un volumen de cloroformo-alcohol isoamílico (24:1). Vortex hasta homogeneizar. Centrifugar a 12.000 RPM durante 5 minutos. Recuperar la fase superior y traspasar a tubos eppendorf de 1,6 ml limpios, colocando 0,6 ml por tubo aproximadamente. • Agregar un volumen de isopropanol absoluto frío y acetato de sodio 3M pH 5,2 a una concentración final de 10% v/v. Incubar en hielo por toda la noche. • Centrifugar a 14.000 RPM por 25 minutos. Descartar sobrenadante. • Lavar con 500 µl de etanol al 70% agitando suavemente. Centrifugar a 14.000 RPM por 10 minutos. Repetir el lavado con etanol. Descartar sobrenadante y secar el pellet a 37°C. • Resuspender el DNA en 50 µl de buffer TE a un solo tubo en dos tandas, cada una con 25 µl de buffer TE. Almacenar en tubos eppendorf de 0,2 ml con 5 µl aproximadamente de DNA a -20°C. Para confirmar la obtención de DNA al final de la extracción, se debió realizar una electroforesis en un gel de agarosa al 0,8%. Para comprobar la presencia de RNA en las muestras luego de la extracción, se añadió 1 µl de RNasa (Ribonucleasa A from bovine pancreas, SIGMA) cuya concentración era de 1,2 mg/ml a uno de los tubos con 5 µl de muestra de DNA, para tener una concentración final de 0,2 mg/ml de RNasa, y se dejó reaccionar a temperatura ambiente por 30 minutos. La RNasa fue diluida en la solución de RNasa mostrada en la Tabla 21, Anexo B. 14 2. 2. 4. T-RFLP El protocolo de T-RFLP usado correspondió al implementado por Gabriela Morales (Morales 2006) en el laboratorio de Biohidrometalurgia de la Escuela de Ingeniería, Universidad de Chile. 2. 2. 4. 1. Amplificación de un fragmento del rDNA 16S Se realizó la amplificación de un fragmento del DNA extraído mediante la reacción de PCR con los primers forward 341f marcado con fluorósforo FAM y reverse 1100r (Anexo A), usando dos diluciones del DNA (1:10 y 1:100) y la muestra sin diluir, en duplicado. En todos los tubos se tuvo un volumen final de 25 µl, correspondiente a la mezcla de reacción, según las condiciones que se presentan en la Tabla 3, 1 µl de la muestra de DNA a amplificar (muestra sin diluir o diluida) y agua desionizada estéril para completar el volumen de reacción. (Morales 2006) Tabla 3. Concentraciones necesarias de los reactivos para la reacción de PCR. Reactivos Partidor forward Partidor reverse Nucleótidos Buffer Taq MgCl2 Taq Polimerasa Concentración final 0,5 µM 0,5 µM 200 µM 1X 2 mM 2,5 U El programa de PCR utilizado se presenta en la Tabla 4. (Morales 2006) Tabla 4. Programa de PCR utilizado. N° de ciclos 1 30 1 Tiempo 3 min. 45 seg. 45 seg. 45 seg. 7 min. - Temperatura [ºC] 95 94 56 72 72 4 Finalmente, se realizó una electroforesis en gel de agarosa al 1,2% para visualizar la obtención de una banda de aproximadamente 780 pb. A partir de éste se eligió la dilución a purificar y digerir, la cual correspondió a la primera dilución, ordenadas de menor a mayor, en la cual se observa sólo la banda de interés en cada uno de los duplicados. 2. 2. 4. 2. Purificación y cuantificación del DNA La purificación de los productos de la reacción de PCR se llevó a cabo utilizando el kit de purificación UltraClean PCR Clean-up DNA purification kit, de MoBio Laboratories, siguiendo el protocolo descrito por el fabricante. 15 La cuantificación del DNA obtenido se realizó mediante la determinación de la absorbancia a una longitud de onda de 260 nm de una dilución 1:60 de la muestra a analizar (1 µl de muestra en 59 µl de agua desionizada estéril) (Morales G. 2006). La concentración de DNA en [ng/µl] se calculó según la siguiente fórmula: Concentración de DNA ng = Absorbancia × Factor de dilución × Factor de conversión µl donde el factor de conversión para DNA de doble hebra es de 50 [ng/µl] de DNA por unidad de absorbancia y el factor de dilución corresponde a 60, con lo que se obtiene que la concentración de DNA es igual a: Concentración de DNA ng = Absorbancia × 3000 µl 2. 2. 4. 3. Digestión de los productos de PCR y electroforesis capilar Para cada muestra, la digestión de los productos de PCR purificados se realizó de forma independiente con las enzimas RsaI (Morales G. 2006) y MspI, en un volumen final de reacción de 20 µl, según las concentraciones mostradas en la Tabla 5 y completado con agua desionizada estéril. La reacción se realizó durante 3 horas a 37ºC para asegurar la completa digestión de los fragmentos. Tabla 5. Concentración de los reactivos en la digestión de los productos de PCR Reactivos Producto de PCR purificado Enzima de restricción Buffer de la enzima de restricción Concentración final 18 [ng/µl] 15 U 1X Posteriormente los fragmentos digeridos fueron enviados a una empresa externa para realizar la electroforesis capilar. La concentración mínima de DNA a enviar recomendada es de 10 [ng/µl] y el volumen mínimo de 5 µl, debido a que este es el volumen necesario para cargar el equipo. 2. 2. 4. 4. Procesamiento de los resultados de la electroforesis capilar Los datos obtenidos del elecroferograma, en forma de tablas, debieron ser procesados para el análisis posterior. En primer lugar fue necesario redondear el tamaño de los TRF, debido a que los largos que se obtienen del electroferograma corresponden a números decimales, producto del tiempo de migración de los distintos TRFs obtenidos, y su tamaño real debe corresponder a un número entero dado por la cantidad de pb que lo componen, por lo tanto se deben redondear dichos valores al número entero más cercano. (Morales 2006) Luego se debieron eliminar los fragmentos cuyo largo fuera menor que 35 pb o mayor que 490 pb, dado que el estándar de tamaño molecular (1Kb) discrimina entre 35 y 500 pb, con mayor error en los extremos. (Morales 2006) 16 Por último, los fragmentos que diferían entre si en 2 pb fueron agrupados, debido a que el error del equipo corresponde a ± 2 pb. Esto se traduce en que la altura y el área asociado al largo de un TRF será la suma de aquellos fragmentos que pueden ser agrupados (Wang. et al. 2004; Li et al. 2007). Una vez realizado este procedimiento, se pudieron comparar los TRFs obtenidos con los largos de los TRFs que se obtuvieron virtualmente a partir del programa Virtual Diegest, de MICA, con lo cual fue posible asociar una especie bacteriana a cada fragmento. 2. 2. 5. Análisis utilizados para comparar las especies bacterianas encontradas Los análisis utilizados para comparar las especies hacterianas encontradas en las muestras estudiadas correspondieron a la determinación de la abundancia relativa de cada TRF obtenido; el cálculo de los índices de diversidad y de Shannon- Wiener; el análisis de cluster y el análisis de componentes principales, cuya determinación se muestra a continuación. 2. 2. 5. 1. Determinación de la abundancia relativa de los TRFs La abundancia relativa (Pi) de cada TRF (i) se calculó como se muestra en la siguiente ecuación (Wang et al. 2004; Jernberg et al. 2005; Li et al. 2007). Abundancia relativa i = Pi = área peak i n ∑ área peak i i Donde i corresponde al largo del TRF y n al total de TRFs obtenidos en una muestra. Se presentan los valores de la abundancia relativa en forma de porcentajes y se consideraron como significativos aquellos TRFs cuya abundancia relativa fuera mayor o igual al 1% (Jernberg et al. 2005; Li et al. 2007), para poder así eliminar o minimizar los TRFs que pudieran ser asociados a ruidos en el electroferograma. 2. 2. 5. 2. Índices de biodiversidad A continuación se presentan las ecuaciones de los índices comúnmente usados para análisis de biodiversidad. 2.2.5.2.1. Índice de diversidad de especies La diversidad de especies (S) corresponde a la cantidad de TRFs únicos, es decir diferentes, encontrados en el perfil correspondiente a cada muestra analizada (Li et al. 2007). Para su cálculo se tomaron en cuenta sólo aquellos TRF cuya abundancia relativa fuera mayor o igual al 1%. 2.2.5.2.2. Índice de Shannon-Wiener El índice de Shannon-Wiener (H) toma en cuenta la diversidad de especies y la proporción de cada una de ellas en la comunidad estudiada, y se calcula para cada condición en estudio como se muestra a continuación (Dunbar et al. 2000; Keylock 2005): 17 n H = −∑ Pi ⋅ ln( Pi ) i Donde i corresponde al largo del TRF, n al total de TRFs obtenidos en una muestra y Pi a la abundancia relativa de cada TRF Se debe destacar que el mínimo valor de este índice es 0, mientras que el máximo corresponde a ln(1/S), donde S es el índice de diversidad de especies (www.mdsg.umd.edu). 2. 2. 5. 3. Análisis de cluster El análisis de cluster consiste en elaborar dendrogramas, gráficos, que muestren la relación existente entre las diversas bacterias asociadas a un TRF o entre las soluciones a partir de una característica. En el estudio de la dinámica de las poblaciones microbianas, la característica analizada corresponde a la presencia o ausencia de un determinado TRF en las soluciones en estudio, cuya abundancia relativa fuera mayor o igual al 1%. Con esto se creó una matriz binaria por cada enzima de restricción utilizada, llamada matriz de caracteres, en la cual se colocó un 1 si el TRF se encuentra en la muestra y 0 si no (Vingron et al. 2002/2003; Meyer et al. 2004). Es importante notar que si se deseaba comparar el comportamiento de las bacterias según su aparición en las soluciones en estudio, las filas correspondían a los distintos TRFs y las columnas a las soluciones, mientras que si se deseaba comparar las distintas soluciones según las bacterias encontradas en ellas, las filas corresponderían a las soluciones y las columnas a los TRFs. A partir de la matriz de caracteres se utilizaron las herramientas estadísticas de Matlab para obtener la figura del dendrograma. En primer lugar se calculó el coeficiente de similitud de Jaccard (sij) entre los distintos TRFs, para luego calcular la distancia genética (dij), la que representa la diferencia entre los distintos fragmentos estudiados, como se muestra a continuación. d ij = 1 − s ij = 1 − nij ni + n j − nij Donde nij corresponde al número de TRFs compartidos entre dos soluciones comparadas, es decir la cantidad de 1 presentes en la matriz de caracteres, y ni y nj corresponden al número de TRFs en la solución i y la j, respectivamente (Ma y Zeng 2004). Luego se determinó la proximidad entre los TRFs a partir de las distancias calculadas utilizando algún método. En este caso se utilizó el método de los “promedios” (average) que provee Matlab, para intentar emular el método de UPGMA (Unweighted pair-group mean aritmetic method). Finalmente, el programa desplegó la figura obtenida para el dendrograma. 18 2. 2. 5. 4. Análisis de componentes principales Para realizar el análisis de componentes principales, se utilizaron las herramientas estadísticas de Matlab. Como datos de entrada para Matlab necesarios para crear el gráfico de componentes principales, se debió fabricar una matriz en la cual se encontrara la abundancia relativa de cada TRF (filas) en cada solución (columnas) (Jernberg et al. 2005). 19 Capítulo 3. Resultados 3. 1. Selección de la segunda enzima a utilizar en los análisis de T-RFLP Siguiendo la metodología expuesta para determinar la segunda enzima de restricción a utilizar (Métodos 2. 2. 1.), se obtuvieron las tablas de las enzimas de restricción y sus sitios de corte de cada fragmento del rDNA 16S amplificado virtualmente para las especies bacterianas consideradas, las que se encuentran en el Anexo D. Al comparar los mapas de restricción obtenidos para las cinco especies bacterianas consideradas, se pudo constatar que, no hay una enzima que permita diferenciar entre A. ferrooxidans y A. thiooxidans exceptuando la RsaI, como propuso Morales (2006), dado el par de partidores universales utilizados y el producto amplificado obtenido. Este resultado se ve restringido a las enzimas consideradas en el programa Webcutter 2.0, las que representan a la gran mayoría de las enzimas de restricción existentes, Basado en lo anterior, se buscó una enzima que, a pesar de no permitir diferenciar entre las distintas especies de Acidithiobacillus, permitiera distinguir otras especies, representando un aporte a la técnica de T-RFLP propuesta para el análisis de ambientes biolixiviantes. Es por esta razón que se escogió MspI como la segunda enzima de restricción a utilizar, con la que se pudo diferenciar de manera clara L. ferrooxidans de Sb. thermosulfidooxidans. A continuación se presenta una tabla resumen con los tamaños de los TRFs de las bacterias comúnmente encontradas en ambientes de biolixiviación, obtenidos con el programa Virtual Diegest, de MICA, para la digestión virtual con las enzimas escogidas, por separado y con ambas al mismo tiempo. Tabla 6. Tamaño en pb de los TRF obtenidos con el programa Virtual Diegest a través de la digestión con las enzimas de restricción RsaI y MspI, de forma independiente y de forma combinada (RsaI+MspI). Enzimas de restricción RsaI MspI RsaI + MspI 121 138 121 121 138 121 551 163 163 249 163 163 309 163 163 146 379 146 146 103 103 141 143 141 141 115 115 Bacterias biolixiviantes Acidimicrobium ferrooxidans Acidiphilium acidophilum Acidithiobacillus caldus Acidithiobacillus ferrooxidans Acidithiobacillus thiooxidans Leptospirillum ferrooxidans Leptospirillum ferriphilium Sulfobacillus acidophilus Sulfobacillus thermosulfidooxidans Para el análisis se consideraron las bacterias comúnmente encontradas en ambientes de biolixiviación y consideradas como las más importantes, y el TRF analizado corresponde a aquel que se alineó con el partidor forward. 20 3. 2. Variación de los parámetros fisicoquímicos en las muestras estudiadas Durante la utilización de la columna como sistema en estudio, se monitoreó la evolución del potencial redox de la solución para cada una de las condiciones de operación propuestas, el cual puede ser relacionado con el crecimiento de microorganismos hierrooxidantes. La Figura 5 presenta los cambios en el potencial en las muestras 4, 5, 6, 7, 8 y 9, las cuales representan diferentes condiciones posibles en ambientes de lixiviación, como concentración de iones metálicos, pH y aire añadido al sistema, que provocan tanto la inhibición de algunas especies bacterianas como la estimulación del crecimiento de otras. 650 Eh [mV] 600 550 500 450 400 350 300 0 3 6 9 12 15 Tiempo [días] Muestra 4 Muestra 5 Muestra 6 Muestra 7 Muestra 8 Muestra 9 Figura 5. Evolución del potencial redox en las muestras 4, 5, 6, 7, 8 y 9 de la mini-columna de biolixiviación operada bajo diferentes condiciones (Tabla 7). La simbología utilizada para cada muestra se encuentra en la parte inferior del gráfico. El valor de los parámetros fisicoquímicos estudiados para cada muestra, se encuentran en la Tabla 7. En las muestras el Fe+2 inicial se agregó como FeSO4, en las muestras 6 y 7 se agregó sulfato como K2SO4, en las soluciones 8 y 9 se agregó aluminio en forma de Al2(SO4)3 y el zinc fue agregado en la muestra 9 como ZnSO4. 21 Tabla 7. Valores de los parámetros estudiados para cada muestra a la que fue sometida la mini-columna. Parámetros Unidades 1 Eh final mV 443 pH inicial 1,6 pHfinal 1,77 Fe+2 inicial ppm 3000 Fe total ppm 3000 inicial Fe+2 final ppm 1421 +3 Fe final ppm 2290,3 Fe total final ppm 3711,3 Cu final ppm 2600 Cu disuelto mg 1820 -2 SO4 inicial g/l 5,2 SO4-2 final g/l 14,8 SO4-2 g/l 9,6 generado Al final ppm Zn final ppm Recuento bact/ml 4,5 * 105 directo Células cél 3,15 * 108 generadas Volumen ml 700 final Aireación no 2 425 1,6 1,82 6000 3 515 1,6 2,3 6000 4 597 1,6 1,45 6000 Muestras 5 592 1,6 1,76 6000 6000 6000 6000 6000 6000 6000 6000 6000 2349,75 3682 6031,75 560 532 10,3 15,8 79,13 5564,42 5643,55 160 156,8 10,3 13,3 5,82 6505,03 6510,85 163,5 155,3 10,3 19,46 6,99 7454,76 7461,75 181,3 166,8 10,3 14,4 10,3 7359,6 7369,9 128,8 122,4 25,3 29,31 33,64 7966,11 7999,75 146,3 139,0 25,3 32,7 39,43 6536,37 6575,8 85 80,8 55,3 59,88 72,96 6902,64 6975,6 61,3 57,0 67,1 73 5,5 3,0 9,1 4,1 4,0 7,4 4,6 5,9 - - - 26,9 0,53 27,1 0,78 45,5 0,61 7450 0,47 7530 7410 5,75 * 106 1,58 * 108 4,26 * 107 1,35 * 107 8,25 * 107 5,0 * 107 7,88 * 107 3,78 * 107 5,46 * 109 1,55 * 1011 4,05 * 1010 1,24 * 1010 7,84 * 1010 4,75 * 1010 7,49 * 1010 3,52 * 1010 950 980 950 920 950 950 950 930 no Si si no si si si si 22 6 608 1,6 1,79 6000 7 583 1,3 1,45 6000 8 614 1,6 1,64 6000 9 604 1,6 1,62 6000 La Figura 6 y la Figura 7 muestran la evolución de la cantidad de cobre lixiviado y la cantidad de ión férrico generado, respectivamente, en función del tiempo. Cobre disuelto [Kg] 3,5 3 2,5 2 1,5 1 0,5 0 0 7 14 21 28 35 42 49 56 63 70 77 84 91 98 Tiempo [días] Fe(III) generado [Kg] Figura 6. Evolución de la cantidad de cobre lixiviado (en [Kg]). 55 50 45 40 35 30 25 20 15 10 5 0 0 7 14 21 28 35 42 49 56 63 70 77 84 91 98 Tiempo [días] Figura 7. Evolución de la cantidad de ión férrico en generado (en [Kg]). La Figura 8 muestra la evolución de la cantidad de sulfato generado en función del tiempo. 23 Sulfato generado [g] 50,0 45,0 40,0 35,0 30,0 25,0 20,0 15,0 10,0 5,0 0,0 0 7 14 21 28 35 42 49 56 63 70 77 84 91 98 Tiempo [días] Figura 8. Evolución de la cantidad de sulfato generado (en [g]). Cabe destacar que en el transcurso del experimento no se midió la temperatura de la solución o la que había al interior de la columna. Al no haber colocado ningún tipo de calefacción a la columna, se considera que ésta se encontraba a temperatura ambiente, aproximadamente, por lo que se podría asociar el crecimiento de microorganismos mesófilos y moderadamente termófilos que se pudieran haber adecuado a las condiciones del medio. 24 3. 3. Obtención de microorganismos, DNA genómico y producto amplificado Se efectuó un recuento directo de la cantidad de células presentes en la solución en todas las muestras, luego de lo cual se procedió con la etapa de extracción de DNA. En la muestra 1 no se pudo obtener DNA genómico, debido a que correspondía a la condición en la cuál se tenía la menor cantidad de células, aproximadamente 4,5 * 105 cél/ml, por lo que no se siguió utilizando esta muestra para los posteriores estudios. La Figura 9 muestra el aumento de la cantidad de células generadas durante el proceso de biolixiviación ocurrido en la columna a partir de la muestra 2. Número de células en solución 1,0E+12 1,0E+11 1,0E+10 1,0E+09 0 7 14 21 28 35 42 49 56 63 70 77 84 91 98 Tiempo [días] Figura 9. Evolución de la cantidad de células generadas a partir de la muestra 2. Otro análisis realizado correspondió a intentar explicar el barrido obtenido en los geles de agarosa luego de la extracción, correspondiente a fragmentos pequeños de DNA según el estándar de tamaño 1 Kb. Para esto se incubaron dos de las muestras, las muestras 3 y 5, con RNasa según se muestra en la metodología, con lo cual se determinó que el barrido correspondía a RNA. (Figura 10). 25 PM verificar si se2 trataría de RNA 3 1 PM 4 12.216 pb 1.018 pb 506 pb Figura 10. Gel de agarosa al 0,8% del DNA genómico de las muestras 3 y 5 . Carril 1: muestra 5 ; carril 2: muestra 5 tratada con RNasa; carril 3: muestra 3; carril 4: muestra 3 tratada con RNasa; PM: Estándar de tamaño molecular 1Kb. Luego se llevó a cabo la etapa de PCR de manera exitosa para las muestras estudiadas, obteniéndose el fragmento amplificado del tamaño esperado para todas las diluciones. A través de un gel de agarosa 1,4% (por ejemplo la Figura 11) se observaron los fragmentos amplificados de cada una de las muestras y se procedió a elegir la dilución con la se harían los análisis posteriores. PM 1 2 3 4 5 6 1.018 pb ± 780 pb 506 pb Figura 11. Gel de 1,4% de agarosa de los productos de PCR de la muestra 4 para distintas diluciones. Carriles 1y 2: amplificado de la muestra 4 sin diluir; carriles 3 y 4: amplificado de la dilución 1:10 de la muestra 4; carriles 5 y 6: amplificado de la dilución 1:100 de la muestra 4; PM: estándar de tamaño molecular 1Kb. 26 Luego se realizó la etapa de purificación y la digestión enzimática con la enzima de restricción RsaI o MspI. En la etapa de purificación se estudiaron dos posibles métodos: purificar la banda del amplificado directamente desde el gel con el kit correspondiente o purificar la solución obtenida luego del PCR con un kit de purificación de productos de PCR. En ambos casos, al hacer un gel de agarosa al 1,4%, se obtuvo la banda esperada purificada, por lo que se esperaba que no hubiera diferencias en la digestión enzimática y en el electroferograma. Es importante destacar que la digestión al utilizar cada enzima fue completa, debido a que al hacer una electroforesis en gel de agarosa al 1,4% de algunas de las muestras digeridas, los fragmentos observados no correspondieron al fragmento entero amplificado con el método de PCR, sino que a fragmentos de menor tamaño. 27 3. 4. Determinación de las poblaciones bacterianas en solución y su comparación mediante diversos análisis. 3. 4. 1. Determinación de la abundancia Relativa de los TRFs Luego del tratamiento de los TRFs obtenidos en el electroferograma para cada muestra, mencionado en Métodos, 2. 2. 5. 1. , se calcularon las abundancias relativas que se muestran en las tablas a continuación. La Tabla 8 y la Tabla 9, presentan los resultados de la digestión con RsaI y MspI respectivamente. Para este análisis sólo se consideraron los TRFs cuya abundancia relativa fuera mayor al 1%, para eliminar o minimizar los errores asociados a ruidos en el electroferograma (Jernberg et al. 2005; Li et al. 2007), o aquellos que pudieran ser relacionados a alguna especie bacteriana asociada a procesos de biolixiviación. En ambas tablas, las muestras corresponden a las distintas condiciones de operación a las que fue sometida la mini-columna de biolixiviación, que se presentan en la Tabla 7. Tabla 8. Abundancias relativas de los TRFs más significativos obtenidos en todas las muestras digeridas con RsaI. Tamaño del TRF(pb) 70 77 85 94 121 126 141 146 213 238 249 267 309 2 3 4 5 6 7 8 9 4,04 2,71 1,08 0,70 1,06 0,19 66,75 12,24 - 7,03 3,47 0,95 2,15 0,39 62,88 5,0 0,57 7,90 10,13 7,67 0,82 1,31 1,44 1,67 0,68 44,57 3,33 6,54 1,33 1,34 0,36 10,83 0,99 0,42 63,72 1,92 4,65 2,38 0,27 2,34 0,30 1,08 88,30 1,61 - 2,12 3,46 0,84 82,47 1,41 5,06 0,21 0,11 1,52 1,14 80,20 2,44 0,28 0,32 0,46 0,78 84,52 2,48 - Total 88,77 82,44 87,39 86,88 93,63 95,57 85,69 88,56 Abundancia relativa (%) en cada muestra 28 Tabla 9. Abundancias relativas (en porcentaje) de los TRFs más significativos obtenidos en todas las muestras para la digestión con MspI. Tamaño del TRF(pb) 70 103 115 130 138 143 148 163 203 325 2 3 4 5 6 7 8 9 7,00 9,66 1,15 1,64 50,27 9,06 2,50 - 1,08 1,09 2,74 70,49 3,31 7,72 2,26 2,06 6,33 48,11 6,18 14,44 2,02 1,71 12,38 3,11 62,76 4,90 7,53 - 84,74 2,65 6,10 - 3,69 1,16 82,20 1,06 4,22 - 2,62 1,59 1,86 80,73 2,38 1,16 1,27 - 1,25 85,55 1,62 1,54 2,09 - Total 81,28 88,69 80,85 90,68 93,49 92,33 91,61 92,05 Abundancia relativa (%) en cada muestra Cada TRF puede ser asociado a una especie bacteriana particular presente en procesos de biolixiviación. Por ejemplo, la Figura 12 muestra la variación de la abundancia relativa de A. ferrooxidans en las muestras estudiadas, basado en los resultados obtenidos con la enzima de restricción RsaI. Por otra parte, hay TRFs que corresponden a más de una especie bacteriana. Al tener dos digestiones independientes con enzimas distintas, es posible descartar algunas bacterias que estarían presentes en el proceso usando el análisis con una sola enzima, pero que no están presentes al analizar los resultados obtenidos por la digestión con otra enzima. De esta manera, se tiene que las bacterias que con mayor probabilidad estarían formando parte de las soluciones estudiadas, corresponden a las que se encuentran en ambos análisis presentadas en la Tabla 10. Abundancia relativa [%] 7 6 5 4 3 2 1 0 2 3 4 5 6 7 8 9 Muestras Figura 12. Variación de la abundancia relativa de A.ferrooxidans según el análisis con la enzima RsaI. 29 Tabla 10. Bacterias presentes en las soluciones a partir del análisis de T-RFLP con RsaI y MspI de forma independiente. Bacterias α proteobacteria Acidimicrobium ferrooxidans Acidiphilum spp. Acidocella spp. Acidomonas methanolica Ferrimicrobium acidiphilium A. ferrooxidans A. thiooxidans Arthrobacter spp. Desulfosporosinus spp. Shewanella spp. Sb. acidophilus Sb. thermosulfidooxidans 4 si Muestras 5 6 si si 7 si 8 si 9 si - si si - si si - si si - si si si si si si si si - si si - si si - si si - si - TRF (pb) RsaI MspI 213 138 2 si 3 si 121 138 - 249 309 126 94 238 141 141 163 163 143 164 163 143 115 si si - 30 A continuación se presentan algunas características generales de las bacterias encontradas en solución: • • • • • • • • • • • Acidithiobacillus ferrooxidans: son preferentemente aeróbicas, quimiolitótrofas, capaces de oxidar Fe+2 y compuestos de azufre, en condiciones anaeróbicas son capaces de reducir Fe+3. Han sido encontradas como el microorganismo dominante en procesos de lixiviación de sulfuros de cobre, especialmente cuando existen altas concentraciones de ferroso. Son mesófilas y el rango de pH en el que se desarrollarían estaría entre 1,8 y 2,0. (Rawlings 1998, Malki et al. 2006, Watling 2006, Ghauri et al. 2007) Acidithiobacillus thiooxidans: aeróbicas, quimilitótrofas, son capaces de oxidar compuestos de azufre reducido. Son mesófilas, probablemente encontrándose hasta los 35°C, y toleran ambiente más ácidos en comparación con A. ferrooxidans, con un rango de pH entre 0,5 y 5,5. (Malki et al. 2006, Watling 2006, Ghauri et al. 2007) Acidiphilium: bacterias heterótrofas, ácido tolerantes, son capaces de oxidar compuestos de azufre de manera autotrófica y de reducir Fe+3 en condiciones aeróbicas o anaeróbicas. Entre ellas, Acidiphilium acidophilum es la única capaz de crecer autotróficamente usando compuestos de azufre reducidos, heterotróficamente usando distintas fuentes de carbono, o mixotróficamente usando carbón orgánico o inorgánico. (Küsel et al. 1999, Rawlings 2002, Malki et al. 2006, Watling 2006) Acidimicrobium ferrooxidans: termófila moderada, con un pH óptimo de 2. Es mixotrófica, hierrooxidante y reductora de Fe+3, y su presencia se encuentra asociada al crecimiento de Sulfobacillus. (Rawlings 1998, Rawlings 2002, Watling 2006, Rawlings y Jonson 2007) Acidocella: bacteria acidófila, heterotrófica, es capaz de crecer a pH inferiores a a 3,5 y tolerar altas concentraciones de aluminio. Esta bacteria ha sido encontrada asociada al crecimiento de A. ferrooxidans y se adhiere fuertemente a los precipitados de ión férrico. (Wakao et al. 2002, Ghauri et al. 2007, página web: www.learner.org/channel/courses/biology/textbook/microb/microb_14.html) Acidomonas methanolica: bacteria heterotrófica reductora de Fe+3 (Rawlings y Jonson 2007). Arthrobacter: bacteria aeróbica, heterótrofa. Es capaz de utilizar una gran variedad de compuestos orgánicos, incluyendo compuestos aromáticos, razón por la cual se encuentra presente en diferentes ambientes, especialmente en muestras de suelo. (Crocker et al. 2000) Desulfosporosinus: bacteria mesofílica, anaeróbica, reductora de sulfato. Es capaz de crecer de manera autotrófica usando hidrógeno, dióxido de carbono y sulfato. (Robertson et al. 2001, Malki et al. 2006) Ferrimicrobium acidiphilium: es mesófila, heterotrófica, capaz de oxidar Fe+2 y reducir Fe+3. Su pH óptimo se encuentra entre pH 1,7 y 1,8.( Watling 2006, Rawlings y Jonson 2007) Shewanella: anaeróbica, heterótrofa, reduce Fe(III) y Mn(IV), y oxida H2., compuestos aromáticos o cadenas cortas de ácidos orgánicos. (Arnold et al. 1990, Roden y Lovley 1993, Küsel et al. 1999, Gadd 2001) Sulfobacillus: Son termófilas moderadas, capaces de crecer autotróficamente, oxidando le ión ferroso o compuestos de azufre reducidos. También son capaces de crecer en ausencia de oxígeno reduciendo el ión férrico y oxidando compuestos de azufre reducido. Su pH óptimo se encuentra entre 1 y 2,5 Se ha encontrado que su crecimiento se encontraría asociado al de Acidimicrobium ferrooxidans. En particular, Sb.thermosulfidooxidans en más activa en la oxidación de Fe+2 y Sb.acidophilus oxida más rápidamente los compuestos de azufre. (Rawlings 2002, Watling 2006, Rawlings y Jonson 2007) 31 3. 4. 2. Comparación de los índices de biodiversidad La Tabla 11 presenta los valores obtenidos para el índice de diversidad de especies por cada enzima de restricción, el que corresponde a la cantidad de TRFs únicos, es decir diferentes, encontrados en cada muestra. Tabla 11. Índice de diversidad de especies (S) de cada muestra según la enzima de restricción utilizada (RsaI o MspI). S Muestra 2 3 4 5 6 7 8 9 RsaI 3 3 7 4 3 4 4 2 MspI 6 5 6 5 3 5 6 5 En la Tabla 12 se muestran los valores obtenidos para el índice de Shannon-Wiener (H) con cada enzima, el que toma en cuenta la diversidad de especies y la proporción de cada una de ellas en la comunidad estudiada, correspondiente a la abundancia relativa. Tabla 12. Índice de Shannon-Wiener (H) de cada muestra según la enzima de restricción utilizada (RsaI o MspI). H Muestra 2 3 4 5 6 7 8 9 RsaI 0,37 0,52 0,90 0,75 0,23 0,49 0,38 0,23 32 MspI 1,00 0,70 1,14 1,00 0,41 0,52 0,51 0,40 3. 4. 3. Comparaciones de las muestras estudiadas 3. 4. 3. 1. Comparación entre los TRF asociados a bacterias según su aparición en las muestras La Figura 13 muestra los dendrogramas obtenidos utilizando los TRFs cuya abundancia relativa sea mayor o igual al 1% y asociados a alguna bacteria de los ambientes ácidos de biolixiviación. (a) (b) Figura 13. Relación de los TRFs significativos (a) obtenidos de la digestión con RsaI, (b) obtenidos de la digestión con MspI. 33 La Figura 14 muestra la comparación entre las bacterias resultantes de la comparación de los resultados de los electroferogramas en cada muestra, a partir de los cuales se determinó que las bacterias realmente presentes en solución corresponden a las de la Tabla 10. Desulfosporosinus spp. Shewanella spp. Acidimirobium ferrooxidans Acidiphilum spp; Acidocella spp Acidomonas methanolica Ferrimicrobium acidiphilium Arthrobacter spp. A.ferrooxidans. α proteobacteria Sb.acidophilus Sb.thermosulfidooxidans A.thiooxidans Distancia Figura 14. Relación de las bacterias encontradas en solución a partir de la Tabla 10. 34 3. 4. 3. 2. Comparación entre las soluciones según la aparición de bacterias en solución La Figura 15 presenta la comparación de las muestras según las bacterias en solución, determinadas con el método de T-RFLP, para cada digestión enzimática. (a) (b) Figura 15. Dendrogramas que muestran la relación entre las soluciones a partir de las poblaciones desarrolladas en ellas (a) según la digestión con RsaI, (b) según la digestión con MspI. La Figura 16 muestra la comparación de las soluciones a partir de la aparición de las bacterias presentadas en la Tabla 10 en la solución. 35 Distancia Figura 16. Dendrograma de la relación entre las soluciones a partir de las poblaciones desarrolladas en ellas, según los datos obtenidos en la Tabla 10. 36 3. 4. 3. 3. Comparación de los TRFs mediante el análisis de componentes principales El resultado del análisis de componentes principales para las muestras estudiadas, se muestra en la Figura 17, correspondiente a los TRFs obtenidos con RsaI, y la Figura 18, correspondiente a los TRFs obtenidos con MspI. Cada punto en las figuras se relaciona con un tamaño de TRF, el cual se muestra al lado de cada uno de los puntos, y representa a las bacterias encontradas en solución. Figura 17. Gráfico del análisis de componentes principales para los TRF obtenidos de la digestión con RsaI. Los números corresponden a los tamaños de los TRF. 37 Figura 18. Gráfico del análisis de componentes principales para los TRF obtenidos de la digestión con MspI. Los números corresponden a los tamaños de los TRF. 38 Capítulo 4. Discusión 4. 1. Selección de la segunda enzima de restricción a utilizar En diversas fuentes se propone que las enzimas de restricción adecuadas para llevar a cabo la técnica de T-RFLP, son aquellas que reconocen un sitio de restricción de 4 pb. (Liu et al. 1997; Bryan et al. 2005). Esto eventualmente permitiría obtener una mayor cantidad de TRFs distintos, debido a que la probabilidad de que cada enzima encuentre más de un sitio de restricción aumentaría, respecto a aquellas que reconocen sitios de 5, 6 o 7 pb. Esta situación dependerá del grado de conservación de los sitios de restricción en los fragmentos analizados (Liu et al. 1997). Al estudiar los TRFs teóricos obtenidos con distintas enzimas de restricción, a partir de los fragmentos del rDNA 16S amplificados teóricamente, se comprobó que aquellas enzimas que reconocían sitios de restricción de un largo mayor a 4 pb producían TRFs de igual tamaño para la mayoría de las bacterias consideradas, o simplemente no cortaban la secuencia, según mostró el programa Virtual Diegest, de MICA. Por otro lado, enzimas que reconocían sitios de restricción de 4 pb también presentaron TRFs teóricos de igual tamaño, lo que sugiere que ese sitio de restricción en particular es altamente conservado en los genes del rDNA 16S de estos microorganismos. Se escogió MspI (C/CGG) como segunda enzima de restricción, teniendo que permite distinguir a L. ferrooxidans y Sb. thermosulfidooxidans de otras bacterias consideradas en procesos de biolixiviación. El uso de esta enzima permite, además, diferenciar L. ferrooxidans de L. ferriphilium y Sb. thermosulfidooxidans de Sb. Acidophilus (Tabla 6). Sin embargo, L. ferriphilium y Sb. Acidophilus no podrían ser completamente diferenciadas del resto de las bacterias, debido a que existe por lo menos una bacteria más cuyo TRF es del mismo tamaño (Tabla 22, Anexo E). De la misma forma, al digerir con RsaI y MspI no es posible distinguir entre algunas especies bacterianas, como se muestra en la Tabla 6. Debido a que su TRF es el mismo, no se sabría con certeza cual o cuales bacterias están presentes en la solución. Cabe destacar que al usar la enzima de restricción RsaI, más de algún TRF asociado a las bacterias en estudio tendría un largo mayor que 490 pb, por lo tanto están fuera del rango del estándar de tamaño de la electroforesis capilar y no serán encontrados en la solución. Al usar MspI las bacterias descartadas con la enzima anterior podrían ser encontradas en solución, complementando la información entregada por el análisis con RsaI. Se ha encontrado en la literatura que el uso dos enzimas de forma simultánea para realizar la digestión de los fragmentos amplificados, aumenta la resolución de la técnica de T-RFLP, lo que se traduce en un aumento de la cantidad de TRF únicos (Liu et al, 1997). En el caso particular de las enzimas utilizadas en este trabajo, su aplicación en conjunto no resulta un gran aporte respecto a la digestión con las dos enzimas de forma separada, debido a que no se lograría la diferenciación entre A. ferrooxiands y A. thiooxidans (Tabla 6). En estas condiciones sigue siendo mejor el uso de RsaI de forma independiente para los ambientes de biolixiviación. Cabe destacar que la base de datos a través de la cual se efectuó la determinación de los TRF en la implementación inicial de la técnica (Morales 2006) y que dio como resultado el uso de la enzima RsaI, fue actualizada en Julio del 2007. Con esta nueva información, se tiene que los TRFs de A.thiooxidans y de A.albertensis (Tabla 22, Anexo E) son del mismo tamaño al digerir con RsaI. Por lo tanto no se cumpliría la premisa inicial de que esta enzima permite distinguir sin lugar a dudas entre A.ferrooxiands y A. thiooxidans. 39 4. 2. Análisis de los parámetros fisicoquímicos de las muestras estudiadas Una primera aproximación para estudiar la dinámica de las distintas poblaciones bacterianas presentes en procesos de biolixiviación, la constituye la Figura 5. En ella se ve cómo afectan las diferentes condiciones del medio en la oxidación de ión ferroso a ión férrico. A medida que aumenta la concentración de sulfato (muestras 6 y 7), de aluminio (muestras 8 y 9) o de zinc (muestra 9), las curvas de Eh muestran una tendencia a retardar su llegada a valores cercanos a los 600 mV, siendo la muestra 9 la que tarda mayor cantidad de tiempo. Esto se explica por la toxicidad del zinc y los niveles de aluminio y sulfato presentes en la columna y la solución, que se traducen en un grado de inhibición de la oxidación de Fe+2 (Tabla 7) y del crecimiento bacteriano (Figura 9) (Dopson et al. 2003). Las muestras 4 y 5, en que la concentración de sulfato inicial es similar y a las cuales no se añadió ningún compuesto en particular al medio (Tabla 7), muestran una marcada diferencia en su potencial redox a partir del tercer día, siendo la muestra 4 la que menos tarda en llegar a 600 mV. La explicación de este fenómeno puede estar relacionada con los valores de pH y la aireación presente en la columna. En la muestra 4 se suministró aire y el pH final fue de aproximadamente 1,4, mientras que en la muestra 5 no se suministró aire y el pH final fue de alrededor de 1,7, a pesar de que las condiciones iniciales eran similares. De esta forma la muestra 4 se considera más favorable, respecto a la muestra 5, para el crecimiento de una mayor cantidad de organismos biolixiviantes (Figura 9 y Tabla 7), en particular de los hierrooxidantes, entre los que se incluyen los Acidithiobacillus y los Leptospirillum (Yahya y Johnson 2002; Rohwerder et al. 2003), lo que explicaría la mayor rapidez y mayor cantidad de oxidación de Fe+2 (Tabla 7). En el caso de las muestras 6 y 7, el párrafo anterior se contradice con lo sucedido. Si bien en ambas muestras fue suministrado aire, el pH de la muestra 6 se mantuvo entre 1,6 y 1,7, aproximadamente, mientras que en la muestra 7 se mantuvo entre 1,3 y 1,4, por lo cual se esperaría que en la muestra 7 hubiese una mayor actividad oxidativa, como se comentó anteriormente. Sin embargo en este caso se tiene mayor concentración de sulfato, respecto de las muestras 4 y 5, la que aumentó aproximadamente el doble para simular una mayor generación de ácido sulfúrico (Tabla 7), lo que junto con el bajo pH pudo haber inhibido a algún tipo de microorganismo tanto en crecimiento como en actividad oxidativa. La muestra 6 representaría la situación más favorable para el crecimiento de A. ferrooxidans y otras especies hierooxidantes, lo que podría traducirse en que este caso presente la más rápida actividad oxidativa (Figura 5). En la muestra 8 (Figura 5) se observa que durante los primeros días los valores del Eh fueron inferiores a los de todas las muestras estudiadas, exceptuando la muestra 9. No obstante, alrededor del día 8 se observa claramente como el Eh es mayor que en las muestras 5, 7 y 9. La explicación de fenómeno podría ser que durante los primeros días se tiene una etapa de ambientación de los microorganismos a los niveles de aluminio adicionados a la solución, pero luego algunos de ellos ven estimulado su crecimiento, dependiendo de la concentración de aluminio en el medio, y otros simplemente toleran la concentración de aluminio presente (Wakao et al 2002). Las muestras 2 y 3 no son presentadas en la Figura 5 debido a que no se realizó un seguimiento significativo de la variación de los valores del Eh. Por otro lado, en la muestra 1 a pesar de poseer dichos valores, estos presentan una pendiente creciente suave debido a corresponder a la etapa de ambientación de la columna, por lo tanto no se alcanzaron valores de Eh superiores a 450 mV en un tiempo considerable para el estudio y fue cambiada, no siendo significativa para este análisis. 40 La gran concentración de cobre obtenida en la solución 1 (Figura 6), en comparación con las otras muestras, se explica debido a que el mineral usado en la columna provenía de un proceso de lixiviación y se encontraba seco. Luego, al hacer circular el medio de cultivo MC, se provocó un lavado del mineral con lo que se recuperó el cobre disuelto que no se había recuperado antes de que se guardara el mineral. En el resto de las muestras la cantidad de cobre disuelto (Figura 6) está estrictamente relacionado con la actividad de las bacterias (su estimulación o su inhibición). En particular se observa que la cantidad de cobre disuelto tiende a disminuir a medida que se añaden iones de aluminio y zinc a las muestras. En la Figura 7 se observa cómo la generación de ión férrico es siempre creciente, asociado a la actividad oxidativa de las bacterias en solución y/o adheridas al mineral de la columna. De la misma forma, la cantidad de sulfato generado es creciente (Figura 8), lo que se asocia a oxidación química o a la actividad de bacterias azufre oxidantes presentes en solución y/o adheridas al mineral. 4. 3. Obtención de microorganismos, DNA genómico y fragmentos amplificados del rDNA 16S En la Figura 9 se observa el aumento de la cantidad de células generadas en el tiempo. En particular, en la muestra 9 la cantidad de células generadas disminuye posiblemente debido a los iones de zinc añadidos al sistema (Tabla 7). Por otra parte, en las muestras en las que no hubo aireación se obtuvo la menor cantidad de células generadas (muestras 2, 3 y 5, Tabla 7), sin que hubiese algún ión adicional en solución. Esto muestra la necesidad de la adición de aire para la obtención de una gran cantidad de microorganismos relacionados con estos ambientes. Al analizar las variaciones de pH en la Tabla 7, se observa que la mayor cantidad de células generadas se tiene en la muestra 3, en la que se tiene el pH más alto analizado (pH 2,3). Sin embargo en esta muestra el potencial redox alcanzó los 515 mV, por lo que los niveles de Fe+3 no habrían alcanzado a afectar a los microorganismos presentes y no se puede asociar una alta actividad de bacterias hierrooxidantes que expliquen la cantidad de células. Por otra parte, las muestras en las que se tiene el menor pH final estudiado (muestras 4 y 7 con un pH final de 1,45), no se obtuvo un cambio sustancial respecto a las otras muestras en la cantidad de células presentes. Es por esto que no se pueden asociar las variaciones de pH al comportamiento general de las muestras en cuanto a la cantidad de células generadas. Como ya se comentó en los resultados, la muestra 1 fue la que presentó la menor concentración de células al realizar el recuento directo al final de cada etapa (Tabla 7) y no se logró extraer su DNA. Debido a esto se especula que se requiere una cantidad de células totales del orden de 1010, por lo que se debería tener 1 litro de solución con una concentración de 107 células/ml para lograr una extracción de DNA exitosa. Por otra parte, la Figura 10 demuestra que se logró la extracción de DNA genómico, y que eventualmente sería necesario añadir al protocolo de extracción una etapa en la que se adicionará RNasa, para así eliminar el RNA obtenido (señal al final de los carriles 1 y 3), permitiendo ver de forma clara en la electroforesis la calidad del DNA extraído, aunque la presencia de DNA no afectó las reacciones de PCR (Figura 11). 41 En la comparación de los métodos de purificación del producto de PCR, en ambos casos se encontró un fragmento, dependiente de la enzima de restricción utilizada, que poseía la mayor abundancia relativa, superior al 40% en todas las muestras. Por otra parte, los electroferogramas resultantes de la purificación desde el gel no presentaban todos los posibles TRFs obtenidos de la digestión enzimática, porque el mayor TRF en ocasiones no superaba los 170 pb, por lo que fueron considerados como resultados no significativos. Fue por estas razones que se decidió llevar a cabo el protocolo de T-RFLP utilizando el kit de purificación de productos de PCR. 4. 4. Análisis de los TRFs encontrados y su relación con las condiciones de la minicolumna Se puede observar en la Tabla 8 y la Tabla 9 que al hacer la digestión con RsaI se tiene una mayor cantidad de TRFs respecto de los obtenidos con MspI (13 con RsaI versus 10 con MspI), lo que proviene, en el caso de RsaI, de contabilizar TRFs que si bien no son significativos (abundancia relativa menor al 1%) pueden ser asociados a alguna bacteria biolixiviante que se esperaría encontrar. Por otra parte, con ambas enzimas de restricción se obtuvo un TRF con la mayor abundancia relativa, sobre el 40%, que se encuentra presente en todas las muestras en estudio. En el caso de RsaI su tamaño es de 213 pb y en el caso de MspI es de 138 pb, los cuales pueden ser asociados a α proteobacterias genéricas. Por otra parte, el TRF de largo 138 con MspI es también asociado a Acidimicrobium ferrooxidans, Acidiphilum spp., Acidocellas spp., y Acidomonas methanolica, todas α proteobacterias heterótrofas (Taxonomy, NCBI), mientras que éstas corresponden al TRF de 121 pb para la digestión con RsaI. Con esta información no parece extraña la posibilidad de que el TRF de 213 pb obtenido con RsaI, corresponda efectivamente alguna α proteobacteria que se desarrolle en ambientes ácidos como el estudiado. En la Tabla 8 se observa la presencia de TRFs de largo 70, 77 y 85 pb al hacer la digestión con RsaI, los cuales pueden ser asociados a alguna bacteria no cultivable, según los resultados arrojados por la digestión virtual. Para estas bacterias no cultivables, su TRF con MspI correspondería a 138 pb, por lo cual no se podría descartar la posibilidad de que efectivamente se hayan desarrollado en el sistema estudiado. Sin embargo, como no se conoce cuales fueron las condiciones en las que fueron obtenidas las bacterias no cultivables que aparecen en la base de datos, es decir el ambiente en el cual se encontraban, es difícil determinar si realmente estos fragmentos corresponden a alguna bacteria o simplemente a errores asociados a la técnica, razón por la cual estos fragmentos no fueron incluidos en los siguientes análisis estadísticos. El mismo argumento es válido en el caso del TRF de largo 325 pb obtenido con la enzima MspI en la Tabla 9, el que corresponde también a alguna bacteria no cultivable. En este caso, su correspondiente TRF con RsaI es de 127 pb, el que puede ser asociado al de tamaño 126 pb. Ambos fragmentos son significativos en las mismas soluciones (en la muestra 3 y la 4) por lo que no se podría descartar del todo su presencia. Contraria a la anterior es la situación de los fragmentos de largo 267 pb con RsaI y 70 pb con MspI. El primero, si bien puede ser relacionado con una bacteria no cultivable, en la digestión virtual no hay más de una bacteria no cultivable asociada a dicho fragmento, por lo que no se confirma su existencia, pudiendo ser resultado de un error de la técnica o debido a que no ha sido detectado por otras investigaciones. El segundo, por otra parte, no corresponde a ninguna bacteria, cultivable o no, según los resultados de la digestión virtual. Tomando en cuenta el error del método de ±2 pb, no existían TRFs de tamaño 70 ±2 pb, por lo que probablemente éste correspondería a algún tipo de error. 42 El análisis de cada situación estudiada debe hacerse de manera individual, comparando la Tabla 8 con la Tabla 9, y viendo en cada caso a qué bacteria correspondería cada TRF según la Tabla 22 (Anexo E). Para este análisis se descartaron los TRFs asociados a bacterias no cultivables y los TRF que no pueden ser asociados a bacterias, como se menciona en el párrafo anterior. Como ya se explicó, los fragmentos de 213 y 138 pb corresponden a α proteobacterias. Sin embargo no se puede descartar la presencia específica de Acidimicrobium ferrooxidans, Acidiphilum spp., Acidocellas spp. o Acidomonas methanolica en las muestras 2, 3, 6 y 9, debido a que son también α proteobacterias y a que su TRF de 121 pb con RsaI estaba presente en dichas muestras, pese a no ser significativo según los criterios utilizados. En las otras condiciones (muestras 4, 5, 7 y 8), el TRF de 121 pb tuvo una abundancia relativa mayor al 1%, por lo que confirmaría la presencia de, al menos una de las bacterias heterotróficas antes mencionadas, importantes por disminuir los niveles de los compuestos orgánicos en solución producidos durante el metabolismo de los microorganismos. El fragmento de 249 pb, obtenido a través de la digestión con RsaI, corresponde a A. ferrooxidans y se encuentra de manera significativa a partir de la muestra 3. Como su TRF con MspI es de 163 pb y se encuentra presente en todas las muestras, permitiría confirmar la presencia a partir de la muestra 3 de esta bacteria considerada como la más común en procesos de biolixiviación. Asimismo, la presencia de actividad oxidativa del ión ferroso a ión férrico de manera importante a partir de la muestra 3, concuerda plenamente con la aparición de A. ferrooxidans en las soluciones. En el caso de A. thiooxidans su TRF de 309 con RsaI sólo es significativo en la muestra 4, pero aparece con una abundancia relativa menor al 1% en las muestras 3, 5, 7 y 8. Al igual que en el caso de A. ferrooxidans, su TRF es de 163 pb, por lo que sólo se podría asegurar la presencia de A. thiooxidans en la muestra 4 y podría especularse que estuviera presente en baja concentración en las otras muestras. El TRF de 126 pb con RsaI corresponde a Arthrobacter spp., cuyo tamaño es de 143 pb con MspI. Como ambos fragmentos se encuentran en el análisis en las muestras 2, 3, 4 y 8, se puede establecer que el microorganismo se encontraba presente en dichas soluciones, lo que concuerda con el resultado obtenido por Gabriela Morales (2006) que encontró esta bacteria asociada a procesos de biolixiviación. El fragmento de largo 141 pb con RsaI puede ser asociado a Sb.acidophilus o a Sb.thermosulfidooxidans, de 143 y 115 pb con MspI respectivamente. Estos tres TRFs se encuentran presentes en el análisis de la solución 4, único en el cual el TRF de 141 pb es significativo, por lo que su presencia se vería confirmada sólo en ese caso. No obstante, el fragmento de 115 pb con MspI se encuentra presente significativamente en las muestras 2, 3, 4, 5, 7 y 8 y el 141 pb de forma no significativa en las 2, 3 y 5, por lo que se puede pensar en la presencia de Sb. thermosulfidooxidans en estas soluciones pero que, debido a errores asociados a la técnica de TRFLP, no haya sido posible detectarlos al digerir RsaI. La presencia de algún Sufobacilli en la muestra 4 concuerda con Rawlings (2002), que postula que el crecimiento de Acidimicrobium ferrooxidans ocurriría en asociación con estas bacterias. Del mismo modo, Yahya y Johnson (2002) comentan que se encuentra una mayor actividad de estas bacterias al crecer en mezcla con otros microorganismos acidófilos, lo que concuerda con los casos en estudio. Por otro lado, el TRF de 94 pb con RsaI se asocia con Desulfosporosinus spp. (bacteria sulfato reductora (Malki et al. 2006)) y con Enterobacter cloacae (heterotrófica neutrófila (Brandl 2001)), ambas asociadas a ambientes de biolixiviación. El tamaño de su TRF al digerir con MspI sería de 164 y 163 43 pb respectivamente, los que se consideran iguales debido al error asociado a la electroforesis capilar. Como ambos TRF obtenidos al digerir con distintas enzimas se encuentran presentes en la muestra 2, la única que presentó el TRF de 94 pb, las dos bacterias podrían ser asociadas al sistema en estudio, pero debido a que el pH es ácido, se descarta la presencia de Enterobacter cloacae dado que su desarrollo es poco probable. El TRF de largo 238 pb con RsaI en las muestras 4, 5 y 7 correspondería, según la digestión virtual, a Shewanella spp., bacteria que reduce el ión férrico (Küsel et al. 1999), o a Pseudoalteromonas spp., bacteria acuática. Como ambas bacterias serían asociadas a un TRF de 163 pb con MspI, el cual se encontró presente en las mismas muestras, se considera que efectivamente Shewanella spp. estaría presente en el estudio, descartando a las Pseudoalteromonas spp. por no pertenecer a un ambiente ácido. Es importante notar el caso de los TRF de 103, 130 y 148 pb obtenidos en la digestión con MspI. El primero es asociado a L. ferriphilium y cuyo TRF con RsaI correspondería a 146 pb. Al no encontrarse el fragmento de 146 pb de manera significativa en alguna de las muestras, sólo estaría presente en la muestra 4 con abundancia relativa menor al 1%, se supondría que no había L. ferriphilium en la solución. Confirmando lo anterior, tampoco se encontraron presentes TRFs que puedan ser asociados a la familia de las Nitrospira, a la que pertenecen los Lesptospirilli (Rawlings 2002). Además, se realizó una reacción de PCR con partidores específicos para L. ferrooxidans, con lo que no se encontró presencia de esta bacteria, reafirmando la suposición anterior. Pese a esto, el hecho de que se pudiese encontrar L ferriphilium en las muestras estudiadas concuerda con algunos trabajos que afirman que en realidad es esta bacteria y no L.ferrooxidans la que comúnmente se encuentra en los procesos de biolixiviación (Rawlings 2002). El TRF de 130 pb obtenido con MspI podría corresponder a Chromobacterium violaceum, Stenotrophomonas maltophila o Psycrobacter glanícola, según los resultados de la digestión virtual, y su fragmento al digerir con RsaI sería de 146 pb, en los dos primeros casos, y de 551 pb. Todas estas bacterias son asociadas a procesos de biolixiviación según Brandl (2001), sin embargo no corresponden a las bacterias más comunes. Las tres bacterias son heterotróficas, y en el caso de Chromobacterium violaceum el principal agente lixiviante correspondería a cianuro (Brandl 2001), por lo cual no correspondería encontrarla en el ambiente estudiado. Asimismo, el TRF de 146 pb no fue significativo en el estudio y el de 551 pb no puede ser detectado dado el rango del electroferograma, así que no se podría asegurar la presencia de alguna de estas bacterias. El TRF de 148 pb obtenido de la digestión con MspI podría ser asociado a Alicyclobacillus disulfidooxidans, mientras que el de 164 pb correspondería a Alicyclobacillus spp. Ambas bacterias tendrían asociado un TRF de 146 pb al digerir con RsaI, por lo que, al igual que en el caso anterior, no se puede asegurar la presencia de estas bacterias en el estudio. A lo largo del análisis se observa que las poblaciones bacterianas no se mantienen de una condición a otra, en los casos en que las soluciones son prácticamente iguales (soluciones 2 y 3 con cada enzima). Además, aparecen como significativas bacterias que no son encontradas frecuentemente en los ambientes de biolixiviación (Desulfosporosinus spp. y Shewanella spp.). Asimismo, al tomar en cuenta el análisis de los TRFs obtenidos con cada enzima en forma separada, no se encuentra una clara tendencia en la inhibición de las bacterias entre las distintas soluciones. Esto concuerda con el hecho de que las comunidades bacterianas estudiadas a escala de laboratorio tienden a presentar las características de sistemas caóticos, atribuibles a predación, competencia por los sustratos y nuevas presiones selectivas impuestas por los cambios de los parámetros fisicoquímicos de los sistemas estudiados (Saikaly et al. 2005). 44 Luego de este razonamiento se puede observar cómo el análisis a partir de cada enzima en forma independiente, puede inducir a suponer la presencia de una u otra bacteria, lo que podría no corresponder con la realidad. Es por esta razón que se considera más válida la información otorgada al comparar la presencia de los TRFs al digerir con cada enzima por separado (Tabla 10). Esta restricción permitiría disminuir los efectos caóticos del análisis de las digestiones independientes, pero a la vez se debe tener en cuenta que el sistema quedaría virtualmente subestimado. Se debe considerar además que este análisis por comparación se encuentra sujeto a los errores asociados a la técnica de T-RFLP, mencionados más adelante, los cuales influirían directamente en el tamaño, la presencia de los TRFs y en su abundancia relativa. La más alta velocidad de oxidación de ión ferroso que se observa en la muestra 4 (Tabla 7 y Figura 5), se ajusta al hecho de que sea en esta condición en la que se encuentre una mayor diversidad de microorganismos de manera significativa. En todos los casos estudiados es aceptable encontrar organismos heterótrofos asociados a estos ambientes, como Acidiphilum y Ferrimicrobium acidiphilium, además de los hierrooxidantes, sulfato reductores o azufre oxidantes, debido a que se encargarían de eliminar los compuestos orgánicos que podrían llegar a ser tóxicos para las otras bacterias (Brierley 2000). La concentración de cobre sólo parece presentar valores inhibitorios en las muestras 1 y 2 para algunos de los microorganismos encontrados, como Sb. thermosulfidooxidans y Acidiphilium angustum (Tabla 1), ninguno de los cuales se encuentra en la muestra 2, si se trabaja con los TRFs encontrados con ambas enzimas (Tabla 10). En la muestra 7, la posible presencia de Acidocella spp. y Acidiphilum spp. se explicaría por la acción estimuladora que podrían provocar en ellas ciertas concentraciones de aluminio agregadas en forma de sulfato de aluminio (Wakao et al. 2002). Por otro lado, no se observa la presencia de especies de Leptospirillum a pesar de que algunas soluciones presentaron las condiciones óptimas para su crecimiento: aire y pH óptimo (entre 1.0 y 1.7). Esta situación podría deberse, en la muestra 2 y 3, al bajo Eh al cual fue recolectada la solución; a que éstas se encontraran adheridas al mineral de la columna en mayor proporción que el resto de los microorganismos (Ghauri et al. 2007) o a que precipitaran junto con el hidróxido férrico, dependiente de los valores de pH en la solución, como sucedería con A.ferrooxidans. En la muestra 9, la concentración de zinc es inhibitoria para Sb. thermosulfidooxidans y Acidiphiliun angustum, según la Tabla 1, lo que se refleja en las bacterias encontradas en esta solución: sólo A.ferrooxidans y alguna α proteobacteria. Como Acidiphilium cryptum y Acidocella aminolytica corresponden a α proteobacterias y no serían inhibidas, como muestra la Tabla 1, probablemente podrían ser éstas las que se encuentren en la solución. Además, en Rawlings (2002) se comenta como las distintas Acidiphilium (bacterias heterótrofas) crecen frecuentemente cercanas a A. ferrooxidans, lo que explicaría su posible presencia en las muestras y en particular en la solución 9. La constante generación de sulfato hace suponer la presencia de bacterias azufre oxidantes o la oxidación química del azufre por medio de Fe+3. Las bacterias contribuirían a la generación de ácido sulfúrico y la mantención de pH bajos. Esto se ajustaría a la aparición de A.thiooxidans, A.ferrooxidans 45 y Sb.thermosulfidooxidans o Sb. acidophilus en la muestra 4, la posible presencia de A.thiooxidans en las muestras 3, 5, 7 y 8, la posible presencia de alguna de las Sulfobacillus en las muestras 2, 3 y 5 (con abundancia relativa menor al 1%) y a la presencia de A.ferrooxidans en todas las muestras excepto la 2. Los bajos niveles de A.thiooxidans y el hecho de no haber detectado otras bacterias azufre oxidantes en solución, pudo deberse a que estas bacterias se encontrasen adheridas al mineral y su concentración en solución no permitiera detectarlas. La Figura 12 presenta la variación de la abundancia relativa de A.ferrooxidans entre las distintas muestras. A partir de ella no es posible establecer una relación entre la concentración de sulfato en solución y la aparición de esta bacteria en las muestras. Sin embargo, se destaca la disminución de su abundancia relativa en las muestras 8 y 9, las que corresponden a la mayor concentración de sulfato y a la presencia de iones de aluminio y zinc, que en conjunto pudieron haber afectado la cantidad de bacterias presentes. Al analizar la Tabla 10 se observa que los parámetros de la muestra 4 (pH, Eh, iones y aireación) favorecen la diversidad bacteriana, al encontrarse mayor cantidad de bacterias distintas en solución. 4. 5. Análisis de la diversidad biológica en la mini-columna 4. 5. 1. Análisis de los índices diversidad de especies y de Shannon-Wiener Al comparar los índices de diversidad de especies obtenidos (Tabla 11), se observa que en la muestra 4, que parece ser una de las que favorece el crecimiento de microorganismos asociados a estos ambientes ácidos, se presenta la mayor cantidad de TRFs únicos y asociados a alguna especie bacteriana para ambas enzimas (7 para RsaI y 6 para MspI). No obstante, se debe destacar que en el caso de la digestión con MspI, las muestras 2 y 8 tienen el mismo número de TRFs únicos que la muestra 4, es decir la máxima cantidad. Por otra parte, al hacer el mismo ejercicio con la muestra 9 en la que hay mayores niveles de inhibición, en la digestión con RsaI se tiene la menor cantidad de TRFs únicos respecto a las otras condiciones estudiadas (2 TRFs), no así en el caso de la MspI (5 TRFs), cuya mínima cantidad de TRFs únicos se obtiene en la muestra 6 (3 TRFs). Pese a esto, no se logra observar en ninguno de los casos una correspondencia entre el aumento de las condiciones inhibitorias y la disminución del índice de diversidad de especies, es decir de los TRF únicos encontrados. El índice de Shannon-Wiener (Tabla 12) muestra, al igual que el índice de diversidad de especies, que la mayor diversidad, es decir el mayor valor del índice, se encuentra en la muestra 4 para la digestión con cada enzima, asociado a las mejores condiciones de crecimiento. En el caso de los resultados obtenidos por la digestión de los fragmentos amplificados del rDNA 16S con RsaI y con MspI, entre las muestras 4 y 5 y 7, 8 y 9 el índice de Shannon-Wiener disminuye su valor al igual que aumentan las condiciones no favorables, solución 5 sin aireación, o inhibitorias en la solución. Al analizar los índices de Shannon-Wiener de las muestras 6 y 9 se observa que su valor es prácticamente igual con ambas enzimas, pero esto no es concluyente debido a que la muestra 9 es aquella que presenta niveles de zinc inhibitorios y la muestra 6 teóricamente no debiera presentar inhibiciones determinantes. 46 Las diferencias arrojadas por los índices de diversidad de especies y de Shannon-Wiener en el análisis provienen directamente de la manera en que se calculan. El primero es un reflejo directo del número de TRFs diferentes asociables a alguna bacteria, mientras que el índice de Shannon-Wiener utiliza el valor de la abundancia relativa para su cálculo. Como la cantidad de TRFs y la abundancia relativa están sujetas a etapas anteriores para su determinación, tendrán asociados los errores de arrastre que se puedan haber generado durante el T-RFLP y el tratamiento de los datos obtenidos del electroferograma. 4. 5. 2. Comparación entre las distintas muestras obtenidas de la mini-columna 4. 5. 2. 1. Comparación entre las bacterias encontradas en solución Los dendrogramas de la Figura 13 y la Figura 14 presentan básicamente cómo se agrupan las bacterias asociadas a cada TRF basados en su presencia en las muestras analizadas. El dendrograma asociado a los TRFs encontrados al hacer la digestión con RsaI (Figura 13 (a)), muestra como los TRFs de 309 y 141 pb, correspondientes a A.thiooxidans o A.albertensis y a Sb.acidophilus o Sb.thermosulfidooxidans respectivamente, se encuentran presentes en las mismas muestras (muestra 4), dada la forma en que fueron obtenidos los dendrogramas. En el caso del dendrograma elaborado a partir de la digestión con MspI (Figura 13 (b)), los fragmentos que presentan mayor cercanía y que se encuentran presentes en iguales condiciones, tienen un tamaño de 138, 143 y 163 pb, y estarían relacionados con los otros fragmentos. Esto concuerda con lo mostrado en el dendrograma a partir de RsaI (Figura 13 (a)), en donde los TRF de 249 y 213 pb, si bien corresponden al segundo par de fragmentos más cercanos en cuanto a su aparición en el análisis de las soluciones, también estarían relacionados con todos los otros fragmentos. Este fenómeno proviene del hecho que las bacterias asociadas a estos TRFs, A. ferrooxidans en el caso del TRF de 249 pb, algún Acidithiobacillus para el de 163 pb, y α proteobacterias en el caso de los fragmentos de 213 y 138 pb, se encuentran presentes en todas o casi todas las muestras estudiadas, por lo que se deben estar relacionados con los microorganismos que van apareciendo en las otras situaciones estudiadas. El TRF de 126 pb para RsaI sería el siguiente en cercanía a los TRF de 249 y 213 pb, coincidiendo con la cercanía del fragmento de 143 pb mostrado en el dendrograma elaborado a partir de los resultados obtenidos con la enzima MspI, dado que ambos representan al Arthrobacter spp.. Sin embargo, esta situación se contradice con el haber encontrado el fragmento de 141 pb alejado del primer grupo (249 y 213 pb), ya que el TRF de 143 también podría corresponder a Sb.acidophilus. Se destaca también el hecho de que el TRF de 121 pb para el análisis con RsaI se encuentre alejado del fragmento de 213 pb (Figura 13 (a)), los que podrían estar relacionados por pertenecer ambos a α proteobacterias. En ambas figuras se muestra como los fragmentos de 94 pb obtenido al digerir con RsaI y de 203 pb al digerir con MspI, se encuentran en la rama más alejada de los microorganismos comúnmente encontrados en biolixiviación, correspondientes a Desulfosporosinus spp. y Thermotrix thiopara respectivamente. En la Figura 14 se presenta el dendrograma resultante del análisis de las digestiones independientes con cada enzima de restricción, el cual es similar al obtenido de la digestión con RsaI. En ella se observa que las bacterias Sulfobacillus y A.thiooxidans se encontrarían estrechamente 47 relacionadas, como ya se ha comentado, debido a que ambas se encuentran presentes sólo en la muestra 4. Esto se reafirma al notar que ambas corresponden a bacterias azufre oxidantes, lo que daría cuenta de la mayor cantidad de sulfato generado. En el caso de A.ferrooxidans y α proteobacterias, la gran relación existente entre ellas (Figura 14), se explica debido a que sus respectivos fragmentos se encuentran presentes en la mayor parte de las muestras analizadas. Estas bacterias también se encontrarían más relacionadas que las otras a Arthrobacter spp. Estos hechos coinciden una vez más con la necesidad de tener bacterias heterótrofas en solución acompañando a alguna bacteria quimiolitótrofa, con el fin de eliminar los compuestos orgánicos presentes en solución. La explicación anterior también se ajusta a la relación entre la aparición de Shewanella spp. y la presencia de Acidimicrobium ferrooxidans, Acidiphilum spp.; Acidocella spp., Acidomonas methanolica o Ferrimicrobium acidiphilium. Esto se debe a que todas serían heterótrofas y algunas de ellas son capaces de reducir Fe+3, por lo que podrían crecer acompañando a las A.ferrooxidans. La bacteria Desulfosporosinus spp. sería aquella menos relacionada al resto de las bacterias encontradas en solución. Lo anterior podría deberse a que es la única bacteria anaeróbica estricta sulfato reductora encontrada en el análisis, detectada en la muestra 2, en la que no hubo aireación y el potencial redox indicaría que los niveles de Fe+3 en solución no llegarían a ser tóxicos. En el análisis de componentes principales se observa que en el caso de la digestión con RsaI, Figura 17, al igual que en el dendrograma, los fragmentos de 141 y 309 pb se encuentran juntos, debido a su presencia sólo en la muestra 4 y que sus abundancias relativas son similares, 1,67% y 1,33% respectivamente. Además el fragmento de 94 pb, que se encuentra cercano a los antes mencionados, podría ser asociado al mismo grupo, lo que no concuerda con los parámetros de las soluciones en que cada uno fue encontrado ni con el resultado del dendrograma. Contraria a esta situación es la de los TRF de 213 y 121 pb, los que se encuentran comparativamente alejados del resto de los puntos observados, por lo que no podrían ser agrupados con ninguno de ellos. Al igual que lo ocurrido en el dendrograma correspondiente, se podría haber esperado que estos dos TRFs se hubiesen encontrado relacionados, ya que ambos son asociados filogenéticamente a α proteobacterias, uno de forma específica (213 pb) y el otro a bacterias de la misma familia (121 pb). En la Figura 18, correspondiente al análisis de componentes principales de los TRF obtenidos de la digestión con MspI, se observa, de manera similar a la de la Figura 17, que dos de los fragmentos se encuentran relativamente alejados del resto, los TRFs de 138 y 163 pb. En particular, no se hubiese esperado que el fragmento de 138 pb no formara un grupo unido al TRF de 143 pb, debido a que se encontraron presentes bajo las mismas condiciones. Como la abundancia relativa de los fragmentos de 213 pb con RsaI y 138 de la digestión con MspI fue siempre mayor al 40%, mientras que la de los otros fue menor que 15% en la mayoría de los casos, se entiende que en el análisis de componentes principales éstos TRFs no se encuentren agrupados a los otros fragmentos. Por otra parte, en la Figura 18 se observa que los puntos no formarían algún grupo definido en comparación con la figura obtenida en el caso de la digestión con RsaI. 48 En ambos análisis de componentes principales, la primera componente principal, correspondiente al eje de las absisas, explica casi por completo la variabilidad del sistema en estudio (sobre un 98%). Según este análisis, en ambos casos todos los TRFs, exceptuando el de 138 y 213 pb cuando corresponda, podrían ser asociados a un mismo grupo, pero esto no permitiría observar cómo las variaciones de las condiciones en la columna afectan en las poblaciones encontradas en solución. Así, se puede establecer que no se observa una relación entre la agrupación efectuada a través de los dendrogramas y del análisis de componentes principales, debido principalmente a que los dendrogramas toman en cuenta la aparición de los microorganismos en cada solución, mientras que el análisis de componentes principales toma en cuenta relaciones a partir de la abundancia relativa de los TRF en cada solución. Es por esta razón que los dendrogramas pueden ser considerados como más representativos en el estudio de la variación de las poblaciones bacterianas. 4. 5. 2. 2. Comparación entre las soluciones según las bacterias encontradas La Figura 15, muestra el resultado del análisis de cluster al observar la presencia de cada TRF en las soluciones estudiadas. En la Figura 15 (a) se observa que las soluciones 5 y 7, y las 3 y 6, presentan las mismas bacterias en solución al hacer la digestión con RsaI. En el caso del pH ningún par de soluciones tiene igual pH final, por lo que se podría pensar que esto no provoca grandes cambios en las poblaciones presentes. Por otra parte, el Eh final en las muestras 5 y 7 fue similar (592 y 583 mV respectivamente), lo que no se cumple en las muestras 3 y 6, por lo que no se puede llegar a alguna relación. Al analizar la Figura 15 (b) se tiene una estrecha relación entre las muestras 3, 5 y 7, que, como se explicó anteriormente, no permiten llegar a alguna relación concluyente. En esta figura y en la Figura 15 (a), se observa que las soluciones 8 y 9 se encontrarían relacionadas, lo que podría explicarse dado que ambas corresponden a las soluciones con mayores concentraciones de iones metálicos que pudieron haber afectado el desarrollo bacteriano. Al comparar ambos dendrogramas en forma general, se observa que no se mantienen las relaciones entre las distintas soluciones al analizar los resultados obtenidos de las distintas digestiones enzimáticas. Al analizar el dendrograma de la Figura 16, obtenido del análisis de las bacterias presentes en cada digestión, se obtiene prácticamente el dendrograma resultante de la digestión con la enzima RsaI. De esta manera no se puede establecer una relación específica entre las soluciones estudiadas, y se podría pensar que los distintos parámetros no influirían en la obtención de las mismas poblaciones bacterianas en una solución, lo que concuerda con la posibilidad de encontrar comportamientos caóticos en los sistemas de laboratorio. (Saikaly et al. 2005) 49 4. 6. Efectos de los errores asociados a la metodología de T-RFLP 4. 6. 1. Asociados a la extracción de DNA En primer lugar, la extracción del DNA depende de la habilidad del experimentador, y es inevitable que en el transcurso de ésta no se pierda DNA. Por esto, si bien no se puede asegurar que el DNA obtenido corresponda al de todos los microorganismos presentes en la solución, éste debería pertenecer al de las bacterias más representativas del ambiente estudiado. 4. 6. 2. Asociados a la reacción de PCR Por otra parte, Egert y Friedrich (2003) postulan que durante la reacción de PCR se podrían producir pseudo-TRF, en muestras de cultivos puros y de muestras extraídas directamente del ambiente, debido a la posibilidad de que en algunos fragmentos amplificados el sitio de restricción deseado se encuentre localizado en un sector que se encuentre como hebra simple. Como las enzimas de restricción, al ser endonucleasas, no cortan hebras simples, éstas reconocerían el siguiente sitio de restricción de doble hebra en la secuencia, el cual podría haberse formado si la hebra se pliega sobre si misma en alguna sección, como se muestra en la Figura 19. Este hecho podría incrementar virtualmente la abundancia relativa de algún fragmento que no se relaciona a alguna bacteria, según los datos obtenidos “in sílico”. Además, el reconocimiento de estos pseudo-TRF dependerá de la enzima de restricción utilizada, ya que se encuentra influido directamente por la posición en la cual se encuentra el sitio de restricción. 4. 6. 3. Asociados a la digestión enzimática En la etapa de digestión podría ocurrir que no hubiera digestión completa, independientemente de la formación de pseudo-TRFs (Egert y Friedrich 2003; Li et al. 2007). La electroforesis de agarosa al 1,4% realizada, en la cual se observó que los fragmentos digeridos fueron menores al tamaño del fragmento amplificado, no permite descartar este hecho. Cabe la posibilidad de que aún hubiese habido digestión parcial del amplificado, es decir que el DNA no hubiera sido cortado en todos los sitios posibles para dar origen a los TRFs más pequeños y esperados teóricamente en el análisis obtenido de la digestión virtual, de hasta 300 pb aproximadamente. 50 (a) (b) Figura 19. (a) Modelo esquemático de la formación de pseudo-TRF, plegamiento parcial de la hebra simple podría o no formar pseudo-TRF. (b) Ejemplo de las estructuras secundarias formadas. (Egert y Friedrich 2003) Por último, en la etapa de electroforesis capilar, el equipo tiene asociado un error, derivado de los posibles ruidos en el momento de la lectura, y ésta además puede depender de los compuestos presentes en la muestra, derivados de la purificación y la digestión del DNA y que podrían eventualmente influir en los resultados del análisis. 51 Capítulo 5. Conclusiones En el trabajo presentado, se utilizó la técnica de T-RFLP para identificar las poblaciones bacterianas presentes en soluciones de biolixiviación y observar si los cambios de los parámetros fisicoquímicos de las soluciones influyen la diversidad bacteriana. A partir del análisis de las tablas de enzimas de restricción y los sitios de corte para cinco de los microorganismos característicos en procesos de biolixiviación, fue posible determinar que, aparte de RsaI, no hay otra enzima de restricción que logre distinguir entre A. ferrooxidans y A. thiooxidans. De esta manera se seleccionó como segunda enzima de restricción a utilizar la enzima MspI, la cual permitiría distinguir entre L. ferrooxidans y Sb. thermosulfidooxidans. En forma paralela, se logró obtener poblaciones microbianas creciendo en una mini-columna de biolixiviación, la cual fue montada en el Laboratorio de Biohidrometalurgia de la Universidad de Chile, con lo que se aseguró la obtención de muestras íntegras de DNA y poder variar las condiciones de crecimiento. En el análisis de los TRFs resultantes de la digestión enzimática con RsaI y MspI en forma separada, se encontraron algunas diferencias a la hora de definir las bacterias presentes en forma significativa, es decir con una abundancia relativa mayor o igual al 1%. Esto pudo deberse a comportamientos caóticos del sistema o a errores en el método de T-RFLP. A partir de lo anterior, se concluyó que es válido considerar como bacterias presentes en la solución, aquellas cuyos TRFs se encuentren presentes en la muestra analizada al digerir con ambas enzimas por separado. Sin embargo, no se puede descartar completamente la presencia de las bacterias cuyo TRF no se encuentre al digerir con una de las enzimas o que tengan una abundancia relativa menor que el 1%, debido a que los errores asociados al método de T-RFLP pueden derivar en una sub o sobreestimación de la cantidad, de la presencia o de la abundancia relativa de cada TRF. La cantidad de ión férrico y de sulfato generado presente en la solución mostró ser creciente en el tiempo, lo que concuerda con la presencia de bacterias hierrooxidantes, como A.ferrooxidans, y con la posibilidad de oxidación química del azufre o de la presencia de bacterias azufre oxidantes, como A.thiooxidans y Sb.thermosulfidooxidans. Al aumentar la concentración de Al(III) y Zn(II) en solución, se observó la disminución de la abundancia relativa de A.ferrooxidans y una disminución de la diversidad bacteriana. En la muestra 8, donde se agregó Al2(SO4)3, se encontraron los TRFs de bacterias que verían estimulado su crecimiento en presencia de Al(III), como Acidocella spp. y Acidiphilum spp. Las variaciones de pH no mostraron ser determinantes en la cantidad de células generadas en solución. Asimismo, el aumento de la concentración de sulfato en las muestras 6 y 7, no muestra una tendencia particular al analizar la abundancia relativa de A.ferrooxidans. Por otro lado, el aire añadido al sistema favoreció la cantidad de microorganismos generados. Los parámetros fisicoquímicos de la muestra 4 mostraron ser los más favorables para la diversidad bacteriana: Eh de 597 mV, pH inicial de 1,6, pH final de 1,4, concentración inicial de FeSO4 de 6 g/l y aire. 52 Se destaca el hecho de que no se detectara ninguna especie de Leptospirillum, bacteria frecuentemente encontrada asociada a procesos de biolixiviación, al digerir con la enzima RsaI, por lo que se supone que no estaría presente en las muestras estudiadas. Esto podría deberse a que estas bacterias se encontrasen adheridas al mineral o a que hubiesen precipitado junto con los hidróxidos de hierro, por lo tanto su presencia en solución no sería detectable. Mediante el análisis del índice de diversidad de especies no se logra observar, en ninguno de los casos, una correspondencia entre el aumento de las condiciones inhibitorias y la disminución del índice, es decir de los TRFs únicos encontrados. En el caso del índice de Shannon-Wiener, se observa una cierta tendencia a disminuir en ciertas soluciones a medida que aumenta la cantidad de iones en solución. Sin embargo, como estos índices dependen de la presencia y la abundancia relativa de los TRFs, se verán afectados por los errores asociados al T-RFLP. A partir de los TRFs con presencia significativa en las muestras, se elaboraron dendrogramas para cada enzima de restricción utilizada. Se pudo observar cierta concordancia entre los dos dendrogramas. Los TRFs asociados a A. ferrooxidans y a α proteobacterias fueron agrupados juntos, y luego estaría asociado a éstos el TRF correspondiente a Arthrobacter spp. para ambos casos. Las dos últimas corresponderían a las bacterias heterótrofas que eliminarían los compuestos orgánicos producidos por otros microorganismos. Por otro lado, A.thiooxidans y las Sb.thermosulfidooxidans y Sb.acidophillus fueron agrupadas juntas, correspondiendo a bacterias azufre oxidantes presentes sólo en la muestra 4. La agrupación de Shewanella spp. y de Acidimicrobium ferrooxidans, Acidiphilum spp., Acidocella spp., Acidomonas methanolica o Ferrimicrobium acidiphilium, se debería a que todas son heterótrofas y algunas de ellas son capaces de reducir Fe+3, por lo que podrían crecer acompañando a las A.ferrooxidans. Las agrupaciones resultantes al analizar las bacterias presentes en cada solución, no permiten ser concluyentes a la hora de establecer una relación entre los parámetros fisicoquímicos estudiados, lo que concuerda con la posibilidad de encontrar comportamientos caóticos en los sistemas de laboratorio. Finalmente, el análisis de componentes principales mostró no ser concluyente agrupar los TRFs asociados a bacterias biolixiviantes según su presencia frente a parámetros fisicoquímicos. Esto se debe principalmente a que su elaboración toma abundancias relativas de los fragmentos considerados, los cuales presentan valores sobre el 40% y el resto menor al 13% en todos los casos. 53 a la hora de determinados en cuenta las dispares, uno Bibliografía Acevedo F. y Gentina J.C. (2005). “Biolixiviación de minerales de cobre”. En: “Fundamentos y perspectivas de las tecnologías biomineras” Editado por Acevedo F. y Gentina J.C. 1a Edición, Ediciones Universitarias de Valparaíso, Valparaíso, 45-61. Arnold R.G., Hoffmann M.R., Dichristina T.J. y Picardal F.W. (1990). “Regulation of Dissimilatory Fe(III) Reduction Activity in Shewanella putrefaciens”. Applied and Environmental Microbiology 56: 2811-2817. Ballester A. (2005). “Mecanismo de biolixiviación”. En: “Fundamentos y perspectivas de las tecnologías biomineras” Editado por Acevedo F. y Gentina J.C. 1a Edición, Ediciones Universitarias de Valparaíso, Valparaíso, 9-24. Brandl H. (2001). “Microbial leaching of metals”. En: Rehm H.J. (Ed.) Biotechnology, Vol. 10. Wiley-VCH, Weinheim, 191-224. Brierley J.A. (2000). “2000 Wadsworth award lecture: Expanding role of microbiology in metallurgical processes”. Mining Engineering 52: 49-53. Bryan C.G., Hallberg K.B., Johnson C.B. (2005). “Microbial populations of tailings spoils at the São Domingos former copper mine”. Proceedings of the16th International Biohydrometallurgy Symposium. Cape Town, South Africa. Crocker F.H.,. Fredrickson J.K., White D.C., Ringelberg D. B. y Balkwill D. L. (2000). “Phylogenetic and physiological diversity of Arthrobacter strains isolated from unconsolidated subsurface sediments”. Microbiology 146: 1295-1310. Dopson M., Baker-Austin C., Koppineedi R., Bond P.L. (2003). “Growth in sulfidic mineral environments: metal resistance mechanisms in acidophilic microorganisms”. Microbiology 149: 1959-1970. Dunbar J., Ticknor L.O., Kuske C.R. (2000). “Assessment of microbial diversity in four southwestern Unated States soils by 16S rRNA gene terminal restriction fragment analysis”. Applied and Environmental Microbiology 66: 2943-2950. Egert M., Friedrich M.W. (2003). “Formation of pseudo-terminal restriction fragments, a PCRrelated bias affecting terminal restriction fragment length polymorphism analysis of microbial community structure”. Applied and Environmental Microbiology 69: 2555-2562. Gadd G.M. (2001). “Microbial metal transformations”. The Journal of Microbiology 39: 83-88. Ghauri M.A., Okibe N., Johnson D.B. (2007). “Attachment of acidophilic bacteria to solid surface: The significance of species and strain variations”. Hydrometallurgy 85: 72-80. Keylock C.J. (2005). “Simpson diversity and the Shannon–Wiener index as special cases of a generalized entropy”. Oikos 109:1, 203–207. 54 Küsel K., Dorsch T., Acker G., Stackebrandt E. (1999). “Microbial reduction of Fe(III) in acidic sediments: isolation of Acidiphilium cryptum JF-5 capable of copuling the reduction of Fe(III) to oxidation of glucose”. Applied and Environmental Microbiology 65: 3633-3640. Jernberg C., Sullivan A., Edlund C., Jansson J.K. (2005). “Monitoring of antibiotic-induced alterations in the human intestinal microflora and detection of probiotic strains by use of terminal restriction fragment length polymorphism”. Applied and Environmental Microbiology 71: 501-506. Li F., Hullar M.A.J., Lampe J.W. (2007). “Optimization of terminal restriction fragment polymorphism (TRFLP) analysis of human gut microbiota”. Journal of Microbiological Methods 68: 303-311. Liu W.-T., Marsh T.L., Cheng H., Forney L.J. (1997). “Characterization of microbial diversity by determining terminal restriction fragment length polymorphisms of genes encoding 16S rRNA”. Applied and Environmental Microbiology 63: 4516-4522. Ma H-W., Zeng A-P (2004). “Phylogenetic comparison of metabolic capacities of organisms at genome level”. Molecular Phylogenetic and Evolution 31: 204-213. Malki M., González-Toril E., Sanz J.L., Gómez F., Rodríguez N., Amils R. (2006). “Importance of the iron cycle in biohydrometallurgy”. Hydrometallurgy 83: 223-228. Meyer A., Franco A.A., Pereira A., Lopes de Souza C. (2004). “Comparison of similarity coefficients used for cluster analysis with dominant markers in maize (Zea mays L)”. Genetics and Molecular Biology 27: 83-91. Morales G. (2006). “Estimación de la biodiversidad bacteriana presente en procesos de biolixiviación mediante el análisis del DNA”, Memoria de título en Ingeniería Civil en Biotecnología, Universidad de Chile. Rawling D.E. (1998). “Industrial practice and the biology of leaching of metals from ores: The 1997 Pan labs lecture”. Journal of Industrial Microbiology and Biotechnology 20: 268-274. Rawling D.E. (2002). “Mining using microbes”. Annual Review Microbiology 56: 65-91. Rawlings D.E. y Johnson D.B. (2007). “The microbiology of biomining: development and optimizationof mineral-oxidizing microbial consortia”. Microbiology 153: 315-324 Robertson W.J., Bowman J.P., Franzmann P. D. y Mee B. J. (2001). “Desulfosporosinus meridiei sp. nov., a sporeforming sulfate-reducing bacterium isolated from gasolene-contaminated groundwater” International Journal of Systematic and Evolutionary Microbiology 51: 133–140. Roden E.E. y Lovley D.R. (1993). “Dissimilatory Fe(III) Reduction by the Marine Microorganism Desulfuromonas acetoxidans”. Applied and Environmental Microbiology 59: 734-742. Rohwerder T., Gehrke T., Kinzler K., Sand W. (2003) “Bioleaching review part A: Prograess in bioleaching: fundamentals and mechanisms of bacterial metal sulfide oxidation”. Applied Microbiology and Biotechnology 63: 239-248. 55 Saikaly P.E., Stroot P.G., Oerther D.B. (2005). “Use of 16S rRNA gene terminal restriction fragment analysis to assess the impact of solids retention time on the bacterial diversity of activated sludge” Applied and Environmental Microbiology 71: 5814-5822. Vingron M., Stoye J., Luz H. (Winter 2002/2003). “Algorithms for phylogenetic reconstructions”. Lecture, Notes and Exercises. Wakao N., Yasuda T., Jojima Y., Yamanaka S., Hiraishi A. (2002). “Enhanced growth of Acidocella facilis and related acidophilic bacteria at high concentration of aluminum”. Microbes and Environments 17: 98-102. Wang M., Ahrné S., Molín G. (2004). “T-RFLP combined with principal component analysis and 16S rRNA gene sequencing: an effectyive strategy for comparison of fecal microbiota in infantsa of different ages”. Journal of Microbiological Methods 59: 53-69. Watling H.R. (2006). “The bioleaching of sulphide minerals with emphasis on cooper sulphides – A review”. Hydrometallurgy 84: 81-108. Yahya A., Johnson D.B. (2002) “Bioleaching of pyrite at low pH and low redox potentials by novel mesophilic Gram-positive bacteria”. Hydrometallurgy 63: 181-188. mica.ibest.uidaho.edu, Página web de Microbial Community Analysis III (MICA), Programa Virtual Diegest (ISPaR), Semestre de Otoño del 2007. rna.lundberg.gu.se/cutter2/, Página web del programa de determinación de mapas de restricción Webcutter 2.0, Mayo del 2007. www.ebi.ac.uk/kalign Página web de Kalign, European Bioinformatics Institute (EBI), Mayo del 2007. www.learner.org/channel/courses/biology/textbook/microb/microb_14.html , Página web: Rediscovering biology, Octubre 2007. www.mdsg.umd.edu, Página web: Maryland Sea Grant Marine Education: Biofilms and biodiversity: how to calculate biodiversity?, 21 de Agosto del 2007. www.ncbi.nlm.nih.gov, Página web de la Nacional Center for Biotechnology Information (NCBI), Secciones Books, Genome y Taxonomy, Mayo del 2007. 56 Anexos Anexo A. Partidores utilizados Los partidores universales utilizados fueron tomados de Morales G. (2006), los cuales corresponden a: • Partidor forward marcado con el fluorósforo FAM: 341f • 5’- CCTACGGGAGGCAGCAG -3’ Partidor reverse: 1100r 5’- GGGTTGCGCTCGTTG -3’ Anexo B. Soluciones utilizadas A continuación se presenta la composición de las soluciones utilizadas en los métodos mencionados. Todas ellas fueron preparadas en agua destilada, excepto el buffer TAE y la solución para la RNasa, preparado en agua desionizada. Tabla 13. Composición de medio MC. Reactivo (NH4)2SO4 MgSO4 * 7H2O K2HPO4 * 3H2O Concentración [g/l] 0,4 0,4 0,056 Tabla 14. Composición del Buffer A2X. Reactivo Tris HCl pH 8,0 EDTA NaCl Citrato de Na CaCl2 Concentración [mM] 200 50 200 2 10 Tabla 15. Composición del Buffer de lavado. Reactivo Buffer A2X Glicerol Agua Destilada Concentración (v/v) 50% 25% 25% 57 Tabla 16. Composición del Buffer TE. Reactivo Tris HCl pH 8,0 EDTA Concentración [mM] 10 1 Tabla 17.Composición del Buffer de carga 6X. Reactivo Glicerol Azul de bromofenol Agua destilada Concentración 30% v/v 0,25% p/v - Tabla 18. Composición del Buffer TAE 50X. Reactivo Tris HCl pH 8,0 Ácido acético glacial EDTA 0,5 M Agua desionizada Concentración 24,2% p/v 5,71% v/v 10% v/v - Tabla 19. Composición del Buffer de almacenamiento. Reactivo Tris HCl pH 7,5 NaCl Concentración [mM] 100 200 Tabla 20. Composición del estándar de peso molecular, 1 Kb. Reactivo Buffer de almacenamiento Estándar de peso molecular 1 Kb Buffer de carga Agua destilada Concentración (v/v) 10% 10% 16,5% - Tabla 21. Composición de la solución de la RNasa. Reactivo Tris HCl (pH 7,5) NaCl Agua desionizada estéril 58 Concentración (mM) 10 15 - Anexo C. Protocolos (a) Protocolo de electroforesis en gel de agarosa al X%. • Pesar X gramos de agarosa, dado el porcentaje de agarosa que se desee en el gel. Disolver en 100 ml de Buffer TAE 1X, en horno microondas por aproximadamente 1 minuto, agitando cada 20 segundos hasta disolver completamente. • Una vez tibia la mezcla, adicionar 10 µl de bromuro de etidio. • Colocar la(s) peineta(s) en la cámara de electroforesis, y luego agregar la cantidad de agarosa necesaria para la formación de los pocillos. • Cuando la agarosa haya gelificado, retirar la(s) peineta(s) y cubrir con Buffer TAE 1X. • Cargar la(s) muestra(s) en el gel. Para cargar, mezclar 5µl de una muestra de DNA + 3µl de Buffer de carga 6X, y el volumen total colocarlo en uno de los pocillos formados por la peineta. • Cargar 5 µl de estándar de peso molecular. • Correr el gel por 40 minutos a 100 Volts (Programa 8). • Retirar el gel de la cámara y colocar en el transiluminador UV para visualizar la migración de las bandas. (b) Protocolo de determinación de Fe+2 y fierro total. Determinación de ferroso en precencia de férrico: • • • • • Tomar 0,1 ml de muestra diluída hasta una concentración de 100 ppm de ferroso, Agregar 1 ml de solución NaF 0,5 M y agitar; Agregar 0,4 ml de solución de fenantrolina y agitar; Agregar 1 ml de agua destilada y agitar; Leer absorbancia a 510 nm contra blanco. Determinación de fierro total: • • • • • Tomar 0,1 ml de muestra diluída hasta una concentración de 100 ppm de ferroso, Agregar 0,1 ml de solución de hidroxilamina y agitar; Agregar 0,4 ml de solución de fenantrolina y agitar; Agregar 1,9 ml de agua destilada y agitar; Leer absorbancia a 510 nm contra blanco. 59 Anexo D. Tablas de las enzimas de restricción, su sitio de restricción y el largo de los fragmentos que se forman (a) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Acidithiobacillus caldus. (776 pb) Enzyme name AatI AccB1I AccII AccIII AciI Recognition sequence agg/cct g/gyrcc Cg/cg t/ccgga ccgc No. cuts 1 2 6 1 12 AclWI AcsI AcyI AfaI AflIII AluI AlwI AlwNI Ama87I AocI ApaI ApoI Asp700I AspLEI AspS9I AsuI AvaI BamHI BanI BanII BbeI BbiII BbvI BcgI BcnI BcoI BglII BmyI BpmI BsaBI BsaHI BsaJI BsaOI BsaWI ggatc r/aatty Gr/cgyc Gt/ac a/crygt Ag/ct ggatc cagnnn/ctg c/ycgrg Cc/tnagg gggcc/c r/aatty gaann/nnttc gcg/c g/gncc g/gncc c/ycgrg g/gatcc g/gyrcc grgcy/c ggcgc/c Gr/cgyc gcagc cgannnnnntgc Cc/sgg c/ycgrg a/gatct gdgch/c ctggag gatnn/nnatc Gr/cgyc c/cnngg cgry/cg w/ccggw 1 2 1 1 1 2 1 1 1 1 1 2 1 4 2 2 1 1 2 2 1 1 2 1 1 1 1 3 2 1 1 6 1 1 Positions of sites 77 496 683 62 187 239 423 527 631 659 63 119 188 248 296 351 394 542 559 594 602 762 122 22 334 497 551 654 521 730 122 712 272 501 591 22 334 77 239 423 499 770 401 587 401 587 272 118 496 683 279 591 500 497 15 716 726 273 272 364 279 591 688 316 373 533 497 185 271 283 455 542 644 556 659 60 Enzyme name Bsc4I Bse118I Bse1I Bse21I Bse8I BseAI BseDI BseNI Bsh1236I Bsh1285I Bsh1365I BshNI BsiEI BsiI BsiMI BsiSI BsiYI BslI BsmFI BsoBI BsoFI Bsp120I Bsp1286I Bsp13I Bsp143I Bsp143II BspEI BspLU11I BsrBRI BsrDI BsrFI BsrI BsrSI BssAI BssSI Bst2UI Bst71I BstDEI BstDSI BstEII BstF5I BstH2I BstI BstMCI BstNI Recognition sequence ccnnnn/nnngg r/ccggy actgg Cc/tnagg gatnn/nnatc t/ccgga c/cnngg actgg Cg/cg cgry/cg gatnn/nnatc g/gyrcc cgry/cg ctcgtg t/ccgga c/cgg ccnnnnn/nngg ccnnnnn/nngg gggac c/ycgrg Gc/ngc g/ggccc gdgch/c t/ccgga /gatc rgcgc/y t/ccgga a/catgt gatnn/nnatc gcaatg r/ccggy actgg actgg r/ccggy ctcgtg Cc/wgg gcagc c/tnag c/crygg g/gtnacc ggatg rgcgc/y g/gatcc cgry/cg Cc/wgg No. cuts 4 1 4 1 1 1 6 4 6 1 1 2 1 1 1 3 4 4 2 1 3 1 3 1 2 1 1 1 1 2 1 4 4 1 1 6 2 2 1 1 2 1 1 1 6 Positions of sites 6 100 288 644 162 227 314 383 489 501 533 659 185 271 283 455 542 644 227 314 383 489 62 187 239 423 527 631 556 533 496 683 556 736 659 163 273 660 7 101 289 645 7 101 289 645 109 760 272 12 60 713 587 279 591 688 659 118 364 500 659 654 533 36 59 162 227 314 383 489 227 314 383 489 162 736 284 340 399 457 543 645 15 716 501 509 185 393 70 492 500 118 556 284 340 399 457 543 645 61 Enzyme name BstOI BstPI BstSFI BstUI BstX2I BstYI Bsu36I BsuRI Cac8I CfoI Cfr10I Cfr13I Cfr42I Cfr9I Csp6I CviJI CvnI DdeI DpnI DpnII DraII DsaI Eco147I Eco24I Eco64I Eco81I Eco88I Eco91I EcoNI EcoO109I EcoO65I EcoRI EcoRII EheI FauI FokI FriOI Fsp4HI GsuI HaeII HaeIII HapII HgaI HhaI Hin1I Recognition sequence Cc/wgg g/gtnacc c/tryag cg/cg r/gatcy r/gatcy cc/tnagg gg/cc gcn/ngc gcg/c r/ccggy g/gncc ccgc/gg c/ccggg g/tac rg/cy cc/tnagg c/tnag ga/tc /gatc rg/gnccy c/crygg agg/cct grgcy/c g/gyrcc cc/tnagg c/ycgrg g/gtnacc cctnn/nnnagg rg/gnccy g/gtnacc g/aattc /ccwgg ggc/gcc cccgc ggatg grgcy/c gc/ngc ctggag rgcgc/y gg/cc c/cgg gacgc gcg/c gr/cgyc No. cuts 6 1 1 6 2 2 1 3 3 4 1 2 1 1 1 10 1 2 2 2 1 1 1 2 2 1 1 1 1 1 1 1 6 1 3 2 2 3 2 1 3 3 1 4 1 Positions of sites 284 340 399 457 543 645 393 668 62 187 239 423 527 631 118 364 118 364 501 77 402 589 179 206 591 239 423 499 770 162 401 587 188 272 550 77 166 277 306 402 521 589 649 720 730 501 501 509 120 366 118 364 587 185 77 279 591 496 683 501 272 393 5 587 393 334 282 338 397 455 541 643 498 542 594 762 70 492 279 591 12 60 713 316 373 500 77 402 589 163 273 660 416 239 423 499 770 497 62 Enzyme name Hin6I HinP1I HinfI HpaII HphI Hsp92I Hsp92II HspAI ItaI KasI Kpn2I KspI Kzo9I MaeII MaeIII MamI MboI MboII MflI MnlI MroI MseI Msp17I MspA1I MspI MspR9I MvaI MvnI MwoI NarI NciI NdeII NlaIII NlaIV NspBII NspI PalI Pme55I PspAI PspALI PspEI PspN4I PspOMI PstI RsaI Recognition sequence g/cgc g/cgc g/antc c/cgg ggtga gr/cgyc catg/ g/cgc gc/ngc g/gcgcc t/ccgga ccgc/gg /gatc a/cgt /gtnac gatnn/nnatc /gatc gaaga r/gatcy cctc t/ccgga t/taa gr/cgyc cmg/ckg c/cgg cc/ngg cc/wgg cg/cg gcnnnnn/nngc gg/cgcc cc/sgg /gatc catg/ ggn/ncc cmg/ckg rcatg/y gg/cc agg/cct c/ccggg ccc/ggg g/gtnacc ggn/ncc g/ggccc ctgca/g gt/ac No. cuts 4 4 1 3 3 1 3 4 3 1 1 1 2 2 2 1 2 3 2 4 1 4 1 1 3 7 6 6 5 1 1 2 3 5 1 2 3 1 1 1 1 5 1 1 1 Positions of sites 237 421 497 768 237 421 497 768 696 163 273 660 331 354 399 497 611 658 719 237 421 497 768 12 60 713 496 659 188 118 364 142 255 251 393 533 118 364 74 158 637 118 364 11 201 327 374 659 211 564 616 752 497 187 163 273 660 273 284 340 399 457 543 645 284 340 399 457 543 645 62 187 239 423 527 631 37 234 435 505 767 497 273 118 364 611 658 719 120 498 588 664 685 187 611 658 77 402 589 77 272 274 393 120 498 588 664 685 587 672 551 63 Enzyme name SacII Sau3AI Sau96I ScrFI SduI SfaNI SfcI Sfr303I SmaI Sse9I SseBI SstII StuI TaqI TfiI ThaI Tru1I Tru9I Tsp45I Tsp509I TspEI TspRI TthHB8I XhoII XmaI XmnI Recognition sequence ccgc/gg /gatc g/gncc cc/ngg gdgch/c gcatc c/tryag ccgc/gg ccc/ggg /aatt agg/cct ccgc/gg agg/cct t/cga g/awtc cg/cg t/taa t/taa /gtsac /aatt /aatt cagtg t/cga r/gatcy c/ccggg gaann/nnttc No. cuts 1 2 2 7 3 1 1 1 1 5 1 1 1 1 1 6 4 4 1 5 5 2 1 2 1 1 Positions of sites 188 118 364 401 587 273 284 340 399 457 543 645 279 591 688 627 668 188 274 22 218 334 577 617 77 188 77 621 696 62 187 239 423 527 631 211 564 616 752 211 564 616 752 393 22 218 334 577 617 22 218 334 577 617 19 384 621 118 364 272 77 64 (b) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Acidithiobacillus ferooxidans. (776 pb) Enzyme name AatI AccB1I AccII AccIII AciI Recognition sequence agg/cct g/gyrcc cg/cg t/ccgga ccgc No. cuts 1 1 4 1 12 AcsI AfaI AflIII AgeI AluI Alw21I AlwNI Ama87I ApaI ApoI Asp700I AspHI AspLEI AspS9I AsuI AvaI BanI BanII Bbv12I BbvI BcgI BcnI BcoI BfaI BglII BlpI BmyI BpmI Bpu1102I BsaJI BsaOI BsaWI Bsc4I Bse118I Bse1I BseAI r/aatty gt/ac a/crygt a/ccggt ag/ct gwgcw/c cagnnn/ctg c/ycgrg gggcc/c r/aatty gaann/nnttc gwgcw/c gcg/c g/gncc g/gncc c/ycgrg g/gyrcc grgcy/c gwgcw/c gcagc cgannnnnntgc cc/sgg c/ycgrg c/tag a/gatct gc/tnagc gdgch/c ctggag gc/tnagc c/cnngg cgry/cg w/ccggw ccnnnn/nnngg r/ccggy actgg t/ccgga 3 4 1 1 2 1 1 1 1 3 1 1 1 1 1 1 1 2 1 3 1 1 1 3 1 1 3 1 1 6 1 2 3 2 1 1 Positions of sites 77 494 62 187 525 631 660 63 188 248 296 351 394 540 557 593 601 682 762 22 334 664 249 309 505 549 655 302 519 730 421 712 272 589 22 334 664 77 421 770 585 585 272 494 279 589 421 15 185 716 726 273 272 311 484 499 364 415 279 421 589 373 415 185 271 283 453 540 639 554 302 660 6 288 644 162 302 227 660 65 Enzyme name BseDI BseNI Bsh1236I Bsh1285I BshNI BsiEI BsiHKAI BsiI BsiMI BsiSI BsiYI BslI BsmFI BsoBI BsoFI Bsp120I Bsp1286I Bsp13I Bsp143I Bsp1720I BspEI BspLU11I BspMI BsrDI BsrFI BsrI BsrSI BssAI BssSI Bst2UI Bst71I BstDEI BstDSI BstF5I BstMCI BstNI BstOI BstSFI BstUI BstX2I BstYI BstZI BsuRI Cac8I CelII Recognition sequence c/cnngg actgg cg/cg cgry/cg g/gyrcc cgry/cg gwgcw/c ctcgtg t/ccgga c/cgg ccnnnnn/nngg ccnnnnn/nngg gggac c/ycgrg gc/ngc g/ggccc gdgch/c t/ccgga /gatc gc/tnagc t/ccgga a/catgt acctgc gcaatg r/ccggy actgg actgg r/ccggy ctcgtg cc/wgg gcagc c/tnag c/crygg ggatg cgry/cg cc/wgg cc/wgg c/tryag cg/cg r/gatcy r/gatcy c/ggccg gg/cc gcn/ngc gc/tnagc No. cuts 6 1 4 1 1 1 1 1 1 4 3 3 1 1 6 1 3 1 1 1 1 1 1 2 2 1 1 2 1 5 3 2 1 1 1 5 5 1 4 1 1 1 4 2 1 Positions of sites 185 271 283 453 540 639 227 62 187 525 631 554 494 554 421 736 660 163 273 303 661 7 289 645 7 289 645 760 272 12 60 182 392 554 713 585 279 421 589 660 364 415 660 655 402 36 59 162 302 227 227 162 302 736 284 340 455 541 640 15 185 716 415 507 185 70 554 284 340 455 541 640 284 340 455 541 640 669 62 187 525 631 364 364 551 77 394 553 587 179 206 415 66 Enzyme name CfoI Cfr10I Cfr13I Cfr42I Cfr9I CfrI Csp6I CviJI Recognition sequence gcg/c r/ccggy g/gncc ccgc/gg c/ccggg y/ggccr g/tac rg/cy No. cuts 1 2 1 1 1 2 4 12 DdeI DpnI DpnII DraII DsaI EaeI EagI EclXI Eco147I Eco24I Eco52I Eco64I Eco88I EcoNI EcoO109I EcoRI EcoRII FauI FokI FriOI Fsp4HI GsuI HaeIII HapII HgaI HhaI Hin6I HinP1I HinfI HpaII HphI Hsp92II HspAI ItaI Kpn2I KspI c/tnag ga/tc /gatc rg/gnccy c/crygg y/ggccr c/ggccg c/ggccg agg/cct grgcy/c c/ggccg g/gyrcc c/ycgrg cctnn/nnnagg rg/gnccy g/aattc /ccwgg cccgc ggatg grgcy/c gc/ngc ctggag gg/cc c/cgg gacgc gcg/c g/cgc g/cgc g/antc c/cgg ggtga catg/ g/cgc gc/ngc t/ccgga ccgc/gg 2 1 1 1 1 2 1 1 1 2 1 1 1 1 1 2 5 2 1 2 6 1 4 4 1 1 1 1 3 4 1 3 1 6 1 1 Positions of sites 770 162 302 585 188 272 392 551 248 308 504 548 77 166 184 277 394 519 553 587 645 650 720 730 415 507 366 364 585 185 392 551 551 551 77 279 589 551 494 272 5 585 334 664 282 338 453 539 638 593 762 70 279 589 12 60 182 392 554 713 373 77 394 553 587 163 273 303 661 415 770 768 768 146 218 696 163 273 303 661 354 610 659 719 768 12 60 182 392 554 713 660 188 67 Enzyme name Kzo9I MaeI MaeII MboI MboII MflI MnlI MroI MseI MspA1I MspI MspR9I MvaI MvnI MwoI NciI NdeII NlaIII NlaIV NspBII NspI PalI PinAI Pme55I PspAI PspALI PspN4I PspOMI PstI RsaI SacII Sau3AI Sau96I ScrFI SduI SfaNI SfcI Sfr303I SmaI Sse9I SseBI SstII StuI TfiI ThaI Recognition sequence /gatc c/tag a/cgt /gatc gaaga r/gatcy cctc t/ccgga t/taa cmg/ckg c/cgg cc/ngg cc/wgg cg/cg gcnnnnn/nngc cc/sgg /gatc catg/ ggn/ncc cmg/ckg rcatg/y gg/cc a/ccggt agg/cct c/ccggg ccc/ggg ggn/ncc g/ggccc ctgca/g gt/ac ccgc/gg /gatc g/gncc cc/ngg gdgch/c gcatc c/tryag ccgc/gg ccc/ggg /aatt agg/cct ccgc/gg agg/cct g/awtc cg/cg No. cuts 1 3 2 1 3 1 4 1 4 1 4 6 5 4 4 1 1 3 2 1 2 4 1 1 1 1 2 1 1 4 1 1 1 6 3 2 1 1 1 5 1 1 1 3 4 Positions of sites 364 311 484 499 142 250 364 74 158 637 364 11 107 327 374 660 211 562 615 752 187 163 273 303 661 273 284 340 455 541 640 284 340 455 541 640 62 187 525 631 37 391 433 767 273 364 610 659 719 496 586 187 610 659 77 394 553 587 302 77 272 274 496 586 585 673 249 309 505 549 188 364 585 273 284 340 455 541 640 279 421 589 627 680 669 188 274 22 334 575 616 664 77 188 77 146 218 696 62 187 525 631 68 Enzyme name Tru1I Tru9I Tsp509I TspEI TspRI XhoII XmaI XmaIII XmnI Recognition sequence t/taa t/taa /aatt /aatt cagtg r/gatcy c/ccggg c/ggccg gaann/nnttc No. cuts 4 4 5 5 3 1 1 1 1 Positions of sites 211 562 615 752 211 562 615 752 22 334 575 616 664 22 334 575 616 664 19 226 384 364 272 551 77 69 (c) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Acidithiobacillus thiooxidans. (776 pb) Enzyme Name AatI AccB1I AccII AciI Recognition sequence agg/cct g/gyrcc cg/cg ccgc No. cuts 1 1 4 13 AcsI AfaI AflIII AgeI AluI AlwNI Ama87I ApaI ApoI Asp700I AspLEI AspS9I AsuI AvaI BanI BanII BbvI BcgI BcnI BcoI BfaI BglII BmyI BpmI BsaJI BsaOI BsaWI Bsc4I Bse118I Bse1I BseDI BseNI Bsh1236I Bsh1285I BshNI BsiEI BsiI r/aatty gt/ac a/crygt a/ccggt ag/ct cagnnn/ctg c/ycgrg gggcc/c r/aatty gaann/nnttc gcg/c g/gncc g/gncc c/ycgrg g/gyrcc grgcy/c gcagc cgannnnnntgc cc/sgg c/ycgrg c/tag a/gatct gdgch/c ctggag c/cnngg cgry/cg w/ccggw ccnnnn/nnngg r/ccggy actgg c/cnngg actgg cg/cg cgry/cg g/gyrcc cgry/cg ctcgtg 2 3 1 1 2 1 1 1 2 1 1 2 2 1 1 2 3 1 1 1 2 1 3 1 6 1 1 3 2 1 6 1 4 1 1 1 1 Positions of sites 77 496 62 187 527 631 63 132 188 248 296 351 394 542 559 594 602 681 762 22 334 309 507 551 654 302 521 730 712 272 591 22 334 77 770 401 587 401 587 272 496 279 591 15 185 716 726 273 272 311 486 364 279 591 688 373 185 271 283 455 542 644 556 302 6 288 644 162 302 227 185 271 283 455 542 644 227 62 187 527 631 556 496 556 736 70 Enzyme Name BsiSI BsiYI BslI BsmFI BsoBI BsoFI Bsp120I Bsp1286I Bsp143I BspLU11I BsrDI BsrFI BsrI BsrSI BssAI BssSI Bst2UI Bst71I BstDEI BstDSI BstMCI BstNI BstOI BstSFI BstUI BstX2I BstYI BstZI BsuRI Cac8I CfoI Cfr10I Cfr13I Cfr42I Cfr9I CfrI Csp6I CviJI Recognition sequence c/cgg ccnnnnn/nngg ccnnnnn/nngg gggac c/ycgrg gc/ngc g/ggccc gdgch/c /gatc a/catgt gcaatg r/ccggy actgg actgg r/ccggy ctcgtg cc/wgg gcagc c/tnag c/crygg cgry/cg cc/wgg cc/wgg c/tryag cg/cg r/gatcy r/gatcy c/ggccg gg/cc gcn/ngc gcg/c r/ccggy g/gncc ccgc/gg c/ccggg y/ggccr g/tac rg/cy No. cuts 3 3 3 1 1 6 1 3 1 1 2 2 1 1 2 1 7 3 2 1 1 7 7 1 4 1 1 1 5 4 1 2 2 1 1 2 3 12 DdeI DpnI DpnII DraII DsaI EaeI c/tnag ga/tc /gatc rg/gnccy c/crygg y/ggccr 2 1 1 1 1 2 Positions of sites 163 273 303 7 289 645 7 289 645 760 272 12 60 182 392 556 713 587 279 591 688 364 654 36 59 162 302 227 227 162 302 736 284 340 399 443 457 543 645 15 185 716 416 509 185 556 284 340 399 443 457 543 645 284 340 399 443 457 543 645 668 62 187 527 631 364 364 553 77 394 402 555 589 133 179 206 591 770 162 302 401 587 188 272 392 553 308 506 550 77 166 184 277 394 402 521 555 589 649 720 730 416 509 366 364 587 185 392 553 71 Enzyme Name EagI EclXI Eco147I Eco24I Eco52I Eco64I Eco88I EcoNI EcoO109I EcoRI EcoRII FauI FriOI Fsp4HI GsuI HaeIII HapII HgaI HhaI Hin6I HinP1I HincII HindII HinfI HpaII HphI Hsp92II HspAI ItaI KspI Kzo9I MaeI MaeII MboI MboII MflI MnlI MseI MspA1I MspI MspR9I MvaI MvnI MwoI NciI Recognition sequence c/ggccg c/ggccg agg/cct grgcy/c c/ggccg g/gyrcc c/ycgrg cctnn/nnnagg rg/gnccy g/aattc /ccwgg cccgc grgcy/c gc/ngc ctggag gg/cc c/cgg gacgc gcg/c g/cgc g/cgc gty/rac gty/rac g/antc c/cgg ggtga catg/ g/cgc gc/ngc ccgc/gg /gatc c/tag a/cgt /gatc gaaga r/gatcy cctc t/taa cmg/ckg c/cgg cc/ngg cc/wgg cg/cg gcnnnnn/nngc cc/sgg No. cuts 1 1 1 2 1 1 1 1 1 1 7 3 2 6 1 5 3 1 1 1 1 1 1 4 3 1 3 1 6 1 1 2 1 1 3 1 5 4 1 3 8 7 4 5 1 Positions of sites 553 553 77 279 591 553 496 272 5 587 334 282 338 397 441 455 541 643 594 682 762 279 591 12 60 182 392 556 713 373 77 394 402 555 589 163 273 303 416 770 768 768 140 140 146 218 663 696 163 273 303 354 611 658 719 768 12 60 182 392 556 713 188 364 311 486 142 364 74 158 637 364 11 107 327 374 421 211 564 616 752 187 163 273 303 273 284 340 399 443 457 543 645 284 340 399 443 457 543 645 62 187 527 631 37 128 391 435 767 273 72 Enzyme Name NdeII NlaIII NlaIV NspBII NspI PalI PinAI Pme55I PspAI PspALI PspN4I PspOMI PstI RsaI SacII Sau3AI Sau96I ScrFI SduI SfaNI SfcI Sfr303I SmaI Sse9I SseBI SstII StuI TaqI TfiI ThaI Tru1I Tru9I Tsp509I TspEI TspRI TthHB8I XhoII XmaI XmaIII XmnI Recognition sequence /gatc catg/ ggn/ncc cmg/ckg rcatg/y gg/cc a/ccggt agg/cct c/ccggg ccc/ggg ggn/ncc g/ggccc ctgca/g gt/ac ccgc/gg /gatc g/gncc cc/ngg gdgch/c gcatc c/tryag ccgc/gg ccc/ggg /aatt agg/cct ccgc/gg agg/cct t/cga g/awtc cg/cg t/taa t/taa /aatt /aatt cagtg t/cga r/gatcy c/ccggg c/ggccg gaann/nnttc No. cuts 1 3 2 1 2 5 1 1 1 1 2 1 1 3 1 1 2 8 3 2 1 1 1 4 1 1 1 1 4 4 4 4 4 4 3 1 1 1 1 1 Positions of sites 364 611 658 719 498 588 187 611 658 77 394 402 555 589 302 77 272 274 498 588 587 672 309 507 551 188 364 401 587 273 284 340 399 443 457 543 645 279 591 688 627 679 668 188 274 22 334 577 617 77 188 77 621 146 218 663 696 62 187 527 631 211 564 616 752 211 564 616 752 22 334 577 617 22 334 577 617 19 226 384 621 364 272 553 77 73 (d) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Leptospirillum ferrooxidans. (778 pb) Enzyme name AatI Acc16I Acc65I AccB1I AccII AciI No. cuts 1 1 1 1 3 11 AcsI AfaI AflIII AhdI AluI Alw26I AlwNI Ama87I ApaI ApoI Asp700I Asp718I AspEI AspLEI AspS9I AsuI AvaI AviII AvrII BanI BanII BbvI BcgI BcoI BfaI BlnI BmyI BpmI BsaJI BsaMI BsaOI Bsc4I Bse118I Bse1I BseDI BseNI Bsh1236I Bsh1285I BshNI BsiEI BsiI 1 4 1 1 2 2 1 1 1 1 1 1 1 2 1 1 1 1 1 1 1 5 1 1 1 1 1 1 5 1 1 5 1 2 5 2 3 1 1 1 1 Positions of sites 77 30 144 144 62 187 630 63 188 334 351 394 518 541 558 593 601 764 334 146 484 534 550 653 657 277 732 303 424 714 102 590 334 77 144 657 31 772 586 586 102 30 643 144 590 15 55 185 521 718 728 102 644 643 590 316 161 185 455 541 643 294 555 6 65 140 541 643 378 227 306 161 185 455 541 643 227 306 62 187 630 555 144 555 738 Recognition sequence agg/cct tgc/gca g/gtacc g/gyrcc cg/cg ccgc r/aatty gt/ac a/crygt gacnnn/nngtc ag/ct gtctc cagnnn/ctg c/ycgrg gggcc/c r/aatty gaann/nnttc g/gtacc gacnnn/nngtc gcg/c g/gncc g/gncc c/ycgrg tgc/gca c/ctagg g/gyrcc grgcy/c gcagc cgannnnnntgc c/ycgrg c/tag c/ctagg gdgch/c ctggag c/cnngg gaatgc cgry/cg ccnnnn/nnngg r/ccggy actgg c/cnngg actgg cg/cg cgry/cg g/gyrcc cgry/cg ctcgtg 74 Enzyme name BsiSI BsiYI BslI BsmAI BsmBI BsmFI BsmI BsoBI BsoFI Bsp120I Bsp1286I BspLU11I BsrDI BsrFI BsrI BsrSI BssAI BssSI BssT1I Bst2UI Bst71I BstDEI BstDSI BstMCI BstNI BstOI BstSFI BstUI BstZI BsuRI Cac8I CfoI Cfr10I Cfr13I Cfr42I CfrI Csp6I CviJI No. cuts 1 5 5 2 1 2 1 1 8 1 1 1 1 1 2 2 1 1 1 3 5 2 2 1 3 3 1 3 1 5 4 2 1 1 1 1 4 15 DdeI DraII DsaI EaeI EagI Eam1105I EclHKI EclXI Eco130I Eco147I Eco24I Eco32I 2 1 2 1 1 1 1 1 1 1 1 2 Positions of sites 379 7 66 141 542 644 7 66 141 542 644 303 424 425 109 762 294 102 12 52 60 182 392 515 555 715 586 590 653 36 378 227 306 227 306 378 738 643 399 457 542 15 55 185 521 718 416 486 161 185 555 399 457 542 399 457 542 239 62 187 630 552 77 271 378 554 588 179 206 308 590 31 772 378 586 188 552 145 483 533 549 77 160 166 184 271 277 310 317 378 394 554 588 648 722 732 416 486 586 161 185 552 552 657 657 552 643 77 590 120 365 Recognition sequence c/cgg ccnnnnn/nngg ccnnnnn/nngg gtctc cgtctc gggac gaatgc c/ycgrg gc/ngc g/ggccc gdgch/c a/catgt gcaatg r/ccggy actgg actgg r/ccggy ctcgtg c/cwwgg cc/wgg gcagc c/tnag c/crygg cgry/cg cc/wgg cc/wgg c/tryag cg/cg c/ggccg gg/cc gcn/ngc gcg/c r/ccggy g/gncc ccgc/gg y/ggccr g/tac rg/cy c/tnag rg/gnccy c/crygg y/ggccr c/ggccg gacnnn/nngtc gacnnn/nngtc c/ggccg c/cwwgg agg/cct grgcy/c gat/atc 75 Enzyme name Eco52I Eco64I Eco88I EcoNI EcoO109I EcoRII EcoRV EcoT14I ErhI Esp3I FauI FriOI Fsp4HI FspI GsuI HaeIII HapII HgaI HhaI Hin6I HinP1I HinfI HpaII HphI Hsp92II HspAI ItaI KpnI KspI MaeI MboII MnlI No. cuts 1 1 1 2 1 3 2 1 1 1 3 1 8 1 1 5 1 2 2 2 2 1 1 2 3 2 8 1 1 1 1 9 MseI MslI MspA1I MspI MspR9I Mva1269I MvaI MvnI MwoI NlaIII NlaIV NspBII NspI PalI PleI Pme55I PspN4I PspOMI 3 1 1 1 3 1 3 3 4 3 3 1 2 5 1 1 3 1 Positions of sites 552 144 102 5 642 586 397 455 540 120 365 643 643 425 541 593 764 590 12 52 60 182 392 515 555 715 30 316 77 271 378 554 588 379 52 626 31 772 29 770 29 770 235 379 267 354 610 657 721 29 770 12 52 60 182 392 515 555 715 148 188 644 74 11 22 201 317 327 498 510 635 746 279 501 615 709 187 379 399 457 542 294 399 457 542 62 187 630 391 435 523 769 610 657 721 146 587 636 187 610 657 77 271 378 554 588 239 77 146 587 636 586 Recognition sequence c/ggccg g/gyrcc c/ycgrg cctnn/nnnagg rg/gnccy /ccwgg gat/atc c/cwwgg c/cwwgg cgtctc cccgc grgcy/c gc/ngc tgc/gca ctggag gg/cc c/cgg gacgc gcg/c g/cgc g/cgc g/antc c/cgg ggtga catg/ g/cgc gc/ngc ggtac/c ccgc/gg c/tag gaaga cctc t/taa caynn/nnrtg cmg/ckg c/cgg cc/ngg gaatgc cc/wgg cg/cg gcnnnnn/nngc catg/ ggn/ncc cmg/ckg rcatg/y gg/cc gagtc agg/cct ggn/ncc g/ggccc 76 Enzyme name RsaI SacII Sau96I ScrFI SduI SfcI Sfr303I Sse9I SseBI SspI SstII StuI StyI TaqI ThaI Tru1I Tru9I Tsp509I TspEI TspRI TthHB8I XmaIII XmnI No. cuts 4 1 1 3 1 1 1 4 1 1 1 1 1 1 3 3 3 4 4 2 1 1 1 Positions of sites 146 484 534 550 188 586 399 457 542 590 239 188 218 334 576 616 77 25 188 77 643 620 62 187 630 279 501 615 279 501 615 218 334 576 616 218 334 576 616 19 418 620 552 77 Recognition sequence gt/ac ccgc/gg g/gncc cc/ngg gdgch/c c/tryag ccgc/gg /aatt agg/cct aat/att ccgc/gg agg/cct c/cwwgg t/cga cg/cg t/taa t/taa /aatt /aatt cagtg t/cga c/ggccg gaann/nnttc 77 (e) Tabla de las enzimas de restricción que cortan el fragmento del rDNA 16S amplificado de Sulfobacillus thermosulfidooxidans. (766 pb) Enzyme name Acc65I AccB1I AccBSI AccII AccIII AciI No. cuts 1 2 1 6 1 14 AcsI AfaI AluI AlwNI Ama87I ApaI ApoI Asp718I AspLEI AspS9I AsuI AvaI AvaII BanI BanII BbsI Bbv16II BbvI BcgI BcnI BcoI BfaI Bme18I BmyI BpiI BpuAI BsaAI BsaJI BsaOI BsaWI Bsc4I Bse118I Bse1I BseAI BseDI BseNI BseRI Bsh1236I Bsh1285I BshNI 1 4 3 1 1 2 1 1 8 6 6 1 1 2 2 2 2 3 1 6 1 2 1 2 2 2 2 6 2 1 4 2 3 1 6 3 1 6 2 2 Positions of sites 139 139 506 345 62 182 302 417 625 665 209 63 183 243 258 291 345 388 507 536 553 588 666 674 752 328 141 478 529 545 93 515 720 702 490 585 680 328 139 40 304 390 417 627 662 696 760 117 126 400 494 581 676 117 126 400 494 581 676 490 494 139 506 585 680 75 115 75 115 15 180 706 716 120 130 278 451 491 501 490 305 480 494 585 680 75 115 75 115 169 251 129 180 374 449 490 536 300 550 209 6 282 502 536 114 136 222 396 647 209 129 180 374 449 490 536 222 396 647 286 62 182 302 417 625 665 300 550 139 506 78 Recognition sequence g/gtacc g/gyrcc gagcgg cg/cg t/ccgga ccgc r/aatty gt/ac ag/ct cagnnn/ctg c/ycgrg gggcc/c r/aatty g/gtacc gcg/c g/gncc g/gncc c/ycgrg g/gwcc g/gyrcc grgcy/c gaagac gaagac gcagc cgannnnnntgc cc/sgg c/ycgrg c/tag g/gwcc gdgch/c gaagac gaagac yac/gtr c/cnngg cgry/cg w/ccggw ccnnnn/nnngg r/ccggy actgg t/ccgga c/cnngg actgg gaggag cg/cg cgry/cg g/gyrcc Enzyme name BsiEI BsiI BsiMI BsiSI No. cuts 2 2 1 11 BsiYI BslI BsmFI BsoBI BsoFI 4 4 2 1 9 Bsp120I Bsp1286I Bsp13I Bsp143I Bsp143II BspEI BspMI BsrBI BsrFI BsrI BsrSI BssAI BssSI Bst2UI Bst71I BstD102I BstDEI BstDSI BstH2I BstMCI BstNI BstOI BstSNI BstUI BstZI BsuRI Cac8I CfoI Cfr10I Cfr13I Cfr42I Cfr9I CfrI Csp6I CviJI 2 2 1 1 1 1 1 1 2 3 3 2 2 4 3 1 2 2 1 2 4 4 1 6 1 8 6 8 2 6 1 1 1 4 18 DdeI DpnI DpnII 2 1 1 Positions of sites 300 550 658 726 209 115 120 129 137 210 269 278 451 491 500 510 7 283 503 537 7 283 503 537 109 750 490 12 60 177 289 386 550 663 672 703 581 676 585 680 209 358 663 209 701 345 114 136 222 396 647 222 396 647 114 136 658 726 334 398 537 639 15 180 706 345 410 524 180 374 663 300 550 334 398 537 639 334 398 537 639 251 62 182 302 417 625 665 547 77 118 128 396 401 549 583 678 42 174 201 585 676 692 40 304 390 417 627 662 696 760 114 136 117 126 400 494 581 676 183 490 547 140 477 528 544 44 77 93 118 128 136 155 179 265 272 291 396 401 515 549 583 678 720 410 524 360 358 79 Recognition sequence cgry/cg ctcgtg t/ccgga c/cgg ccnnnnn/nngg ccnnnnn/nngg gggac c/ycgrg gc/ngc g/ggccc gdgch/c t/ccgga /gatc rgcgc/y t/ccgga acctgc gagcgg r/ccggy actgg actgg r/ccggy ctcgtg cc/wgg gcagc gagcgg c/tnag c/crygg rgcgc/y cgry/cg cc/wgg cc/wgg tac/gta cg/cg c/ggccg gg/cc gcn/ngc gcg/c r/ccggy g/gncc ccgc/gg c/ccggg y/ggccr g/tac rg/cy c/tnag ga/tc /gatc Enzyme name DraII DsaI EaeI EagI EclXI Eco105I Eco24I Eco47I Eco52I Eco64I Eco88I EcoNI EcoO109I EcoRI EcoRII FauI FriOI Fsp4HI No. cuts 2 2 1 1 1 1 2 1 1 2 1 1 2 1 4 4 2 9 HaeII HaeIII HapII 1 8 11 HgaI HgiEI HhaI Hin6I HinP1I HinfI HpaII 2 1 8 8 8 1 11 HphI Hsp92II HspAI ItaI 2 2 8 9 Kpn2I KpnI KspI Kzo9I MaeI MaeII MboI MboII MnlI MroI MseI MslI MspA1I MspI 1 1 1 1 2 3 1 4 7 1 3 1 1 11 Positions of sites 581 677 180 374 547 547 547 251 585 680 494 547 139 506 490 5 581 677 328 332 396 535 637 536 588 678 752 585 680 12 60 177 289 386 550 663 672 703 663 77 118 128 396 401 549 583 678 115 120 129 137 210 269 278 451 491 500 510 410 621 494 40 304 390 417 627 662 696 760 38 302 388 415 625 660 694 758 38 302 388 415 625 660 694 758 22 115 120 129 137 210 269 278 451 491 500 510 278 348 605 709 38 302 388 415 625 660 694 758 12 60 177 289 386 550 663 672 703 209 143 183 358 305 480 168 190 250 358 29 74 114 369 11 152 269 284 311 321 415 209 261 610 742 653 182 115 120 129 137 210 269 278 451 491 500 510 80 Recognition sequence rg/gnccy c/crygg y/ggccr c/ggccg c/ggccg tac/gta grgcy/c g/gwcc c/ggccg g/gyrcc c/ycgrg cctnn/nnnagg rg/gnccy g/aattc /ccwgg cccgc grgcy/c gc/ngc rgcgc/y gg/cc c/cgg gacgc g/gwcc gcg/c g/cgc g/cgc g/antc c/cgg ggtga catg/ g/cgc gc/ngc t/ccgga ggtac/c ccgc/gg /gatc c/tag a/cgt /gatc gaaga cctc t/ccgga t/taa caynn/nnrtg cmg/ckg c/cgg Enzyme name MspR9I No. cuts 10 MvaI MvnI MwoI NciI NdeII NlaIII NlaIV NspBII NspI PalI PspAI PspALI PspN4I PspOMI RsaI SacII Sau3AI Sau96I ScrFI 4 6 6 6 1 2 6 1 1 8 1 1 6 2 4 1 1 6 10 SduI Sfr303I SinI SmaI SnaBI Sse9I SstII TaqI TfiI ThaI Tru1I Tru9I Tsp509I TspEI TspRI TthHB8I XmaI XmaIII 2 1 1 1 1 4 1 1 1 6 3 3 4 4 2 1 1 1 Positions of sites 120 130 278 334 398 451 491 501 537 639 334 398 537 639 62 182 302 417 625 665 229 385 429 668 702 757 120 130 278 451 491 501 358 605 709 135 141 495 508 582 678 182 605 77 118 128 396 401 549 583 678 490 492 135 141 495 508 582 678 581 676 141 478 529 545 183 358 117 126 400 494 581 676 120 130 278 334 398 451 491 501 537 639 585 680 183 494 492 251 213 328 571 611 183 615 22 62 182 302 417 625 665 261 610 742 261 610 742 213 328 571 611 213 328 571 611 395 598 615 490 547 81 Recognition sequence cc/ngg cc/wgg cg/cg gcnnnnn/nngc cc/sgg /gatc catg/ ggn/ncc cmg/ckg rcatg/y gg/cc c/ccggg ccc/ggg ggn/ncc g/ggccc gt/ac ccgc/gg /gatc g/gncc cc/ngg gdgch/c ccgc/gg g/gwcc ccc/ggg tac/gta /aatt ccgc/gg t/cga g/awtc cg/cg t/taa t/taa /aatt /aatt cagtg t/cga c/ccggg c/ggccg Anexo E. Digestión virtual A continuación se presenta una tabla con los tamaños de los TRFs obtenidos de la digestión virtual. Tabla 22. Tamaños de los TRFs obtenidos de la digestión virtual de las bacterias asociadas a procesos de biolixiviación Acetobacter methanolicus Acetobacter spp. Acidimicrobium ferrooxidans Acidiphilium acidophilum Acidiphilium angustum Acidiphilium cryptum Acidiphilium symbioticum Acidithiobacillus caldus Acidithiobacillus albertensis Acidithiobacillus capsulatus Acidithiobacillus concretivorus Acidithiobacillus delicatus Acidithiobacillus denitrificans Acidithiobacillus ferrooxidans Acidithiobacillus intermedius Acidithiobacillus kabobis Acidithiobacillus neapolitanus Acidithiobacillus novelius Acidithiobacillus organoparus Acidithiobacillus perometabolis Acidithiobacillus pumbophilus Acidithiobacillus rubellus Acidithiobacillus tepidarius Acidithiobacillus thiooxidans Acidithiobacillus thioparus Acidithiobacillus versutus Acidobacterium capsulatum Acidocella spp. Acidomonas methanolica Alicyclobacillus spp. Alicyclobacillus disulfidooxidans Arthrobacter spp. Aureobacterium liquifaciens Bacillus coagulans Bacillus licheniformis Bacillus megaterium Bacillus polymyxa Chromobacterium violaceum Comamonas testosteroni Crenothrix spp. Desulfosporosinus spp. Enterobacter aglomerans Enterobacter cloacae RsaI 121 121 121 121 121 121 551 309 146 146 249 147 309 117 121 121 121 146 146 126 147 117 117 146 102 94 94 Msp I 138 138 138 138 138 138 163 163 163 163 163 163 163 148 123 138 138 164 148 143 102 216 215 130 132 164 163 82 Temperatura Termófilo moderado Mesófilo Mesófilo Mesófilo Termófilo moderado Mesófilo Mesófilo Mesófilo Mesófilo Mesófilo - Termófilo moderado Mesófilo - Termófilo moderado Ferrimicrobium acidiphilium Gallionella spp. Kingella kingae Lactobacillus acidophilus Leptospirillum ferrooxidans Leptospirillum ferriphilium Leptospirillum thermoferooxidans Leptospirillum spp. Leptotrix discophora Metallogenium spp. Ochrobacterium anthropi Propionibacterium acnes Pseudomonas cepacia Pseudomonas putida Psychrobacter glacincola Serratia ficaria Shewanella spp. Siderocapsa spp. Staphilococcus latis Staphilococcus spp Stenotrophomonas maltophila Sulfobacillus acidophilus Sulfobacillus thermosulfidooxidans Thermothrix thiopara Thiobacillus prosperus Thiomonas cuprinus Thiomonas spp. RsaI 121 146 146 552 146 146 Msp I 138 103 163 215 379 103 146 146 525 464 317 551 551 238 146 146 141 141 135 552 146 103 163 102 143 163 130 163 163 215 130 143 115 203 163 163 83 Temperatura Mesófilo Mesófilo Mesófilo Mesófilo Termófilo moderado Termófilo moderado Mesófilo Mesófilo Mesófilo 84