Tema 7. Estructura y organización del ADN Objetivos
Anuncio

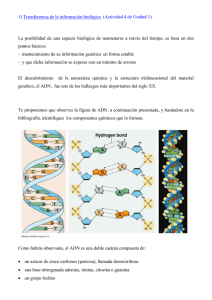
Tema 7. Estructura y organización del ADN Genética CC. Mar 2005-06 Objetivos •Examinar las propiedades que debe tener el material hereditario. •Comprender la estructura y organización del material hereditario. •Entender la replicación del ADN. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 2 Material hereditario • El material hereditario debe … – Contener de una forma estable la información genética. – Transmitir la información fielmente. – Poder cambiar, permitiendo la variación, adaptación y evolución. • El material hereditario lo forman: – ¿Proteínas? – ¿Ácidos nucleicos (ADN y ARN)? Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 3 Búsqueda del material hereditario 1890 August Weismann propone que hay una sustancia en el núcleo que controla las características de los individuos. 1900 Los cromosomas contienen la información hereditaria. Más tarde se comprueba que los cromosomas están hechos de ácidos nucleicos (ADN y ARN) y proteínas. 1928 Experimento de transformación de Griffith. 1944 Experimento de transformación de Avery, MacLeod y McCarty. 1953 Experimento del bacteriófago de Hershey y Chase. 1953 Watson y Crick proponen la estructura en doble hélice del ADN. 1956 Gierer y Schramm demuestran que el ARN es el material hereditario de algunos virus. 1957 Fraenkel-Conrat y Singer confirman el experimento de Gierer y Schramm. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 4 Experimento de transformación de Griffith • Streptococcus pneumoniae: agente transforma cepa IIR (no virulenta) en IIIS (virulenta). Figura. Experimento de transformación de Griffith (1928). Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 5 Experimento de transformación de Avery, MacLeod y McCarty • Lisaron bacterias IIIS y aislaron sus componentes. • El ADN es el agente transformante. Figura. Oswald Avery Figura. Experimento de transformación de Avery, MacLeod y McCarty (1944). Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 6 Bacteriófagos Figura. Fago T2. Figura. Ciclo lítico del fago T2. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 7 Experimento de Hershey y Chase • Marcan fagos T2 – DNA (32P) – Proteínas (35S) • Infectan E. coli. • 32P aparece en las bacterias y progenie. Alfred Hershey, Nobel 1969 Figura. Experimento de Hershey y Chase (1953) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 8 Virus ARN • Gierer y Schramm (1956): ARN provoca enfermedad del virus del mosaico del tabaco (TMV). • Fraenkel-Conrat y Singer (1957): TMV (RNA A, proteínas B) + TMV (RNA B, proteínas A) = TMV A. Figura. Experimento de Fraenkel-Conrat y Singer. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 9 Estructura ADN (I) • ADN y ARN son polímeros formados por cadenas de nucleótidos: – azúcar pentosa: desoxirribosa (ADN) o ribosa (ARN). – base nitrogenada. – grupo fosfato. Figura. Estructura de la desoxirribosa y ribosa. Figura. Grupo fosfato. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 10 Estructura ADN (II) • Purinas: adenina (A) y guanina (G). • Pirimidinas: citosina (C), timina (T) y uracilo (U). • ADN: A, C, G y T. • ARN: A, C, G y U. Figura. Bases nitrogenadas. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 11 Estructura ADN (III) • Nucleósido = pentosa (C1) –enlace covalente– base (purina N9, pirimidina N1). • Nucleótido = pentosa (C5) + grupo fosfato. • Cadenas polinucleotídicas: nucleótido (fosfato) –enlace fosfodiéster 5’-3’– nucleótido (C3). Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 12 Estructura del ADN • Estructura descubierta por James Watson y Francis Crick en 1953. • Basada en dos fuentes de información principales: – Estudios de composición de bases de Erwin Chargaff. – Estudios de difracción de rayos X de Rosalind Franklin y Maurice Wilkins. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 13 Reglas de Chargaff • Chargaff (1949): – – – – (A + G) / (C + T) = 1 A/T = 1 G/C = 1 (A + T) / (G + C) = %GC • Contenido GC varía a escala evolutiva: – 50-60% procariotas – 45% eucariotas Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 14 Difracción de rayos X • Los rayos X son difractados de una forma particular según la disposición de los átomos en una molécula. • Franklin y Wilkins (1953), ADN: – hélice no sencilla – diámetro de 2nm – dos regularidades de 0.34 y 3.4 nm a lo largo del eje de la molécula Figura. Difracción de rayos X. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 15 Modelo de Watson y Crick (1953) 1. Dos cadenas polinucleotídicas enlazadas en una doble hélice dextrógira. 2. Las cadenas son antiparalelas, una está orientada en dirección 5’-3’ y la otra en dirección 3’-5’. 3. El esqueleto azúcar-fosfato se sitúa en la parte externa, mientras que las bases nitrogenadas se orientan perpendicularmente hacia el eje interno. 4. Las bases complementarias en cadenas opuestas forman enlaces de hidrógeno. A se empareja con T (puentes 2H), y C con G (puentes 3H). 5. Pares de bases 0.34 nm aparte. Una vuelta completa de la hélice requiere 3.4 nm (10 bases por vuelta). 6. Las vértebras de azúcar-fosfato forman surcos mayores y menores. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 16 Modelo de la doble hélice ADN-B Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 17 Un descubrimiento revolucionario Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 18 Protagonistas • Crick, Watson y Wilkins recibieron el Nobel en 1962. Rosalind Franklin James D. Watson Francis H. Crick Maurice H. F. Wilkins Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 19 Tipos diferentes de ADN Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 20 Estructura del ARN • El ARN es de cadena única, y puede adoptar estructuras complejas, incluso intramoleculares. • El ARN utiliza como azúcar la ribosa. • El ARN utiliza U en vez de T. • El ARN puede catalizar importante reacciones biológicas. tRNA rRNA Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 21 Organización del ADN en cromosomas • El DNA de la célula se organiza cromosomas. • El cromosoma o juego de cromosomas que contiene todo el DNA de un organismo se denomina genoma. • En procariotas el genoma es usualmente un cromosoma circular único. • En eucariotas el genoma es un juego haploide completo de cromosomas contenidos en el núcleo celular. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 22 Cromosomas virales • Virus = ácidos nucleicos + proteínas. • Material genético: – ADN!1, ADN!2, ARN!1, ARN!2 – Circular o linear – Uno o varios cromosomas Los fagos pares son ADN!1. El genoma de T4 abarca 168 kb. En una población los distintos genomas son permutaciones circulares de los mismos genes. Algunos genes se repiten al principio y final del cromosoma, lo que se denomina redundancia terminal. Fago "X174 infecta E. coli. Tiene un único cromosoma circular de ADN de 5386 nucleótidos. Forma icosaédrica. El fago # es parecido a los fagos T. Genoma ADN!2. Presenta extremos pegajosos complementarios. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 23 Cromosomas procarióticos • La mayoría ADN!2 circular. • En Bacteria y Archaea el cromosoma se localiza una masa densa (nucleoide). • E. coli: 1 cromosoma de 4.6 Mb – Superenrrollamiento – Lazos Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 24 Valor C • Valor C = cantidad total de ADN en el genoma haploide de una especie. • Varía entre las especies sin relación con complejidad estructural u organizativa (paradoja del valor C). Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 25 Cromatina • La cromatina es el complejo de ADN y proteínas cromosómicas. Parecida en todos los eucariotas. • En la cromatina encontramos proteínas histonas y no histonas. • Las histonas son comunes, pequeñas, básicas (+) – – – – Papel fundamental en el empaquetamiento de la cromatina. Cinco tipos principales H1, H2A, H2B, H3 y H4, En peso, histonas/ADN =1 en todos los eucariotas. Muy conservadas evolutivamente (H4 de vacas y guisantes se diferencian en 2 aminoácidos). • Las proteínas no histonas son a menudo acídicas (-) – Difiere mucho entre los tipos celulares y entre los diferentes organismos. – En peso, no histona / ADN = 50-100%. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 26 Empaquetamiento ADN (I) • El genoma de un única célula humana (3.4 ! 109 pb) debería de medir 2.3 metros. • Empaquetamiento a varios niveles: – Enrollamiento del ADN (145 pb) sobre octámeros de histonas (2 ! H2A, H2B, H3 y H4) para formar los nucleosomas. – Conexión de los nucleosomas mediante ADN de unión (9-115 pb) asociado a H1. – Empaquetamiento de los nucleosomas en fibras de cromatina de 30 nm. – Formación de dominios en lazo. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 27 Empaquetamiento ADN (II) Nucleosoma Fibras de 30 nm Nucleosomas unidos por ADN de unión + H1 Dominios en lazo Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 28 Empaquetamiento ADN (III) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 29 Eucromatina y heterocromatina • Eucromatina: sigue los ciclos habituales de condensación (máxima en metafase)y descondensación durante el ciclo celular. – La mayoría del genoma de una célula activa es eucromatina. – Transcripción activa. – Secuencias no repetidas. • Heterocromatina: siempre condensada. – Transcripción inactiva. – La heterocromatina constitutiva está presente en todas las células en posiciones idénticas cromosomas homólogos. Básicamente ADN repetitivo. Ej. Regiones centroméricas. – La heterocromatina facultativa varía en estado de célula a célula, durante el desarrollo, y a veces, entre cromosomas homólogos. Ej. corpúsculos de Barr. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 30 ADN centromérico y telomérico • En el centrómero se pueden encontrar secuencias de ADN especiales (CEN en levaduras) que interactúan con los microtúbulos. • Las secuencias teloméricas mantienen la estabilidad de los cromosomas y intervienen en su replicación: – Secuencias simples teloméricas: secuencias repetidas en tándem en los extremos con funciones esenciales estabilizadoras. – Secuencias asociadas a telómeros, de forma más interna, algo más complejas y con función desconocida. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 31 Propiedades físico-químicas de los ácidos nucleicos • Densidad de los ácidos nucleicos. • Desnaturalización de los ácidos nucleicos: temperatura de fusión (Tm). • Absorbancia a 260 nm. • Cinética de renaturalización: Curvas Cot. • Hibridación de los ácidos nucleicos. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 32 Densidad y desnaturalización • $G+C $densidad del ADN. • Desnaturalización: paso de cadena doble a sencilla. Absorbancia a 260 nm – $G+C $estabilidad del ADN (+puentes 3H). – Temperatura de fusión (Tm): Tº necesaria para desnaturalizar la mitad del ADN de una mezcla. – $G+C $Tm Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 33 Absorbancia UV a 260 nm • El menor grado de absorción se produce en estado de doble hélice. • La absorción aumenta cuando se produce la desnaturalización pasando a estado de hélice sencilla (efecto hipercrómico, aumento de la absorbancia). • Si degradamos este ADN de hélice sencilla a nivel de nucleótidos libres, de nuevo aumenta la absorbancia. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 34 Cinética de la reasociación (I) • La renaturalización es el paso de hélice sencilla a doble hélice. • Curvas Cot o de renaturalización. • Cot ! = tiempo en que se ha reasociado ! del ADN. – Directamente proporcional a la complejidad del ADN del organismo. – $secuencias repetidas $velocidad renaturalización Figura. Curva Cot. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 35 Cinética de la reasociación (II) • Las curvas Cot de virus y bacterias tienen un sólo punto de inflexión (secuencias únicas). • Las curvas Cot de eucariotas poseen varios puntos de inflexión: – Cot ! bajos (10-4 a 10-1): secuencias altamente repetidas – Cot ! 10–102: secuencias moderadamente repetidas. – Cot ! > 103 : secuencias únicas o de bajo número de copias. Figura. Curvas Cot. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 36 Hibridación • Las técnicas de desnaturalización renaturalización permiten la hibridación ADN+ADN o ADN+ARN. • Se puede identificar el gen que ha dado lugar a un determinado ARN mensajero. • Las técnicas de hibridación "in situ" permiten localizar la posición de un gen de un cromosoma eucariótico. – ADN marcado con fluorescencia (FISH). – Marcaje del genoma (GISH). Figura. FISH Figura. GISH Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 37 Secuencias únicas y repetidas de ADN • ADN de secuencia única: una o pocas copias. – Genes codificantes – ADN procariota – ~65% genoma humano • ADN repetitivo: – Moderadamente repetitivo (pocas o hasta 105 copias) o altamente repetitivo (105 – 107 copias). – Disperso (SINEs y LINEs; función desconocida) o en tándem (centrómeros y telómeros; ~65% genoma humano; microsatélites). Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 38 Modelos de replicación del ADN • Semiconservativo: cada molécula de la progenie conserva una de las hebras parentales. • Conservativo: las hebras parentales sirven como un único molde para la síntesis de nuevas dobles hélices. • Dispersivo: fragmentos de doble hélice parental sirven como moldes para la síntesis de nuevos segmentos de doble cadena. Las nuevas hélices están formadas por segmentos parentales y de la progenie. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 39 Experimento de Meselson y Stahl (1958) • Obtuvieron evidencia experimental de que el modelo semiconservativo era el correcto. • Marcan ADN de E. coli con 15N. • 15N y 14N (normal) se pueden separar mediante centrifugación en gradientes de densidad. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 40 Experimento de Meselson y Stahl (1958) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 41 ADN polimerasa I • Arthur Kornberg (1955): la ADN polimerasa I lleva a cabo la síntesis de ADN. • Los componentes necesarios son: – La ADN polimerasa I – Desoxirribonucleósidos trifosfato (dNTPs = dATP, dCTP, dGTP, dTTP), precursores de los nucleótidos. – Un molde de ADN. – Iones de magnesio (Mg2+), que optimizan la actividad de la ADN polimerasa. Figura. Arthur Kornberg (Nobel 1959). Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 42 Reacción de la polimerasa I de ADN (I) • Formación de un enlace fosfodiéster en el 3’-OH de la desoxirribosa y el fosfato 5’ del dNTP. – La energía proviene de la liberación de 2 de los 3 fosfatos. – La cadena que crece actúa como cebador en la reacción. • La polimerasa encuentra el dNTP complementario al nucleótido de la cadena molde y lo incorpora – 800 nucleótidos por segundo. – 1 error cada 10-6 nucleótidos. • La dirección de la síntesis es 5’-3’. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 43 Reacción de la polimerasa I de ADN (II) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 44 Reacción de la polimerasa I de ADN (III) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 45 Polimerasas de ADN procarióticas • E. coli: 3 polimerasas de ADN (I, II, y II). • Actividad exonucleásica 3’-5’: corrección de errores (tasa error final 10-9). • La polimerasa de ADN I presenta además actividad exonucleásica 5’-3’. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 46 Replicación en procariotas • Un único origen de replicación (oriC en E. coli, ~245 pb). • En este sitio el ADN se desnaturaliza en cadenas sencillas produciendo una horquilla de replicación. • Replicación bidireccional. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 47 Inicio de la replicación en E. coli (I) 1. 2. 3. 4. 5. 6. 7. Una girasa (tipo de topoisomerasa) relaja el superenrollamiento del ADN. Proteínas iniciadoras se unen al origen de replicación. Una helicasa de ADN se une a las proteínas iniciadoras. La helicasa avanza hidrolizando ATP desenrollando la doble cadena. Las proteínas de unión al ADN de cadena sencilla (SSB), previniendo que estas cadenas se emparejen de nuevo. La primasa de ADN (polimerasa de ADN) se une a la helicasa formando el primosoma. La primasa sintetiza un cebador de ARN (11-12 nt.), al cual puede añadir nucleótidos la polimerasa de ADN III en dirección 5’-3’. El cebador de RNA es más tarde eliminado y reemplazado por ADN por acción de la polimerasa de ADN I. El hueco es sellado por una ligasa. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 48 Inicio de la replicación en E. coli (II) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 49 Replicación semidiscontinua (I) • Para mantener la polaridad 5’-3’, el ADN ha de sintetizarse en direcciones opuestas en ambas hebras molde. • Hebra líder: síntesis en la misma dirección que el movimiento de la horquilla de replicación. – Síntesis continua. – Sólo necesita 1 cebador • Hebra retardada: síntesis en dirección opuesta al movimiento de la horquilla de replicación. – Síntesis discontinua (fragmentos de Okazaki). – Necesita varios cebadores de ARN. – Eventualmente los fragmentos de Okazaki son conectados entre sí por la acción de la polimerasa I de ADN (que reemplaza el cebador de ARN por ADN) y una ligasa de ADN, que une los fragmentos. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 50 Replicación semidiscontinua (II) ADN superenrrollado relajado por la girasa y desenrollado por la helicasa + proteínas: 5’ Proteínas SSB Fragmentos de Okazaki ATP 1 Polimerasa III 2 3 Hebra retardada Helicasa + Proteínas Iniciadoras 3’ primasa Polimerasa III % 5’ Cebador ARN 3’ Pares de bases 5’ Cebador de ARN reemplazado por la polimerasa I & hueco sellado por la ligasa 3’ Hebra líder Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 51 Replicación semidiscontinua (III) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 52 Replicación semidiscontinua (IV) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 53 Replisoma • Las proteínas que intervienen en la replicación forma un complejo denominado replisoma. • La hebra retardada puede plegarse: – las polimerasas de las dos hebras forman un complejo. – Se acercan los fragmentos de Okazaki. El primosoma puede ser reusado en la misma horquilla. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 54 Replicación ADN circular E. coli 1. Dos horquillas de replicación resultan en una estructura en forma de zeta (&). 2. A medida que se separan las hebras, se forma superenrrollamiento positivo en algún otro lugar de la molécula. 3. Las topoisomerasas alivian la tensión en los superenrrollamientos, permitiendo que las dos hebras continúen separándose. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 55 Replicación en círculos rodantes • 1. 2. 3. 4. Varios fagos de ADN, como "X174. Corte en la hebra +. El extremo 5’ la hebra + se desplaza para formar un horquilla. La cadena intacta sirve como molde. La síntesis en la la hebra + desplazada ocurre de forma discontinua hacia el final de la cadena. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 56 Replicación del ADN en eucariotas • La replicación el ADN es similar en procariotas y en eucariotas (varios cromosomas). • En cada ciclo celular, cada cromosoma debe de ser duplicado fielmente durante la fase S. Tres puntos de chequeo: – Punto START (G1; en levaduras): se comprueba si la célula es suficientemente grande y el ambiente favorable, para pasar a la fase S. – Punto de chequeo de la G2: se decidirá si la célula entra en mitosis, para lo que todo el DNA tiene que estar replicado. – Punto de chequeo durante la mitosis: los cromosomas deben de estar propiamente ligados al huso mitótico para que se complete la mitosis. – En estos chequeos intervienen ciertas proteínas denominadas ciclinas, y quinasas dependientes de ciclinas. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 57 Polimerasas de ADN eucarióticas • No se sabe mucho sobre estas enzimas, pero al menos 5 han sido identificadas en mamíferos: – Polimerasa ' (alfa): nuclear, replicación del ADN, sin función correctora. – Polimerasa ( (beta): nuclear, reparación del ADN, sin función correctora. – Polimerasa ) (gamma): mitocondrial, replicación del ADN, con función correctora. – Polimerasa * (delta): nuclear, replicación del ADN, con función correctora. – Polimerasa + (epsilon): nuclear, reparación del ADN (?),con función correctora. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 58 Replicón eucariótico • Cada cromosoma eucariótico es una doble cadena linear de ADN. • Existen varios orígenes de replicación (no simultáneos). • La región de replicación se denomina replicón o unidad de replicación. – El replicón se mueve de forma más lenta que en procariotas. – Los fragmentos de Okazaki son más pequeños (100-150 pb). Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 59 Orígenes de replicación • En levaduras se han identificado secuencias específicas que parecen conferir la habilidad de replicación autónoma (secuencias de replicación autónoma o ARSs). • Se cree que por lo menos algunas de estas secuencias corresponden con orígenes de replicación. • En eucariotas más complejos los orígenes de replicación no están bien definidos. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 60 Replicación de los telómeros (I) • Al eliminar los cebadores de los extremos, los huecos no pueden ser cebados por otros fragmentos => los cromosomas se acortaría en cada ronda de replicación. • Secuencias repetidas en tándem en los telómeros. • La telomerasa (transcriptasa reversa), que contiene proteínas y ARN complementario a la repetición terminal, añade repeticiones en el extremo del cromosoma, rellenando el hueco dejado por el cebador. • El resultado de la acción de la telomerasa es el alargamiento del cromosoma. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 61 Replicación de los telómeros (II) Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 62 Ensamblaje del nuevo ADN en nucleosomas • La replicación está coordinada con la formación de nuevos nucleosomas. • Se requiere la formación de nuevas histonas. • Los nucleosomas se forman casi de forma inmediata durante la replicación. Figura. Ensamblaje de nucleosomas. Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 63 Replicación del ADN mitocondrial • Modelo del lazo de desplazamiento o lazo D. • Hélice ligera (L) y pesada (H). • Primero se inicia la replicación unidireccional de la nueva hélice L. • Cuando se ha sintetizado, unos 2/3 de la hélice L, comienza la síntesis de la nueva hélice H, en otro origen y en una sola dirección, opuesta a la de síntesis de la hélice L. Figura. Replicación del ADN mitocondrial Genética CC Mar 2005/6 • D. Posada, Universidad de Vigo 64