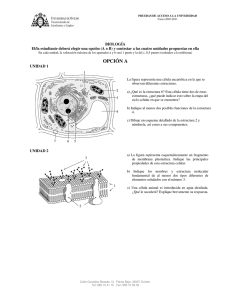
segmento del adn del herpevirus de la enfermedad de marek
Anuncio

k OFICINA ESPAÑOLA DE PATENTES Y MARCAS 19 k kInt. Cl. : C12N 15/38 11 Número de publicación: 2 110 999 6 51 ESPAÑA C07K 14/055 C12N 7/04 A61K 39/255 C12N 15/86 C12Q 1/70 k TRADUCCION DE PATENTE EUROPEA 12 kNúmero de solicitud europea: 91915865.9 kFecha de presentación : 19.08.91 kNúmero de publicación de la solicitud: 0 502 146 kFecha de publicación de la solicitud: 09.09.92 T3 86 86 87 87 k 54 Tı́tulo: Segmento del ADN del Herpesvirus de la Enfermedad de Marek que codifica la glicoproteı́na gE. k 30 Prioridad: 24.08.90 US 572711 25.07.91 US 736335 238 Administration Building East Lansing, Michigan 48824, US k 72 Inventor/es: Velicer, Leland F.; k 74 Agente: Ungrı́a López, Javier 45 Fecha de la publicación de la mención BOPI: 01.03.98 ES 2 110 999 T3 k 73 Titular/es: Michigan State University 45 Fecha de la publicación del folleto de patente: 01.03.98 Aviso: k Brunovskis, Peter y Coussens, Paul M. k En el plazo de nueve meses a contar desde la fecha de publicación en el Boletı́n europeo de patentes, de la mención de concesión de la patente europea, cualquier persona podrá oponerse ante la Oficina Europea de Patentes a la patente concedida. La oposición deberá formularse por escrito y estar motivada; sólo se considerará como formulada una vez que se haya realizado el pago de la tasa de oposición (art◦ 99.1 del Convenio sobre concesión de Patentes Europeas). Venta de fascı́culos: Oficina Española de Patentes y Marcas. C/Panamá, 1 – 28036 Madrid ES 2 110 999 T3 DESCRIPCION Antecedentes de la invención 5 10 15 (1) Campo de la invención La presente invención se refiere a segmentos del genoma del Herpesvirus de la Enfermedad de Marek, de su región corta única (US ) que codifica la glicoproteı́na gE y a nuevas glicoproteı́nas producidas a partir de ellos. En particular, la presente invención se refiere a segmentos de ADN que contienen los genes que codifican estos antı́genos glicoproteicos y que contienen secuencias promotoras potenciales hasta 400 nucleótidos 5’ de cada gen, segmentos que son útiles para sondar el herpesvirus de la enfermedad de Marek, como una fuente posible de promotores del virus de la enfermedad de Marek (MDV), para la expresión génica con el fin de producir las glicoproteı́nas que a su vez pueden ser utilizadas para producir anticuerpos que reconozcan los tres antı́genos glicoproteicos, y en el caso de los últimos dos genes, para sitios de inserción potenciales para genes foráneos y como sitios posibles para la inactivación génica con el fin de atenuar aislados de campo de MDV para fines de vacunación. (2) Técnica anterior 20 25 30 35 40 45 50 55 60 El MDV es un herpesvirus oncogénico de los pollos, que se sabe que causa linfomas de células T y desmielinación de los nervios periféricos. La enfermedad resultante, enfermedad de Marek (MD), fue el primer trastorno linfomatoso que acontece de forma natural que fue controlado eficazmente mediante vacunación, utilizando bien el herpesvirus de pavos (HVT) relacionado antigénicamente, además de apatogénico, o bien aislados de campo del MDV atenuados. Debido a las propiedades biológicas similares, especialmente a su linfotropismo, el MDV ha sido clasificado como un miembro de la subfamilia de los herpesvirus gamma (Roizman, B., y col., Intervirology 16:201-217 (1981)). De las tres subfamilias de los herpesvirus, los herpesvirus gamma exhiben diferencias particularmente acusadas con respecto a la composición y organización del genoma. Por ejemplo, el virus B-linfotrópico de Epstein-Barr (EBV) de los humanos tiene un genoma de 172,3 kpb con un contenido de G+C del 60%, está unido por repeticiones directas de 0,5 kpb terminales y contiene un conjunto caracterı́stico de repeticiones internas en tándem de 3,07 kpb (Baer, R., y col., Nature (London) 310:207-211 (1984)). El herpesvirus saimiri (HVS), un herpesvirus T-linfotrópico de los monos del nuevo mundo y de los vertebrados inferiores, tiene una secuencia codificadora rica en A+T (112 kpb; 36% de G+C; esto es ADN-L) sin ninguna redundancia interna a gran escala, pero en lugar de ello contiene más de 30 reiteraciones de una secuencia de 1,44 kpb con un 71% de G+C en los extremos del genoma (ADN-H) (Banker, A.T., y col., J. Virol. 55:133-139 (1985)). A pesar de las diferencias estructurales entre EBV y HVS, los genomas de estos dos virus codifican proteı́nas serológicamente relacionadas y comparten una organización común de secuencias codificadoras que difiere de la de los herpesvirus alfa neutrópicos, ejemplificados por el virus del herpes simple (HSV) y por el virus de la varicela-zóster (VZV) (Camerion, K.R., y col., J. Virol. 61:2063-2070 (1987); Davison, A.J., y col., J. Gen. Virol. 68:1067-1079 (1987); Davison, A.J., y col., J. Gen. Virol. 67:597-611 (1986); Davison, A.J., y col., J. Gen. Virol. 76:1759-1816 (1986); Davison, A.J., y col., J. Gen. Virol. 64:1927-1942 (1983); Gompels, U.A., J. of Virol. 62:757-767 (1988); y Nichols, J., y col., J. of Virol. 62:3250-3257 (1988)). En contraste con otros herpesvirus gamma, el MDV tiene una estructura genómica que se asemeja estrechamente a la de los herpesvirus alfa (Cebrian, J., y col., Proc. Natl. Acad. Sci. USA 79:555-558 (1982); y Fukuchi, K., y col., J. Virol. 51:102-109 (1984)). Los miembros de esta última subfamilia tienen estructuras genómicas similares que constan de segmentos largos (L) y cortos (S) unidos covalentemente. Cada segmento contiene un único (U) segmento (UL , US ) flanqueado por un par (terminales e interna) de regiones repetidas invertidas (TRL , IRL ; TRS ; respectivamente). Los herpesvirus alfa incluyen los HSV y VZV humanos, el virus de la pseudorrabia porcina (PRV), el herpesvirus bovino (BHV) y el herpesvirus equino (EHV). Debido a que el MDV contiene extensas secuencias repetidas que flanquean su región UL su estructura genómica se asemeja mucho a la del HSV (Cebrian, J., y col., Proc. Natl. Acad. Sci. USA 79:555-558 (1982); y Fukuchi, K., y col., J. Virol. 51:102-109 (1984)). Estudios recientes (Buckmaster, A.E., y col., J. Gen. Virol. 69:2033-2042 (1988)) han mostrado que los dos herpesvirus gamma, MDV y HVT, parecen tener una similitud mayor con los herpesvirus alfa, VZV y HSV, que con el herpesvirus gamma, EBV. Esto se basaba en una comparación de numerosos clones de MDV y HVT aislados aleatoriamente a nivel de los aminoácidos pronosticados; las secuencias individuales no sólo mostraban una mayor afinidad con los genes de los herpesvirus alfa que con los genes de los herpesvirus gamma, sino que se encontró que los dos genomas vı́ricos eran generalmente colineales con VZV, al menos con respecto a la región larga única (UL ). Tal colinealidad de los genes 2 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 de UL se extiende a otros herpesvirus alfa tales como HSV-1, HSV-2, EHV-1 y PRV según se evidenció por el análisis de las secuencias (McGeoch, D.J., y col., J. Gen. Virol. 69:1531-1574 (1988)) y por los experimentos de hibridación de ADN (Davison, A.J., y col., J. Gen. Virol. 64:1927-1942 (1983)). Muchos de estos genes de UL son compartidos por otros herpesvirus, incluyendo los herpesvirus beta y gamma (Davison, A.J., y col., J. Gen. Virol. 68:1067-1079 (1987)). La organización y comparación de tales genes ha sugerido el acontecimiento pasado de reordenaciones a gran escala para explicar la divergencia de los herpesvirus a partir de un antepasado común. Desgraciadamente, tal hipótesis no explica la presencia de los genes del componente S de los herpesvirus alfa (corto único, US , y corto con repeticiones asociadas invertidas/terminales, IRS , TRS ) que parecen únicos de los miembros de esta subfamilia (Davison, A.J., y col., J. Gen. Virol. 68:1067-1079 (1987); Davison, A.J., y col., J. Gen. Virol. 67:597-611 (1986); y McGeoch, D.J., y col., J. Mol. Biol. 181:1-13 (1985)). La secuencia de ADN y la organización de los genes en un fragmento de EcoR1 de 5,5 kpb que mapea en la región US de la cepa RBIB de MDV fue descrita por Ross, Binns y Pastorek (Ross, L.J.N., y col., Journal of General Virology 72:949-954 (1991)). Las propiedades y las relaciones evolutivas de cuatro de los polipéptidos pronosticados fueron también descritas (Ross, L.J.N. y M.M. Binns, Journal of General Virology 72:939-947 (1991)). En ese fragmento encontraron los homólogos de US2, US3, US6 (gD) y US7 (gI) del HSV, ası́ como un gen especı́fico del MDV. Para este último, sólo estaba presente parte del gen. Estos informes confirman la presencia de cuatro genes en US del MDV, y la relación evolutiva propuesta anteriormente. Es importante destacar que no se describió ninguna evidencia para US8 (gE), ni para los genes a la izquierda de US2. La región US de los herpesvirus alfa además de su unicidad comparada con los herpesvirus beta y gamma, es particularmente interesante debido a las notables diferencias en su contenido y organización genética dentro de esta última subfamilia (por ejemplo US de HSV-1=13,0 kpb, 12 genes, McGeoch, D.J., y col., J. Mol. Biol. 181:1-13 (1985); US de VZV=5,2 kpb, 4 genes, Davison, A.J., y col., J. Gen. Virol. 76:1759-1816 (1986)). En el caso de HSV-1, se ha encontrado que 11 de los 12 genes de US son prescindibles para la replicación en cultivo celular (Longnecker, R., y col., Proc. Natl. Acad. Sci. USA 84:4303-4307 (1987)). Esto ha sugerido la implicación potencial de estos genes en la patogénesis y/o en la latencia (Longnecker, R., y col., Proc. Natl. Acad. Sci. USA 84:4303-4307 (1987); Meignier, B., y col., Virology 162:251-254 (1988); y Weber, P.C., y col., Science 236:576-579 (1987)). En el informe de Buckmaster y col. (Buckmaster, A.E., y col., J. Gen. Virol. 69:2033-2042 (1988)), con la excepción de la identificación de secuencias parciales de MDV homólogas a la proteı́na alfa 22 (US1) temprana inmediata de HSV y la proteı́na quinasa de serina-treonina (US3), no se determinó el contenido, localización y organización de los homólogos del componente S del MDV. Además, a pesar de la presencia de al menos cuatro genes de glicoproteı́nas de US del HSV (dos en VZV), tales homólogos no fueron identificados. En la solicitud N◦ de Serie 07/229.011 registrada el 5 de Agosto de 1988, incluyendo a Leland F. Velicer, uno de los presentes inventores, el segmento de ADN del herpesvirus de la Enfermedad de Marek conteniendo posiblemente el gen que codifica el complejo antigénico B glicoproteico (gp100, gp60, gp49) fue identificado pero no secuenciado. El antı́geno B es un complejo glicoproteico importante porque puede producir al menos una inmunidad protectora parcial, y de este modo el segmento de ADN del MDV puede ser utilizado para sondas, como una fuente posible de promotores en la región reguladora 5’ del gen, y para la expresión génica con el fin de producir las glicoproteı́nas, que a su vez pueden ser utilizadas para producir anticuerpos que reconozcan los antı́genos glicoproteicos. Sin embargo, no habı́a discusión de las glicoproteı́nas de la presente invención. Estas glicoproteı́nas del antı́geno B no están codificadas por la región US y por tanto son de una región diferente del genoma del MDV. En la solicitud N◦ de Serie 07/526.790, registrada el 17 de Mayo de 1987 por Leland F. Velicer, el segmento de ADN del herpesvirus MDV conteniendo el gen que codifica el antı́geno A glicoproteico (gp57-65) está descrito pero no secuenciado. Este segmento de ADN del MDV es útil como sonda, como una fuente posible de promotores en la región reguladora 5’ del gen, y para producir anticuerpos por la secuencia de acontecimientos descrita anteriormente. Este ADN es también importante porque se sabe actualmente que el antı́geno A es un homólogo de gC del HSV, un gen no esencial para la replicación en cultivo celular. Debido a que esa propiedad se aplica también muy probablemente al homólogo del MDV, puede ser útil como un sitio para la inserción de genes foráneos. Sin embargo, no habı́a discusión de las glicoproteı́nas de la presente invención. Esta glicoproteı́na tampoco está codificada por la región US y es por tanto de una región diferente del genoma del MDV. Otras glicoproteı́nas están codificadas por el genoma del herpesvirus de la enfermedad de Marek. En la solicitud N◦ de serie 07/572.711, registrada el 24 de Agosto de 1990 por Leland F. Velicer y col., se describe el ADN del MDV que contiene los genes que codifican las glicoproteı́nas gD, gI y parte de gE 3 ES 2 110 999 T3 5 del MDV, con las secuencias de nucleótidos del MDV para los genes de gD y gI completos y para parte del gE (homólogos del MDV de los genes del HSV US6, US7, US8, respectivamente). Este segmento de ADN del MDV es útil como sonda, como una fuente posible de promotores en la región reguladora 5’ del gen, y para producir anticuerpos por la secuencia de acontecimientos descrita anteriormente. La presente invención está dirigida en particular al gen completo (US8) que codifica la glicoproteı́na gE. Objetos 10 15 Es un objeto de la presente invención proporcionar ADN secuenciado que codifica la glicoproteı́na gE. Es además un objeto de la presente invención proporcionar segmentos de ADN que codifican este antı́geno glicoproteico y que contienen secuencias promotoras potenciales hasta 400 nucleótidos 5’ del gen; los cuales son útiles como sondas de ADN, como una fuente posible de promotores del MDV, para producir anticuerpos que reconozcan el antı́geno y como sitios de inserción para genes foráneos y como sitios posibles para la inactivación génica con el fin de atenuar aislados de campo de MDV para fines de vacunación. Estos y otros objetos serán cada vez más aparentes por referencia a la descripción siguiente y a las figuras. En las figuras 20 Las Figuras 1A a C muestran la localización en el mapa, la estrategia de secuenciación y la organización de los marcos de lectura abierta (ORFs) del MDV: La Figura 1A incluye la estructura genómica del MDV y los mapas de restricción que describen el área secuenciada. 25 30 35 40 45 50 La Figura 1B incluye la localización en el mapa y la estrategia de secuenciación. Los recuadros definen los clones plasmı́dicos con insertos unidos a BamHI, EcoRI o SalI que fueron utilizados para generar los moldes de M13mp18 y 19 para la secuenciación del ADN. Las flechas hacia la derecha y hacia la izquierda definen secuencias derivadas de la hebra superior e inferior, respectivamente. Los sitios de los enzimas de restricción están identificados como: B = BamHI, E = EcoRI, Nc = NcoI, Ns = NsiI, S = SalI y P = PstI. Las secuencias derivadas de bibliotecas aleatorias (Sau3A, TaqI, RsaI), los fragmentos de restricción clonados especı́ficos, las bibliotecas digeridas con Bal 31 o utilizando oligonucleótidos derivados sintéticamente están indicados por a, b, c y d, respectivamente. La Figura 1C incluye la organización de los ORFs de US de MDV. Los números se refieren a homólogos sobre la base de la relación con la nomenclatura de los ORFs de US del HSV-1 (McGeoch, D.J., y col., J. Mol. Biol. 181:1-13 (1985)). Los recuadros representan la situación de los ORFs del MDV. Las flechas definen la dirección de la transcripción/traducción. Los nombres de los ORFs se muestran sobre los recuadros. Las señales de poliadenilación potencial en las hebras superior e inferior están resaltadas por AATAAA y AAATAA, respectivamente. SORF1 y SORF2 son ORFs del componente S especı́fico del MDV dados nombres arbitrarios. La Figura 2 muestra la secuencia de nucleótidos y de aminoácidos pronosticada. La secuencia de nucleótidos se da como la hebra 5’ a 3’ hacia la derecha solamente (numerada de 1 a 10350). Las secuencias de aminoácidos pronosticadas dirigidas hacia la derecha y hacia la izquierda se muestran por encima y por debajo de las secuencias de nucleótidos correspondientes en el código de una sola letra, respectivamente. El nombre de cada ORF se da a la izquierda de la primera lı́nea de la secuencia de aminoácidos. Las secuencias de aminoácidos están numeradas desde la primera M (código de tres letras) (ATG en el ADN) en el extremo N hasta el último aminoácido en el extremo C, que precede al codon de terminación (identificado por un ∗). Los sitios potenciales de consenso TATA situados dentro de los 400 nucleótidos del ATG están subrayados y definidos como sitios que contienen al menos seis de siete similitudes con las secuencias consenso TATA(AT)A(AT) definidas por Corden y col. (Corden, B., y col., Science 209:1406-1414 (1980)). Los subrayados más largos de siete nucleótidos se refieren a áreas que contienen sitios de consenso TATA que solapan. 55 60 La Figura 3A muestra la alineación de los homólogos del componente S mostrando regiones seleccionadas que exhiben una conservación de aminoácidos máxima. Los huecos han sido introducidos para maximizar la alineación de aminoácidos idénticos según se describe en Métodos. La secuencia consenso (cons) indica los residuos que son compartidos por al menos todos los virus excepto uno y están indicadas por letras mayúsculas. En alineaciones entre más de dos secuencias, los asteriscos (∗) indican los residuos conservados por todos los virus. Los números de los aminoácidos (con respecto a 5’-ATG) de las regiones alineadas correspondientes están listados antes y después de cada secuencia. 4 ES 2 110 999 T3 5 10 15 20 25 La Figura 3B muestra los análisis de matrices de puntos que representan las homologı́as globales entre las comparaciones de homólogos del segmento S de MDV-herpesvirus alfa seleccionadas. Se generaban puntos donde se encontraban al menos 15 aminoácidos idénticos o similares sobre una longitud de ventana móvil de 30. Las diagonales resultantes ilustran las regiones que muestran la mayor conservación. Los números de los aminoácidos (con respecto a 5’-ATG) de las secuencias correspondientes se representan encima y a la derecha de cada gráfica. La Figura 4 muestra una comparación de la organización genómica global de los ORFs del componente S disponibles (Audonnet, J.-C., y col., J. Gen. Virol. 71:2969-2978 (1990); McGeoch, D.J., y col., J. Gen. Virol. 68:19-38 (1987); Tikoo, S.K., y col., J. Virol. 64:5132-5142 (1990); Van Zijl, M., y col., J. Gen. Virol. 71:1747-1755 (1990); Zhang, G., y col., J. Gen. Virol. 71:2433-2441 (1990); Cullinane, A.A., y col., J. Gen. Virol. 69:1575-1590 (1988); Davison, A.J., y col., J. Gen. Virol. 76:1759-1816 (1986); McGeoch, D.J., y col., J. Mol. Biol. 181:1-13 (1985); Petrovskis, E.A., y col., Virology 159:193-195 (1987); Petrovskis, E.A., y col., J. Virol. 60:185-193 (1986); y Petrovskis, E.A., y col., J. Virol. 59:216223 (1986)). Los números encima de cada ORF se refieren a homólogos sobre la base de la relación con la nomenclatura de los ORFs de US del HSV-1 (McGeoch, D.J., y col., J. Mol. Biol. 181:1-13 (1985)). Las denominaciones de polipéptidos alternativos comunes a cada sistema están listadas debajo de aquellos ORFs en los que era aplicable. Las barras sólidas de caja alta y caja baja se refieren a los ORFs dirigidos hacia la derecha y hacia la izquierda, respectivamente. La flechas se refieren a sitios de unión IRS -US y/o US -TRS identificados. El gen 4 del PRV es homólogo al US4 del HSV-2, en lugar de al US4 del HSV-1. La Figura 5 muestra la secuencia de etapas necesarias para producir un segmento completo de ADN del herpesvirus de la enfermedad de Marek que codifica la glicoproteı́na gI y la parte de gE incluida en la solicitud registrada el 24 de Agosto de 1990. Descripción general 30 35 La presente invención se refiere a un segmento de ADN que codifica el precursor de la glicoproteı́na gE entre la secuencia de nucleótidos 8488 y 9978 del ADN del herpesvirus de la enfermedad de Marek, y que contiene secuencias promotoras potenciales hasta 400 nucleótidos 5’ de cada gen, según se muestra en la Figura 2 (y se identifica como parte de la SEC ID N◦ :1) y a subfragmentos del ADN que reconocen el ADN. Además, la presente invención se refiere a los nuevos precursores de glicoproteı́nas que son producidos por expresiones de los genes de los segmentos de ADN. Además la presente invención se refiere a los promotores génicos potenciales del MDV, que están localizados en los 400 nucleótidos 5’ de cada secuencia codificadora. 40 Descripción especı́fica 45 La presente invención muestra un análisis de la secuencia de un tramo de ADN de 10,35 kpb que abarca la mayorı́a de la región US del MDV. En total se identificaron siete homólogos de US del MDV, incluyendo tres genes de glicoproteı́nas y dos marcos de lectura abierta especı́ficos del MDV adicionales. Ejemplo 1 Materiales y métodos 50 Plásmidos Recombinantes, subclonaje de M-13 y secuenciación del ADN 55 60 EcoR1-0 y EcoR1-I de MDV de la cepa patógena GA fueron clonados previamente en pBR328 (Gibbs, C.P., y col., Proc. Natl. Acad. Sci. USA 81:3365-3369 (1984)), (Silva, R.F., y col., J. Virol. 54:690-696 (1985)) y se hicieron disponibles por R.F. Silva, USDA Avian Disease and Oncology Lab, East Lansing, MI, donde se mantienen estos clones. BamHI-A y BamHI-P1 de la cepa GA fueron clonados previamente en pACYC184 y pBR322, respectivamente (Fukuchi, K., y col., J. Virol. 51:102-109 (1984)) y suministrados amablemente por M. Nonoyama, Showa University Research Institute, St. Petersburg, FL. El clon GA-02 de la cepa GA, un clon de EMBL-3 conteniendo un inserto de SalI de MDV parcialmente digerido, que contiene BamHI-A, -P1, y secuencias flanqueantes 5’ y 3’ adicionales (proporcionado amablemente por P. Sondermeier, Intervet Intl. B. V., Boxmeer, Paı́ses Bajos) fue utilizado para extender al análisis hacia la derecha de los fragmentos de EcoR1 y BamH1 anteriores. Este clon del fago fue utilizado para 5 ES 2 110 999 T3 5 10 15 20 25 30 generar subclones de pUC18 con insertos unidos a SalI más pequeños (pSP18-A, pSP18-B y pSP18-C) que contienen la región 3’ flanqueante de BamHI-P1. Estos clones (Figura 1B) fueron utilizados para generar subclones de M13mp18 y -19 para utilización como moldes para la secuenciación de nucleótidos. Se realizaron preparaciones de plásmidos a pequeña y a gran escala utilizando el procedimiento de lisis alcalina (Maniatis, T., y col., Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory, Cold Spring Harbor, New York (1982)). Los subclones de los fagos M13mp18 y M13mp19 para ser utilizados como moldes para la secuenciación fueron generados utilizando subfragmentos de restricción especı́ficos determinados por mapeo de restricción o por la utilización de conjuntos de ADN vı́rico digeridos con Sau3A, Taq I o RsaI ligados en los sitios únicos de BamHI, AccI o SmaI del ADN de M13 RF, respectivamente. En algunos casos se obtuvieron clones por deleción de M13 que solapaban por digestiones preparativas con Bal31 de los sitios de restricción de AccI, NaeI o NsiI en EcoR1-0 por el método de Poncz y col. (Poncz, M., y col., Proc. Natl. Acad. Sci. USA 79:4298-4302 (1982)). Se utilizaron métodos estándar (Maniatis, T., y col., Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory, Cold Spring Harbor, New York (1982)) para las digestiones de restricción, electroforesis en gel, purificación de los fragmentos de ADN a partir de geles de agarosa, ligaduras y rellenado de los salientes en 5’ con el fragmento Klenow. Los productos de M13 ligados fueron transformados en células huésped JM107 competentes con CaCl2 y se añadieron a una superficie de agar B fundido conteniendo 10 l de IPTG 100 mM, 50 l de X-gal al 2% y 200 l de un cultivo fresco de JM101 de una noche. Estos contenidos se plaquearon posteriormente en placas de agar B y se incubaron a 37◦C durante una noche. Las placas recombinantes (claras) se utilizaron luego para infectar 5 ml de medio YT diluido 1:50 con un cultivo de una noche de JM101 y se rotaron a 37◦ C durante 6 horas. Las células resultantes fueron aglutinadas por centrifugación durante 5 minutos a temperatura ambiente y se retiraron los sobrenadantes y se almacenaron a 4◦C para conservar cepas vı́ricas de cada clon recombinante. Utilizando los sobrenadantes recuperados, se aisló ADN del fago M13 de hebra sencilla para ser utilizado como molde en la secuenciación de ADN por el método de la terminación de la cadena didesoxi según las instrucciones del Manual de Instrucciones para el Clonaje/Secuenciación con Didesoxi de M13 suministrado por Bethesda Research Laboratories. Los fagos M13mp recombinantes fueron posteriormente seleccionados mediante electroforesis de ADN vı́rico de hebra sencilla purificado en minigeles de agarosa al 1% y seleccionando aquellos moldes que mostraban una movilidad reducida en comparación con el ADN control de M13mp18 de hebra sencilla. 35 40 45 50 55 La secuenciación del ADN con moldes de M13 de hebra sencilla fue realizada por el método de la terminación de la cadena didesoxi (Sanger, F.S., y col., Proc. Natl. Acad. Sci. USA 74:5463-5467 (1977)) empleando la ADN polimerasa T7 modificada, SequenaseTM (United States Biochemical Corp., Cleveland, Ohio). Un resumen de la estrategia para la secuenciación está incluido en la Figura 1B. Para las reacciones de secuenciación del ADN, se emplearon las instrucciones especı́ficas etapa por etapa proporcionadas con el kit para secuenciación con SequenaseTM . Brevemente, los moldes de M13 de hebra sencilla fueron unidos primero con el cebador oligonucleotı́dico sintético universal de M13 por incubación a 65◦ C durante 2 minutos seguido por enfriamiento lento hasta que la temperatura de incubación estaba por debajo de 30◦C. Después de la adición de las mezclas apropiadas de desoxi y didesoxinucleótidos trifosfato (dNTPs y ddNTPs, respectivamente), de desoxiadenosina 5’-(alfa-tio) trifosfato marcada radioactivamente (35 SdATP, 1000-1500 Ci/mmol; NEN-DuPont) y del enzima SequenaseTM , se inició la sı́ntesis de las hebras complementarias marcadas radioactivamente a partir del cebador fijado. Cuatro reacciones de sı́ntesis separadas se terminaron cada una por la incorporación del ddNTP especı́fico (ddATP, ddGTP, ddTTP o ddCTP) utilizado en cada tubo. Los productos de reacción fueron sometidos a electroforesis a través de geles de poliacrilamida 7%/urea 50%/Tris-Borato-EDTA y las cadenas marcadas fueron visualizadas por autorradiografı́a. Ambas hebras fueron secuenciadas al menos una vez. Esto fue facilitado por la utilización de 16 oligonucleótidos 17-meros sintéticos generados sobre la base de secuencias determinadas previamente y sustituyeron al cebador universal bajo condiciones de reacción similares a las anteriores (0,5 pmoles/reacción) de acuerdo con la aproximación general descrita por Strauss (Strauss, E.C., y col., Anal. Biochem. 154:353-360 (1986)). Análisis de los datos de las secuencias 60 Las secuencias fueron ensambladas y analizadas en un microordenador personal IBM System 2/Modelo 50 utilizando los paquetes de programas para análisis de secuencias IBI/Pustell (Pustell, J., y col., Nucl. Acids Res. 14:479-488 (1986)) y Genepro (Versión 4.10; Riverside Scientific Enterprises, Seattle, WA) o programas obtenidos del University of Wisconsin Genetics Computer Group (GCG; Devereaux, J., 6 ES 2 110 999 T3 5 10 15 20 25 y col., Nucl. Acids Res. 12:387-395 (1984)) y ejecutados en un miniordenador VAX 8650. Las búsquedas de las bases de datos de la National Biochemical Research Foundation-Protein (NBRF-Protein, Emisión 21,0, 6/89) se realizaron con el programa FASTA del GCG (Pearson, W.R., y col., Proc. Natl. Acad. Sci. USA 85:2444-2448 (1988)) que utiliza: (1) una modificación del algoritmo de Wilbur y Lipman (Wilbur, W.J., y col., Proc. Natl. Acad. Sci. USA 80:726-730 (1983)) para localizar las regiones de similitud; (2) un sistema de puntuación basado en PAM250 (Dayhoff, M.O., y col., p. 345-352. En M.O. Dayhoff (ed.), Atlas of protein sequence and structure, vol. 5, Supl. 3. National Biomedical Research Foundation, Washington, D.C. (1978)) y (3) el procedimiento de alineación de Smith y Waterman (Smith, T.F., y col., Adv. Appl. Mathematics 2:482-489 (1981)) para unir, cuando fuera posible, las regiones con la mayor puntuación que no solapaban con el fin de derivar una alineación y su puntuación optimizada resultante. Se generaron representaciones gráficas de homologı́a de matrices de puntos utilizando el programa DOTPLOT del GCG con el archivo de salida del COMPARE del GCG. Este último crea un archivo de los puntos de similitud entre dos secuencias de aminoácidos pronosticadas para las que se eligieron una longitud de ventana de 30 y una severidad de 15 (en las que las sustituciones de aminoácidos conservadoras son puntuadas como positivas). Utilizando el programa GAP del GCG, se alinearon secuencias de aminoácidos especı́ficas utilizando el algoritmo de Needleman y Wunsch (Needleman, S.B., y col., J. Mol. Biol. 48:443-453 (1970)); después de la inserción de huecos (para maximizar el número de coincidencias) se determinó el porcentaje de residuos de aminoácidos idénticos y similares. Con el fin de crear alineaciones múltiples utilizando el GAP, se crearon archivos de salida de secuencias del MDV con huecos después de comparaciones sucesivas con el GAP entre la secuencia del MDV y sus secuencias homólogas (en orden de homologı́a descendente). Estos archivos de salida fueron utilizados como secuencias de entrada para ejecuciones posteriores del GAP hasta que la alineación de estas secuencias con huecos ya no pudo expandirse más por la adición de nuevos huecos. Después de la alineación, se mostraron los archivos de salida con huecos y se calculó una secuencia consenso utilizando el programa PRETTY del GCG. Con el fin de conseguir resultados óptimos, se empleó en algunos casos una edición manual (utilizando el LINEUP del GCG). Resultados 30 35 40 La secuencia de ADN de 10.350 nucleótidos presentada (Figura 2) parece abarcar la mayorı́a de la región corta única (US ) del genoma del MDV (GA). Un resumen de la estrategia de secuenciación está incluido en Materiales y Métodos y se representa en la Figura 1B. Esta secuencia abarca los fragmentos de US , EcoR1-0, EcoR1-I y se extiende hasta un sitio de SalI 1,55 kpb corriente abajo del extremo 3’ de BamHI-P1 (Figuras 1A y 1B). Fukuchi y col. (Fukuchi, K., y col., J. Virol. 51:102-109 (1984)) han mapeado previamente la unión IRS -US hasta un fragmento de Bgl I de 1,4 kb situado en el segundo de cinco subfragmentos de EcoR1 de BamHI-A (Figura 1B). De este modo, a la secuencia aquı́ presentada deberı́an faltarle entre 2,6 y 4,0 kb de la región US 5’-proximal, asumiendo que la situación de la unión IRS -US anterior puede ser confirmada independientemente. Debido a que la región secuenciada no se extiende a una distancia suficiente corriente abajo de BamHI-P1, la unión US -TRS del MDV no ha sido todavı́a definida con precisión (Davison, A.J., y col., J. Gen. Virol. 76:1759-1816 (1986)). Para VZV, EHV-4 y HSV-1, este lı́mite está situado alrededor de 100 pb corriente arriba, o 1,1 y 2,7 kb corriente abajo, respectivamente, del codon de terminación de sus homólogos de US8 respectivos (Cullinane, A.A., y col., J. Gen. Virol. 69:1575-1590 (1988); Davison, A.J., y col., J. Gen. Virol. 76:1759-1816 (1986) y McGeoch, D.J., y col., J. Gen. Virol., 69:1531-1574 (1988)). 45 50 55 60 Se encontró que el contenido global de G+C de la región secuenciada era del 41%, algo inferior a los valores de G+C genómicos del MDV del 46% (Lee, L.F., y col., J. Virol. 7:289 (1971)). Las frecuencias observadas de dinucleótidos CpG en la secuencia completa, o en las regiones codificadoras solamente, no diferı́a significativamente de las esperadas a partir de sus composiciones de mononucleótidos (datos no mostrados). Este resultado concuerda con los obtenidos a partir de los herpesvirus alfa, mientras que contrasta con los obtenidos de los herpesvirus gamma, tales como el HVS rico en A+T y el EBV rico en G+C, que son ambos deficientes en dinucleótidos CpG (Honess, R.W., y col., J. Gen. Virol. 70:837-855 (1989)). La región secuenciada contiene 9 ORFs completos para codificar probablemente proteı́nas (Fig. 1C, las bases para los nombres se dan posteriormente). Esta predicción estaba basada en: (1) comparaciones de la homologı́a y de la organización posicional con otros genes de los herpesvirus alfa y (2) presencia de TATA y de secuencias consenso de poliadenilación potenciales (Birnstiel, M.L., y col., Cell 41:349-359 (1985) y Corden, B., y col., Science 209:1406-1414 (1980)) y (3) posesión de contextos favorables para la iniciación de la traducción (Kozak, M., J. Cell. Biol. 108:229-241 (1989)). Esta identificación estaba guiada además por la observación de que los herpesvirus alfa tales como HSV y VZV tienden a contener regiones codificadoras relativamente estrechamente empaquetadas, no ayustadas y generalmente que no 7 ES 2 110 999 T3 5 10 15 20 25 solapan (Davison, A.J., y col., J. Gen. Virol. 76:1759-1816 (1986); McGeoch, D.J., y col., J. Gen. Virol. 69:1531-1574 (1988); McGeoch, D.J., y col., J. Mol. Biol. 181:1-13 (1985) y McGeoch, D.J., y col., J. Gen. Virol. 68:19-38 (1987)). Tales genes, especialmente los de las regiones US , comparten a menudo señales de poliadenilación, dando lugar por ello a familias de ARNm 3’-coterminales (Rixon, F.J., y col., Nucl. Acids Res. 13:953-973 (1985)). Los métodos para detectar las regiones que codifican proteı́nas sobre la base de la utilización de tablas de frecuencia de codones derivadas de MDV (utilizando éstas y secuencias del MDV publicadas previamente, Binns, M.M., y col., Virus res. 12:371-382 (1989); Ross, L.J.N., y col., J. Gen. Virol. 70:1789-1804 (1989) y Scott, S.D., y col., J. Gen. Virol. 70:3055-3065 (1989)) o el análisis de la predisposición composicional (utilizando los programas CODONPREFERENCE y TESTCODE del GCG) fueron muy poco concluyentes, sugiriendo que el MDV posee predisposiciones de codones y composicionales relativamente bajas en comparación con las pronosticadas sobre la base de su composición de mononucleótidos. Sin embargo, utilizando el programa FRAMES del GCG, junto con la tabla anterior de frecuencia de codones derivada del MDV, los 9 ORFs identificados muestran claramente un patrón significativamente bajo de utilización de codones poco comunes, que contrasta intensamente con el observado en todas las otras regiones potencialmente traducibles (datos no mostrados). Las secuencias de aminoácidos pronosticadas de los ORFs pronosticados (comenzando a partir del primer codon ATG) están mostradas con relación a la secuencia de nucleótidos de la Figura 2. Los sitios TATA potenciales dentro de los 400 nucleótidos del codon de iniciación están subrayados. Los ORFs propuestos y las situaciones de las señales de poliadenilación potenciales, la identificación de los nucleótidos del contexto de ATG -3, +4 (Kozak, M., J. Cell Biol. 108:229-241 (1989)), ası́ como las longitudes, las masas moleculares relativas y los puntos isoeléctricos pronosticados de los productos traduccionales pronosticados se muestran en la Tabla 1. Un resumen de los datos del MDV se muestra en la Tabla 1, con la situación de los ORFs, las señales de poliadenilación pronosticadas utilizadas, los nucleótidos del contexto traduccional, las longitudes, los tamaños moleculares relativos y los puntos isoeléctricos de los productos de la traducción pronosticados. TABLA 1 30 Tamañob Sitio de Nucleótidos Molecular Inicio Final Poliadenilación del Contexto Longitud Pronosticado pIc del ORF del ORF Pronosticado de ATG -3, +4 (aa) (kDa) Pronosticado a 35 Nombre US1 US10 SORF1 US2 US3 SORF2 US6 US7 US8 40 45 248 1077 2884 3923 4062 5353 5964 7282 8488 784 1715 1832 3114 5240 5793 7172 8346 9978 1777 1777 1790 1790 5394 5904 10040 10040 10040 A,A G,G A,A A,G A,G C,G G,G G,T A,T 179 213 351 270 393 147 403 355 497 20,4 23,6 40,6 29,7 43,8 16,7 42,6d 38,3d 53,7d 6,5 8,2 8,2 7,6 6,1 9,8 10,3d 6,7d 8,0d a 50 55 60 Nucleótidos enumerados con relación a las posiciones -3, +4, respectivamente; la numeración comienza con la A del codon ATG (AUG) como posición +1; a los nucleótidos 5’ a ese sitio se les asignan números negativos. b En ausencia de modificaciones post-traduccionales. c Calculado utilizando el programa del GCG, ISOELECTRIC. d Basado en las secuencias que siguen al sitio de corte del péptido señal pronosticado. En ausencia de información previa concerniente a estos ORFs del MDV, y para simplificar la identificación, han sido nombrados (Figura 1C, Tabla 1) sobre la base de las relaciones homólogas con los ORFs de US codificados por HSV-1 (McGeoch, D.J., y col., J. Mol. Biol. 181:1-13 (1985)). Cuando sea apropiado, las letras MDV precederán al nombre del homólogo para indicar el origen del ORF. Los dos ORFs especı́ficos del MDV han sido denominados arbitrariamente SORF1 y SORF2, sobre la base de su situación en el componente S. 8 ES 2 110 999 T3 5 10 15 20 25 30 De acuerdo con el modelo de barrido para la traducción, la subunidad ribosómica de 40S se une inicialmente al extremo 5’ del ARNm y luego migra, deteniéndose en el primer codon AUG (ATG) en un contexto favorable para iniciar la traducción (Kozak, M., J. Cell Biol. 108:229-241 (1989)). Sin embargo, en ausencia de la nucleasa S1 y/o del análisis de extensión de los cebadores, los sitios de inicio de la traducción definitivos no pueden ser pronosticados con exactitud. Sin embargo, los sitios de iniciación probables están enumerados en la Tabla 1; éstos se refieren a la situación del primer codon ATG dentro del marco encontrado en el marco de lectura abierta principal. Según Kozak (Kozak, M., J. Cell Biol. 108:229-241 (1989)), mientras haya una purina en la posición -3, las desviaciones del resto del consenso sólo perjudican marginalmente la iniciación. En ausencia de tal purina, sin embargo, una guanina en la posición +4 es esencial para una traducción eficaz. La Tabla 1 muestra que todos los ORFs, excepto el SORF2, contienen el importante residuo de purina en la posición -3. No obstante, en el caso de SORF2, una guanina compensadora en la posición +4 está en efecto presente. En el caso del US1 del MDV, dos sitios cap transcripcionales han sido identificados tentativamente por análisis de protección a la nucleasa S1 en 5’ (datos no mostrados). Estos sitios parecen estar situados 18 y 25 nucleótidos corriente abajo de una secuencia TATATAA en la posición 200 y 207, respectivamente (Figura 2). Sobre la base de los datos de S1 en 3’, este transcrito utiliza una señal de poliadenilación situada justo corriente abajo de la región codificadora US10 (Tabla 1, datos no mostrados). Los análisis de transferencia Northern comparativos de la región US indican que el transcrito de US1 del MDV parece ser el transcrito más importante expresado a tiempos tardı́os (72 h) post-infección cuando se observan extensos efectos citopáticos (datos no mostrados). Los estudios de inhibición con ácido fosfonoacético han indicado que el US1 del MDV, en contraste con su equivalente US1 del HSV-1 inmediato temprano, está regulado como un gen de clase tardı́a (datos no mostrados). Utilizando el programa de ordenador FASTA (Pearson, W.R., y col., Proc. Natl. Acad. Sci. USA 85:2444-2448 (1988)) con un valor K-tuple de 1, cada una de las 9 secuencias de aminoácidos pronosticadas fue sometida a selección frente a la base de datos NBRF-Proteı́na (Emisión 21,0, 6/89), y frente a las secuencias génicas (11) del segmento S del EHV-4 recientemente publicadas. Se consideró que las puntuaciones del FASTA optimizadas mayores de 100 indicaban generalmente un grado significativo de similitud de aminoácidos. Los resultados de este análisis están en la Tabla 2. TABLA 2 Comparaciones emparejadas de los homólogos del componente S del MDV y de los herpesvirus alfa 35 US1 40 % Similares % idénticos 45 50 55 Puntuaciones del PASTA US10 Virus MDV HSV-1 VZV PRV EHV-4 MDV HSV-1 VZV EHV-4 MDV HSV-1 VZV PRV EHV-4 47/26 43/27 51/33 48/30 47/26 49/29 43/25 50/29 43/27 49/29 51/35 54/36 51/33 43/25 51/35 56/41 48/30 50/29 54/36 56/41 - 45/24 40/24 45/24 49/27 40/24 49/27 a a a 45/29 49/27 55/32 45/29 49/27 55/32 a - MDV HSV-1 VZV PRV EHV-4 891 101 160 218 208 101 2,047 119 201 150 160 218 119 201 1,378 340 340 1,724 359 525 208 150 359 525 1,308 1,071 134 147 a 251 134 1,617 123 a 180 147 123 978 a 191 251 180 191 a 1,312 Longitud (aa) 179 420 ∗273 213 312 180 259 278 60 9 364 ES 2 110 999 T3 TABLA 2 (Continuación) US2 5 % Similares % idénticos 10 Puntuaciones del PASTA 15 US3 MDV HSV-1 PRV MDV HSV-1 VZV PRP 51/33 a 48/26 a 51/33 a 50/31 a 48/26 50/31 a a 56/38 54/33 55/33 a 56/38 57/41 59/36 a 54/33 57/41 58/35 a 55/33 59/36 58/35 a 1,421 335 a ∗∗168 a 335 1,554 a 112 a ∗∗118 112 a 1,240 a 1,931 611 616 563 a 611 2,409 717 620 a 616 717 1,960 595 a 563 620 595 1,948 a 270 291 256 393 481 393 390 20 US6 25 % Similares % idénticos 30 35 40 Puntuaciones del PASTA US7 Virus MDV MSV-1 PRV EHV-1 BHV-1 MDV HSV-1 VZV PRV EHV-1 MDV HSV-1 VZV PRV EHV-1 BHV-1 42/21 b 44/23 43/21 42/33 MDV HSV-1 VZV PRV EHV-4 BHV-1 2,068 211 279 246 211 1,999 294 253 b b b b 279 294 2,116 428 246 253 428 1,995 291 304 730 494 Longitud (aa) 403 42/21 b 47/27 44/22 50/28 394 44/23 47/27 b 51/30 57/38 43/21 44/22 b 51/30 52/30 402 395 42/33 50/28 b 57/38 52/30 - 39/22 46/23 43/25 41/23 a 39/22 43/24 41/26 42/23 a 46/23 43/24 47/25 46/29 a 43/25 41/26 47/25 51/30 a 41/23 42/23 46/29 51/30 a 291 1,816 145 228 184 242 304 145 1,880 234 188 249 b 228 234 1,705 198 298 730 188 188 198 1,652 274 494 242 249 298 274 1,979 2,148 a a a a a 417 355 390 354 350 424 US8 MDV HSV-1 VZV PRV EHV-1 45 % Similares % idénticos 50 44/22 43/22 46/28 47/22 a 44/22 46/27 49/28 41/23 a 43/22 46/27 49/29 50/28 a 46/28 49/28 47/25 54/34 a 47/22 41/23 46/29 54/34 a 2,489 192 376 ∗∗243 399 192 2,751 357 257 274 Puntuaciones 376 357 3,171 329 468 del PASTA ∗∗217 257 329 2,923 417 399 274 468 417 2,821 a a a a a 55 497 60 10 550 623 577 552 ES 2 110 999 T3 a existencia de homólogos no determinada b 5 c la longitud real diferirá algo, debido a que el codon de iniciación probable no está definido d 10 15 20 25 30 35 40 45 50 55 60 no hay un homólogo presente en el genoma puntuación diferente cuando se invierte el orden de comparación Aunque SORF1 y SORF2 no parecen compartir ninguna homologı́a significativa con ninguna de las secuencias de la base de datos (datos no mostrados), aparte del US3 del MDV, se encontró que los otros seis ORFs (US1, 10, 2, 6, 7 y 8 del MDV; Tablas 1, 2) eran homólogos a los genes del segmento S de los herpesvirus alfa exclusivamente (Tabla 2). Debido a que el ORF de US3 representa un miembro de la superfamilia de las proteı́na quinasas de serina-treonina (Hanks, S.K., y col., Science 241:42- (1988)), se obtuvo un número de puntuaciones por encima de 150 relativamente grande. Sin embargo, estas puntuaciones fueron 3-4 veces más bajas que las obtenidas en comparaciones con los homólogos de US3 de HSV, PRV y VZV. Con el fin de comparar con las homologı́as del segmento S de los herpesvirus alfa previamente establecidas, se incluyen todas las comparaciones posibles del FASTA entre los siete grupos de secuencias relacionadas con los herpesvirus alfa. Se utilizó el programa GAP en comparaciones de parejas similares para generar alineaciones óptimas con el fin de determinar el porcentaje total de aminoácidos idénticos y similares compartidos por las dos secuencias. Según se muestra en la Tabla 2, las comparaciones de homologı́a entre los ORFs del segmento S del MDV y sus equivalentes de herpesvirus alfa fueron comparables a las observadas previamente entre los otros mismos homólogos del segmento S de los herpesvirus alfa. En algunos casos se encontró que los ORFs del MDV estaban más relacionados con los homólogos de los herpesvirus alfa que esos mismos homólogos lo estaban con sus otros equivalentes de los herpesvirus alfa (comparar las homologı́as del US1 de MDV/EHV-4 frente a las del HSV-1/EHV-4 y del US10 de MDV/EHV-4 frente a las del HSV-1/EHV-4). Además, a pesar del hecho de que el VZV carece de homólogos de US2 y US6, el MDV, aunque considerado formalmente un herpesvirus gamma, posee claramente homólogos de US2 y US6. Los resultados de las alineaciones múltiples limitadas para cada uno de los siete homólogos en las áreas que muestran la mejor conservación están representados en la Figura 3A. Las representaciones gráficas de homologı́a por matrices de puntos que representan las homologı́as totales entre las comparaciones de los homólogos del segmento S de MDV-herpesvirus alfa seleccionadas están incluidas en la Figura 3B. (Utilizando una longitud de ventana móvil de 30 aminoácidos, en la que se generan puntos cuando se encuentra que al menos 15 aminoácidos son idénticos o similares). Las diagonales resultantes ilustran las regiones que muestran la mayor conservación. Tales regiones incluyen y en algunos casos se extienden sobre las regiones representadas en la Figura 3A. Se hicieron intentos más sensibles para identificar otras proteı́nas relacionadas no detectadas con FASTA utilizando los programas PROFILE y PROFILESEARCH del GCG. La utilización de estos programas permite comparaciones con bases de datos que se basan en la información disponible a partir de estudios estructurales y, en este caso, de la información implı́cita en las alineaciones de los ORFs del componente S relacionados (incluyendo secuencias del MDV utilizando GAP) (Gribskov, M., y col., Proc. Natl. Acad. Sci. USA 84:4355-4358 (1987)); no obstante, tales análisis no se extendieron sobre los grupos de proteı́nas relacionadas aquı́ descritas. Se ha encontrado que los homólogos de las glicoproteı́nas de los herpesvirus contienen generalmente patrones similares de residuos de cisteı́na conservados. Al comparar los homólogos de gB de siete herpesvirus diferentes incluidos en las subclases de herpesvirus alfa, beta y gamma, existe una conservación completa de 10 residuos de cisteı́na (Ross, L.J.N., y col., J. Gen. Virol. 70:1789-1804 (1989)). El US6 (gD) del HSV-1 contiene 7 residuos de cisteı́na: seis parecen crı́ticos para un plegamiento correcto, para la estructura antigénica y para el grado de procesamiento del oligosacárido (Wilcox, W.C., y col., J. Virol. 62:1941-1947 (1988)). No sólo este mismo patrón general de cisteı́nas está conservado en los homólogos de gD del HSV-2 (McGeoch, D.J., y col., J. Gen. Virol. 68:19-38 (1987)) y del PRV (Petrovskis, E.A., y col., J. Virol. 59:216-223 (1986)), sino que se conserva también en el homólogo de gD del MDV (no se muestra la alineación completa). La Figura 3A representa las porciones de los patrones de conservación de cisteı́nas observados entre los homólogos de US6 (gD), US7 (gI) y US8 (gE) (en cuyo caso se muestran 4, 3 y 6 residuos de cisteı́na conservados, respectivamente). Mientras que los homólogos de US8 de MDV, VZV, PREV y EHV-1 (Audonnet, J.-C., y col., J. Gen. Virol. 71:2969-2978 (1990); Davison, A.J., y col., J. Gen. Virol. 76:1759-1816 (1986) y Petrovskis, E.A., y col., J. Virol. 60:185-193 (1986)) comparten todos un patrón similar de cuatro residuos de cisteı́na conservados próximos a sus extremos amino, los equivalentes de HSV-1 y -2 llevan sólo dos de éstos (McGeoch, D.J., J. Gen. Virol. 71:2361-2367 (1990); 11 ES 2 110 999 T3 5 10 15 datos no mostrados). Es bastante posible que el único patrón de cuatro cisteı́nas conservadas pudiera facilitar la formación de estructuras secundarias y terciarias diferentes que podrı́an transmitir consecuencias funcionales importantes. Éstas podrı́an estar reflejadas por hallazgos que muestran que la gE del HSV-1 tiene actividad de receptor Fc (Johnson, D.C., y col., J. Virol. 62:1347-1354 (1988)), mientras que sus equivalentes de PRV y VZV no la tienen (Edson, C.M., y col., Virology, 161:599-602 (1987) y Zuckerman, F.A., y col., J. Virol. 62:4622-4626 (1988)). Una inspección cuidadosa de las regiones N-terminales de los homólogos de gD, gI y gE del MDV ha revelado que contienen los tres bloques de construcción básicos de las secuencias peptı́dicas señal: una región N-terminal básica, cargada positivamente (región n), una región hidrofóbica central (región h) y una región terminal más polar (región c) que parece definir el sitio de corte (von Heijne, G., J. Mol. Biol. 184:99-105 (1985)). Utilizando un método recientemente mejorado para predecir los sitios de corte de las secuencias señal (von Heijne, G., Nucl. Acids Res. 14:4683-4690 (1986)), la Tabla 3 muestra la posición probable de estos sitios, la situación del dominio transmembrana hidrofóbico y del dominio citoplásmico cargado cerca del extremo C-terminal y la situación de los sitios potenciales de N-glicosilación. La Tabla 3 muestra los datos de las glicoproteı́nas de US del MDV sobre los sitios de corte del péptido señal pronosticados y las situaciones de los dominios transmembrana y citoplásmico y de los sitios potenciales de N-glicosilación (con respecto al codon de iniciación ATG). 20 TABLA 3 Nombre Sitio de Corte del Péptido Señal Pronosticado Dominio Transmembrana Dominio Citoplásmico Sitios de N-glicosilación US6 US7 US8 G30 -D31 S18 -I19 T18 -A19 358-374 269-288 394-419 375-403 289-355 420-497 87, 138, 230, 306 147, 167, 210, 245, 253 60, 133, 148, 203, 229, 277, 366, 388 25 30 35 40 45 50 55 60 Igual que los otros homólogos de gI, el equivalente del MDV contiene un dominio citoplásmico relativamente largo. Sin embargo, en contraste con los otros homólogos de gD, el péptido señal de gD del MDV contiene una región n relativamente larga (18 residuos), que está inusualmente muy cargada (+4; Figura 2) considerando un valor medio global de +1,7 entre los eucariotes, que generalmente no varı́a con la longitud (von Heijne, G., J. Mol. Biol. 184:99-105 (1985)). Aunque existe un codon de metionina más distal directamente antes del codon de iniciación (como en el homólogo de gD del PRV, Petrovskis, E.A., y col., J. Virol. 59:216-223 (1986)) el modelo de barrido para la traducción (Gribskov, M., y col., Proc. Natl. Acad. Sci. USA 84:4355-4358 (1987)) favorece la utilización del codon de iniciación más 5’-proximal (en la posición 5964, Figura 2). Un apoyo adicional está basado en un contexto de traducción global que parece al menos tan bueno como, si no mejor que, el correspondiente al ATG corriente abajo. A pesar de tal predicción, la situación de un sitio cap posible del ARNm entre los dos sitios ATG, que podrı́a imposibilitar tal predicción, no puede ser excluida en este punto. Un punto final concerniente a gD del MDV requiere mención. Utilizando la secuencia de ADN de 10.350 nucleótidos como sonda para someter a selección las bases de datos de ácidos nucleicos GenBank (62,0, 12/89) y EMBL (19,0, 5/89) con el programa de ordenador FASTA (K-tuple=6), una puntuación optimizada de 1027, correspondiente a una identidad de nucleótidos del 91,5% en un solapamiento de 342 pb entre las secuencias codificadoras de gD del MDV (6479-6814; aa#173-aa#284; Figura 2) y un segmento de ADN del MDV de 467 pb informado previamente (Wen, L.-T., y col., J. Virol. 62:3764-3771 (1988)). Se ha informado que esta última secuencia contiene un segmento de 60 pb protegido contra la digestión con ADNasa mediante la unión de un antı́geno nuclear del MDV (MDNA) de 28 kD expresado solamente en células linfoblastoides transformadas con MDV infectadas “latentemente”. En vista de las similitudes entre MDV y VZV, estos autores sugirieron que MDNA puede funcionar de una manera análoga a la de EBNA-1 en la inmortalización de células de primates. En su informe, Wen y col.(Wen, L.-T., y col., J. Virol. 62:3764-3771 (1988)) mapearon el sitio de unión del MDNA al mismo subfragmento de EcoRI de BamHI-A en el que está situado gD del MDV (EcoRI-I, Figura 1). Aunque nuestra secuencia que cubre esta región es consistente con un ORF completo, ininterrumpido que contiene todos los rasgos caracterı́sticos de una glicoproteı́na, y que muestra una homologı́a significativa con gD del HSV, su secuencia contiene alrededor de 140 bases de una secuencia 5’-proximal no relacionada con ninguna 12 ES 2 110 999 T3 determinada a partir de nuestro fragmento de EcoR1-I de 5,3 kpb o de sus secuencias colindantes de 3,5 kb. La secuencia de 327 pb restante (que contiene el supuesto sitio de unión del antı́geno nuclear) aunque se asemeja claramente a nuestra secuencia codificadora de gD, después de la traducción por ordenador fue incapaz de producir un ORF más largo de 30 aa. 5 Discusión 10 15 20 25 30 35 40 45 50 55 60 Datos recientes han mostrado que a pesar de la clasificación del MDV como un herpesvirus gamma, sobre la base de las propiedades linfotrópicas compartidas con otros miembros de esta subfamilia, su estructura genómica (Cebrian, J., y col., Proc. Natl. Acad. Sci. USA 79:555-558 (1982) y Fukuchi, K., y col., J. Virol. 51:102-109 (1984)) y la organización genética de su región UL principalmente (Buckmaster, A.E., y col., J. Gen. Virol. 69:2033-2042 (1988)) se asemeja más estrechamente a la de los herpesvirus alfa neurotrópicos. Además, en los casos en los que se encontraron secuencias polipeptı́dicas conservadas entre las tres subfamilias de herpesvirus (por ejemplo los genes UL ), se observaron consistentemente puntuaciones de homologı́a significativamente mayores frente a los equivalentes de los herpesvirus alfa que frente a los de los herpesvirus beta o gamma respectivos (Davison, A.J., y col., J. Gen. Virol. 67:597611 (1986); Buckmaster, A.E., y col., J. Gen. Virol. 69:2033-2042 (1988); Ross, L.J.N., y col., J. Gen. Virol. 70:1789-1804 (1989) y Scott, S.D., y col., J. Gen. Virol. 70:3055-3065 (1989)). Se ha encontrado previamente que los genes del segmento S de los herpesvirus alfa son únicos para los miembros de esta subfamilia taxonómica (Davison, A.J., y col., J. Gen. Virol. 68:1067-1079 (1987) y Davison, A.J., y col., J. Gen. Virol. 67:597-611 (1986)). La identificación de siete homólogos del MDV de genes del segmento S de los herpesvirus alfa en este estudio es consistente con la idea de que MDV comparte una relación evolutiva más estrecha con los herpesvirus alfa que con los herpesvirus gamma. Esto está apoyado además por el análisis de la frecuencia de dinucleótidos que es incapaz de mostrar una falta de la supresión de CpG como se observó entre todos los herpesvirus gamma hasta ahora ası́ estudiados (Efstathiou, S., y col., J. Gen. Virol. 71:1365-1372 (1990) y Honess, R.W., y col., J. Gen. Virol. 70:837-855 (1989)). La situación anterior se asemeja a una similar observada con el herpesvirus-6 humano (HHV-6), en cuyo caso su linfotropismo T sugirió una clasificación provisional como un herpesvirus gamma (Lopez, C., y col., J. Infect. Dis. 157:1271-1273 (1988)). Sin embargo, un análisis genético posterior ha mostrado una relación mayor entre el HHV-6 y el herpesvirus beta, citomegalovirus humano (HCMV; Lawrence, G.L., y col., J. Virol. 64:287-299 (1990)). Una comparación de la organización genética de los genes del segmento S de los herpesvirus alfa se presenta en la Figura 4. La organización de estos genes varı́a enormemente en algunos casos en la longitud, organización y grado de homologı́a globales. Sin embargo, las disposiciones génicas globales presentadas son consistentes con un modelo para justificar la divergencia de los herpesvirus alfa a partir de un antepasado común mediante varios acontecimientos de recombinación homóloga que tienen como resultado la expansión o contracción de las regiones repetidas invertidas y una pérdida o ganancia concomitante de gen(es) de US . En el caso de VZV, faltan seis homólogos del segmento S en comparación con HSV-1 (US2, US4, US5, US6, US11, US12). Algunos genes, tales como los homólogos de US1, muestran divergencias particulares en la secuencia y en la longitud. Comparados con HSV-1, los homólogos de US1 de MDV, VZV y EHV-4 carecen de aproximadamente 120 aa de secuencia comparable con la porción 5’-proximal de US1 del HSV-1 (alfa 22). Sobre la base del análisis de transferencia Northern, el análisis de protección a la nucleasa S1 y los estudios de inhibición con ácido fosfonoacético, en contraste con su equivalente del HSV-1 inmediato temprano relativamente no caracterizado, el gen US1 del MDV parece estar regulado como un gen de clase tardı́a abundantemente expresado (datos no mostrados). En contraste con los demás herpesvirus alfa, el MDV contiene dos ORFs aparentemente especı́ficos del MDV. Además, la región US del MDV parece contener de 2,6 a 4,0 kb aproximadamente de secuencias 5’-proximales adicionales. Sobre la base de una comparación de la Figura 4 y de la consideración del esquema de recombinación con expansión-contracción, parece probable que haya genes de US especı́ficos del MDV adicionales. Debido a que el MDV ha sido considerado mucho tiempo como un herpesvirus gamma, gran parte del trabajo previo que interpreta sus propiedades ha procedido por analogı́a con la asociación entre el EBV y las células B (Nonoyama, M., p. 333-341. En B. Roizman (ed.), The herpesviruses, vol. 1, Plenum Press (1982) y Wilbur, W.J., y col., Proc. Natl. Acad. Sci. USA 80:726-730 (1983)). Debido a una relación genética más estrecha con los herpesvirus alfa, y teniendo en cuenta el análisis anterior del HHV-6, nosotros estamos de acuerdo con Lawrence y col. (Lawrence, G.L., y col., J. Virol. 64:287-299 (1990)) en que es poco probable que las propiedades linfotrópicas de MDV y HVT estén determinadas por moléculas homólogas a EBV y en que una delimitación de las diferencias moleculares entre el MDV y los herpesvirus alfa neurotrópicos serı́a más provechosa para explicar las diferencias biológicas observadas que el empleo de analogı́as basadas en las propiedades de los herpesvirus gamma tales como EBV y HVS. 13 ES 2 110 999 T3 5 10 15 20 Para explicar tales diferencias, la región US de MDV puede ser particularmente importante. Con pocas excepciones, cada gen del componente L del HSV-1 posee un equivalente en VZV (McGeoch, D.J., y col., J. Gen. Virol. 69:1531-1574 (1988)); un número considerable de éstos están relacionados también con los genes de los herpesvirus beta y gamma (29 de 67 equivalentes del EBV con respecto a los genes de UL del VZV; Davison, A.J., y col., J. Gen. Virol. 68:1067-1079 (1987)). En contraste, los segmentos S de HSV-1 y de VZV difieren significativamente en tamaño y parecen estar entre las partes menos relacionadas de los dos genomas (Davison, A.J., y col., J. Gen. Virol. 67:597-611 (1986) y Davison, A.J., y col., J. Gen. Virol. 64:1927-1942 (1983)). Estudios recientes han mostrado que 11 de los 12 marcos de lectura abierta contenidos en el componente S del HSV-1 son prescindibles para el crecimiento en cultivo de células (Longnecker, R., y col., Proc. Natl. Acad. Sci. USA 84:4303-4307 (1987) y Weber, P.C., y col., Science 236:576-579 (1987)). El mantenimiento y evolución de tal grupo de genes prescindibles sugiere la presencia de funciones relevantes para la supervivencia de los virus en su nicho ecológico especı́fico en el huésped natural o en el huésped animal de laboratorio, más que la presencia de funciones necesarias para la replicación (Longnecker, R., y col., Proc. Natl. Acad. Sci. USA 84:4303-4307 (1987) y Weber, P.C., y col., Science 236:576-579 (1987)). Consistentes con tal hipótesis son los hallazgos de que mutantes del HSV que llevan deleciones especı́ficas de genes del componente S diferentes eran significativamente menos patógenos y exhibı́an una capacidad reducida para el establecimiento de la latencia en ratones (Meignier, B., y col., Virology 162:251-254 (1988)). Con respecto a esto último, existe evidencia que sugiere que el ARN transcrito de la región US del HSV puede estar implicado en el establecimiento y mantenimiento de un sistema de latencia in vitro empleando células fibroblastos de pulmón de feto humano (Scheck, A.C., y col., Intervirology 30:121-136 (1989)). Tomadas juntas, la(s) anterior(e)s evidencia(s) sugiere(n) papel(es) potencialmente importante(s) para los genes de US del MDV en el tropismo tisular, latencia y/o inducción de la transformación celular. 25 30 35 40 45 50 55 60 Una consideración de los tres homólogos gD, gI y gE identificados en esta invención da lugar a otras dos cuestiones de relevancia para el desarrollo de futuras vacunas. Los 11 genes de la región US del HSV-1 prescindibles para el crecimiento en cultivo de células descritos anteriormente incluyen US7 (gI) y US8 (gE) del HSV-1 (Longnecker, R., y col., Proc. Natl. Acad. Sci. USA 84:4303-4307 (1987) y Weber, P.C., y col., Science 236:576-579 (1987)). Asumiendo que los homólogos del MDV tienen las mismas propiedades, estos genes pueden ser útiles como sitios para la inserción de genes foráneos. Además puede ser muy probable que los dos mismos homólogos del MDV, y especialmente US8 (gE), estén implicados en las cuestiones relacionadas con la patogenicidad introducidas anteriormente. Especı́ficamente gE del HSV parece jugar un papel en la capacidad del HSV-1 para establecer infecciones letales y latencia en ratones (Meignier, B., y col., Virology 162:251-254 (1988)). Además, los homólogos gI y gE del PRV de cerdos juegan un claro papel en la virulencia del PRV para pollos de 1 dı́a de edad y para cerdos jóvenes (Mettenleiter, Thomas C., y col., Journal of Virology, p. 4030-4032 (Dic. 1987)). Asumiendo que las mismas consideraciones son ciertas para los homólogos de US7 (gI) y US8 (gE) del MDV, puede ser posible inactivar uno de estos genes o ambos a partir de aislados muy virulentos de MDV que causen epidemias no prevenidas por las vacunas actuales, y crear con ello un virus vacuna atenuado relacionado más estrechamente con el virus de campo que causa las epidemias de la enfermedad. Una consideración más de los tres homólogos (gD, gI y gE) identificados en esta invención da lugar a otra interesante cuestión. Se sabe que los viriones infecciosos del MDV completamente envueltos son producidos solamente en las células epiteliales del folı́culo de las plumas (Payne, L.N., p. 347-431. En B. Roizman (ed.), The herpesviruses, vol. 1. Plenum Press (1982)). Debido a esto, los estudios del MDV han tenido que basarse en cultivos celulares de fibroblastos limitados que promueven solamente la propagación de las infecciones asociadas a células in vitro. Durante los últimos 20 años, los estudios dirigidos a la identificación de antı́genos de superficie inmunogénicos se han basado en este sistema de cultivo in vitro y en total solamente dos antı́genos glicoproteicos (antı́geno A/homólogo gC; antı́geno B) han sido identificados y caracterizados de forma rutinaria (Binns, M.M., y col., Virus Res. 12:371-382 (1989); Coussens, P.M., y col., J. Virol. 62:2373-2379 (1988); Isfort, R.J., y col., J. Virol. 59:411-419 (1986); Isfort, R.J., y col., J. Virol. 57:464-474 (1986) y Sithole, I., y col., J. Virol. 62:4270-4279 (1988)). Esto es a pesar de los hallazgos de los tres homólogos gD, gI y gE del MDV de la presente invención y de dos homólogos de glicoproteı́nas adicionales (gB y gH, Buckmaster, A.E., y col., J. Gen. Virol. 69:2033-2042 (1988) y Ross, L.J.N., y col., J. Gen. Virol. 70:1789-1804 (1989)). Aunque es probable que los sueros de pollos inmunes (ICS) procedentes de pájaros infectados de forma natural reaccionen con muchos, si no con todos, los antı́genos de superficie codificados por el MDV, estos sueros policlonales complejos serı́an útiles solamente hasta el punto en que la expresión/procesamiento del antı́geno en cultivo de células semiproductivo se asemeje a la de las células epiteliales del folı́culo de las plumas. El análisis de transferencia Northern utilizando sondas especı́ficas para gD del MDV sugiere que el ARNm de gD del MDV o no se expresa o se expresa pobremente en las células DEF en el momento en que se 14 ES 2 110 999 T3 5 10 observan extensos efectos citopáticos (datos no mostrados). A la luz del hecho de que el VZV carece de un homólogo gD y de que está fuertemente asociado a las células, será interesante ver si se encuentra que el bloqueo de la formación del virión del MDV en células fibroblastos de aves primarios se correlaciona con la falta de expresión (en estas células) de una glicoproteı́na, tal como gD, y/o de otro(s) gen(es) del componente S. Debido a que la protección contra MD conferida por cepas de MDV (serotipo 2) o HVT (serotipo 3) atenuadas parece tener una base inmunológica, existe un interés considerable en la identificación de antı́genos comunes. A la vista de esta invención que identifica siete homólogos de US del MDV con respecto a los genes de US del HSV (el último de los cuales está claramente menos relacionado con el MDV que el HVT), serı́a sorprendente si se probara que la información anterior que mostraba una falta de homologı́a entre las regiones US de MDV-HVT (Igarashi, T., y col., Virology 157:351-358 (1987)) fuera correcta. Tales resultados negativos pueden reflejar las limitaciones concernientes a las estimaciones de homologı́a basadas en la hibridación, en lugar de en los estudios del análisis de secuencias. 15 El Ejemplo 2 muestra el clonaje molecular de una construcción que contiene el ADN que codifica el gen US7 (gI) del MDV completo y parte del gen US8 (gE) del MDV. Como puede verse, esto se realiza utilizando segmentos de ADN que abarcan la región codificadora de gI y parte de la región codificadora de gE. 20 Ejemplo 2 Clonaje molecular de una construcción que contiene el ADN que codifica el US7 (gI) completo del MDV y parte del US8 (gE) del MDV 25 30 35 40 45 50 55 60 La construcción de un clon recombinante (pKS-MDgI1.59) conteniendo la secuencia codificadora de US7 (gI) del MDV completa y una porción de la secuencia codificadora de US8 (gE) del MDV requiere dos clones del MDV preexistentes, pKS-MDgD1.75 y p19P1 (Fig. 5). El pKS-MDgD1.75 es un plásmido recombinante que contiene el subfragmento NcoI-SstII de 1,75 kpb de EcoR1-I del MDV ligado en el sitio de SmaI-SstII del vector de clonaje, pBluescript KS-. Este clon contiene la secuencia codificadora completa de US6 (gD) del MDV y secuencias adicionales en el extremo 3’ que codifican los primeros 39 aminoácidos (aa) de gI del MDV. El p19P1 es un plásmido recombinante que contiene el subfragmento BamHI-P1 de 1,5 kpb del MDV clonado en el único sitio de BamHI del pUC19. Este clon contiene la secuencia codificadora completa de gI del MDV, excepto los 9 primeros aa de su secuencia señal. Además, en el extremo 3’, el p19P1 contiene los primeros 104 aa de la región codificadora de US8 (gE) del MDV. Para generar pKS-MDgI1.59, el pKS-MDgD1.75 es cortado primeramente con HincII, que corta una vez en el sitio de clonaje múltiple del vector pBluescript y una vez 180 pb aproximadamente corriente arriba del extremo SstII del inserto. Esto tiene como resultado dos fragmentos: un fragmento (1,6 kpb) consta primariamente de secuencias del inserto que codifican US6 (gD) del MDV; el fragmento más grande (3,1 kpb) consta de secuencias del vector pBluescript, además de 180 pb aproximadamente que codifican el extremo N de gI del MDV. El fragmento de 3,1 kb es purificado en gel y ligado consigo mismo por medio de los dos extremos HincII. El plásmido recombinante resultante, pKS-MDgI0.18, es luego cortado con SstI (en el sitio de clonaje múltiple, justo corriente abajo del sitio de SstII). Antes de la digestión posterior con SstII, los extremos SstI cohesivos se hacen extremos redondeados con ADN polimerasa T4. El fragmento SstII-SstI (redondeado) de 3,1 kpb resultante de pMDgI0.18 es purificado en gel y utilizado en la etapa final de ligadura para crear pKS-MDgI1.59. Mientras tienen lugar las manipulaciones enzimáticas de pKS-MDgD1.75 y pKS-MDgI0.18, p19P1 es cortado con HindIII, que corta justo corriente abajo de la secuencia codificadora parcial de US8 (gE) del MDV en el sitio de clonaje múltiple de pUC19. Antes de la digestión con SstIId, los extremos de HindIII cohesivos se hacen extremos redondeados utilizando el fragmento Klenow. El fragmento SstII-HindIII (redondeado) más pequeño (1,4 kpb) contiene una mayorı́a de la secuencia codificadora de US7 (gI) del MDV, además de 312 nucleótidos en el extremo 3’ que codifican el extremo 5’ de gE del MDV. Este fragmento SstII-HindIII (redondeado) de 1,4 kpb es purificado en gel y ligado al fragmento SstII-SstI (redondeado) de 3,1 kpb de pKS-MDgD0.18. El recombinante resultante, pKS-MDgI1.59, contiene la secuencia codificadora completa para gI del MDV y una porción de la secuencia codificadora de gE N-terminal. La digestión de pKS-MDgI1.59 con KpnI produce dos fragmentos; el fragmento más pequeño de 1,15 kpb contiene la secuencia codificadora completa para gI del MDV. El Ejemplo 3 muestra el subclonaje molecular de una construcción que contiene el gen US8 (gE) completo del MDV. 15 ES 2 110 999 T3 Ejemplo 3 Clonaje molecular de una construcción que codifica el US8 (gE) completo del MDV 5 10 La construcción de un clon recombinante (p18-MDgE2.53) conteniendo la secuencia codificadora completa de US8 (gE) del MDV requiere un clon distinto de los clones de BamHI o EcoR1 utilizados previamente. El clon GA-02 de la cepa GA, un clon de EMBL-3 que contiene un inserto de SalI de MDV parcialmente digerido, que contiene BamHI-A, -P1, y secuencias flanqueantes 5’ y 3’ adicionales (proporcionado amablemente por P. Sondermeier, Intervet Intl. B. V., Boxmeer, Paı́ses Bajos) fue utilizado para extender el análisis 3’ de los fragmentos EcoR1-I y BamH1-P1. Subfragmentos de SalI más pequeños situados en el extremo 3’ de este inserto del MDV en los clones del fago fueron purificados en gel y ligados a pUC18 linealizado con SalI (pSP18-A, pSP18-B y pSP18-C, Fig. 1B). El subclon de pUC18, pSP18-A contiene la secuencia codificadora completa de US8 (gE) del MDV y se denomina p18-MDgE2.53 a efectos del depósito en la ATCC. 15 Índice de definición de las letras de la Figura 2. La Tabla 4 muestra los aminoácidos con sus sı́mbolos de una letra y de tres letras. TABLA 4 20 25 30 35 40 A C D E F G H I K L Ala Cys Asp Glu Phe Gly His Ile Lys Leu Alanina Cisteı́na Ácido Aspártico Ácido Glutámico Fenilalanina Glicina Histidina Isoleucina Lisina Leucina M N P Q R S T V W Y Met Asn Pro Gln Arg Ser Thr Val Trp Tyr Metionina Asparragina Prolina Glutamina Arginina Serina Treonina Valina Triptófano Tirosina Cuando los segmentos de ADN que codifican las glicoproteı́nas gI y gE son alterados por mutagénesis por inserción, dirigida a un sitio o por deleción, puede reducirse la patogenicidad del MDV. Además, los segmentos de ADN que codifican las gI y gE no esenciales pueden ser utilizados como sitios para la inserción de segmentos de ADN foráneo que codifiquen proteı́nas que son antigénicamente activas con el fin de producir una vacuna recombinante. Depósito en la ATCC El gen para US6 del MDV (gD del MDV) ha sido depositado en un plásmido (phagemid) pKSMDgD1.75, como ATCC 40855, en la American Type Culture Collection, Rockville, MD, 20852, EE.UU. 45 50 El gen para US7 del MDV (gI del MDV) ha sido depositado en un plásmido (phagemid) pKSMDgI1.59, como ATCC 75040, en la American Type Culture Collection, Rockville, MD, 20852, EE.UU. El gen para US8 del MDV (gE del MDV) ha sido depositado en un plásmido p18-MDgE2.53, como ATCC 75039, en la American Type Culture Collection, Rockville, MD, 20852, EE.UU. Se adjuntan los Listados de Secuencia para las Secuencias ID Nos 1, 2 y 3 según se describieron previamente en la solicitud. 55 60 16 ES 2 110 999 T3 APÉNDICE I (1) INFORMACIÓN GENERAL (i) Solicitante: 5 10 Leland F. Velicer, Peter Brunovskis y Paul Coussens (ii) Tı́tulo de la Invención: Segmento de ADN del Herpesvirus de la Enfermedad de Marek que Codifica las Glicoproteı́nas gD, gI y gE (iii) Número de Secuencias: 3 (iv) Dirección para Correspondencia: 15 20 (A) (B) (C) (D) (E) (F) Destinatario: Ian C. McLeod Calle: 2190 Commons Parkway Ciudad: Okemos Estado: Michigan Condado: Ingham Zip: 48864 (v) Forma Legible por Ordenador: (A) Tipo de Medio: 25 30 disco magnético flexible 1.44 Mb 3 1/2” (B) Ordenador: IBM PS2, Modelo 50 (C) Sistema Operativo: MS-DOS 5.0 (D) Programa: PC-Write 3.02 (viii) Información sobre el Apoderado/Agente: (A) Nombre: Ian C. McLeod (B) Número de Registro: 20.931 (C) Número de Referencia/Expediente: MSU 4.1-132 35 (ix) Información para Telecomunicación: (A) Teléfono: (517) 347-4100 (B) Telefax: (517) 347-4103 40 (2) Información para la SEC ID N◦ : 1 (i) Caracterı́sticas de la Secuencia: 45 (A) (B) (C) (D) Longitud: 10.350 pares de bases Tipo: ácido nucleico Tipo de Hebra: doble Topologı́a: lineal (ii) Tipo de Molécula: ADN genómico 50 (iii) HIPOTÉTICA: Si (v) ANTISENTIDO: No (vi) FUENTE ORIGINAL: 55 (A) Organismo: MDV, cepa GA (vii) FUENTE INMEDIATA: (A) Biblioteca: gemómica 60 (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID N◦ :1: 17 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 GAATTCCTTG AAATTGGAGT GAAATCTTTA GGGAGGGAGG TTTACCATTG TGGAGAATAT ATAGAGCAAG TAGTACATTA GGGGCTGGGT TAAAGACCAA GTAATTTTTG ACCGGATATC ACGTGATGTA AATTCTAGCA ATTATTGTTC CTAGCAGAAG ATAAAAGCTG GTAGCTATAT AATACAGGCC AAAGTCTCCA AATTACACTT GAGCAGAAAA CCTGCTTTCG GCTCCATCGG AGGCAAC ATG AGT CGT GAT CGA GAT CGA GCC AGA CCC GAT ACA CGA TTA Met Ser Arg Asp Arg Asp Arg Ala Arg Pro Asp Thr Arg Leu 1 5 10 TCA TCG TCA GAT AAT GAG AGC GAC GAC GAA GAT TAT CAA CTG CCA CAT Ser Ser Ser Asp Asn Glu Ser Asp Asp Glu Asp Tyr Gln Leu Pro His 15 20 25 30 TCA CAT CCG GAA TAT GGC AGT GAC TCG TCC GAT CAA GAC TTT GAA CTT Ser His Pro Glu Tyr Gly Ser Asp Ser Ser Asp Gln Asp Phe Glu Leu 35 40 45 AAT AAT GTG GGC AAA TTT TGT CCT CTA CCA TGG AAA CCC GAT GTC GCT Asn Asn Val Gly Lys Phe Cys Pro Leu Pro Trp Lys Pro Asp Val Ala 50 55 60 CGG TTA TGT GCG GAT ACA AAC AAA CTA TTT CGA TGT TTT ATT CGA TGT Arg Leu Cys Ala Asp Thr Asn Lys Leu Phe Arg Cys Phe Ile Arg Cys 65 70 75 CGA CTA AAT AGC GGT CCG TTC CAC GAT GCT CTT CGG AGA GCA CTA TTC Arg Leu Asn Ser Gly Pro Phe His Asp Ala Leu Arg Arg Ala Leu Phe 80 85 90 GAT ATT CAT ATG ATT GGT CGA ATG GGA TAT CGA CTA AAA CAA GCC GAA Asp Ile His Met Ile Gly Arg Met Gly Tyr Arg Leu Lys Gln Ala Glu 95 100 105 110 TGG GAA ACT ATC ATG AAT TTG ACC CCA CGC CAA AGT CTA CAT CTG CGC Trp Glu Thr Ile Met Asn Leu Thr Pro Arg Gln Ser Leu His Leu Arg 115 120 125 AGG ACT CTG AGG GAT GCT GAT AGT CGA AGC GCC CAT CCT ATA TCC GAT Arg Thr Leu Arg Asp Ala Asp Ser Arg Ser Ala His Pro Ile Ser Asp 130 135 140 ATA TAT GCC TCC GAT AGC ATT TTT CAC CCA ATC GCT GCG TCC TCG GGA Ile Tyr Ala Ser Asp Ser Ile Phe His Pro Ile Ala Ala Ser Ser Gly 145 150 155 ACT ATT TCT TCA GAC TGC GAT GTA AAA GGA ATG AAC GAT TTG TCG GTA Thr Ile Ser Ser Asp Cys Asp Val Lys Gly Met Asn Asp Leu Ser Val 160 165 170 GAC AGT AAA TTG CAT TAA CTATCCAGAC TTGAAGAGAA AGCTCTTATT Asp Ser Lys Leu His Fin 175 ATATAATTTT AATTGTTAGA CATAGAGCCG ACATTCTTTG ATCTATCTAA TGAGATAAAA TAATAGATTT TGGATTTATT TGTCATGATC TGTTGCAACA AACGCTGACC CCCCCCATCC ATGAAGGGGC GTGTCAAATA ACGTGTTGCC TTTTTGTTGT ATATGAAGAT ATTTAATGTG GCGTTGAGCC TAATGAGAGG AGAACGTGTT TGAATACTGG AGACGAGCGC CGTGTAAGAT TAAAACATAT TGGAGAGGT ATG GCC ATG TGG TCT CTA CGG CGC AAA TCT Met Ala Met Trp Ser Leu Arg Arg Lys Ser 1 5 10 AGC AGG AGT GTG CAA CTC CGG GTA GAT TCT CCA AAA GAA CAG AGT TAT Ser Arg Ser Val Gln Leu Arg Val Asp Ser Pro Lys Glu Gln Ser Tyr 15 20 25 GAT ATA CTT TCT GCC GGC GGG GAA CAT GTT GCG CTA TTG CCT AAA TCT Asp Ile Leu Ser Ala Gly Gly Glu His Val Ala Leu Leu Pro Lys Ser 30 35 40 GTA CGC AGT CTA GCC AGG ACC ATA TTA ACC GCC GCT ACG ATC TCC CAG Val Arg Ser Leu Ala Arg Thr Ile Leu Thr Ala Ala Thr Ile Ser Gln 45 50 55 GCT GCT ATG AAA GCT GGA AAA CCA CCA TCG TCT CGT TTG TGG GGT GAG Ala Ala Met Lys Ala Gly Lys Pro Pro Ser Ser Arg Leu Trp Gly Glu 60 65 70 ATA TTC GAC AGA ATG ACT GTC ACG CTT AAC GAA TAT GAT ATT TCT GCT 18 60 120 180 240 289 337 385 433 481 529 577 625 673 721 769 817 877 937 997 1057 1106 1154 1202 1250 1298 1346 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 Ile Phe Asp Arg Met Thr Val Thr Leu Asn Glu 75 80 85 TCG CCA TTC CAC CCG ACA GAC CCG ACG AGA AAA Ser Pro Phe His Pro Thr Asp Pro Thr Arg Lys 95 100 TTA CGG TGT ATT GAA CGT GCT CCT CTT ACA CAC Leu Arg Cys Ile Glu Arg Ala Pro Leu Thr His 110 115 CGG TTT ACT ATC ATG ATG TAT TGG TGT TGT CTT Arg Phe Thr Ile Met Met Tyr Trp Cys Cys Leu 125 130 TGT ACT GTT TCG CGC TTA TAT GAG AAG AAT GTC Cys Thr Val Ser Arg Leu Tyr Glu Lys Asn Val 140 145 GTA GGT TCG GCA ACG GGC TGT GGA ATA AGT CCA Val Gly Ser Ala Thr Gly Cys Gly Ile Ser Pro 155 160 165 TCT TAT TGG AAA CCT TTA TGT CGT GCC GTC GCT Ser Tyr Trp Lys Pro Leu Cys Arg Ala Val Ala 175 180 GCA ATC GGT GAT GAT GCT GAA TTG GCA CAT TAT Ala Ile Gly Asp Asp Ala Glu Leu Ala His Tyr 190 195 GAA TCG CCA ACA GGA GAC GGG GAA TCC TAC TTA Glu Ser Pro Thr Gly Asp Gly Glu Ser Tyr Leu 205 210 AATTATTAAT AGGATTTTAG GAAAAACTGC TACTAACGTT TTTTCAATAA GGCATTACAG TGTTGTCATG ATTGTATGTA GATTACTTCG ATTGAAACTT TGTCTAAATG TCTGTAGGAT ATCGAGGCGG ACGTAAATGG AGATTGCGGC AAATGTAGGG CAACATCCAT TCGACTCATC GGCCTGCGTC CAAATGGATA TTATGACATT AGAAGATCGA TGGTGAATAG TGGGATCTAT TGCATGATAT GCAATGTTCC CGGTTAGGTT TGATAAGATC ACTCCTCTTC AGAAGAATCA TTTATTTTAT GTCCACTGTC TCAATCGATT CGCTTGCATT TGCGTGCAGC ATGTCTTGAT CCGGCAGGCC TAAGGGTGTT CTATACTCGC ACACAGGTAG GAGCTACCTC TATTGCCCCG CTAAGGACAT TTCTTGCAGA TTCGTGTATT GTGTCGATCA TAACCCTTGT TGATTCCTAT TTTCCAGATG AAATGAAAAC AATGCGGGCA AAAATGGTCC CATCTCTCAC ATCCCAAGTT CTATAGAATA TTCTCCACTG TTTCTGTAAA ATTTGTGATA GTTTCAATCG AAAACATTTT TATAGGCAGA CCAGATAACC ATTTGACACC ACATATCCTT TAGATCCCTC GTTAGTAGAT ATGGTACATA AAAGGCCTAA ATTGAACGAT TCCTTCTGTG AATTCATCAA CAACCACATG TCTTTCTCGG TGGCTTACCA AATCGTCCTC TTGGTATATC CATTGACTCT GCTCATCGTT GTCTTTCAAA TGCGCTCGAT TAGAAGTATA TGGAAGATAG CCTGGATACA TAAGTGATCT TAATATACAA ATTATACGTG ACACTATAGC GACGGTTGTA GTGTATACGC CCCATCATGT AATTATATCT AATTGGTAGC AGCTAATGAC TACCGGCTCT ACATTTTTTC TGTATTCGTG CGAACCGGAA TTGCAATCGC ATCTCTATCT TCTTTCTTGC AATCTGCCGG GTGTACTACT CATTTGAGGT GGTTCGATTT GGTGGGGACC CGAGGATTTT GTATACACAT ACCATATCAC TCTTCTGGGG TGTCGAACTT CGGTTCCCAT GTAGATGTCA GGAATGGCCC ACGGCATACC GGACCAGGTC CCAGACACTT GGCAAAGGAA TACATTCGAG CGCAATGGCA CATATATCTG CTATGTGGAG CATTACCAGA AACTTCAGAT TCCAACATCA TGCCATTCTG TGGAACATCC TGCAACATCT TCAAATAGCC GTTCCGGCCA ATCCGGTACC ACGAACTCCA GTTCCATCTG GGTCGATGTT GCCGAGGAAG AATTAACATG GGTTTGGCAA Tyr Asp Ile Ser Ala 90 ATT GTA GGC CGG GCT Ile Val Gly Arg Ala 105 GAA GAA ATG GAC ACT Glu Glu Met Asp Thr 120 GGA CAT GCT GGA TAC Gly His Ala Gly Tyr 135 CGT CTT ATG GAC ATA Arg Leu Met Asp Ile 150 CTC CCC GAA ATA GAG Leu Pro Glu Ile Glu 170 ACT AAG GGG AAT GCA Thr Lys Gly Asn Ala 185 CTG ACA AAT CTT CGG Leu Thr Asn Leu Arg 200 TAA CTAATCGCAC Fin GTTTAAATAA TTATATGGGG TTTACTATTC GTGCTGGTAC TGTTGATGTA ATCCATGCTA ATGTATGGTT CTTGGATATT GGCATTTCCT AGCAAGAACC CTGTATTGTC GGAAAGCATT CACCTGTTTC ACCAGTTTCG GTCCATCATG GTGTATATCA TCTCTCTCGG CCAAAAATTT CATATCATCG TGTTGAATCT AGAAGGGTTT GCGATGCACC AAGTAGGTCT ACTTTCCTGT AACATTTTCC CCGGAGGTTT TGTCGCAAAA AGAGAGTTTG TGATTGCAAG CCGCCCCAAC AATATCCAGA GCACTATAAA GTGGCTTTGT AACGGAATAG 19 TAAAATTTTA TATGCATGAG ATTAGTCTGG ATAAGACCTC CCTTGTAAAG TTCTCAATAT CTATAATACA CCAGTTTCTG ATGCTATCAT ACGGCATATC ATGAACATAT GTGGTCCAGT ATCTTCAATG GTAAGATCAG GCAAAAAATC AACGATGTAA GCTTCCATAC ACATTAGTAA AACATTGTAG CTCCTGATGT GTTATTGCAC TAATCGTAAT GTCGAATAAC CGCAGTGTAA ACAACAGAAT TAGAGGATTG ATGCGCTCTA AATATTGTCG TAACCTTTTT TATCCACAAG TAGAACATCC CGAATCCCTA CCTTACTATC GTCTGCAGCT 1394 1442 1490 1538 1586 1634 1682 1728 1788 1848 1908 1968 2028 2088 2148 2208 2268 2328 2388 2448 2508 2568 2628 2688 2748 2808 2868 2928 2988 3048 3108 3168 3228 3288 3348 3408 3468 3528 3588 3648 3708 3768 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 CTGGCGATTA AATATCCATA AGAAGTGTGA TATTGTTAAT AGGGTTAAAA TGGGCACACC AAGTAGATGC CTATAGTTAT AACTGGGCCG CTAGGTAATA GAA TGT GGC ATT TCT Glu Cys Gly Ile Ser 5 ACC TAC GGA ATT ATA Thr Tyr Gly Ile Ile 20 TTT GAT ACT TTT CCC Phe Asp Thr Phe Pro 35 GTG GAC GAT GTG AAG Val Asp Asp Val Lys 50 TTG TCA CCT TTT GGG Leu Ser Pro Phe Gly 70 GAC ATT GAT GCA GTT Asp Ile Asp Ala Val 85 TCG TTA CCG CCC GGA Ser Leu Pro Pro Gly 100 GGG GAT AAT ACC AAG Gly Asp Asn Thr Lys 115 AAA ACC CTT GGG AGT Lys Thr Leu Gly Ser 130 TCC ATA ATT AGA TTA Ser Ile Ile Arg Leu 150 ATG GTA ATG CCT AAA Met Val Met Pro Lys 165 ATG GGA CCA TTG CCA Met Gly Pro Leu Pro 180 CTT GGA GCA TTG GCA Leu Gly Ala Leu Ala 195 GTA AAA ACT GAA AAT Val Lys Thr Glu Asn 210 GGG GAC TTT GGG GCA Gly Asp Phe Gly Ala 230 AAA TGT TAT GGA TGG Lys Cys Tyr Gly Trp 245 CTT GCA CTT GAT CCA Leu Ala Leu Asp Pro 260 TTA GTT CTG TTT GAG Leu Val Leu Phe Glu 275 CACATCATCC AGCATCTCTA CATGGACACA GTCTGATCTC GACTGGATGT TGTATTTGTT GATCTTCCTG CCCATCTTCA CAAATCTTAT CTTCGACTCC TCG TCG AAA GTA CAC Ser Ser Lys Val His 10 CAT AAC AGC ATC AAT His Asn Ser Ile Asn 25 GAC AGT ACC GAT AAC Asp Ser Thr Asp Asn 40 ACT GAG AGC TCT CCC Thr Glu Ser Ser Pro 55 AAC GAT GGA AAT GAA Asn Asp Gly Asn Glu 75 TCA GCT GTG CGA ATG Ser Ala Val Arg Met 90 TCT GAA GGG TAT ATC Ser Glu Gly Tyr Ile 105 AGA AAA GTC ATT GTG Arg Lys Val Ile Val 120 GAA ATT GAT ATA TTA Glu Ile Asp Ile Leu 135 GTT CAT GCT TAT AGA Val His Ala Tyr Arg 155 TAC AAA TGC GAC TTG Tyr Lys Cys Asp Leu 170 CTA AAT CAA ATA ATT Leu Asn Gln Ile Ile 185 TAT ATC CAC GAA AAG Tyr Ile His Glu Lys 200 ATA TTT TTG GAT AAA Ile Phe Leu Asp Lys 215 GCA TGT AAA TTA GAT Ala Cys Lys Leu Asp 235 AGT GGA ACT CTG GAA Ser Gly Thr Leu Glu 250 TAC TGT ACA AAA ACT Tyr Cys Thr Lys Thr 265 ATG TCA GTA AAA AAT Met Ser Val Lys Asn 280 CCATACATTG GCAATCGATC CCTCCACCAA ACTCTGGTAG GGAGGCAGAA GAC TCT AAA Asp Ser Lys GGT ACG GAT Gly Thr Asp 30 GCG GAA GTG Ala Glu Val 45 GAG TCC CAA Glu Ser Gln 60 TCC CCC GAA Ser Pro Glu CAG TAT AAC Gln Tyr Asn TAT GTT TGT Tyr Val Cys 110 AAA GCT GTG Lys Ala Val 125 AAA AAA ATG Lys Lys Met 140 TGG AAA TCG Trp Lys Ser TTT ACG TAC Phe Thr Tyr ACG ATA GAA Thr Ile Glu 190 GGT ATA ATA Gly Ile Ile 205 CCT GAA AAT Pro Glu Asn 220 GAA CAT ACA Glu His Thr ACC AAT TCG Thr Asn Ser CTTTATAAGG GCATTCATCT TAATCTTTTT AATATGAAAC ACG ATG Met 1 ACT AAT ACT Thr Asn Thr 15 ACG ACG TTG Thr Thr Leu 4112 4160 ACG GGG GAT Thr Gly Asp 4208 TCT GAA GAT Ser Glu Asp 65 ACG GTG ACG Thr Val Thr 80 ATT GTT TCA Ile Val Ser 95 ACA AAG CGT Thr Lys Arg 4256 ACT GGT GGC Thr Gly Gly 4448 TCT CAC CGC Ser His Arg 145 ACA GTT TGT Thr Val Cys 160 ATA GAT ATC Ile Asp Ile 175 CGG GGT TTG Arg Gly Leu 4496 CAT CGT GAT His Arg Asp 4688 GTA GTA TTG Val Val Leu 225 GAT AAA CCC Asp Lys Pro 240 CCT GAA CTG Pro Glu Leu 255 AGT GCA GGA Ser Ala Gly 4736 GAT ATA TGG Asp Ile Trp 270 ATA ACC TTT TTT GGC AAA Ile Thr Phe Phe Gly Lys 285 20 3828 3888 3948 4008 4064 4304 4352 4400 4544 4592 4640 4784 4832 4880 4928 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 CAA Gln 290 CAA Gln GTA AAC GGC TCA GGT TCT CAG CTG AGA TCC ATA ATT AGA TGC CTG Val Asn Gly Ser Gly Ser Gln Leu Arg Ser Ile Ile Arg Cys Leu 295 300 305 GTC CAT CCG TTG GAA TTT CCA CAG AAC AAT TCT ACA AAC TTA TGC Val His Pro Leu Glu Phe Pro Gln Asn Asn Ser Thr Asn Leu Cys 310 315 320 AAA CAC TTC AAG CAG TAC GCG ATT CAG TTA CGA CAT CCA TAT GCA ATC Leu His Phe Lys Gln Tyr Ala Ile Gln Leu Arg His Pro Tyr Ala Ile 325 330 335 CCT CAG ATT ATA CGA AAG AGT GGT ATG ACG ATG GAT CTT GAA TAT GCT Pro Gln Ile Ile Arg Lys Ser Gly Met Thr Met Asp Leu Glu Tyr Ala 340 345 350 ATT GCA AAA ATG CTC ACA TTC GAT CAG GAG TTT AGA CCA TCT GCC CAA Ile Ala Lys Met Leu Thr Phe Asp Gln Glu Phe Arg Pro Ser Ala Gln 355 360 365 GAT ATT TTA ATG TTG CCT CTT TTT ACT AAA GAA CCC GCT GAC GCA TTA Asp Ile Leu Met Leu Pro Leu Phe Thr Lys Glu Pro Ala Asp Ala Leu 370 375 380 385 TAC ACG ATA ACT GCC GCT CAT ATG TAA ACACCCGTCA AAAATAACTT Tyr Thr Ile Thr Ala Ala His Met Fin 390 CAATGATTCA TTTTATAATA TATACTACGC GTTACCTGCA ATAATGACAA CATTCGAAGT CTTTGAAGAT TCGCAGACCT TTTTTGCGA ATG GCA CCT TCG GGA CCT ACG CCA Met Ala Pro Ser Gly Pro Thr Pro 1 5 TAT TCC CAC AGA CCG CAA ATA AAG CAT TAT GGA ACA TTT TCG GAT TGC Tyr Ser His Arg Pro Gln Ile Lys His Tyr Gly Thr Phe Ser Asp Cys 10 15 20 ATG AGA TAT ACT CTA AAC GAT GAG AGT AAG GTA GAT GAT AGA TGT TCA Met Arg Tyr Thr Leu Asn Asp Glu Ser Lys Val Asp Asp Arg Cys Ser 25 30 35 40 GAC ATA CAT AAC TCC TTA GCA CAA TCC AAT GTT ACT TCA AGC ATG TCT Asp Ile His Asn Ser Leu Ala Gln Ser Asn Val Thr Ser Ser Met Ser 45 50 55 GTA ATG AAC GAT TCG GAA GAA TGT CCA TTA ATA AAT GGA CCT TCG ATG Val Met Asn Asp Ser Glu Glu Cys Pro Leu Ile Asn Gly Pro Ser Met 60 65 70 CAG GCA GAG GAC CCT AAA AGT GTT TTT TAT AAA GTT CGT AAG CCT GAC Gln Ala Glu Asp Pro Lys Ser Val Phe Tyr Lys Val Arg Lys Pro Asp 75 80 85 CGA AGT CGT GAT TTT TCA TGG CAA AAT CTG AAC TCC CAT GGC AAT AGT Arg Ser Arg Asp Phe Ser Trp Gln Asn Leu Asn Ser His Gly Asn Ser 90 95 100 GGT CTA CGT CGT GAA AAA TAT ATA CGT TCC TCT AAG AGG CGA TGG AAG Gly Leu Arg Arg Glu Lys Tyr Ile Arg Ser Ser Lys Arg Arg Trp Lys 105 110 115 120 AAT CCC GAG ATA TTT AAG GTA TCT TTG AAA TGT GAA TCA ATT GGC GCT Asn Pro Glu Ile Phe Lys Val Ser Leu Lys Cys Glu Ser Ile Gly Ala 125 130 135 GGT AAC GGA ATA AAA ATT TCA TTC TCA TTT TTC TAA CATTATAATA Gly Asn Gly Ile Lys Ile Ser Phe Ser Phe Phe Fin 140 145 TATCAGATCG TTTCTTATAT ACTTATTTTC ATCGTCGGGA TATGACTAAC GTATACTAAG TTACAAGAAA CAACTGCTTA ACGTCGAACA TAACGGAAAT AAAAATATAT ATAGCGTCTC CTATAACTGT TATATTGGCA CCTTTTAGAG CTTCGGT ATG AAT AGA TAC AGA TAT Met Asn Arg Tyr Arg Tyr -30 -25 GAA AGT ATT TTT TTT AGA TAT ATC TCA TCC ACG AGA ATG ATT CTT ATA Glu Ser Ile Phe Phe Arg Tyr Ile Ser Ser Thr Arg Met Ile Leu Ile -20 -15 -10 ATC TGT TTA CTT TTG GGA ACT GGG GAC ATG TCC GCA ATG GGA CTT AAG 21 4976 5024 5072 5120 5168 5216 5263 5323 5376 5424 5472 5520 5568 5616 5664 5712 5760 5806 5866 5926 5981 6029 6077 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 Ile Cys Leu Leu -5 AAA GAC AAT TCT Lys Asp Asn Ser 10 AAC CTC CGG GCT Asn Leu Arg Ala 25 GAT ACA CTT GAC Asp Thr Leu Asp AAT TGT AGT TTT Asn Cys Ser Phe 60 CTT CTC AGT TTA Leu Leu Ser Leu 75 GCA TGG TTT GTC Ala Trp Phe Val 90 GAA TAT GCC AAC Glu Tyr Ala Asn 105 AAG TCC CTA GGA Lys Ser Leu Gly GAT CGA GAT GAA Asp Arg Asp Glu 140 AGT GGA TTA TAT Ser Gly Leu Tyr 155 AGT GAC ATA TTA Ser Asp Ile Leu 170 GGG ATA AAG GAT Gly Ile Lys Asp 185 GGT ACA ACA CGG Gly Thr Thr Arg CAT GAA GAA AAT His Glu Glu Asn 220 CTA CCA ATC GTC Leu Pro Ile Val 235 AGA AGT TTT AGA Arg Ser Phe Arg 250 AAT GAC GTC AAA Asn Asp Val Lys 265 GTG GAT GGT TAC Val Asp Gly Tyr GCA CCA TAC ATA Ala Pro Tyr Ile 300 AGT CAA TCA CCT Ser Gln Ser Pro 315 Leu Gly Thr Gly Asp 1 CCG ATC ATT CCC ACA Pro Ile Ile Pro Thr 15 ACT CTC AAT GAA TAC Thr Leu Asn Glu Tyr 30 AAT TCA TAT GAG ACA Asn Ser Tyr Glu Thr 45 GCT GTT TTG AAT CCA Ala Val Leu Asn Pro 65 CTG TTG ATG GGA CGA Leu Leu Met Gly Arg 80 TTG GGC AGA GCA TGT Leu Gly Arg Ala Cys 95 TGC TCT ACT AAT GAA Cys Ser Thr Asn Glu 110 TGG TGG GAT AGA AGA Trp Trp Asp Arg Arg 125 TTG AAA TTG ATT ATT Leu Lys Leu Ile Ile 145 ACG CGT TTA ATA ATT Thr Arg Leu Ile Ile 160 CTG ACT GTT AAA GGA Leu Thr Val Lys Gly 175 AAC AAA CTA TGC AAA Asn Lys Leu Cys Lys 190 CTG TTA GAC ATG GTG Leu Leu Asp Met Val 205 GTG AAG CAG TGG CTT Val Lys Gln Trp Leu 225 GTC GAA ACA TCT ATG Val Glu Thr Ser Met 240 GAT TCA TAT TTA AAA Asp Ser Tyr Leu Lys 255 ATG ACA TCG GCC ACT Met Thr Ser Ala Thr 270 ACT GGA CTC ACT AAT Thr Gly Leu Thr Asn 285 ACT AAA CGA CCG ATA Thr Lys Arg Pro Ile 305 AAA ATA TCA ACA GAA Lys Ile Ser Thr Glu 320 Met Ser Ala Met 5 TTA CAT CCG AAA Leu His Pro Lys 20 AAA ATC CCG TCT Lys Ile Pro Ser 35 AAA CAC GTA ATA Lys His Val Ile 50 TTT GGC GAT CCG Phe Gly Asp Pro CGC AAA Arg Lys GGG AGA Gly Arg CCA TTT Pro Phe 115 TAT GCA Tyr Ala 130 GCA GCA Ala Ala ATT AAT Ile Asn ACA TGT Thr Cys CCG TTC Pro Phe 195 CGA ACA Arg Thr 210 GAA CGA Glu Arg CAA CAA Gln Gln TCA CCT Ser Pro ACT AAT Thr Asn 275 CGG CCC Arg Pro 290 ATC TCT Ile Ser AAA AAA Lys Lys Gly Leu Lys GGT AAT GAA Gly Asn Glu 6125 CCA CTG TTT Pro Leu Phe 40 TAT ACG GAT Tyr Thr Asp 55 AAA TAT ACG Lys Tyr Thr 70 TAT GAT GCT CTA GTA Tyr Asp Ala Leu Val 85 CCA ATT TAT TTA CGT Pro Ile Tyr Leu Arg 100 GGA ACT TGT AAA TTA Gly Thr Cys Lys Leu 120 ATG ACG AGT TAT ATC Met Thr Ser Tyr Ile 135 CCC AGT CGT GAG CTA Pro Ser Arg Glu Leu 150 GGA GAA CCC ATT TCG Gly Glu Pro Ile Ser 165 AGT TTT TCG AGA CGG Ser Phe Ser Arg Arg 180 AGT TTT TTT GTC AAT Ser Phe Phe Val Asn 200 GGA ACC CCG AGA GCC Gly Thr Pro Arg Ala 215 AAT GGT GGT AAA CAT Asn Gly Gly Lys His 230 GTC TCA AAT TTG CCG Val Ser Asn Leu Pro 245 GAC GAC GAT AAA TAT Asp Asp Asp Lys Tyr 260 AAC ATT ACC ACC TCC Asn Ile Thr Thr Ser 280 GAG GAC TTT GAG AAA Glu Asp Phe Glu Lys 295 GTC GAG GAG GCA TCC Val Glu Glu Ala Ser 310 TCC CGA ACG CAA ATA Ser Arg Thr Gln Ile 325 6173 22 6221 6269 6317 6365 6413 6461 6509 6557 6605 6653 6701 6749 6797 6845 6893 6941 6989 7037 ES 2 110 999 T3 5 10 15 20 ATA ATT TCA Ile Ile Ser 330 GGG TCT GGT Gly Ser Gly 345 GAT AGA CGT Asp Arg Arg CTA GTT GTT CTA TGC GTC ATG TTT Leu Val Val Leu Cys Val Met Phe 335 ATA TGG ATC CTT CGC AAA CAC CGC Ile Trp Ile Leu Arg Lys His Arg 350 355 CGT CCA TCA AGA CGG GCA TAT TCC Arg Pro Ser Arg Arg Ala Tyr Ser 365 370 CACGTGTTTG GTATGGGCGT GTCGCTATAG TGCATAAGAA ACATTATATA GCTTCTTTGG TCAGATAGAC GGCGTGTGTG CTA Leu -15 GTT Val GTT Val TTA Leu 25 GAA Glu 50 GCG Ala 30 GGC Gly 35 CCT Pro TCA Ser 40 ATG Met 130 TGG Trp 45 CGC Arg 50 GGC Gly ACT Thr 55 TAC Tyr 210 AAT Asn 60 ACC CAA TTA TTA TTT TGG Gln Leu Leu Phe Trp -10 TAT ACT GGA ACA TCT Tyr Thr Gly Thr Ser 5 GCG TTC CGC GGA TTA Ala Phe Arg Gly Leu 20 TTC CTG GGC GAC CAG Phe Leu Gly Asp Gln 35 ATC TTG AAA TGG GAT Ile Leu Lys Trp Asp 55 ACA TCA TAT ATG GAT Thr Ser Tyr Met Asp 70 TGT AGA GAC GCT GTG Cys Arg Asp Ala Val 85 TTT CCC GAA AAG GGA Phe Pro Glu Lys Gly 100 GAT ACA GGC AGC TAT Asp Thr Gly Ser Tyr 115 AGC GAT ATA TTT AGA Ser Asp Ile Phe Arg 135 GCC TGT AAT CAC TCT Ala Cys Asn His Ser 150 TAT GTC GAC CGT ATG Tyr Val Asp Arg Met 165 AAT TTG CTG GAC AGT Asn Leu Leu Asp Ser 180 CCC CAA TCC ATT TCC Pro Gln Ser Ile Ser 195 GAT AAT TCG GGA ACA Asp Asn Ser Gly Thr 215 AAC AAT TCC CAT GTC Asn Asn Ser His Val 230 GTT TTA AAA TAT ACC ATC CGC CTC TTT CGA Ile Arg Leu Phe Arg -5 GTT ACG TTA TCA ACG Val Thr Leu Ser Thr 10 GAT AAA ATG GTG AAT Asp Lys Met Val Asn 25 ACT CGG ACC AGT TCT Thr Arg Thr Ser Ser 40 GAA GAA TAT AAA TGC Glu Glu Tyr Lys Cys 60 TGT CCT GCT ATA GAC Cys Pro Ala Ile Asp 75 GTA TAT GCT CAA CCT Val Tyr Ala Gln Pro 90 ACA TTG TTG AGA ATT Thr Leu Leu Arg Ile 105 TAC ATA CGT GTA TCT Tyr Ile Arg Val Ser 120 ATG GTT GTT ATT ATA Met Val Val Ile Ile 140 GCT AGT TCA TTT CAG Ala Ser Ser Phe Gln 155 GCC TTT GAA AAT TAT Ala Phe Glu Asn Tyr 170 GAC TCG GAA TTG CAT Asp Ser Glu Leu His 185 ACA GAT ATT AAT ATT Thr Asp Ile Asn Ile 200 ATT TAT TCA CCT ACG Ile Tyr Ser Pro Thr 220 GAT GCA ATG AAT TCG Asp Ala Met Asn Ser 235 CTT CCA AGG CTT ATT TGT Cys 340 AAA Lys TTC ATT GTA ATC Phe Ile Val Ile 7085 ACG GTG ATG TAT Thr Val Met Tyr 360 CGC CTA TAA Arg Leu Fin 7133 GTTGACTACA TTGATCAATG ATTGCG ATG TAT GTA Met Tyr Val GGC ATC TGG TCT ATA Gly Ile Trp Ser Ile 1 GAC CAA TCT GCT CTT Asp Gln Ser Ala Leu 15 GTA CGC GGC CAA CTT Val Arg Gly Gln Leu 30 TAT ACA GGA ACG ACG Tyr Thr Gly Thr Thr 45 TAT TCC GTT CTA CAT Tyr Ser Val Leu His 65 GCC ACG GTA TTC AGA Ala Thr Val Phe Arg 80 CAT GGT AGA GTA CAA His Gly Arg Val Gln 95 GTC GAA CCC AGA GTA Val Glu Pro Arg Val 110 CTC GCT GGA AGA AAT Leu Ala Gly Arg Asn 125 AGG AGT AGC AAA TCT Arg Ser Ser Lys Ser 145 GCC CAT AAA TGT ATT Ala His Lys Cys Ile 160 CTG ATT GGA CAT GTA Leu Ile Gly His Val 175 GCA ATT TAT AAT ATT Ala Ile Tyr Asn Ile 190 GTA ACG ACT CCA TTT Val Thr Thr Pro Phe 205 GTT TTT AAT TTG TTT Val Phe Asn Leu Phe 225 ACT GGT ATG TGG AAT Thr Gly Met Trp Asn 240 TAC TTT TCT ACG ATG 23 7175 7235 7290 7338 7386 7434 7482 7530 7578 7626 7674 7722 7770 7818 7866 7914 7962 8010 8058 8106 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 Thr Val Leu Lys 245 ATT GTA CTA TGT Ile Val Leu Cys 260 TGC CGC TCT CCC Cys Arg Ser Pro 275 GAG GCC CCA CTC Glu Ala Pro Leu 290 TAT AAT GTA AAG Tyr Asn Val Lys Tyr Thr Leu Pro Arg Leu Ile Tyr Phe Ser Thr Met 250 255 ATA ATA GCA TTG GCA ATT TAT TTG GTC TGT GAA AGG Ile Ile Ala Leu Ala Ile Tyr Leu Val Cys Glu Arg 265 270 CAT CGT AGG ATA TAC ATC GGT GAA CCA AGA TCT GAT His Arg Arg Ile Tyr Ile Gly Glu Pro Arg Ser Asp 280 285 ATC ACT TCT GCA GTT AAC GAA TCA TTT CAA TAT GAT Ile Thr Ser Ala Val Asn Glu Ser Phe Gln Tyr Asp 295 300 305 GAA ACT CCT TCA GAT GTT ATT GAA AAG GAG TTG ATG Glu Thr Pro Ser Asp Val Ile Glu Lys Glu Leu Met 310 315 320 GAA AAA CTG AAG AAG AAA GTC GAA TTG TTG GAA AGA GAA GAA TGT GTA Glu Lys Leu Lys Lys Lys Val Glu Leu Leu Glu Arg Glu Glu Cys Val 325 330 335 TAG GTTTGAGAAA CTATTATAGG TAGGTGGTAC CTGTTAGCTT AGTATAAGGG Fin GAGGAGCCGT TTCTTGTTTT AAAGACACGA ACACAAGGCC GTAAGTTTTA TATGTGAATT TTGTGCATGT CTGCGAGTCA GCGTCATA ATG TGT GTT TTC CAA ATC CTG ATA Met Cys Val Phe Gln Ile Leu Ile -15 ATA GTG ACG ACG ATC AAA GTA GCT GGA ACG GCC AAC ATA AAT CAT ATA Ile Val Thr Thr Ile Lys Val Ala Gly Thr Ala Asn Ile Asn His Ile -10 -5 1 5 GAC GTT CCT GCA GGA CAT TCT GCT ACA ACG ACG ATC CCG CGA TAT CCA Asp Val Pro Ala Gly His Ser Ala Thr Thr Thr Ile Pro Arg Tyr Pro 10 15 20 CCA GTT GTC GAT GGG ACC CTT TAC ACC GAG ACG TGG ACA TGG ATT CCC Pro Val Val Asp Gly Thr Leu Tyr Thr Glu Thr Trp Thr Trp Ile Pro 25 30 35 AAT CAC TGC AAC GAA ACG GCA ACA GGC TAT GTA TGT CTG GAA AGT GCT Asn His Cys Asn Glu Thr Ala Thr Gly Tyr Val Cys Leu Glu Ser Ala 40 45 50 CAC TGT TTT ACC GAT TTG ATA TTA GGA GTA TCC TGC ATG AGG TAT GCG His Cys Phe Thr Asp Leu Ile Leu Gly Val Ser Cys Met Arg Tyr Ala 55 60 65 70 GAT GAA ATC GTC TTA CGA ACT GAT AAA TTT ATT GTC GAT GCG GGA TCC Asp Glu Ile Val Leu Arg Thr Asp Lys Phe Ile Val Asp Ala Gly Ser 75 80 85 ATT AAA CAA ATA GAA TCG CTA AGT CTG AAT GGA GTT CCG AAT ATA TTC Ile Lys Gln Ile Glu Ser Leu Ser Leu Asn Gly Val Pro Asn Ile Phe 90 95 100 CTA TCT ACG AAA GCA AGT AAC AAG TTG GAG ATA CTA AAT GCT AGC CTA Leu Ser Thr Lys Ala Ser Asn Lys Leu Glu Ile Leu Asn Ala Ser Leu 105 110 115 CAA AAT GCG GGT ATC TAC ATT CGG TAT TCT AGA AAT GGG ACG AGG ACT Gln Asn Ala Gly Ile Tyr Ile Arg Tyr Ser Arg Asn Gly Thr Arg Thr 120 125 130 GCA AAG CTG GAT GTT GTT GTG GTT GGC GTT TTG GGT CAA GCA AGG GAT Ala Lys Leu Asp Val Val Val Val Gly Val Leu Gly Gln Ala Arg Asp 135 140 145 150 CGC CTA CCC CAA ATG TCC AGT CCT ATG ATC TCA TCC CAC GCC GAT ATC Arg Leu Pro Gln Met Ser Ser Pro Met Ile Ser Ser His Ala Asp Ile 155 160 165 AAG TTG TCA TTA AAA AAC TTT AAA GCA TTA GTA TAT CAC GTG GGA GAT Lys Leu Ser Leu Lys Asn Phe Lys Ala Leu Val Tyr His Val Gly Asp 170 175 180 ACT ATC AAT GTC TCG ACG GCG GTT ATA CTA GGA CCT TCT CCG GAG ATA 24 8154 8202 8250 8298 8346 8399 8459 8511 8559 8607 8655 8703 8751 8799 8847 8895 8943 8991 9039 9087 9135 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 Thr Ile Asn 185 TTC ACA TTG Phe Thr Leu 200 AAG TTC GTC Lys Phe Val 215 GAG TGT ATT Glu Cys Ile Val Ser Thr Ala Val Ile Leu Gly Pro Ser Pro Glu Ile 190 195 GAA TTT AGG GTG TTG TTC CTC CGT TAT AAT CCA ACG TGC Glu Phe Arg Val Leu Phe Leu Arg Tyr Asn Pro Thr Cys 205 210 ACG ATT TAT GAA CCT TGT ATA TTT CAC CCC AAA GAA CCA Thr Ile Tyr Glu Pro Cys Ile Phe His Pro Lys Glu Pro 220 225 230 ACT ACT GCA GAA CAA TCG GTA TGT CAT TTC GCA TCC AAC Thr Thr Ala Glu Gln Ser Val Cys His Phe Ala Ser Asn 235 240 245 ATT GAC ATT CTG CAG ATA GCC GCC GCA CGT TCT GAA AAT TGT AGC ACA Ile Asp Ile Leu Gln Ile Ala Ala Ala Arg Ser Glu Asn Cys Ser Thr 250 255 260 GGG TAT CGT AGA TGT ATT TAT GAC ACG GCT ATC GAT GAA TCT GTG CAG Gly Tyr Arg Arg Cys Ile Tyr Asp Thr Ala Ile Asp Glu Ser Val Gln 265 270 275 GCC AGA TTA ACA TTC ATA GAA CCA GGA ATT CCT TCC TTT AAA ATG AAA Ala Arg Leu Thr Phe Ile Glu Pro Gly Ile Pro Ser Phe Lys Met Lys 280 285 290 GAT GTC CAG GTA GAC GAT GCT GGA TTG TAT GTG GTT GTG GCT TTA TAC Asp Val Gln Val Asp Asp Ala Gly Leu Tyr Val Val Val Ala Leu Tyr 295 300 305 310 AAT GGA CGT CCA AGT GCA TGG ACT TAC ATT TAT TTG TCA ACG GTG GAA Asn Gly Arg Pro Ser Ala Trp Thr Tyr Ile Tyr Leu Ser Thr Val Glu 315 320 325 ACA TAT CTT AAT GTA TAT GAA AAC TAC CAC AAG CCG GGA TTT GGG TAT Thr Tyr Leu Asn Val Tyr Glu Asn Tyr His Lys Pro Gly Phe Gly Tyr 330 335 340 AAA TCA TTT CTA CAG AAC AGT AGT ATC GTC GAC GAA AAT GAG GCT AGC Lys Ser Phe Leu Gln Asn Ser Ser Ile Val Asp Glu Asn Glu Ala Ser 345 350 355 GAT TGG TCC AGC TCG TCC ATT AAA CGG AGA AAT AAT GGT ACT ATC ATT Asp Trp Ser Ser Ser Ser Ile Lys Arg Arg Asn Asn Gly Thr Ile Ile 360 365 370 TAT GAT ATT TTA CTC ACA TCG CTA TCA ATT GGG GCG ATT ATT ATC GTC Tyr Asp Ile Leu Leu Thr Ser Leu Ser Ile Gly Ala Ile Ile Ile Val 375 380 385 390 ATA GTA GGG GGT GTT TGT ATT GCC ATA TTA ATT AGG CGT AGG AGA CGA Ile Val Gly Gly Val Cys Ile Ala Ile Leu Ile Arg Arg Arg Arg Arg 395 400 405 CGT CGC ACG AGG GGG TTA TTC GAT GAA TAT CCC AAA TAT ATG ACG CTA Arg Arg Thr Arg Gly Leu Phe Asp Glu Tyr Pro Lys Tyr Met Thr Leu 410 415 420 CCA GGA AAC GAT CTG GGG GGC ATG AAT GTA CCG TAT GAT AAT ACA TGC Pro Gly Asn Asp Leu Gly Gly Met Asn Val Pro Tyr Asp Asn Thr Cys 425 430 435 TCT GGT AAC CAA GTT GAA TAT TAT CAA GAA AAG TCG GCT AAA ATG AAA Ser Gly Asn Gln Val Glu Tyr Tyr Gln Glu Lys Ser Ala Lys Met Lys 440 445 450 AGA ATG GGT TCG GGT TAT ACC GCT TGG CTA AAA AAT GAT ATG CCG AAA Arg Met Gly Ser Gly Tyr Thr Ala Trp Leu Lys Asn Asp Met Pro Lys 455 460 465 470 ATT AGG AAA CGC TTA GAT TTA TAC CAC TGA TATGTACATA TTTAAACTTA Ile Arg Lys Arg Leu Asp Leu Tyr His Fin 475 ATGGGATATA GTATATGGAC GTCTATATGA CGAGAGTAAA TAAACTGACA ATGCAAATGA AGCTGATCTA TATTGTGCTT TATATTGGGA CAAACCACTC GCACAAGCTC ATTCAACACA TCCACTCTTG CTATTAAATT CCCCATTATA TAACAATACT GACATAACAC TCATATTAAG GGGAGAAAAT AAATATGCAT GGCCGATCAT ATTTTATTGA GATCCGAAAA TATATCATGC AAATAAGCAT GTTCTAGCAC CACTGCAACA TGTGGTTTAT CGATTTCCGG AAAGAATAGT TGAACCATTG CCTCCGAGCA GTTGGCGATC CGTTGACCTG CAGGTCGAC 25 9183 9231 9279 9327 9375 9423 9471 9519 9567 9615 9663 9711 9759 9807 9855 9903 9951 10001 10061 10121 10181 10241 10301 10350 ES 2 110 999 T3 (3) Información para la SEC ID N◦ : 2 5 10 15 20 25 30 35 40 45 50 55 60 (i) Caracterı́sticas de la Secuencia: (A) Longitud: 10.350 pares de bases (B) Tipo: ácido nucleico (C) Tipo de hebra: doble (D) Topologı́a: lineal (ii) Tipo de Molécula: (A) Descripción: ADN genómico (iii) HIPOTÉTICA: Si (iv) ANTISENTIDO: Si (vi) FUENTE ORIGINAL: (A) Organismo: MDV, cepa GA (vii) FUENTE INMEDIATA: (A) Biblioteca: genómica (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID N◦ :2: GTCGACCTGC CGGAAATCGA TTTCGGATCT TGTTATGTCA AGCTTGTGCG GTCAGTTTAT ATGTACATAT CCAAGCGGTA AACTTGGTTA TGGTAGCGTC ACGCCTAATT TGATAGCGAT CGAGCTGGAC TTTATACCCA TGACAAATAA CAATCCAGCA GAATGTTAAT CCCTGTGCTA GAAATGACAT ACAAGGTTCA AAATTCCAAT AGTATCTCCC GTGGGATGAG GCCAACCACA GATACCCGCA TAGGAATATA ATCGACAATA TCCTAATATC TTCGTTGCAG TGGTGGATAT TATGTTGGCC CATTATGACG TTCGTGTCTT ACCTATAATA CTTCAGTTTT ATCATATTGA TTCACCGATG CAATGCTATT TTTTAAAACG AAACAAATTA AGGTCAACGG TAAACCACAT CAATAAAATA GTATTGTTAT AGTGGTTTGT TTACTCTCGT CAGTGGTATA TAACCCGAAC CCAGAGCATG ATATATTTGG AATATGGCAA GTGAGTAAAA CAATCGCTAG AATCCCGGCT ATGTAAGTCC TCGTCTACCT CTGGCCTGCA CAATTTTCAG ACCGATTGTT TAAATCGTGA GTGAATATCT ACGTGATATA ATCATAGGAC ACAACATCCA TTTTGTAGGC TTCGGAACTC AATTTATCAG AAATCGGTAA TGATTGGGAA CGCGGGATCG GTTCCAGCTA CTGACTCGCA TAAAACAAGA GTTTCTCAAA TCCATCAACT AATGATTCGT TATATCCTAC ATACATAGTA GTATTCCACA AAAACCGTAG ATCGCCAACT GTTGCAGTGG TGATCGGCCA ATAATGGGGA CCCAATATAA CATATAGACG AATCTAAGCG CCATTCTTTT TATTATCATA GATATTCATC TACAAACACC TATCATAAAT CCTCATTTTC TGTGGTAGTT ATGCACTTGG GGACATCTTT CAGATTCATC AACGTGCGGC CTGCAGTAGT CGAACTTGCA CCGGAGAAGG CTAATGCTTT TGGACATTTG GCTTTGCAGT TAGCATTTAG CATTCAGACT TTCGTAAGAC AACAGTGAGC TCCATGTCCA TCGTTGTAGC CTTTGATCGT GACATGCACA AACGGCTCCT CCTATACACA CCTTTTCAAT TAACTGCAGA GATGGGGAGA CAATCATCGT TACCAGTCGA GTGAATAAAT GCTCGGAGGC TGCTAGAACA TGCATATTTA ATTTAATAGC AGCACAATAT TCCATATACT TTTCCTAATT CATTTTAGCC CGGTACATTC GAATAACCCC CCCTACTATG GATAGTACCA GTCGACGATA TTCATATACA ACGTCCATTG CATTTTAAAG GATAGCCGTG GGCTATCTGC AATACACTCT CGTTGGATTA TCCTAGTATA AAAGTTTTTT GGGTAGGCGA CCTCGTCCCA TATCTCCAAC TAGCGATTCT GATTTCATCC ACTTTCCAGA CGTCTCGGTG AGAATGTCCT CGTCACTATT AAATTCACAT CCCCTTATAC TTCTTCTCTT AACATCTGAA AGTGATGAGT GCGGCACCTT AGAAAAGTAA ATTCATTGCA TGTTCCCGAA AATGGTTCAA TGCTTATTTG TTTTCTCCCC AAGAGTGGAT AGATCAGCTT ATATCCCATT TTCGGCATAT GACTTTTCTT ATGCCCCCCA CTCGTGCGAC ACGATAATAA TTATTTCTCC CTACTGTTCT TTAAGATATG TATAAAGCCA GAAGGAATTC TCATAAATAC AGAATGTCAA GGTTCTTTGG TAACGGAGGA ACCGCCGTCG AATGACAACT TCCCTTGCTT TTTCTAGAAT TTGTTACTTG ATTTGTTTAA GCATACCTCA CATACATAGC TAAAGGGTCC GCAGGAACGT ATCAGGATTT ATAAAACTTA TAAGCTAACA TCCAACAATT GGAGTTTCCT GGGGCCTCAT TCACAGACCA ATAAGCCTTG TCGACATGGG TTATCGTAAA 26 CTATTCTTTC CATGATATAT TTAATATGAG GTGTTGAATG CATTTGCATT AAGTTTAAAT CATTTTTTAG GATAATATTC GATCGTTTCC GTCGTCTCCT TCGCCCCAAT GTTTAATGGA GTAGAAATGA TTTCCACCGT CAACCACATA CTGGTTCTAT ATCTACGATA TGTTGGATGC GGTGAAATAT ACAACACCCT AGACATTGAT TGATATCGGC GACCCAAAAC ACCGAATGTA CTTTCGTAGA TGGATCCCGC TGCAGGATAC CTGTTGCCGT CATCGACAAC CTATATGATT GGAAAACACA CGGCCTTGTG GGTACCACCT CGACTTTCTT TTACATTATA CAGATCTTGG AATAAATTGC GAAGGGTATA AATTGTTATT ATGGAGTCGT 60 120 180 240 300 360 420 480 540 600 660 720 780 840 900 960 1020 1080 1140 1200 1260 1320 1380 1440 1500 1560 1620 1680 1740 1800 1860 1920 1980 2040 2100 2160 2220 2280 2340 2400 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 TACAATATTA CGAGTCACTG GTCGACATAG AGATTTGCTA GAGAGATACA CAATGTTCCC GTCTCTACAG ATGTAGAACG ATAAGAACTG TTTATCTAAT TCCAGTATAA TACATACATC ATCAATGTAG GGCGGGAATA TGCGAAGGAT CTAGTGAAAT TGGATGCCTC CCTCGGGCCG TGGCCGATGT CTCTAAAACT GATGTTTACC TTCCTGTTCG TGCATAGTTT GTAATATGTC TTAGCTCACG TCATTGCATA CATTAGTAGA AGACAAACCA GCGTATATTT TTACGTGTTT ATTCATTGAG GAGAATTGTC TTATAAGAAT TATTCATACC TTTTATTTCC CATATCCCGA AAATGAGAAT CTTAAATATC TAGACCACTA CTTACGAACT TAATGGACAT TAAGGAGTTA TCTCATGCAA AGGTCCCGAA TCATTATTGC CGGGTGTTTA AAAGAGGCAA TTTTTGCAAT TTGCATATGG AATTGTTCTG GAGAACCTGA ACAGAACTAA GTTCAGGCGA GTTCATCTAA AAAATATATT ATGCTCCAAG TATCTATGTA TCCATCTATA CAATTTCACT TATTATCCCC AAACAATGTT ATATCTGTGG TCCAGCAAAT CGAATACATT CTCCTTATAA CGTATGTAAT TTTTCGGGAA CCTCTGAATA GAATAGCATT GTCCGAGTCT CCGCGGAACG ACTATAGACC GCAATCACAC TCAACTTCTT TGCCCGTCTT CCATATACCA TATTATTTGC CTCGACAGAG ATTAGTGAGT CATTTTGACG TCTCGGCAAA ACCATTTCGT CACCATGTCT GTTATCCTTT ACTCGAAATG ACTGGGTGCT TCTTCTATCC GCAGTTGGCA TGCTACTAGA CGGATCGCCA TGTCTCATAT AGTAGCCCGG TTTCTTAAGT CATTCTCGTG GAAGCTCTAA GTTATGTTCG CGATGAAAAT GAAATTTTTA TCGGGATTCT TTGCCATGGG TTATAAAAAA TCTTCCGAAT TGTATGTCTG TCCGAAAATG GGTGCCATTC AGGTAACGCG CATATGAGCG CATTAAAATA AGCATATTCA ATGTCGTAAC TGGAAATTCC GCCGTTTACT TCCTGCACTC ATTGGTTTCC TTTACATGCT TTCAGTTTTT CAAACCCCGT CGTAAACAAG AGCATGAACT CCCAAGGGTT ACGCTTTGTA ATACTGCATT AAATGGATTG TGCCTACATG TATGGGCCTG TAACAACCAT AGCTGCCTGT AAGGTTGTAC CCGTGGCGTC TATATTCTTC GGTCGCCCAG CAACAAGAGC AGATGCCTCG ACGCCGTCTA ATGCACTATA GATGGACGAC GACCCGATTA GTTCGGGATT ATTATCGGTC CCAGTGTAAC TCATTATATT TTTGAGACTT TCAAGCCACT AACAGCCGTG ATCCCCCGTC GGTTCTCCAT GCAATAATCA CACCATCCTA TATTCACGTA GCATCATATT AATGGATTCA GAATTGTCAA AGGTTTTCAT CCCATTGCGG GATGAGATAT AAGGTGCCAA ACGTTAAGCA AAGTATATAA TTCCGTTACC TCCATCGCCT AGTTCAGATT CACTTTTAGG CGTTCATTAC AACATCTATC TTCCATAATG GCAAAAAAGG TAGTATATAT GCAGTTATCG TCTTGGGCAG AGATCCATCG TGAATCGCGT AACGGATGGA TGTTTGCCAA CATATATCAG AGAGTTCCAC GCCCCAAAGT ACATCACGAT TCTATCGTAA TCGCATTTGT AATCTAATTA TTGCCACCAG CAAACATAGA CGCACAGCTG GGGAGTAATA TCCAATCAGA AAATGAACTA TCTAAATATA ATCTGATACT TCTACCATGA TATAGCAGGA ATCCCATTTC GAATAAAAGT AGATTGGTCC AAAGAGGCGG TCTGACCAAA GCGACACGCC GTCTATCATA CAATGAAACA TTTTTTCTGT GTTTAGTTAT CATCCACGGA TATCGTCGTC GTTGCATAGA GCTTCACATT TTGTACCATT TCGAAAAACT TAATAATTAT ATTTCAATTC GGGACTTTAA AATAAATTGG TGCGTCGTCC AAACAGCAAA GTGTATCAAA TACCTTTCGG ACATGTCCCC ATCTAAAAAA TATAACAGTT GTTGTTTCTT GAAACGATCT AGCGCCAATT CTTAGAGGAA TTGCCATGAA GTCCTCTGCC AGACATGCTT ATCTACCTTA CTTTATTTGC TCTGCGAATC TATAAAATGA TGTATAATGC ATGGTCTAAA TCATACCACT ACTGCTTGAA CTTGCAGGCA AAAAGGTTAT TTTTTGTACA TCCATCCATA CCCCCAATAC GTATTATACC TTATTTGATT ATTTAGGCAT TGGAGCGGTG TCACAGCTTT TATACCCTTC AAACTGCATC TTATAAATTG TAATTTTCAA GCAGAGTGAT TCGCTCATAT CTGGGTTCGA GGTTGAGCAT CAATCCATAT AAGATTTCCG TGGCCGCGTA GTTGATAACG ATCCAAAATA GAAGCTATAT CATACCAAAC CATCACCGTT AAACATGACG TGATATTTTA GTATGGTGCT GGTGGTAATG AGGTGATTTT TGTTTCGACG TTCTTCATGG GACAAAAAAA ACATGTTCCT TAAACGCGTA ATCTCGATCG TTTACAAGTT TCTCCCACAT CATCAACAGT ACTACAATTA CAGTGGAGAC ATGTAATGTG AGTTCCCAAA AATACTTTCA ATAGGAGACG GTAACTTAGT GATATATTAT GATTCACATT CGTATATATT AAATCACGAC TGCATCGAAG GAAGTAACAT CTCTCATCGT GGTCTGTGGG TTCAAAGACT ATCATTGAAG GTCAGCGGGT CTCCTGATCG CTTTCGTATA GTGTTTGCAT TCTAATTATG ATTTTTTACT GTATGGATCA ACATTTGGGT TACATTTTCA CTTTTCGTGG TAGTGGCAAT TACCATACAA AGACATTTTT CACAATGACT AGATCCGGGC AATGTCCGTC 27 CATGCAATTC AGGCCATACG TACAGGCCCA TTCTTCCAGC CAATTCTCAA ATACCACAGC ATGATGTCGC TCGTTCCTGT CATTCACCAT TAACAGATGT ATAATTGTAG AATGTCATTG ACGTGTTATA TTGCGGTGTT CATAGAACAA GGTGATTGAC TTCTCAAAGT TTATTAGTAG AAATATGAAT ACGATTGGTA GCTCTCGGGG CTGAACGGTT TTAACAGTCA TATAATCCAC ATATAACTCG CCAAATGGTT GCTCTGCCCA AAACTGAGAA TCCGTATATA GGGATTTTGT GGAATGATCG AGTAAACAGA TATCTGTATC CTATATATAT ATACGTTAGT AATGTTAGAA TCAAAGATAC TTTCACGACG TTCGGTCAGG GTCCATTTAT TGGATTGTGC TTAGAGTATA AATATGGCGT TCGAATGTTG TTATTTTTGA TCTTTAGTAA AATGTGAGCA ATCTGAGGGA AAGTTTGTAG GATCTCAGCT GACATCTCAA AGTGCAAGCA TTATCTGTAT GGTTTATCCA ATATATGCCA GGTCCCATGA ACTGTCGATT TTTAATATAT TTTCTCTTGG GGTAACGATG ACCGTTTCGG 2460 2520 2580 2640 2700 2760 2820 2880 2940 3000 3060 3120 3180 3240 3300 3360 3420 3480 3540 3600 3660 3720 3780 3840 3900 3960 4020 4080 4140 4200 4260 4320 4380 4440 4500 4560 4620 4680 4740 4800 4860 4920 4980 5040 5100 5160 5220 5280 5340 5400 5460 5520 5580 5640 5700 5760 5820 5880 5940 6000 6060 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 GGCATTCATT TCCATCGTTC CCAAAAGGTG ACAAATCTTC AGATTGGGAC TCGGGAGAGC TCTCAGTCTT CACATCGTCC ACATCCCCCG TCACTTCCGC GTTATCGGTA CTGTCGGGAA AAGTATCAAA CAACGTCGTA TCCGTACCAT TGATGCTGTT ATGTATAATT CCGTAGGTAG TATTAGTTTT AGAGTCGTGT ACTTTCGACG AAGAAATGCC ACATTCCATC GTTTCTGCCT CCGGAGTCGA AGACATCCAG TCTATTACCT AGTTTTAACC CTGTTTCATA TTCTACCAGA GTATAAGATT TGGAGATCAG ACCGGCCCAG TTATTAACAA TAAAAAAGAT TATTGGTGGA GGTGAAG ATG GGT GTG TCC ATG ATA ACT ATA GTC ACA CTT CTA GAT GAA Met Gly Val Ser Met Ile Thr Ile Val Thr Leu Leu Asp Glu 1 5 10 TGC GAT CGA TTG CCA GGA AGA TCT AGA GAT GCT GCA TCT ACT TTA TGG Cys Asp Arg Leu Pro Gly Arg Ser Arg Asp Ala Ala Ser Thr Leu Trp 15 20 25 30 ATA TTC CTT ATA AAG CAA TGT ATG GAA CAA ATA CAG GAT GAT GTG GGT Ile Phe Leu Ile Lys Gln Cys Met Glu Gln Ile Gln Asp Asp Val Gly 35 40 45 GTG CCC ATA ATC GCC AGA GCT GCA GAC CTA TTC CGT TTT GCC AAA CCC Val Pro Ile Ile Ala Arg Ala Ala Asp Leu Phe Arg Phe Ala Lys Pro 50 55 60 ATG TTA ATT CTT CCT CGG CAA CAT CGA CCG ATA GTA AGG ACA AAG CCA Met Leu Ile Leu Pro Arg Gln His Arg Pro Ile Val Arg Thr Lys Pro 65 70 75 CCA GAT GGA ACT GGA GTT CGT GGT ACC GGA TTG GCC GGA ACT AGG GAT Pro Asp Gly Thr Gly Val Arg Gly Thr Gly Leu Ala Gly Thr Arg Asp 80 85 90 TCG TTT ATA GTG CGG CTA TTT GAA GAT GTT GCA GGA TGT TCC ACA GAA Ser Phe Ile Val Arg Leu Phe Glu Asp Val Ala Gly Cys Ser Thr Glu 95 100 105 110 TGG CAG GAT GTT CTA TCT GGA TAT TTG ATG TTG GAA TCT GAA GTT TCT Trp Gln Asp Val Leu Ser Gly Tyr Leu Met Leu Glu Ser Glu Val Ser 115 120 125 GGT AAT GCT CCA CAT AGC TTG TGG ATA GTT GGG GCG GCA GAT ATA TGT Gly Asn Ala Pro His Ser Leu Trp Ile Val Gly Ala Ala Asp Ile Cys 130 135 140 GCC ATT GCG CTC GAA TGT ATT CCT TTG CCA AAA AGG TTA CTT GCA ATC Ala Ile Ala Leu Glu Cys Ile Pro Leu Pro Lys Arg Leu Leu Ala Ile 145 150 155 AAA GTG TCT GGG ACC TGG TCC GGT ATG CCG TGG GCC ATT CCC GAC AAT Lys Val Ser Gly Thr Trp Ser Gly Met Pro Trp Ala Ile Pro Asp Asn 160 165 170 ATT CAA ACT CTC TTG ACA TCT ACA TGG GAA CCG AAG TTC GAC ACC CCA Ile Gln Thr Leu Leu Thr Ser Thr Trp Glu Pro Lys Phe Asp Thr Pro 175 180 185 190 GAA GAT AGA GCG CAT TTT TGC GAC AGT GAT ATG GTA TGT GTA TAC AAA Glu Asp Arg Ala His Phe Cys Asp Ser Asp Met Val Cys Val Tyr Lys 195 200 205 ATC CTC GGG TCC CCA CCC AAT CCT CTA AAA CCT CCG GAA ATC GAA CCA Ile Leu Gly Ser Pro Pro Asn Pro Leu Lys Pro Pro Glu Ile Glu Pro 210 215 220 CCT CAA ATG AGT AGT ACA CCC GGC AGA TTA TTC TGT TGT GGA AAA TGT Pro Gln Met Ser Ser Thr Pro Gly Arg Leu Phe Cys Cys Gly Lys Cys 225 230 235 TGC AAG AAA GAA GAT AGA GAT GCG ATT GCA ATT CCG GTT CGT TAC ACT Cys Lys Lys Glu Asp Arg Asp Ala Ile Ala Ile Pro Val Arg Tyr Thr 240 245 250 GCG ACA GGA AAG TCA CGA ATA CAG AAA AAA TGT AGA GCC GGT AGT CAT Ala Thr Gly Lys Ser Arg Ile Gln Lys Lys Cys Arg Ala Gly Ser His 255 260 265 270 TAG CTGTTATTCG ACAGACCTAC TTGCTACCAA TTAGATATAA TTACATGATG Fin GGGCGTATAC ACATTACGAT TAGGTGCATC GCTACAACCG TCGCTATAGT GTCACGTATA ATTTGTATAT TAGTGCAATA ACAAACCCTT CTAGATCACT TATGTATCCA GGCTATCTTC CATATACTTC TAACATCAGG AGAGATTCAA CAATCGAGCG CATTTGAAAG ACAACG ATG 28 6120 6180 6240 6300 6360 6420 6469 6517 6565 6613 6661 6709 6757 6805 6853 6901 6949 6997 7045 7093 7141 7189 7237 7290 7350 7410 7469 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 60 AGC AGA GTC AAT GCT Ser Arg Val Asn Ala 5 CGA TTT GGT AAG CCA Arg Phe Gly Lys Pro 20 GTG GTT GTT GAT GAA Val Val Val Asp Glu 35 CGA GAG AGA TTA GGC Arg Glu Arg Leu Gly 50 ATT ACA TCG TTT GAT Ile Thr Ser Phe Asp 70 TGG TCT GCC TAT AGA Trp Ser Ala Tyr Arg 85 GAA ACT ATC ACA AAT Glu Thr Ile Thr Asn 100 TGG AGA ATA TTC TAT Trp Arg Ile Phe Tyr 115 AAA CAG GTG GGA CCA Lys Gln Val Gly Pro 130 AAC TGG ACC ACA ATG Asn Trp Thr Thr Met 150 CAC AAT ACA CGA AAT His Asn Thr Arg Asn 165 CTT AGC GGG GCA ATA Leu Ser Gly Ala Ile 180 CCT GTG TGC GAG TAT Pro Val Cys Glu Tyr 195 GGA AAT GCC ATC AAG Gly Asn Ala Ile Lys 210 ACA GAA ACT GGA ATA Thr Glu Thr Gly Ile 230 TCT GAA GAG GAG TTG Ser Glu Glu Glu Leu 245 AAC CGG GAA CAT TGC Asn Arg Glu His Cys 260 GAT CCC ACT ATT CAC Asp Pro Thr Ile His 275 ACA TCA ACA TAT CCA Thr Ser Thr Tyr Pro 290 GGA GGT CTT ATG TAC Gly Gly Leu Met Tyr 310 ACA ATG TTC GAT GAT Thr Met Phe Asp Asp 10 CCG AGA AAG ATT ACT Pro Arg Lys Ile Thr 25 TTC ACA GAA GGA ATC Phe Thr Glu Gly Ile 40 CTT TTA TGT ACC ATA Leu Leu Cys Thr Ile 55 ATA CAC AAG GAT ATG Ile His Lys Asp Met 75 TTT TTT GCC ATG ATG Phe Phe Ala Met Met 90 TTT ACA GAA ACT GAT Phe Thr Glu Thr Asp 105 AGA ACT TGG GAT GTG Arg Thr Trp Asp Val 120 TTT TTG CCC GCA TTG Phe Leu Pro Ala Leu 135 CTT TCC ATA GGA ATC Leu Ser Ile Gly Ile 155 ATG TTC ATG ACA ATA Met Phe Met Thr Ile 170 GAG GTA GCT CGA TAT Glu Val Ala Arg Tyr 185 AGA ACA CCC TTA GGC Arg Thr Pro Leu Gly 200 ACA TGC TGC ACG CAA Thr Cys Cys Thr Gln 215 TCC AAG GAC AGT GGA Ser Lys Asp Ser Gly 235 TAT TAT AGA ACC ATA Tyr Tyr Arg Thr Ile 250 ATA TCA TGC AAT ATT Ile Ser Cys Asn Ile 265 CAT CGA TCT TCT AAT His Arg Ser Ser Asn 280 TTT GGA CGC AGG CCG Phe Gly Arg Arg Pro 295 CAG CAC CCC TAC ATT Gln His Pro Tyr Ile 315 ATG GAT ATA Met Asp Ile AAT GTA AAT Asn Val Asn 30 GTT CAA TGT Val Gln Cys 45 TCT ACT AAC Ser Thr Asn 60 TGG TGT CAA Trp Cys Gln GAC AAA ATG Asp Lys Met CTT ACC GAA Leu Thr Glu 110 AGA GAT GCA Arg Asp Ala 125 TTT TCA TTT Phe Ser Phe 140 AAC AAG GGT Asn Lys Gly CAG TCT GCA Gln Ser Ala GCC GTG GTT Ala Val Val 190 CTG CCG GAT Leu Pro Asp 205 ATG CAA GCG Met Gln Ala 220 CAT AAA ATA His Lys Ile CAT GAT CTT His Asp Leu GAG AAT AGC Glu Asn Ser 270 GTC ATA ACT Val Ile Thr 285 ATG AGT CGA Met Ser Arg 300 TGC CGC AAT Cys Arg Asn 29 Met 1 CCA AGA GGA Pro Arg Gly 15 TTT TGG CAT Phe Trp His 7517 7565 ATG GAA GCC Met Glu Ala 7613 GAG GGA TCT Glu Gly Ser 65 ATG GTT ATC Met Val Ile 80 TTT TCG ATT Phe Ser Ile 95 ACT GGT CAG Thr Gly Gln 7661 TTG AAG ATG Leu Lys Met 7853 CAT CTG GAA His Leu Glu 145 TAT GAT CGA Tyr Asp Arg 160 AGA AAT GTC Arg Asn Val 175 CTT GCT CTA Leu Ala Leu 7901 GAT AGC ATA Asp Ser Ile 8093 AAT CGA TTG Asn Arg Leu 225 AAT GAT TCT Asn Asp Ser 240 ATC AAA CCT Ile Lys Pro 255 ATG GAT ATA Met Asp Ile 8141 TTA CAA GGT Leu Gln Gly 8333 ATG GAT GTT Met Asp Val 305 CTC CAT TTA Leu His Leu 320 8381 7709 7757 7805 7949 7997 8045 8189 8237 8285 8429 ES 2 110 999 T3 5 10 15 20 25 30 35 CGT CCG CCT CGA TCC AGA CTA ATG AAT AGT AAA Arg Pro Pro Arg Ser Arg Leu Met Asn Ser Lys 325 330 AGA CAA AGT TTC AAT CGA AGT AAT CCT CAT GCA Arg Gln Ser Phe Asn Arg Ser Asn Pro His Ala 340 345 TACATACAAT CATGACAACA CTGTAATGCC TTATTGAAAA AACGTTAGTA GCAGTTTTTC CTAAAATCCT ATTAATAATT GATTCCCCGT CTCCTGTTGG CGATTCCCGA AGATTTGTCA TCATCACCGA TTGCTGCATT CCCCTTAGTA GCGACGGCAC GACTCTATTT CGGGGAGTGG ACTTATTCCA CAGCCCGTTG AGACGGACAT TCTTCTCATA TAAGCGCGAA ACAGTACAGT CACCAATACA TCATGATAGT AAACCGAGTG TCCATTTCTT TCAATACACC GTAAAGCCCG GCCTACAATT TTTCTCGTCG GAAGCAGAAA TATCATATTC GTTAAGCGTG ACAGTCATTC AAACGAGACG ATGGTGGTTT TCCAGCTTTC ATAGCAGCCT AATATGGTCC TGGCTAGACT GCGTACAGAT TTAGGCAATA GCAGAAAGTA TATCATAACT CTGTTCTTTT GGAGAATCTA CTAGATTTGC GCCGTAGAGA CCACATGGCC ATACCTCTCC GGCGCTCGTC TCCAGTATTC AAACACGTTC TCCTCTCATT TATCTTCATA TACAACAAAA AGGCAACACG TTATTTGACA GGGTCAGCGT TTGTTGCAAC AGATCATGAC AAATAAATCC ATTAGATAGA TCAAAGAATG TCGGCTCTAT GTCTAACAAT TTTCTCTTCA AGTCTGGATA GTTAATGCAA TTTACTGTCT TTTTACATCG CAGTCTGAAG AAATAGTTCC CGAGGACGCA ATCGGAGGCA TATATATCGG ATATAGGATG GGCGCTTCGA CCTGCGCAGA TGTAGACTTT GGCGTGGGGT CAAATTCATG TTTTAGTCGA TATCCCATTC GACCAATCAT ATGAATATCG ATCGTGGAAC GGACCGCTAT TTAGTCGACA TCGAATAAAA ATCCGCACAT AACCGAGCGA CATCGGGTTT CCATGGTAGA ATTAAGTTCA AAGTCTTGAT CGGACGAGTC ACTGCCATAT TTGATAATCT TCGTCGTCGC TCTCATTATC TGACGATGAT TCGATCTCGA TCACGACTCA TGTTGCCTCC GATGGAGCCG GTGTAATTTG GAGACTTTGG CCTGTATTAT ATAGCTACCA ACAATAATTG CTAGAATTTA CATCACGTGA TATCCGGTCA CCAGCCCCTA ATGTACTACT TGCTCTATAT ATTCTCCACA AAGATTTCAC TCCAATTTCA AGGAATTC ATC CTA CAG ACA TTT Ile Leu Gln Thr Phe 335 TAC CCC ATA TAA Tyr Pro Ile Fin 350 TAAAATTTTA TTATTTAAAC GTGCGATTAG TTATAAGTAG GATAATGTGC CAATTCAGCA GACATAAAGG TTTCCAATAA CCGAACCTAC TATGTCCATA ATCCAGCATG TCCAAGACAA CGTGTGTAAG AGGAGCACGT GGTCTGTCGG GTGGAATGGC TGTCGAATAT CTCACCCCAC GGGAGATCGT AGCGGCGGTT GCGCAACATG TTCCCCGCCG CCCGGAGTTG CACACTCCTG AATATGTTTT AATCTTACAC AGGCTCAACG CCACATTAAA CGCCCCTTCA TGGATGGGGG AAAATCTATT ATTTTATCTC TAAAATTATA TAATAAGAGC ACCGACAAAT CGTTCATTCC GCGATTGGGT GAAAAATGCT CTATCAGCAT CCCTCAGAGT ATAGTTTCCC ATTCGGCTTG AATAGTGCTC TCCGAAGAGC CATCGAAATA GTTTGTTTGT GGACAAAATT TGCCCACATT TCCGGATGTG AATGTGGCAG AATCGTGTAT CGGGTCTGGC AAAGCAGGTT TTCTGCTCAA GCTTTTATCT TCTGCTAGGA AAAATTACTT GGTCTTTAAC ATGGTAAACC TCCCTCCCTA (4) Información para la SEC ID N◦ : 3 40 45 (i) Caracterı́sticas de la Secuencia: (A) (B) (C) (D) Longitud: 497 aminoácidos Tipo: péptido Tipo de Hebra: sencilla Topologı́a: lineal (ii) Tipo de Molécula: (A) Descripción: polipéptido 50 (iv) FUENTE ORIGINAL: (A) Organismo: MDV, cepa GA (vii) FUENTE INMEDIATA: 55 (A) Biblioteca: genómica (xi) DESCRIPCIÓN DE LA SECUENCIA: SEC ID N◦ :3: 60 Met Cys Val Phe Gln Ile Leu Ile Ile Val Thr Thr Ile Lys Val Ala -15 -10 -5 Gly Thr Ala Asn Ile Asn His Ile Asp Val Pro Ala Gly His Ser Ala 1 5 10 30 8477 8522 8582 8642 8702 8762 8822 8882 8942 9002 9062 9122 9182 9242 9302 9362 9422 9482 9542 9602 9662 9722 9782 9842 9902 9962 10022 10082 10142 10202 10262 10322 10350 ES 2 110 999 T3 5 10 15 20 25 30 35 40 45 50 55 Thr Thr Thr Ile Pro 15 Thr Glu Thr Trp Thr 35 Gly Tyr Val Cys Leu 50 Gly Val Ser Cys Met 65 Lys Phe Ile Val Asp 80 Leu Asn Gly Val Pro 95 Leu Glu Ile Leu Asn 115 Tyr Ser Arg Asn Gly 130 Gly Val Leu Gly Gln 145 Met Ile Ser Ser His 160 Ala Leu Val Tyr His 175 Ile Leu Gly Pro Ser 195 Phe Leu Arg Tyr Asn 210 Cys Ile Phe His Pro 225 Ser Val Cys His Phe 240 Ala Arg Ser Glu Asn 255 Thr Ala Ile Asp Glu 275 Gly Ile Pro Ser Phe 290 Leu Tyr Val Val Val 305 Tyr Ile Tyr Leu Ser 320 Tyr His Lys Pro Gly 335 Ile Val Asp Glu Asn 355 Arg Arg Asn Asn Gly 370 Ser Ile Gly Ala Ile 385 Ile Leu Ile Arg Arg 400 Glu Tyr Pro Lys Tyr 415 Asn Val Pro Tyr Asp 435 Gln Glu Lys Ser Ala 450 Trp Leu Lys Asn Asp 465 His Fin Arg Tyr Pro Pro Val 20 Trp Ile Pro Asn His 40 Glu Ser Ala His Cys 55 Arg Tyr Ala Asp Glu 70 Ala Gly Ser Ile Lys 85 Asn Ile Phe Leu Ser 100 Ala Ser Leu Gln Asn 120 Thr Arg Thr Ala Lys 135 Ala Arg Asp Arg Leu 150 Ala Asp Ile Lys Leu 165 Val Gly Asp Thr Ile 180 Pro Glu Ile Phe Thr 200 Pro Thr Cys Lys Phe 215 Lys Glu Pro Glu Cys 230 Ala Ser Asn Ile Asp 245 Cys Ser Thr Gly Tyr 260 Ser Val Gln Ala Arg 280 Lys Met Lys Asp Val 295 Ala Leu Tyr Asn Gly 310 Thr Val Glu Thr Tyr 325 Phe Gly Tyr Lys Ser 340 Glu Ala Ser Asp Trp 360 Thr Ile Ile Tyr Asp 375 Ile Ile Val Ile Val 390 Arg Arg Arg Arg Arg 405 Met Thr Leu Pro Gly 420 Asn Thr Cys Ser Gly 440 Lys Met Lys Arg Met 455 Met Pro Lys Ile Arg 470 Val Asp Gly Thr Leu 25 Cys Asn Glu Thr Ala 45 Phe Thr Asp Leu Ile 60 Ile Val Leu Arg Thr 75 Gln Ile Glu Ser Leu 90 Thr Lys Ala Ser Asn 105 Ala Gly Ile Tyr Ile 125 Leu Asp Val Val Val 140 Pro Gln Met Ser Ser 155 Ser Leu Lys Asn Phe 170 Asn Val Ser Thr Ala 185 Leu Glu Phe Arg Val 205 Val Thr Ile Tyr Glu 220 Ile Thr Thr Ala Glu 235 Ile Leu Gln Ile Ala 250 Arg Arg Cys Ile Tyr 265 Leu Thr Phe Ile Glu 285 Gln Val Asp Asp Ala 300 Arg Pro Ser Ala Trp 315 Leu Asn Val Tyr Glu 330 Phe Leu Gln Asn Ser 345 Ser Ser Ser Ser Ile 365 Ile Leu Leu Thr Ser 380 Gly Gly Val Cys Ile 395 Thr Arg Gly Leu Phe 410 Asn Asp Leu Gly Gly 425 Asn Gln Val Glu Tyr 445 Gly Ser Gly Tyr Thr 460 Lys Arg Leu Asp Leu 475 60 31 Tyr 30 Thr Leu Asp Ser Lys 110 Arg Val Pro Lys Val 190 Leu Pro Gln Ala Asp 270 Pro Gly Thr Asn Ser 350 Lys Leu Ala Asp Met 430 Tyr Ala Tyr ES 2 110 999 T3 REIVINDICACIONES 5 1. Un método para preparar un segmento de ADN con un gen que codifica el precursor de la glicoproteı́na E (gE) del MDV proporcionando una secuencia de ADN situada entre los nucleótidos 8488 y 9978 de la secuencia del ADN del herpesvirus de la enfermedad de Marek, identificada como parte de la SEC ID N◦ :1 y conteniendo secuencias promotoras potenciales hasta 400 nucleótidos 5’ del gen ası́ como subfragmentos del ADN, que reconocen selectivamente el ADN cuando está en forma de sonda, de una manera conocida per se. 10 2. El método de acuerdo con la reivindicación 1, en el que la glicoproteı́na codificada contiene 497 aminoácidos. 3. Un método para preparar un precursor de la glicoproteı́na gE, en el que una proteı́na que comprende la secuencia 15 20 25 30 35 40 45 50 55 60 Met Cys Val Phe -15 Gly Thr Ala Asn 1 Thr Thr Thr Ile 15 Thr Glu Thr Trp Gly Tyr Val Cys 50 Gly Val Ser Cys 65 Lys Phe Ile Val 80 Leu Asn Gly Val 95 Leu Glu Ile Leu Tyr Ser Arg Asn 130 Gly Val Leu Gly 145 Met Ile Ser Ser 160 Ala Leu Val Tyr 175 Ile Leu Gly Pro Phe Leu Arg Tyr 210 Cys Ile Phe His 225 Ser Val Cys His 240 Ala Arg Ser Glu 255 Thr Ala Ile Asp Gly Ile Pro Ser 290 Leu Tyr Val Val 305 Tyr Ile Tyr Leu 320 Tyr His Lys Pro 335 Gln Ile Leu Ile Ile -10 Ile Asn His Ile Asp 5 Pro Arg Tyr Pro Pro 20 Thr Trp Ile Pro Asn 35 Leu Glu Ser Ala His 55 Met Arg Tyr Ala Asp 70 Asp Ala Gly Ser Ile 85 Pro Asn Ile Phe Leu 100 Asn Ala Ser Leu Gln 115 Gly Thr Arg Thr Ala 135 Gln Ala Arg Asp Arg 150 His Ala Asp Ile Lys 165 His Val Gly Asp Thr 180 Ser Pro Glu Ile Phe 195 Asn Pro Thr Cys Lys 215 Pro Lys Glu Pro Glu 230 Phe Ala Ser Asn Ile 245 Asn Cys Ser Thr Gly 260 Glu Ser Val Gln Ala 275 Phe Lys Met Lys Asp 295 Val Ala Leu Tyr Asn 310 Ser Thr Val Glu Thr 325 Gly Phe Gly Tyr Lys 340 Val Thr Thr Ile Lys -5 Val Pro Ala Gly His 10 Val Val Asp Gly Thr 25 His Cys Asn Glu Thr 40 Cys Phe Thr Asp Leu 60 Glu Ile Val Leu Arg 75 Lys Gln Ile Glu Ser 90 Ser Thr Lys Ala Ser 105 Asn Ala Gly Ile Tyr 120 Lys Leu Asp Val Val 140 Leu Pro Gln Met Ser 155 Leu Ser Leu Lys Asn 170 Ile Asn Val Ser Thr 185 Thr Leu Glu Phe Arg 200 Phe Val Thr Ile Tyr 220 Cys Ile Thr Thr Ala 235 Asp Ile Leu Gln Ile 250 Tyr Arg Arg Cys Ile 265 Arg Leu Thr Phe Ile 280 Val Gln Val Asp Asp 300 Gly Arg Pro Ser Ala 315 Tyr Leu Asn Val Tyr 330 Ser Phe Leu Gln Asn 345 32 Val Ala Ser Ala Leu Tyr 30 Ala Thr 45 Ile Leu Thr Asp Leu Ser Asn Lys 110 Ile Arg 125 Val Val Ser Pro Phe Lys Ala Val 190 Val Leu 205 Glu Pro Glu Gln Ala Ala Tyr Asp 270 Glu Pro 285 Ala Gly Trp Thr Glu Asn Ser Ser 350 ES 2 110 999 T3 5 10 15 Ile Val Asp Glu Asn 355 Arg Arg Asn Asn Gly 370 Ser Ile Gly Ala Ile 385 Ile Leu Ile Arg Arg 400 Glu Tyr Pro Lys Tyr 415 Asn Val Pro Tyr Asp 435 Gln Glu Lys Ser Ala 450 Trp Leu Lys Asn Asp 465 His Fin Glu Ala Ser Asp Trp 360 Thr Ile Ile Tyr Asp 375 Ile Ile Val Ile Val 390 Arg Arg Arg Arg Arg 405 Met Thr Leu Pro Gly 420 Asn Thr Cys Ser Gly 440 Lys Met Lys Arg Met 455 Met Pro Lys Ile Arg 470 Ser Ser Ser Ser Ile 365 Ile Leu Leu Thr Ser 380 Gly Gly Val Cys Ile 395 Thr Arg Gly Leu Phe 410 Asn Asp Leu Gly Gly 425 Asn Gln Val Glu Tyr 445 Gly Ser Gly Tyr Thr 460 Lys Arg Leu Asp Leu 475 Lys Leu Ala Asp Met 430 Tyr Ala Tyr es preparada de una manera conocida per se por tecnologı́a de ADN recombinante. 20 4. Un método para reducir la patogenicidad o virulencia de un herpesvirus de la enfermedad de Marek por el que se altera un gen que codifica la glicoproteı́na E. 25 30 35 5. Un método para producir un vector vı́rico vacuna, para utilización in vivo, o un vector de expresión in vitro, para una proteı́na que produce anticuerpos contra la enfermedad de Marek, que comprende la etapa de insertar en la vacuna o vector un segmento de ADN que contiene todo o parte de un gen que codifica la glicoproteı́na gE del herpesvirus de la enfermedad de Marek. 6. Un método para proporcionar genes foráneos a un herpesvirus de la enfermedad de Marek, que comprende la inserción del gen foráneo en una región del ADN del herpesvirus que codifica la proteı́na gE no esencial. 7. Un método para preparar un segmento de ADN, en el que el segmento de ADN codifica una parte del precursor de la glicoproteı́na E (gE) del MDV situado entre los nucleótidos 8488 y 8799 de la secuencia de ADN del herpesvirus de la enfermedad de Marek e identificado como parte de la SEC ID N◦ :1, donde el segmento de ADN contiene opcionalmente una región reguladora 5’ con el gen de hasta 400 pb de longitud según se muestra en la Fig. 2, y subfragmentos del ADN que reconocen selectivamente el ADN cuando está en forma de sonda, en el que el segmento de ADN es preparado de una manera conocida per se. 40 8. Un método para preparar un precursor de la glicoproteı́na gE, en el que una proteı́na que comprende la secuencia: 45 50 55 60 Met Cys Val Phe -15 Gly Thr Ala Asn 1 Thr Thr Thr Ile 15 Thr Glu Thr Trp Gly Tyr Val Cys 50 Gly Val Ser Cys 65 Lys Phe Ile Val 80 Gln Ile Leu Ile Ile -10 Ile Asn His Ile Asp 5 Pro Arg Tyr Pro Pro 20 Thr Trp Ile Pro Asn 35 Leu Glu Ser Ala His 55 Met Arg Tyr Ala Asp 70 Asp Ala Gly Ser 85 Val Thr Thr Ile Lys -5 Val Pro Arg Gly His 10 Val Val Asp Gly Thr 25 His Cys Asn Glu Thr 40 Cys Phe Thr Asp Leu 60 Glu Ile Val Leu Arg 75 Val Ala Ser Ala Leu Tyr 30 Ala Thr 45 Ile Leu Thr Asp es preparada de una manera conocida per se. 9. Un método para preparar una región reguladora 5’ para la glicoproteı́na gE, en el que la secuencia situada entre los nucleótidos 8088 y 8487 de la secuencia mostrada en la Fig. 2 es preparada de una 33 ES 2 110 999 T3 manera conocida per se. 5 10. Un método para producir una sonda que tiene especificidad para el ADN del herpesvirus de la enfermedad de Marek, en el que una sonda que reconoce selectivamente el segmento de ADN de la reivindicación 1 es preparada de una manera conocida per se. 10 15 20 25 30 35 40 45 50 55 60 NOTA INFORMATIVA: Conforme a la reserva del art. 167.2 del Convenio de Patentes Europeas (CPE) y a la Disposición Transitoria del RD 2424/1986, de 10 de octubre, relativo a la aplicación del Convenio de Patente Europea, las patentes europeas que designen a España y solicitadas antes del 7-10-1992, no producirán ningún efecto en España en la medida en que confieran protección a productos quı́micos y farmacéuticos como tales. Esta información no prejuzga que la patente esté o no incluı́da en la mencionada reserva. 34