Clase 7 vier 1-4-11
Anuncio
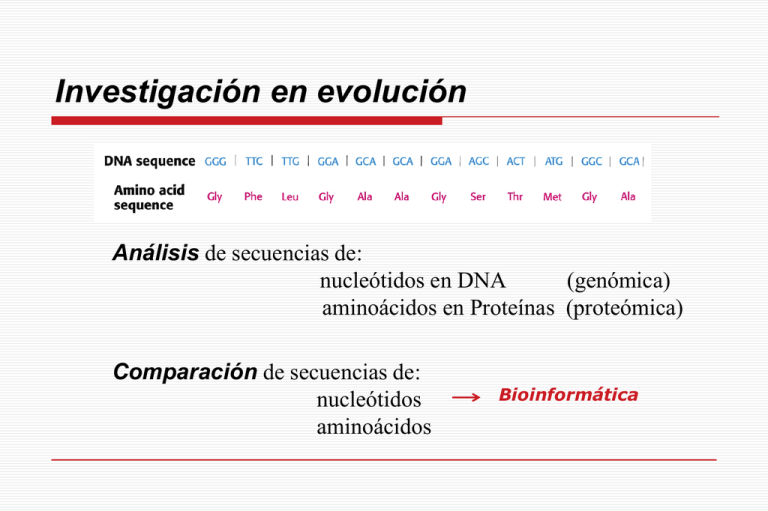
Investigación en evolución Análisis de secuencias de: nucleótidos en DNA (genómica) aminoácidos en Proteínas (proteómica) Comparación de secuencias de: nucleótidos aminoácidos Bioinformática Análisis de secuencias nucleótidos en DNA 1- Patrón de fragmentos de restricción 2’,3’- dd de CTP 2’,3’- dd de ATP a) Hidrólisis catalizada por enzimas de restricción b) Electroforesis 2- Secuenciación de ADN (Método de Sanger) (interrupción controlada de la replicación) 1 hebra de interés de ADN + + + + ADN polimerasa ++ ++ Cebador ++ ++ 4 NTPs ++ ++ 2’,3’- didesoxi de ATP + - - 2’,3’- didesoxi de TTP - + - 2’,3’- didesoxi de GTP - -+ 2’,3’- didesoxi de CTP - -- + 2’,3’- dd de GTP 2’,3’- dd de TTP Cebador 3’ GTCGATGATACGACT 5’ 5’ CAGCTACTATGCTGA 3’ Las nuevas hebras de DNA son separadas y sometidas a electroforesis Determinación de secuencia de aa en una proteína Obtención de péptidos pequeños por hidróslisis enzimática En cada péptido 1- Cuantificación de aa 2- Determinación de aa N-terminal 3- Secuenciación Obtención de péptidos pequeños por hidróslisis enzimática y no enzimática Obtención de péptidos pequeños por hidróslisis enzimática. Ejemplo 1- Cuantificación de aa: (determina cuáles y cuánto de c/aa pero no la secuencia) a) Hidrólisis ácida del péptido; b) derivatización (por ej. con nihindrina, fluorescamina); c) HPLC para separación y cuantificación de los derivados de aa derivatizados 2- Determinación del aa N-terminal (marcaje c/cloruro de dabsilo, hidrólisis) 3- Secuenciación (Met. Químico= Degradación de Edman) (Mét.Físico= MALDI-TOFF) Análisis de secuencias de aa en Proteínas Secuenciación de Edman separa el residuo amino terminal y se obtiene PTH-aa MALDI-TOFF Espectrometría de masas Investigación de la evolución Similitud en la estructura Antecesor común? ortólogos (homólogos presentes en distintos organismos igual función y que han evolucionado mediante descendencia directa) parálogos (homólogos presentes en un mismo organismo que codifican proteínas con similar o distinta función,). Análisis de homologías: Análisis estadístico de secuencias alineadas Contar número de identidades para cada alineamiento Técnicas de calificación de homologías: - Identidades: +10 puntos -Inseción de huecos (para compensar desajustes por deleciones de nucleótidos): -25 puntos El alineamiento es significativo? Alineamientos para muchas secuencias barajadas Hemoglobina y mioglobina humanas Análisis para secuencias verdaderas La homologia del 25,9% es estadisticamente significativa Matriz de sustitución BLOSUM-62 Para análisis de parentescos lejanos Se analiza sustituciones en bloques de secuencias alineadas Enmarcado (arriba): aa sustituido Rojo: aa cargados Verde: aa polares Azul: aa grandes e hidrofobicos Negro: otros Alineamiento con inserción de un hueco. 147 aa totales, 38 aa emparejados – 25,9% similitud Alineamientos con anotación de sustituciones “conservadoras” (en amarillo), poseen calificación superior a cero s/matriz Blosum-62 Distribución de calificaciones de alineamientos de mioglobina humana y leghemoglobina de altramuz P=1/20 P=1/300 p-= probabilidad de que alineamiento sea debido al azar Blosum 62 aporta mayor valor estadístico IDENTIDADES >25% Secuencias probablemente homólogas 15-25%: análisis de significación estadística del alineamiento <15% no descarta la posible homología (analizar estructuras tridimensionales). Comparación de estructuras: terciaria se conserva más que la primaria Chaperona Citoesqueleto Homología en secuencia (estr.1ria) sólo del 15.6% pero alta en estr.3ria Alinear secuencias consigo mismas permiten detectar motivos repetidos GRAFICO AUTODIAGONAL Puntos: aa alineados Diagonal: alineamiento consigo misma Lineas // a la diagonal: repeticiones El gen que codifica a la proteína evolucionó mediante la duplicación del gen que codificaba a un único dominio. EVOLUCION: Divergente: mismo ancestro Convergente: no tienen antepasado común pero evolucionan hacia una estructura semejante cuando realizan igual actividad bioquimica Proteasas de serina: En el sitio activo confluyen Ser, His,Asp En el resto, las estructuras son muy diferentes entre si. ANALISIS DE SECUENCIAS DE RNA Nucleotidos en posiciones 9 y 22 mantienen capacidad de formar parejas de Watson y Crick CONSTRUCCION DE ARBOLES EVOLUTIVOS 1- Analisis de homologías 2- Calibrar tiempos de divergencia con registros de fósiles Comparacion de secuencias de DNA PCR+secuenciación EVOLUCION EN EL LABORATORIO RNAs sintetizados por química combinatoria - Moleculas de RNA que unen ATP con alta afinidad se transcriben inversamente a DNA - Amplificar por PCR - Transcripción a RNA - Despues de 8 generaciones: secuenciación - 17 secuencias; 16 con igual estructura; Afinidad KdATP= 50 µM MOLECULA DE RNA QUE SE UNE AL ATP Estructura secundaria conservada Dos regiones helicoidales con bases emparejadas de Watson y Crick Hueco adonde puede unirse el anillo de adenina











