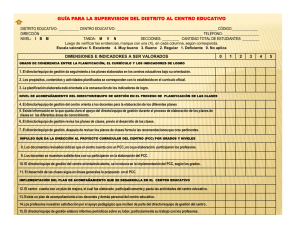
universidad de chile
Anuncio

UNIVERSIDAD DE CHILE Campus Sur Instituto de Nutrición y Tecnología de los Alimentos Facultad de Ciencias Agronómicas Facultad de Ciencias Forestales Facultad de Ciencias Veterinarias y Pecuarias PROGRAMA DE DOCTORADO EN CIENCIAS SILVOAGROPECUARIAS Y VETERINARIAS “ANÁLISIS MOLECULAR DE LOS GENES all1765 Y all2320 QUE CODIFICAN PROTEÍNAS EXCLUSIVAS DE CIANOBACTERIAS FILAMENTOSAS” DINKA MANDAKOVIC SEYLER Tesis para optar al Grado Académico de Doctor en Ciencias Silvoagropecuarias y Veterinarias PROFESOR GUÍA: DRA. LUZ MÓNICA VÁSQUEZ PÉREZ SANTIAGO, CHILE 2013 PROGRAMA DE DOCTORADO EN CIENCIAS SILVOAGROPECUARIAS Y VETERINARIAS “ANÁLISIS MOLECULAR DE LOS GENES all1765 Y all2320 QUE CODIFICAN PROTEÍNAS EXCLUSIVAS DE CIANOBACTERIAS FILAMENTOSAS” DINKA MANDAKOVIC SEYLER Tesis para optar al Grado Académico de Doctor en Ciencias Silvoagropecuarias y Veterinarias COMITÉ DE TESIS Calificación Dra. Luz Mónica Vásquez Pérez (Director de Tesis) Dr. Jaime Romero (Consejero) Dr. José Pizarro (Consejero) Dr. Roberto Vidal (Consejero) Dr. Guillermo Figueroa (Presidente Comisión Examen de Grado) Financiamiento CONICYT Número de registro SANTIAGO, CHILE 2013 Firma AGRADECIMIENTOS El desarrollo de esta tesis estuvo lleno de las más diversas emociones, sin dejar muchos minutos para rutinas o aburrimientos. El deseo de experimentar estas diversas sensaciones de forma frecuente en mi vida fue una de las razones principales por las cuales decidí ser científico. Por ese motivo, mi primer agradecimiento es a mis padres, quienes no sólo me permitieron soñar con esta idea de ser una científica “loca”, sino que me ayudaron a hacer este sueño realidad. Quiero agradecer en especial a mi mamá, por ser un ejemplo a seguir en el ámbito profesional y familiar, y por estar siempre presente cuando más te he necesitado. Gracias por ser MI mamá. Quiero agradecer a mi familia, especialmente a mis hermanos, que confiaron siempre en mí y me apoyaron en la idea de seguir perfeccionándome. Gracias por hacerme sentir orgullosa de mis logros. Quiero agradecer a la Dra. Mónica Vásquez, por ser una guía excepcional y muy dedicada. Gracias por el apoyo y la confianza permanente. Quiero agradecer a los integrantes y amigos de los MV y BD labs, por la ayuda siempre dispuesta y especialmente por las notables conversas y pelambres durante los almuerzos. Las extrañaré chiquillas. Quiero agradecer a mis amigas de la vida, en especial a la Pati, a la Dani, a la ovejita Paula y a la Panchita, porque siempre están ahí. ¡Las adoro! Quiero agradecer a la mayor alegría que nació durante el desarrollo de esta tesis, mi proyecto más satisfactorio y el que me ha brindado más enseñanzas, mi Ferrán Valentino. Gracias por haber llegado a mi vida. Te amo hijo. Finalmente, quiero agradecer a mi Negrito, por ser mi amor, mi soporte y mi crítico más constructivo. Gracias por acompañarme en este proceso, y aprovecho de agradecerte por hacerme inmensamente feliz junto con la Francisquita, niña hermosa por dentro y por fuera y hermana excepcional. Este tesis fue realizada gracias a la ayuda financiera de: Beca de Doctorado CONICYT, Beca de Apoyo tesis Doctoral CONICYT, Becas de ayuda viajes del Programa de Doctorado de Ciencias Silvoagropecuarias y Veterinarias de la Universidad de Chile. ÍNDICE RESUMEN ............................................................................................................................................................... 4 ABSTRACT ............................................................................................................................................................... 5 INTRODUCCIÓN ...................................................................................................................................................... 6 REVISIÓN BIBLIOGRÁFICA ....................................................................................................................................... 8 1. COMUNICACIÓN Y ADHESIÓN INTERCELULAR EN ORGANISMOS MULTICELULARES. .............................................................. 8 2. MULTICELULARIDAD EN CIANOBACTERIAS. ............................................................................................................... 9 2.1 Transferencia molecular mediante periplasma continuo ........................................................................... 12 2.2 Comunicación directa entre células del filamento por proteínas localizadas en conexiones intercelulares .. 13 2.2.1 sepJ.................................................................................................................................................................... 15 2.2.2 fraD.................................................................................................................................................................... 18 3. GENES EXCLUSIVOS DE CIANOBACTERIAS FILAMENTOSAS. .............................................................................................. 21 HIPÓTESIS ............................................................................................................................................................. 23 OBJETIVO GENERAL .............................................................................................................................................. 23 OBJETIVOS ESPECÍFICOS........................................................................................................................................ 23 MATERIALES Y MÉTODOS ..................................................................................................................................... 24 1. CEPAS BACTERIANAS Y CONDICIONES DE CRECIMIENTO DE LOS CULTIVOS ........................................................................... 24 2. RT-PCR EN TIEMPO REAL Y ANÁLISIS NORTHERN BLOT PARA MEDIR EXPRESIÓN GÉNICA ........................................................ 24 2.1 Análisis Northern blot ............................................................................................................................... 24 2.2 Análisis RT-PCR en tiempo real.................................................................................................................. 25 3. GENERACIÓN DE PROTEÍNAS DE FUSIÓN CON GFP Y DE ANTICUERPOS PARA LOCALIZAR LAS PROTEÍNAS EN LA CÉLULA ................... 26 3.1 Generación de proteínas de fusión con GFP............................................................................................... 26 3.1.1 All1765- GFP-mut2 y All1765-sf-GFP.................................................................................................................... 26 3.1.2 All2320- GFP-mut2 ............................................................................................................................................. 28 3.2 Generación de anticuerpos ....................................................................................................................... 29 3.2.1 Generación de anticuerpo anti-All1765 ............................................................................................................... 30 3.2.2 Generación de anticuerpo anti-All2320 ............................................................................................................... 30 3.2.3 Análisis Western Blot .......................................................................................................................................... 31 3.2.4 Análisis por inmunofluorescencia ........................................................................................................................ 32 3.3 Microscopía confocal................................................................................................................................ 32 4. GENERACIÓN DE CEPAS MUTANTES Y ANÁLISIS DE SUS RESPECTIVOS FENOTIPOS .................................................................. 33 4.1 Generación de cepas mutantes ................................................................................................................. 33 4.1.1 Cepa mutante del gen all1765 por deleción (Δall1765) ........................................................................................ 33 4.1.2 Cepa mutante del gen all2320 por interrupción con cassette de resistencia a antibiótico (all2320::C.S3) .............. 35 4.2 Microscopía óptica ................................................................................................................................... 38 4.3 Microscopía electrónica de transmisión .................................................................................................... 38 RESULTADOS......................................................................................................................................................... 40 1.- ALL1765 ......................................................................................................................................................... 40 1.1 Analizar la presencia y abundancia del transcrito de all1765 e identificar la localización subcelular de la proteína All1765 en Anabaena sp. PCC 7120. .................................................................................................. 40 1.2 Determinar el papel de All1765 en la estructura del filamento y en la diferenciación celular de Anabaena sp. PCC 7120. ...................................................................................................................................................... 52 2.- ALL2320 ......................................................................................................................................................... 59 2 2.1 Analizar la presencia y abundancia del transcrito de all2320 e identificar la localización subcelular de la proteína All1765 en Anabaena sp. PCC 7120. .................................................................................................. 59 2.2 Determinar el papel de All2320 en la estructura del filamento y en la diferenciación celular de Anabaena sp. PCC 7120. ...................................................................................................................................................... 70 DISCUSIÓN ............................................................................................................................................................ 75 1.- ALL1765 ......................................................................................................................................................... 75 2.- ALL2320 ......................................................................................................................................................... 81 CONCLUSIONES ..................................................................................................................................................... 89 BIBLIOGRAFÍA ....................................................................................................................................................... 90 ANEXOS ................................................................................................................................................................ 99 3 RESUMEN La multicelularidad en cianobacterias se produce por la facultad de algunas especies para formar filamentos que contienen células diferenciadas con funciones especializadas. Entre estas células diferenciadas se encuentran las células vegetativas, que son las encargadas de realizar la fotosíntesis, y los heterocistos, que realizan la fijación de nitrógeno. Cómo se forma exactamente el filamento y cómo se genera el intercambio intercelular de moléculas es aún una incógnita. Hay evidencias que indican que el periplasma y la membrana externa serían continuos a lo largo del filamento, como también hay antecedentes que señalan la existencia de septosomas, que son conjuntos de proteínas en las conexiones intercelulares que podrían permitir el intercambio de compuestos y que mantienen la integridad de la estructura filamentosa. Durante este estudio, se caracterizaron dos genes, all1765 y all2320, los cuales mediante un análisis bioinformático previo de genómica comparativa realizado en nuestro laboratorio, resultaron ser exclusivos de cianobacterias filamentosas. La caracterización incluyó aspectos como: expresión génica en condiciones que afectan la diferenciación del filamento, localización subcelular y el fenotipo generado a partir de cepas mutantes de estos genes en comparación con la cepa silvestre. Se seleccionaron estos genes porque los análisis in silico indicaron que codifican proteínas que contienen dominios de transmembrana, y dado que nos interesaba entender su papel en la formación del filamento, parecía una condición deseable, pudiendo por ejemplo cumplir un rol en mantener la integridad del filamento. Los resultados obtenidos indicaron que ambas proteínas efectivamente participan en mantener la estructura normal del filamento, pero no tienen un rol en adhesión célula-célula, en intercambio molecular o en diferenciación celular. all1765 se asoció a una función en el proceso de fragmentación, la cual es una de las etapas del crecimiento celular descrito previamente en cianobacteria filamentosas. all2320, en cambio, tiene una participación esencial en la orientación del anillo de división celular de Anabaena sp. PCC 7120. En esta proteína llama la atención su polarización en sólo una de las células en división, lo cual demuestra que la división celular en Anabaena sp. PCC 7120 no es un proceso de división binaria como ocurre en la mayoría de las bacterias. Palabras Clave: Multicelularidad, Cianobacteria, Dominios de transmembrana, Septosomas. 4 ABSTRACT Multicellularity in cyanobacteria appears from their capacity to form filaments that contain differentiated cells with specialized functions, like the ability to carry out photosynthesis by vegetative cells and nitrogen fixation by heterocysts. Exactly how the filament is formed and how the intercelular molecular exchange between cells occurs is unclear. There is evidence that show that the periplasm and the outer membrane would be continuous along the filament, and also antecedents that demonstrate the existence of septosomes, which are groups of proteins located at the intercellular connections that could allow intercellular transfer of compounds and that maintain the integrity of this filamentous structure. During this study, I characterized two genes, all1765 and all2320, which by a previous comparative genomic bioinformatic analysis done in our laboratory, resulted to be exclusive of filamentous cyanobacteria. The characterization included aspects like: gene expression in conditions that affect filament differentiation, subcellular localization and the phenotype generated from mutants of these genes when compared to the wild-type. These genes were selected because the in silico analyses showed that they codify proteins that contain transmembrane domains, and because it was interesting to understand their role in filament formation, it seemed to be a desirable condition, for instance, fulfilling a role in sustaining filament integrity. The results showed that these genes effectively participated in sustaining the normal structure of the filament but had no role in cell-cell adhesion, molecular exchange or cell differentiation. all1765 was associated to fragmentation, which is one step of cellular growth previously observed in filamentous cianobacteria. all2320, on the other hand, has a fundamental participation in the orientation of the division ring of Anabaena sp. PCC 7120. In the last protein, it is remarkable its polarization in only one of the cells in division, which demonstrates that cellular division in Anabaena sp. PCC 7120 is not a binary division process like occurs in most bacteria. Keywords: Multicellularity, Cyanobacteria, Transmembrane domains, Septosomes. 5 INTRODUCCIÓN Las cianobacterias son bacterias Gram-negativas que colonizan normalmente ambientes acuáticos, sin embargo se pueden encontrar en prácticamente todos los hábitat de la Tierra. Hace aproximadamente 2800 millones de años, las cianobacterias fueron las responsables del inicio de la fotosíntesis oxigénica en nuestro planeta, es decir, fueron las responsables de la aparición de oxígeno en nuestra atmósfera (Schwartzman y cols., 2008). Para llevar a cabo este cambio en la atmósfera terrestre, fue necesaria una gran biomasa de estos microorganismos, y la forma de alcanzar esta biomasa fue mediante la adquisición eficiente de nutrientes por parte de las cianobacterias. Esto lo consiguieron algunas cianobacterias desarrollando la capacidad de fijar nitrógeno, generando nitrógeno combinado o disponible. La fotosíntesis y la fijación de nitrógeno son procesos incompatibles, ya que el oxígeno generado en la fotosíntesis inactiva irreversiblemente a la enzima que cataliza la fijación de nitrógeno, la nitrogenasa, con lo que se inhibe la fijación de nitrógeno (Fay, 1992). Por lo tanto, las cianobacterias debieron desarrollar estrategias para poder llevar a cabo estas dos reacciones al mismo tiempo y de forma eficiente. La primera estrategia fue realizar fotosíntesis en el día y fijación de nitrógeno por la noche, mientras que la segunda fue desarrollar multicelularidad, formando un filamento que contiene células diferenciadas que poseen funciones especializadas y que se comunican entre sí. Entre estos tipos celulares se encuentran las células vegetativas, que son las encargadas de realizar la fotosíntesis, y los heterocistos, que fijan el nitrógeno (Flores y Herrero, 2010). Hay evidencias que indican que la formación del filamento y el intercambio de moléculas estaría dada por una continuidad del periplasma y de la membrana externa a lo largo del filamento (Flores y cols., 2006; Mariscal y cols., 2007; Mariscal y Flores, 2010). Por otro lado, existen antecedentes que indican la presencia de proteínas que estarían localizadas en las conexiones intercelulares formando estructuras denominadas “septosomas”, las cuales conservan la integridad del filamento y permitirían una comunicación directa entre sus células (Wilk y cols., 2011; Bauer y cols., 1995; Flores y cols., 2007; Nayar y cols., 2007; Mariscal y Flores, 2010; Merino-Puerto y cols., 2010). Durante este estudio, se caracterizaron los genes exclusivos de cianobacterias filamentosas all1765 y all2320 obtenidos del estudio genómico comparativo realizado en nuestro 6 laboratorio (Stucken y cols., 2010). La caracterización involucró la cuantificación de la expresión génica en condiciones que afectan la diferenciación del filamento, localización subcelular y el fenotipo generado a partir de cepas mutantes de estos genes en comparación con la cepa silvestre. Se seleccionaron estos genes porque los análisis in silico indicaron que codifican proteínas hipotéticas que contienen dominios de transmembrana y coiled-coil, por lo cual podrían estar localizadas en la membrana citoplasmática, que es la región de la célula en donde se encuentran los septosomas, y que por lo tanto, podían tener un rol en mantener la integridad del filamento. 7 REVISIÓN BIBLIOGRÁFICA 1. Comunicación y adhesión intercelular en organismos multicelulares. La aparición de multicelularidad en la historia de la vida se presume que requirió de la confluencia de factores ambientales, ecológicos y genéticos, y que se encuentra asociada con un incremento en el número de genes relacionados con diferenciación celular, comunicación célulacélula y adhesión en comparación a organismos unicelulares (Rokas, 2008). Algunos procariontes, como las Myxobacterias, son considerados organismos multicelulares en ciertas etapas de su vida ya que tienen la habilidad de agregarse cuando hay una baja en el contenido de nutrientes en el medio, formando lo que se conoce como cuerpos fructíferos. En estos cuerpos, las bacterias se diferencian en esporas que pueden sobrevivir en circunstancias hostiles extremas hasta que mejoran las condiciones ambientales (Kaiser y cols., 2010). Otra bacteria que es considerada un organismo multicelular es Streptomyces coelicolor. Esta bacteria filamentosa Gram positiva, presenta un ciclo de vida en el que hay formación de hifas vegetativas y de hifas aéreas, siendo sólo estas últimas generadoras de esporas, y por lo tanto, teniendo estas hifas una función diferenciada en la reproducción (Chater, 2001). La comunicación intercelular generada en esta bacteria es mediante autoinductores, que son moléculas señalizadoras difusibles que permiten, entre otras acciones, informar a las bacterias de la presencia de otros miembros de su misma especie para así poder controlar la densidad celular de la población (Chater, 2001). Ciertos eucariontes, como las algas verdes, pueden existir en forma de organismos unicelulares, formando colonias o como organismos multicelulares. Algunas especies de algas que forman grandes colonias son las que pertenecen al género Volvox. En Volvox, las células individuales que se encuentran formando colonias, están conectadas entre sí por finos puentes citoplasmáticos, lo que les permite batir su flagelo de forma sincronizada para mover la colonia completa en la misma dirección (Green y cols., 1981). Asimismo, las células de Volvox presentan especialización de funciones, encontrándose por ejemplo células reproductivas ubicadas en un extremo de la colonia y que tienen la capacidad de dividirse para formar nuevas pequeñas colonias (Schmitt, 2003). Dada esta cooperación y especialización observada en las células de Volvox, este género tiende a ser considerado un organismo multicelular. 8 Para formar un organismo multicelular, las células deben estar conectadas entre sí. Los eucariontes han desarrollado numerosas formas de lograr esta comunicación. Como se describe anteriormente, las células de Volvox no se separan completamente durante la división celular, lo que permite que se mantengan conectadas por puentes citoplasmáticos. En plantas superiores, las células se mantienen conectadas por estructuras denominadas plasmodesmos. Los plasmodesmos son canales proteicos que permiten llevar a cabo una dinámica intercelular mediante el transporte de nutrientes, hormonas, proteínas reguladoras y moléculas de ARN entre células adyacentes (Cilia & Jackson, 2004). Las plantas terrestres han desarrollado complejos plasmodesmos, en donde una forma aprisionada del retículo endoplasmático es centralmente localizada dentro de puentes generados por la membrana citoplasmática, proporcionando continuidad entre las células adyacentes (Lucas & Ham, 2009). En células animales adyacentes, su comunicación viene dada por canales llamados “gap junctions”, los cuales se encuentran formados por proteínas formadoras de poros llamadas conexinas, que atraviesan las bicapas lipídicas de ambas células y que permiten el intercambio de iones y pequeñas moléculas (Kumar & Gilula, 1996). El tipo de organización y comunicación celular presente en plantas y animales también se observa en cianobacterias filamentosas. Las cianobacterias filamentosas son procariontes multicelulares cuyas células permanecen adheridas luego de la división celular lo que lleva a la formación de filamentos. En intervalos regulares de estos filamentos, células individuales se diferencian y adquieren funciones especializadas (Flores & Herrero, 2010). Al igual a lo observado en organismos eucariontes, las células del filamento de cianobacterias se comunican entre sí mediante mecanismos que permiten el intercambio intercelular de moléculas, como también permiten mantener la integridad del filamento (Bauer y cols., 1995; Flores y cols., 2006; Flores y cols., 2007; Mariscal y cols., 2007; Nayar y cols., 2007; Mariscal & Flores, 2010; Merino-Puerto y cols., 2010). 2. Multicelularidad en cianobacterias. Las cianobacterias pueden clasificarse según su morfología en cinco subsecciones, siendo la I y II correspondientes a unicelulares, la III son aquellas que forman filamentos sin células diferenciadas, mientras que la IV y V son aquellas cianobacterias filamentosas que poseen células 9 diferenciadas, en dónde en el caso de la subsección V, además presentan ramificaciones, es decir, se dividen en más de un plano (Rippka y cols., 1979). Las cianobacterias de las subsecciones IV y V son entonces cianobacterias filamentosas que desarrollan multicelularidad, es decir, que presentan células diferenciadas, con funciones especializados y que se comunican entre sí. Dentro de las funciones celulares especializadas se encuentran la fijación de nitrógeno y la fotosíntesis, que como ya se ha indicado, son procesos incompatibles ya que el oxígeno producido durante la fotosíntesis inactiva a la enzima nitrogenasa, que es la encargada de fijar el nitrógeno (Fay, 1992). Las células vegetativas son las encargadas de realizar la fotosíntesis, mientras que los heterocistos son los encargados de fijar el nitrógeno (Flores & Herrero, 2010) (Figura 1a). Las células vegetativas tienen una membrana citoplasmática, una pared de peptidoglicano y una membrana externa, que es la organización tradicional de bacterias Gram-negativas (Wolk, 1973) (Figura 1c). Entre la membrana citoplasmática y la membrana externa se encuentra la región conocida como periplasma, mientras que el espacio periplásmático entre una y otra célula del filamento se conoce como septo. A su vez, los heterocistos contienen estas mismas tres capas y dos más, que son propias de este tipo celular. Éstas son la capa de glicolípidos y la capa de polisacáridos (Figura 1b). La capa de glicolípidos permite mantener un ambiente micro-óxico dentro del heterocisto, lo que evita la inactivación de la nitrogenasa por parte del oxígeno, y la capa de polisacáridos mantendría la integridad de la capa de glicolípidos (Mariscal & Flores, 2010). Para llevar a cabo el intercambio intercelular de metabolitos y para mantener la integridad del filamento, se postula que el periplasma y que la membrana externa serían continuos a lo largo de esta estructura (Flores y cols., 2006; Mariscal y cols., 2007; Mariscal & Flores, 2010) (Figura 1c). Asimismo, existen evidencias de proteínas localizadas en las conexiones intercelulares que permitirían una comunicación directa entre células del filamento y que tienen función en mantener la integridad de esta estructura en cianobacterias (Bauer y cols., 1995; Flores y cols., 2007; Nayar y cols., 2007; Mariscal & Flores, 2010; Merino-Puerto y cols., 2010; Wilk y cols., 2011) (Figura 1c, cuadro naranjo). 10 Figura 1: Micrografías electrónicas de las células diferenciadas en el filamento de Anabaena sp. PCC 7120 (a) Filamento de Anabaena sp. PCC 7120 señalando heterocisto y células vegetativas. Flecha roja indica magnificación de la sección del heterocisto. Flecha azul indica magnificación de la sección del filamento entre células vegetativas. (b) Sección de un heterocisto mostrando la capa de glicolípido (HGL) y la capa de polisacárido (HP). CM: Membrana citoplasmática, PG: Pared de peptidoglicano, OM: Membrana externa. (c) Sección del filamento entre dos células vegetativas señalando CM, PG y OM. Cuadro naranjo indica localización de las proteínas que forman las conexiones intercelulares. Esta sección del filamento permite advertir que la membrana externa sería continua a lo largo del filamento. Figuras extraídas de Flores y cols., 2006. 11 . 2.1 Transferencia molecular mediante periplasma continuo A pesar que el mecanismo por el cual ocurre la transferencia intercelular de compuestos en el filamento de las cianobacterias es desconocido, uno de los conductos sugeridos para este suceso es la existencia de un periplasma continuo a lo largo del filamento (Flores y cols., 2006; Mariscal y cols., 2007; Mariscal & Flores, 2010). La hipótesis de este mecanismo indica que para proveer movimiento de moléculas entre diferentes tipos celulares del filamento de la cianobacteria Anabaena sp. PCC 7120, el periplasma no sólo debe ser físicamente continuo, sino también funcionalmente continuo entre células vegetativas y entre células vegetativas y heterocistos. En el estudio realizado por Mariscal y colaboradores el año 2007, estos investigadores utilizaron una cepa de Anabaena sp. PCC 7120 que expresaba el gen de la proteína fluorescente verde GFP (en inglés, green fluorescent protein) con un péptido señal que permite su translocación o exportación de la célula mediante el sistema twin-arginine translocation system (TAT) y clivaje por la enzima señal peptidasa I. El sistema TAT es conocido porque permite translocar proteínas plegadas a través de la membrana citoplasmática (Robinson & Bolhuis, 2004). Cuando esta cepa era sujeta a privación de nitrógeno disponible en el medio, GFP era producido en el heterocisto ya que estaba fusionado al promotor de un gen que se activa en este tipo celular en ausencia de nitrógeno disponible. Al momento de expresarse, era exportado mediante el sistema TAT y liberado al periplasma. La fluorescencia emitida por GFP fue observada en la periferia a lo largo de todas las células del filamento. En este mismo estudio, realizaron una sustitución en el sistema de translocación y utilizaron la enzima señal peptidasa II en vez de la peptidasa I, lo que resultó en el anclaje de GFP en la membrana citoplasmática. En esta nueva cepa, GFP se acumuló en la periferia de los heterocistos, y no fue capaz de pasar a las células vegetativas adyacentes. Estos resultados indicaron que GFP se mueve por el periplasma de heterocistos a células vegetativas en el filamento de la cianobacteria, lo que indica la efectiva existencia de un periplasma continuo. Sin embargo, posteriormente Zhang y colaboradores (2008), utilizando un enfoque experimental similar, concluyeron que GFP no atraviesa el septo entre las células del filamento producto de la existencia de barreras en el periplasma que impiden la difusión de GFP. Ellos observaron fluorescencia de GFP en la periferia de las células que generaba esta proteína, pero no detectaron 12 señal de GFP distribuyéndose a las células vegetativas vecinas. La razón de esta discrepancia puede deberse a que el grupo de Zhang fusionó GFP a un péptido señal de la proteína TorA de Escherichia coli, que a pesar de ser un péptido señal heterólogo al usado en el sistema TAT, podría no ser eficientemente procesado en Anabaena sp. PCC 7120. Por lo tanto, la existencia de un periplasma continuo a lo largo del filamento de Anabaena sp. PCC 7120 es un tópico aún en discusión. 2.2 Comunicación directa entre células del filamento por proteínas localizadas en conexiones intercelulares El segundo mecanismo por el cual ocurriría la transferencia intercelular de metabolitos en el filamento de las cianobacterias es mediante uniones célula-célula. En el estudio realizado por Wilk y colaboradores el año 2011, utilizaron la técnica de tomografía electrónica para analizar la ultraestructura del septo entre células vegetativas del filamento de Anabaena sp. PCC 7120, con el fin de visualizar en detalle la estructura de las conexiones intercelulares que previamente habían sido observadas por microscopía electrónica (Flores y cols., 2006). Luego de este análisis, demostraron la presencia de conexiones proteicas que se fusionaban con las membranas citoplasmáticas de células adyacentes en el filamento de la cianobacteria, estructuras que denominaron “septosomas” (Figura 2). Existen ciertos genes que codifican proteínas que se localizan en la membrana citoplasmática en las conexiones intercelulares del filamento, las cuales se postula podrían ser parte de los septosomas ya que cuando los genes que las codifican se encuentran mutados en la cianobacteria filamentosa Anabaena sp. PCC 7120, se observa un fenotipo en donde el filamento se fragmenta, lo cual afecta la multicelularidad y la viabilidad del cultivo. Algunos de estos genes son sepJ y fraD (Flores y cols., 2007; Nayar y cols., 2007; Merino-Puerto y cols., 2010). 13 Figura 2. Visualización de septosomas en una sección de contacto célula-célula en el filamento de Anabaena sp. PCC 7120. (a) Micrografía electrónica de una sección de contacto célula-célula en el filamento de Anabaena sp. PCC 7120. Las flechas indican una conexión célula-célula en el septo entre dos células vegetativas. Tamaño barra 100 nm. (b) Tomografía electrónica de estructuras localizadas en una región de contacto célula-célula en el filamento de Anabaena sp. PCC 7120. Septosomas y membranas citoplasmáticas (morado), tilacoides (verde) y membrana externa (azul). Tamaño barra 200 nm. Figuras extraídas de Wilk y cols., 2011. 14 2.2.1 sepJ sepJ codifica la proteína SepJ de 751 aminoácidos, la cual presenta en su extremo carboxilo terminal un dominio homólogo a permeasas bacterianas localizado en la membrana citoplasmática, y una región extra citoplasmática con una región coiled-coil que permitiría su interacción con otras proteínas (Flores y cols., 2007). SepJ es considerada una proteína de función desconocida, pero cuando el gen que la codifica se encuentra mutado en Anabaena sp. PCC 7120, se produce la fragmentación del filamento (Flores y cols., 2007; Nayar y cols., 2007). En la figura 3b podemos observar que a las 24 horas posteriores al crecimiento de la mutante en sepJ, cultivada en un medio que no posee nitrógeno disponible, es decir, en un medio en donde ocurre la diferenciación de heterocistos en el filamento, se observan filamentos más pequeños que los observados en la cepa silvestre (Figura 3a). A las 72 horas de crecimiento de la mutante en medio sin nitrógeno disponible, se observan principalmente células individuales en el medio de cultivo (Figura 3c). Este mismo fenotipo fragmentado se observó en estas mutantes crecidas en un medio en presencia de nitrógeno disponible (Flores y cols., 2007). Esto nos indica que la fragmentación del filamento en la mutante en sepJ es independiente del medio de cultivo en que es crecida la cianobacteria. Estos antecedentes permiten concluir que la proteína SepJ tiene función en mantener la integridad del filamento en Anabaena sp. PCC 7120. 15 Figura 3: Fenotipo de la cepa Anabaena sp. PCC 7120 silvestre y mutante del gen sepJ. (a) Cepa silvestre 24 horas de crecimiento en ausencia de nitrógeno disponible. Flechas negras señalan heterocistos en el filamento. (b) Cepa mutante del gen sepJ luego de permanecer durante 24 horas en ausencia de nitrógeno disponible. (c) Cepa mutante del gen sepJ luego de permanecer 72 horas en ausencia de nitrógeno disponible. Figuras (a)-(c) extraídas de Nayar y cols., 2007. Otra característica de la mutante en sepJ es que no desarrolla heterocistos completos y funcionales, por lo que la fijación de nitrógeno no es factible en esta mutante (Flores y cols., 2007). Por otra parte, y para ver si esta mutante era capaz de difundir compuestos solubles a lo largo del filamento, Mullineaux y colaboradores (2008) utilizaron la técnica de Recuperación de fluorescencia después de destrucción fotoquímica (en inglés, Fluorescence Recovery After Photobleaching o FRAP) para observar la difusión de calceína a lo largo del filamento de la mutante con respecto a la cepa silvestre (Figura 4). Observaron que la cepa mutante no recuperó la fluorescencia en la célula a la que se le realizó la destrucción fotoquímica de la fluorescencia, la cual fue proporcionada por la calceína que previamente había sido incorporada a todas las células del filamento. Por el contrario, la cepa silvestre recuperó la fluorescencia a los 11 segundos en la célula a la cual se le realizó el photobleaching. Esto indica que SepJ está asociada al intercambio de moléculas a lo largo del filamento. Sin embargo no se tiene claridad si esta proteína participa en el transporte de moléculas de forma directa, asociada a la formación de un canal, o de forma indirecta, siendo parte de proteínas que mantienen la cohesividad entre las células del filamento. 16 Figura 4: Recuperación de fluorescencia después de destrucción fotoquímica (FRAP) en la difusión de calceína a lo largo de filamentos de Anabaena sp. PCC 7120 cepa silvestre (WT) y cepa mutante para el gen sepJ (ΔsepJ). Microscopía de fluorescencia en donde se observan las células del filamento fluorescentes por encontrarse cargadas con calceína. Las mediciones de FRAP fueron realizadas en células vegetativas crecidas por 16 horas en medio libre de nitrato. Flechas blancas indican la célula que fue sometida a la destrucción fotoquímica en WT y ΔsepJ. t: tiempo en segundos post destrucción fotoquímica de la célula señalada. Figura extraída de Mullineaux y cols., 2008. Flores y colaboradores el año 2007 localizaron a la proteína SepJ en la célula (Figura 5). Esto lo lograron generando una proteína de fusión SepJ con la proteína fluorescente verde GFP. Observaron que SepJ se localiza en los septos entre todas las células del filamento, por lo tanto su ubicación está asociada a la localización de las conexiones intercelulares (Figura 5a). Además, se observó que SepJ forma un anillo en el centro de la célula durante la división celular (Figura 5b), al igual que la proteína encargada de dirigir la división celular en procariontes FtsZ (Sakr y cols., 2006). Esto señala que SepJ podría estar cumpliendo un rol importante durante el proceso de división celular en cianobacterias filamentosas. 17 Figura 5: Localización subcelular de SepJ-GFP en Anabaena sp. PCC 7120. (a) Microscopía óptica (panel izquierdo), microscopía de fluorescencia señalando autofluorescencia de cianobacteria (panel central), microscopía de fluorescencia señalando SepJ producto de fusión con GFP señalando localización de SepJ en los septos (panel derecho). Medios de cultivo con amonio (cuadro superior), con nitrato (cuadro central) y con nitrógeno atmosférico (cuadro inferior). Tamaño barra 6 µm. (b) Microscopía óptica (panel superior), microscopía de fluorescencia señalando autofluorescencia de cianobacteria (panel central), microscopía de fluorescencia señalando SepJ producto de fusión con GFP señalando localización de SepJ en el centro de la célula formando un anillo durante división celular (panel inferior). Tamaño barra 3 µm. Figura extraída de Flores y cols., 2007. 2.2.2 fraD FraD, codificada por fraD, es otra proteína que mantiene la integridad del filamento. Esta es una proteína formada por 344 aminoácidos que presenta en su estructura cinco dominios de transmembrana y dos regiones coiled-coil, y se encuentra localizada en la membrana citoplasmática. Cuando el gen que codifica FraD se encuentra mutado en Anabaena sp. PCC 7120, se observa una fragmentación del filamento (Merino-Puerto y cols., 2010), pero a diferencia de lo ocurrido en la mutante de sepJ, la mutante del gen fraD sólo presenta este fenotipo en ausencia de nitrógeno disponible en el medio de cultivo. 18 Por otra parte, en este mismo estudio se observó que a diferencia de lo observado en la mutante de sepJ, la mutante en fraD desarrolla heterocistos completos y funcionales, pero no es capaz de crecer en medio sólido en ausencia de nitrógeno disponible en el medio de cultivo. Por lo tanto, es probable que el paso de nitrógeno fijado por los heterocistos a través del filamento esté siendo afectado en esta mutante. Luego, para averiguar si esta mutante logra difundir compuestos solubles a lo largo del filamento, estos investigadores utilizaron la técnica de FRAP para analizar la difusión de calceína en esta estructura (Figura 6). Al igual que lo ocurrido en la mutante del gen sepJ, esta mutante no recupera la fluorescencia en la célula a la que se le realizó el photobleaching, por lo tanto FraD es una proteína necesaria para la difusión de compuestos solubles a través del filamento. Figura 6: Recuperación de fluorescencia después de destrucción fotoquímica (FRAP) en la difusión de calceína a lo largo de filamentos de Anabaena sp. PCC 7120 cepa silvestre (wt) y cepa mutante para el gen fraD (ΔfraD). Microscopía de fluorescencia en donde Pre-bleach indica células del filamento fluorescentes por encontrarse cargadas con calceína previa destrucción fotoquímica. Las mediciones de FRAP fueron realizadas en células vegetativas. La fluorescencia de calceína fue monitoreada a diferentes tiempos posteriores a la destrucción fotoquímica (0, 12 y 27 segundos). Flechas blancas al tiempo 0 segundos señalan células sometidas a destrucción fotoquímica. Figura extraída de Merino-Puerto y cols., 2010. 19 Merino-Puerto y colaboradores (2010), utilizando la metodología de localización subcelular mediante la creación de la proteína de fusión GFP-FraD, observaron que FraD, al igual que SepJ, se localiza en los septos entre las células del filamento (Figura 7). Esto revela que la localización de FraD se encuentra asociada a las conexiones intercelulares presentes a lo largo del filamento. Figura 7: Localización subcelular de GFP-FraD en Anabaena sp. PCC 7120. Microscopía de fluorescencia señalando FraD producto de fusión con GFP señalando localización de FraD en los septos. Medios de cultivo con nitrato (cuadro superior) y con nitrógeno atmosférico (cuadro inferior). Figura extraída de Merino-Puerto y cols., 2010. 20 3. Genes exclusivos de cianobacterias filamentosas. Nuestro laboratorio, en colaboración con el grupo de los Dr. Cembella y Glockner, secuenció los genomas de las dos cianobacterias filamentosas con los genomas más pequeños hasta ahora descritos: Cylindrospermopsis raciborskii CS-505 y Raphidiopsis brokii D9 (Stucken y cols., 2010). Con estos genomas disponibles, se compararon bioinformaticamente todas las secuencias hasta ahora disponibles de cianobacterias, obteniéndose un grupo de 10 genes exclusivos de cianobacterias filamentosas (genes siempre presentes en el genoma de todas las cianobacterias filamentosas hasta ahora descritas y su identidad de secuencia aminoacídica es superior al 30%). Dentro de estos 10 genes, se encuentran hetR, patU3 y hetZ, los cuales poseen una función en el desarrollo de heterocistos. Sin embargo, al encontrarse presentes en las cianobacterias filamentosas que desarrollan y también en las que no desarrollan heterocistos, podrían tener una función más general. La proteína HetR es considerada la reguladora maestra en la diferenciación de heterocistos (Buikema & Haselkorn, 2001). HetR se une a la región reguladora de su propio gen para aumentar su trascripción junto con la de otros genes. Por otra parte, PatU3 tiene un rol en el patrón de formación de heterocistos (Zhang y cols., 2007), mientras que HetZ se encuentra asociada al desarrollo de heterocistos (Zhang y cols., 2007). Otro gen de este set de genes exclusivos de cianobacterias filamentosas resultó ser el que codifica la previamente descrita proteína FraD, que posee un rol en mantener la integridad del filamento (Merino-Puerto y cols., 2010). Los otros seis genes codifican proteínas hipotéticas, y han sido denominados all1765, all2320, all1729, all2344, alr0202 y alr4863 de acuerdo al locus en el cual se encuentran en el genoma de Anabaena sp. PCC 7120. Al ser proteínas hipotéticas conservadas, no se conoce la función que estarían cumpliendo en cianobacterias filamentosas. Dentro de estos seis genes, dos (all1765 y all2320) codifican proteínas que presentan en su estructura dominios de transmembrana (Figura 8). El gen all1765 codifica una proteína de 185 aminoácidos que presenta un dominio de transmembrana y además un sitio de metilación amino terminal, que por lo que se conoce, proteínas con este sitio incluyen proteínas que forman los pili de las vías de secreción tipo II y IV, sistemas que permiten el transporte de proteínas (y en el caso del tipo IV también material genético) del citoplasma al periplasma. Por otra parte, el gen all2320 codifica una proteína de 197 aminoácidos que presenta un dominio de transmembrana y una región coiled-coil, que permitiría su interacción con otras proteínas. Estas características los hace 21 genes interesantes de estudio ya que las proteínas que codifican podrían estar localizadas en la membrana citoplasmática donde se encuentran las conexiones intercelulares al igual que las proteínas FraD y SepJ, que como ya se ha demostrado, son proteínas que participan en mantener la integridad del filamento. Figura 8: Estructura de proteínas All1765 y All2320 exclusivas de cianobacterias filamentosas. Mediante el Programa bioinformático SMART se obtuvo la estructura secundaria de las proteínas All1765 y All2320. All1765 consta de un domino de transmembrana con sitio de metilación amino terminal y All2320 presenta un dominio de transmembrana y una región coiled-coil. 22 HIPÓTESIS Las proteínas codificadas por los genes all1765 y all2320 están localizadas en las conexiones intercelulares del filamento de la cianobacteria Anabaena sp. PCC 7120 y son necesarias para mantener su estructura (integridad del filamento y diferenciación). OBJETIVO GENERAL Determinar la localización subcelular y participación en la integridad del filamento y diferenciación celular de las proteínas All1765 y All2320 en Anabaena sp. PCC 7120. OBJETIVOS ESPECÍFICOS 1.- Analizar la presencia y abundancia del transcrito de all1765 y all2320 e identificar la localización subcelular de las proteínas All1765 y All2320 en Anabaena sp. PCC 7120. 1.1 Evaluar la abundancia relativa de los transcritos de all1765 y all2320 en deprivación de nitrógeno disponible mediante Northern Blot y RT-PCR en tiempo real. 1.2 Obtener proteínas de fusión All1765-GFP y All2320-GFP y generar anticuerpos contra All1765 y All2320 para localizar la proteína en la célula. 2.- Determinar el papel de All1765 y All2320 en la estructura del filamento y en la diferenciación celular de Anabaena sp. PCC 7120. 2.1 Obtener cepas mutantes de los genes all1765 y all2320 y analizar el fenotipo obtenido (integridad del filamento y diferenciación). 23 MATERIALES Y MÉTODOS 1. Cepas bacterianas y condiciones de crecimiento de los cultivos La cepa de cianobacteria que se utilizará en este estudio es Anabaena sp. PCC 7120 (obtenida a través del Dr. Enrique Flores, CSIC, España). Esta es una cianobacteria filamentosa autótrofa de agua dulce que diferencia células a lo largo del filamento, formando heterocistos cada 10 o 15 células vegetativas aproximadamente. Es considerada una cepa modelo de cianobacterias ya que presenta un eficiente sistema de conjugación que permite una factible manipulación genética. Su genoma se encuentra secuenciado (número de acceso GenBank BA000019), el cual consta de un cromosoma circular de 6,41 Mpb y de 6 plásmidos que tienen tamaños entre 408 kpb y 5 kpb. Anabaena sp. PCC 7120 (cepa silvestre y cepas generadas en este estudio) fueron crecidas en medio BG11o (libre de nitrógeno disponible) (Anexo 1) o BG11 (el cual contiene NaNO3 como fuente de nitrógeno), a 30°C bajo luz blanca (25 to 75 μE m-2 s-1), en medios líquidos en agitación (90 to 100 r.p.m.) o en medios solidificados con agar Difco 1%, a excepción de las muestras tomadas para realizar los análisis de RT-PCR en tiempo real y Western blot. En estos casos, Anabaena sp. PCC 7120 fue crecida en cultivos líquidos a 25 °C bajo luz blanca (25 to 75 μE m-2 s-1) en matraces estáticos en medio MLAo (libre de nitrógeno disponible) (Anexo 2), de acuerdo al protocolo de Castro y colaboradores (2004), o con la adición de 2 mM NH 4Cl como fuente de nitrógeno (MLAn). 2. RT-PCR en tiempo real y análisis Northern blot para medir expresión génica 2.1 Análisis Northern blot Para realizar los análisis por Northern blot, el ARN total fue aislado de Anabaena sp. PCC 7120 utilizando RNeasy Plant Mini Kit (Qiagen Sciences) de acuerdo a las instrucciones del fabricante. Muestras de 15 mL fueron colectadas de cultivos crecidos durante 0, 3, 6, 9, 12 y 24 horas en BG11o para realizar las extracciones de ARN. El tiempo 0 es el cultivo inicial que se encuentra en inicio de fase exponencial de crecimiento en BG11 (DO 750 nm 0,2 - 0,3), del cual se obtuvo los cultivos experimentales de las horas siguientes previo lavado por filtros de 8 µm de poro para realizar el cambio de medio. La integridad de los ARN fue analizada mediante electroforesis de 5 µg de cada muestra en geles de agarosa 1 % (p/v) en TAE 1 X, corridos a 80 V por 90 min, 24 teñidos con bromuro de etidio y visualizados en un transiluminador UV. 10 µg de ARN de cada muestra fueron cargados en cada carril en geles de agarosa 1 % denaturantes por formaldehído. Las sondas de ADN fueron generadas por PCR usando ADN genómico de Anabaena sp. PCC 7120 y los partidores all1765-5 y all1765-6 para la sonda del gen all1765 y all2320-4 y all2320-2 para el gen all2320. Las hibridaciones fueron realizadas como lo describe Paz-Yepes y Herrero (2007). Como control de carga del ARN y de eficiencia de la transferencia, los filtros fueron hibridados con la sonda del gen rnpB. Las sondas fueron marcadas con el kit DNA labeling (Reay to Go GE Healthcare) y dCTP [α-32P]. Las áreas radioactivas en las membranas fueron visualizadas y cuantificadas usando un sistema Cyclone storage phosphor (Packard). La cuantificación de la expresión de cada gen (pixeles) fue medida en triplicado biológico para cada condición. 2.2 Análisis RT-PCR en tiempo real Para realizar los análisis por RT-PCR en tiempo real, el ARN total fue aislado de Anabaena sp. PCC 7120 utilizando “RNeasy Plant Mini Kit” (Qiagen Sciences) de acuerdo a las instrucciones del fabricante. Muestras de 15 mL fueron colectadas de cultivos crecidos durante 0, 3, 6, 12, 24 y 48 horas en MLAn o MLAo para realizar las extracciones de ARN. El tiempo 0 es el cultivo inicial que se encuentra en inicio de fase exponencial de crecimiento en MLAn (DO 750 nm 0,2 - 0,3), del cual se obtuvieron los cultivos experimentales previo lavado del medio por filtros de 8 µm de poro. La integridad de los ARN fue analizada mediante electroforesis de 5 µg de cada muestra en geles de agarosa 1 % (p/v) en TAE 1 X (Anexo 3), corridos a 80 V por 90 min, teñidos con bromuro de etidio y visualizados en un transiluminador UV. 10 µg de ARN extraídos de cada muestra fueron tratados con la enzima desoxirribonucleasa (DNasa) utilizando TURBO DNA-free™ Kit (Ambion®), según especificaciones del fabricante. Se retrotranscribió 1µg del ARN (tratado con DNasa) para obtener cADN (ADN complementario). Para esto se utilizó el sistema ImProm-II™ Reverse Transcription System (Promega®) con partidores al azar según instrucciones del fabricante. El cADN sintetizado se utilizó como templado para cuantificar los transcritos por medio de qPCR (Light Cycler 480, Roche), utilizando el kit LightCycler 480 SYBR Green I Master (Roche) según las especificaciones del frabricante. En este kit, se usa SYBR Green como fluorocromo de unión a la doble hebra. Los partidores utilizados en este experimento y en todos los experimentos posteriores en los que fueron necesarios, se diseñaron 25 usando el programa Primer 3 Input versión 0.4.0. Para calcular las veces de cambio en la abundancia relativa de los transcritos, se usó el modelo matemático para cuantificación relativa en RT-PCR en tiempo real descrito por Pfaffl (2001), en donde los blancos controles son las muestras crecidas en MLAn y los blancos muestrales son las muestras crecidas en MLAo. Las veces de cambio de la abundancia relativa de los transcritos de tres réplicas biológicas y tres réplicas técnicas de los tiempos de muestreo en cada tratamiento fueron analizados por ANOVA, y en los casos en donde se obtuvieron diferencias significativas, las medias se compararon por medio del test de Tukey (p < 0,05). Como control experimental, la expresión génica bajo estas mismas condiciones fue observada para hglD. Además, células vegetativas y heterocistos de 100 filamentos de cada réplica biológica fueron contados en cada condición. El número promedio de heterocistos sobre el número total de células de las tres muestras biológicas fue promediado en todos los tiempos de muestreo. 3. Generación de proteínas de fusión con GFP y de anticuerpos para localizar las proteínas en la célula 3.1 Generación de proteínas de fusión con GFP 3.1.1 All1765- GFP-mut2 y All1765-sf-GFP Los programas bioinformáticos que permiten predecir la localización subcelular y topología de proteínas de bacterias Gram-negativas (http://www.expasy.org/tools), no presentan consenso en la disposición de la proteína All1765 en la célula (Figura 13). Por este motivo, se generaron dos tipos de cepas con proteínas All1765 fusionadas en su extremo carboxilo terminal con GFP: una que tradujera proteínas fusionadas con GFP-mut2, la cual es una versión de la proteína fluorescente que pliega bien en el citoplasma (Flores y cols., 2007; Merino-Puerto y cols., 2010) y otra que tradujera proteínas fusionadas con “super folder GFP” (sf-GFP), la cual es otra versión de GFP que pliega de forma correcta también en el periplasma celular, pero que hasta ahora no había sido probada en cianobacterias sino sólo en Escherichia coli (Dinh & Bernhardt, 2011). ADN de Anabaena sp. PCC 7120 fue aislado por el método CTAB mencionado previamente. E. coli cepa DH5α fue utilizada para realizar las construcciones de los plásmidos. E. coli cepa HB101, usada para realizar las conjugaciones con la cianobacteria en la conjugación triparental, 26 fue crecida en medio LB (Anexo 4) y suplementada, cuando correspondió, con antibióticos a concentraciones estandarizadas (Ausubel y cols., 2008). Para la construcción de las proteínas de fusión All1765-GFP-mut2 y All1765-sf-GFP, un fragmento de 546 pb de all1765 fue amplificado por PCR utilizando como templado ADN genómico de Anabaena sp. PCC 7120 y los partidores all1765-5 y all1765-6, en donde all1765-5 incluye un sitio NheI y all1765-6 incluye un sitio SacI. Se realizó la extracción de ADN nuclear de Anabaena sp. PCC 7120 utilizando el protocolo que utiliza sales de bromuro de cetil-trimetil amonio (CTAB) tal como lo describieron Del Sal y colaboradores (1989) y Doyle y Doyle (1987). La amplificación por PCR de este y todos los fragmentos genómicos generados en este trabajo, se llevaron a cabo en un termociclador TECHNE® TC-312 o Eppendorf® Mastercycler en volúmenes de reacción de 25 µL, con una concentración final de 1,5 mM MgCl2 Invitrogen®, 1X tampón PCR Buffer 10 X (-MgCl2) Invitrogen®, 1 U Taq ADN polimerasa Invitrogen®, 0,2 µM partidores y 0,2 mM de cada desoxinucleótido (Promega®). El programa que se utilizado en el termociclador dependió principalmente de los partidores diseñados para cada caso. Para visualizar los productos amplificados se realizaron electroforesis en geles de agarosa al 1% (p/v) en TAE 1 X y tinción con bromuro de etidio o GelRed™ (Biotium). El fragmento de 546 pb de all1765 amplificado fue clonado en el vector de clonamiento pMBL-T para secuenciarlo. El clonamiento de este fragmento y de todos los que fueron requeridos en este trabajo se realizaron primero mediante la ligación del ADN en pMBL-T utilizando la enzima T4 ADN ligasa (Fermentas) durante aproximadamente 15 horas a 22 ºC. Luego, los vectores de clonamiento con los insertos de interés, fueron transferidos a E. coli DH5α quimiocompetentes mediante quimiotransformación. Las células quimiocompetentes son células previamente tratadas para que de forma eficiente, mediante un choque térmico, puedan adquirir un plásmido. Esta es una forma rápida y fácil de incorporar el plásmido de interés en la bacteria huésped y así lograr su replicación. La generación de células de E. coli DH5α quimiocompetentes y la quimiotransformación se realizaron de acuerdo al protocolo descrito por Ausubel y colaboradores (2002). Se verificó la presencia del inserto en los clones por selección en placas de agar LB suplementado con ampicilina (100 µg/ml), IPTG (0,1 mM) y X-Gal (40 µg/mL). Luego, se realizó la extracción de ADN plasmidial a aquellas colonias seleccionadas mediante el sistema Wizard® Plus SV Minipreps DNA Purification System (Promega®), según instrucciones del fabricante. 27 Posteriormente a la verificación de la secuencia del fragmento de 546 pb de all1765 en pMBL-T, éste fue transferido como un fragmento SacI/NheI al plásmido movilizable pRL277 (plásmido derivado del vector pBR322; Black y cols., 1993), resistente a Estreptomicina y Espectinomicina (SmR/SpR), que previamente había sido digerido con las mismas enzimas de restricción, y que ya contenía el gen gfp-mut2. Esto generó una fusión entre gfp-mut2 y el último aminoácido de all1765. Luego, se utilizó este mismo vector para generar la fusión de all1765 con sf-gfp mediante la digestión del vector con las enzimas BamHI y NheI, con lo cual se liberó el gen gfpmut2 y en ese sitio se ligó el gen sf-gfp. Ambos vectores generados, fueron transferidos a Anabaena por conjugación triparental. La conjugación triparental es una conjugación bacteriana que requiere tres cepas, por esto su nombre “triparental”. Primero, requiere una cepa “ayudante”, que es la cepa que contiene el plásmido conjugativo necesario para llevar a cabo la conjugación. Esta cepa asiste en la transferencia del plásmido que se quiere movilizar que se encuentra en la cepa “donadora” hacia la cepa “receptora”. De esta forma, en este trabajo la cepa “ayudante” fue E. coli que contenía el plásmido conjugativo pRL443, el cual contiene un cassette de resistencia a ampicilina (Merino-Puerto y cols., 2010). La cepa “donadora” fue E. coli, previamente transformada con el pásmido movilizable (SmR/SpR) que contenía la construcción all2320-gfpmut2. Esta cepa también se quimiotransformó con el plásmido de metilación pRL623 (Elhai y cols., 1997), para evitar que el plásmido movilizable fuera digerido por enzimas de restricción que se encuentran en la cepa “receptora”, que fue Anabaena sp. PCC 7120. El protocolo que se utilizó para llevar a cabo la conjugación triparental fue realizado de acuerdo a lo descrito por Elhai y colaboradores (1997). De esta forma, clones SmR/SpR, y que por lo tanto debieran haber incorporado el vector con all1765-gfp-mut2 o all1765-sf-gfp por recombinación simple, fueron seleccionados, siendo su inserción en el genoma de la cianobacteria verificada por PCR utilizando los partidores all1765-1 (partidor forward) y all1765-2 (partidor reverse), ambos complementarios a secuencias localizadas fuera de la construcción para verificar su inserción en todos los cromosomas de la cianobacteria (segregación), y el partidor gfp4 (partidor reverse), a modo de verificar la recombinación de la construcción en el lugar correcto del genoma. 3.1.2 All2320- GFP-mut2 Diversos programas bioinformáticos que permiten predecir la localización subcelular y topología de proteínas de bacterias Gram-negativas (http://www.expasy.org/tools), incluyendo los 28 programas Sosui y TopPred 0.01, sugieren que la región amino terminal de All2320 es extracitoplasmática, y que la región carboxilo terminal se encuentra en el citoplasma, por lo que se decidió generar sólo cepas con proteínas All2320 fusionadas en su extremo carboxilo terminal con GFP-mut2. ADN de Anabaena sp. PCC 7120 fue aislado por el método CTAB mencionado previamente. E. coli cepa DH5α fue utilizada para realizar las construcciones de los plásmidos. E. coli cepa HB101, usada para realizar las conjugaciones con la cianobacteria en la conjugación triparental, fue crecida en medio LB y suplementada, cuando correspondió, con antibióticos a concentraciones estandarizadas (Ausubel y cols., 2008). Para la construcción de la proteína de fusión All2320-GFP-mut2, un fragmento de 484 pb de all2320 fue amplificado por PCR utilizando como templado ADN genómico de Anabaena sp. PCC 7120 y los partidores all2320-4 y all2320-2, en donde all2320-2 incluye un sitio NheI. El fragmento de 484 pb de all2320 se amplificó por PCR y fue clonado en el vector de clonamiento pMBL-T. Para identificar aquellos clones que poseían el inserto correcto, se realizó una selección colorimétrica (exploración azul-blanco) de las colonias crecidas y se realizó la extracción de ADN plasmidial a aquellas colonias seleccionadas. Luego de verificar la secuencia del fragmento de 484 pb de all2320 en pMBL-T, éste fue transferido como un fragmento SacI/NheI al plásmido movilizable pRL277, que previamente había sido digerido con las mismas enzimas de restricción, y que ya contenía el gen gfp-mut2. Esto generó una fusión entre gfp-mut2 y el último aminoácido de all2320. El vector all2320-gfpmut2 fue transferido a Anabaena por conjugación triparental. De esta forma, clones SmR/SpR, y que por lo tanto debieran haber incorporado el vector con all2320-gfp-mut2 por recombinación simple, fueron seleccionados, siendo su inserción en el genoma de la cianobacteria verificada por PCR utilizando los partidores all2320-1 (partidor forward) y all2320-9 (partidor reverse), ambos complementarios a secuencias localizadas fuera de la construcción para verificar su inserción en todos los cromosomas de la cianobacteria (segregación), y el partidor gfp4 (partidor reverse), a modo de verificar la recombinación de la construcción en el lugar correcto del genoma. 3.2 Generación de anticuerpos 29 3.2.1 Generación de anticuerpo anti-All1765 Para la generación del anticuerpo anti-All1765, se seleccionó un minipéptido de 8 aminoácidos mediante análisis in silico de la proteína y se sintetizó en una empresa externa (Genscript). El análisis consistió en la selección de un péptido que se encontrara expuesto en la proteína y que no arrojara identidad aminoacídica con otras proteínas de la cianobacteria. Luego, estos minipéptidos fueron inyectados a grupos de dos conejos de acuerdo al procedimiento estándar del Bioterio del Departamento de Ciencias Biológicas de la Pontificia Universidad Católica de Chile. De esta forma, 500 µg de minipéptidos fueron administrados por inyección con el adyuvante completo Freund (CFA) para guiar la respuesta inmune, seguidos por tres inyecciones en los días 30, 60 y 90 con la misma cantidad de la proteína en el adyuvante incompleto Freund. Los sueros totales fueron colectados 4 meses posteriores a la última inyección. 3.2.2 Generación de anticuerpo anti-All2320 Para la generación del anticuerpo anti-All2320, la región codificante del gen fue amplificada usando ADN aislado de Anabaena sp. PCC 7120. El producto de PCR amplificado fue insertado en los sitios XhoI y HindIII del vector pET23b (Novagen) para producir el plásmido pETall2320-His. Este vector de expresión generó una proteína de fusión All2320-His que contenía la secuencia codificante de All2320 (secuencia aminoacídica: SHHHHHHN) en su extremo carboxilo terminal. La secuencia nucleotídica de este vector de expresión construido fue confirmada por secuenciación. La expresión de la proteína de fusión All2320-His fue inducida en células de E. coli cepa BL21 que contenían el plásmido pET-all2320-His por previa transformación de la bacteria, mediante la adición de 1 mM isopropyl-b-D-thiogalactopyranoside (IPTG) por 3 horas a 37°C cuando la densidad óptica del cultivo a 600 nm estuvo en 0,4. Las células colectadas de un cultivo de 1000 mL fueron resuspendidas en 10 mL del buffer de lisis 1 (8 M Urea, 0,01 M Tris, 0,1 M NaCl, 1 % tritón x-100, pH 8, 100 µg/ml lisozima y cóctel inhibitorio de proteasa) (Complete Mini, Roche) y luego fraccionadas usando un homogeneizador ultrasónico a 40 % de potencia y 70 % de pulso, en hielo por 22 minutos (Omni-Receptor 250, Omni International Inc.). La muestra soluble fue cargada en una columna Ni-MAC (Novagen) pre-equilibrada con buffer Binding, donde posteriormente las proteínas que se unieron fueron eluídas con 250 mM imidazol en buffer Elute (300 mM NaCl, 50 mM fosfato de sodio, pH 8). La 30 proteína eluída fue posteriormente purificada mediante electroelusión por el eluidor Bio-Rad electro, de acuerdo a las instrucciones del fabricante. La muestra eluída fue cargada en dos geles SDS-PAGE para electroforesis, en donde sólo uno fue teñido. Usando el gel teñido como marcador, bandas individuales de 30 KDa fueron cortadas del gel no teñido. Estas bandas fueron cargadas en un electroelutor Bio-Rad que contenía membranas con un poro de 12-15 kDa, con buffer de elusión (el buffer Tris/glicina fue también usado para realizar la electroforesis del gel). Un voltaje constante de 30 V fue aplicado por 8 - 10 horas a 4 °C. Las proteínas eluídas fueron colectadas de la membrana y guardadas a -20 °C. Para la generación del anticuerpo, la proteína fue dializada usando PBS 1X a pH 7. Grupos de dos conejos fueron inmunizados con la proteína All2320 purificada de acuerdo al procedimiento estándar del Bioterio del Departamento de Ciencias Biológicas de la Pontificia Universidad Católica de Chile. De esta forma, 500 µg de la proteína purificada fue administrada con el adyuvante completo Freund (CFA) (solución acuosa con la proteína, junto con un aceite mineral, un agente dispersante y con una suspensión de Mycobacterium muertos por calor) para guiar la respuesta inmune, seguidos por tres inyecciones en los días 30, 60 y 90 con la misma cantidad de la proteína con el adyuvante incompleto Freund (igual al adyuvante completo Freund pero sin la suspensión de Mycobacterium). Los sueros totales fueron colectados 4 meses posteriores a la última inyección. 3.2.3 Análisis Western Blot Para los análisis por Western Blot, 15 µL de muestras de cultivo de Anabaena sp. PCC 7120 crecidas en MLA y durante 48 horas en MLAo, fueron mezcladas con 5 µL de buffer de carga (6 % SDS, 15 %, 2-mercaptoetanol, 30 % glicerol, y 0,3 mg/ml bromofenol blue en 188 mM TrisHCl, pH 6,8), calentados a 90 °C por 10 minutos y separados por geles SDS-PAGE a 12 % TrisTricina. Las proteínas separadas en los geles fueron transferidas electroforéticamente a membranas de celulosa a 300 mA por 60 minutos. La membrana, fue bloqueada con 5 % de leche descremada en buffer TS-T (50 mM Tris, 150 mM NaCl, 0.05 % Tween20, pH 7,5) durante 16 horas a temperatura ambiente. Después de lavar la membrana con buffer TS-T, el anticuerpo (1:400) fue adicionado, diluido en buffer TS-T que contenía 5 % de leche e incubado por 2 horas a temperatura ambiente. La unión de los anticuerpos fue detectada por el anticuerpo secundario 31 horseradish peroxidasa-conjugado anti-conejo IgG (Invitrogen), seguido por un sistema de detección quimioluminiscente (Pierce), de acuerdo a instrucciones del fabricante. 3.2.4 Análisis por inmunofluorescencia 20 µL de un cultivo de la cianobacteria que se encontraba en fase exponencial de crecimiento (DO 750 nm 0,3), fue esparcido en un vidrio de poli-Lycina para microscopía (Sigma-Aldrish) y secado a 50 °C por 20 minutos. Los spots secados por aire fueron fijados en etanol 70 % (Merck) por 30 minutos a -20 °C. Los vidrios fueron nuevamente secados por aire y posteriormente lavados tres veces con PBS 0,1 % Tween20 por 2 minutos. Luego, los spots fueron bloqueados con PBS 0,1 % BSA 3 % (suero bovino albumina) Tween20 por 15 minutos a temperatura ambiente. Los vidrios fueron incubados con el anticuerpo primario diluido 1:100 en PBS 0,1 % BSA 3 % Tween20 por 1,5 horas a 4 °C en una cámara húmeda. Después de lavar el exceso de suero tres veces con PBS 0,1 % Tween20 por 2 minutos, los spots fueron incubados con el anticuerpo secundario Alexa Fluor488 goat anti conejo IgG en PBS concentración final 10 µg/ml (Invitrogen, USA) por 45 minutos a 4 °C en una cámara húmeda. Posteriormente, los spots fueron nuevamente lavados, se les adicionaron 20 µl del reactivo Prolong antifade (Invitrogen) y un cubre-objeto sellado fue asentado sobre el spot. 3.3 Microscopía confocal Luego de obtener los cultivos de Anabaena sp. PCC 7120 transformados con las construcción all1765-gfp-mut2, all1765-sf-gfp, all2320-gfp-mut2 o las inmunofluorescencias preparadas con los anticuerpos anti-All1765 y anti-All2320, se observaron los filamentos por microscopía confocal (Olympus SV 1000) con el fin de localizar las proteínas All1765 y All2320 en la célula. Para observar la fluorescencia emitida por las proteínas fusionadas con GFP-mut2 o sf-GFP, las muestras fueron excitadas con irradiación a 488 nm con un láser de ion argón. La emisión de la fluorescencia de GFP fue monitoreada a longitudes de onda entre 500 y 540 nm. Los anticuerpos generados utilizaron Alexa Fluor 488 como anticuerpo secundario, el cual se excitó a 488 nm y emitió fluorescencia entre 500 y 540 nm. Para visualizar la autofluorescencia dada por la clorofila presente en las células vegetativas de los filamentos de la cianobacteria, las muestras fueron excitadas a 520 a 560 nm y la emisión de la fluorescencia fue monitoreada a 630-700 nm. 32 4. Generación de cepas mutantes y análisis de sus respectivos fenotipos 4.1 Generación de cepas mutantes 4.1.1 Cepa mutante del gen all1765 por deleción (Δall1765) ADN de Anabaena sp. PCC 7120 fue aislado por el método CTAB mencionado previamente. E. coli cepa DH5α fue utilizada para realizar las construcciones de los plásmidos. E. coli cepa HB101, usada para realizar las conjugaciones con la cianobacteria en la conjugación triparental, fue crecida en medio LB y suplementada, cuando correspondió, con antibióticos a concentraciones estandarizadas (Ausubel y cols., 2008). Para mutar all1765 mediante la deleción de gran parte de su secuencia del genoma de Anabaena sp. PCC 7120 (deleción del 92,5 % del gen), se realizó la estrategia esquematizada en la Figura 9. Primero, fragmentos de ADN río arriba y río abajo de la región central del gen, fueron amplificados por PCR usando como templado el ADN de la cianobacteria y los partidores all1765-1/all1765-3 y all1765-4/all1765-2 (Tabla 1). Los productos de PCR fueron mezclados, diluidos y re-amplificados usando los partidores all1765-1 y all1765-2, obteniendo un fragmento amplificado de ADN que contempla gran parte del gen all1765 delecionado. Este fragmento fue clonado en pMBL-T, secuenciado e insertado en el plásmido pRL277. Este plásmido es derivado del vector pBR322 (Black y cols., 1993), el cual puede ser movilizado por conjugación, contiene un cassette de resistencia a Estreptomicina y Espectinomicina (SmR/SpR) y permite la selección positiva de doble recombinantes en bacterias Gram-negativas mediada por la expresión del gen sacB, cuya expresión se induce por sacarosa (Cai y Wolk, 1990). 33 Figura 9. Estrategia para la generación de la cepa mutante del gen all1765 mediante deleción (Δall1765). Para delecionar el gen all1765, fragmentos de ADN fueron amplificados usando los partidores all1765-1/all1765-3 y all1765-2/all1765-4. Los productos de PCR obtenidos (1+3 y 4+2) fueron mezclados, diluidos y re-amplificados utilizando los partidores all1765-1/all1765-2, obteniendo como amplificado, un fragmento de ADN en el que gran parte de all1765 se encuentra delecionado. Este fragmento fue clonado en el vector pMBL-T, digerido por las enzimas de restricción SacI y SpeI y transferido al plásmido movilizable pRL277, el cual fue transferido por conjugación triparental (metodología que necesita de los plásmidos de metilación pRL623 y conjugativo pRL443) desde E. coli a Anabaena PCC 7120. 34 sacB codifica la enzima levansacarasa, proteína secretora propia de la bacteria Gram-positiva Bacillus subtilis, que resulta letal cuando es expresada en bacterias Gram-negativas, incluyendo cianobacterias. Esta letalidad se genera porque levansacarasa se acumula en la membrana celular de bacterias Gram-negativas producto de la ineficaz exportación de la proteína. Por lo tanto, si la cianobacteria es crecida en medio con sacarosa, se podrán seleccionar las colonias que hayan sufrido de doble recombinación del plásmido en la región del genoma cianobacteriano de interés con el fin de eliminar el gen sacB, y así evitar la muerte de la cianobacteria. Es por este motivo que después de la conjugación triparental, los clones fueron crecidos en medio BG11 suplementado con 5 % sacarosa (Suc). Posteriormente, las colonias Suc R fueron analizadas por PCR para asegurar la presencia de clones que tuvieran el gen all1765 delecionado utilizando los partidores all1765-1 (partidor forward) y all1765-2 (partidor reverse), ambos complementarios a secuencias localizadas fuera de la deleción y así verificar la mutación en todos los cromosomas de la cianobacteria (segregación), y los partidores all1765-1 y (partidor forward) y all1765-8 (partidor reverse), en dónde este último es complementario a un segmento de la región delecionada del gen all1765, para así verificar la mutación en el lugar correcto del genoma. 4.1.2 Cepa mutante del gen all2320 por interrupción con cassette de resistencia a antibiótico (all2320::C.S3) ADN de Anabaena sp. PCC 7120 fue aislado por el método CTAB mencionado previamente. E. coli cepa DH5α fue utilizada para realizar las construcciones de los plásmidos. E. coli cepa HB101, usada para realizar las conjugaciones con la cianobacteria en la conjugación triparental, fue crecida en medio LB y suplementada, cuando correspondió, con antibióticos a concentraciones estandarizadas (Ausubel y cols., 2008). Para mutar el gen all2320 del genoma de Anabaena sp. PCC 7120 mediante su interrupción por un cassette de resistencia a antibiótico, se realizó la estrategia esquematizada en la Figura 10. Primero, fragmentos de ADN internos y río arriba de la región central del gen, fueron amplificados por PCR usando como templado el ADN de la cianobacteria y los partidores all2320-1/all2320-3 y all2320-4/all2320-2, en donde all2320-3 y all2320-4 fueron diseñados complementarios en 10 pb e incluyeron un sitio ClaI (Tabla 1). Los productos de PCR fueron mezclados, diluidos y re-amplificados usando los partidores all2320-1 y all2320-2, obteniendo un fragmento amplificado de all2320 con un sitio interno ClaI a 120 pb del codón de inicio del gen. Este fragmento fue clonado en pMBL-T, secuenciado y en su sitio ClaI, se le insertó un cassette 35 C.S3 de resistencia a los antibióticos Estreptomicina y Espectinomicina (SmR/SpR) (Elhai & Wolk, 1988a) para inactivar all2320. Luego, el fragmento que contenía C.S3, fue insertado en el plásmido pRL278. Este plásmido es derivado del vector pBR322 (Black y cols., 1993), el cual puede ser movilizado por conjugación, contiene un cassette de resistencia a Neomicina (Nm R) y también incluye el gen sacB para selección positiva de las cepas mutantes por doble recombinación. Después de la conjugación triparental, los clones SmR/SpR fueron crecidos en medio BG11 suplementado con 5 % sacarosa (Suc). Posteriormente, las colonias Suc R fueron analizadas por PCR a modo de encontrar clones que tuvieran el gen all2320 interrumpido por C.S3 utilizando los partidores all2320-1 (partidor forward) y all2320-2 (partidor reverse), ambos complementarios a secuencias localizadas fuera de la inserción de C.S3 para verificar su inserción en todos los cromosomas de la cianobacteria (segregación), y los partidores all2320 extCS3F (partidor forward) y CS3rev (partidor reverse), en dónde este último es complementario a una región del cassette C.S3, para así verificar la inserción del cassette en el lugar correcto del genoma. 36 Figura 10. Estrategia para la generación de la cepa mutante en all2320 mediante interrupción del gen (all2320::C.S3). Para inactivar el gen all2320, fragmentos de ADN fueron amplificados usando los partidores all2320-1/all2320-3 y all2320-2/all2320-4. Los productos de PCR obtenidos (1+3 y 4+2) fueron mezclados, diluidos y re-amplificados utilizando los partidores all2320-1/all2320-2, obteniendo un fragmento amplificado de all2320 con un sitio interno de corte por la enzima de restricción ClaI. Este fragmento fue clonado en el vector pMBL-T, el cassette C.S3 (SmR/SpR) fue insertado en el sitio ClaI y la construcción, previamente digerida por las enzimas de restricción XhoI y SpeI desde el vector pMBL-T, fue transferida al plásmido movilizable pRL278, el cual fue transferido por conjugación triparental (metodología que necesita de los plásmidos de metilación pRL623 y conjugativo pRL443) desde E. coli a Anabaena PCC 7120. 37 4.2 Microscopía óptica Para visualizar los filamentos por microscopía óptica, cultivos de Anabaena sp. PCC 7120 cepas silvestre y mutantes crecidos en BG11 (en presencia de Sm y Sp para all2320::C.S3) fueron colectados, lavados y resuspendidos en BG11 o BG11o (en presencia de Sm y Sp para all2320::C.S3). Luego de 48 horas de crecimiento, las muestras fueron observadas por microscopía óptica (Nikon Eclipse TS 100). Dentro de los análisis fenotípicos, se realizó un conteo de las células contenidas en 50 filamentos de cultivos de Anabaena sp. PCC 7120 cepa silvestre y cepa mutante en all1765, crecidos en presencia y ausencia de nitrógeno disponible a 30 °C, a las 0, 24, 48 y 72 horas (cada conteo se realizó en triplicado). El tiempo 0 es el cultivo inicial que se encuentra en inicio de fase exponencial de crecimiento en BG11 (DO 750 nm 0,2 - 0,3), del cual se obtuvieron los cultivos experimentales previo lavado del medio por filtros de 8 µm de poro. Las medias obtenidas a cada tiempo y en cada condición fueron comparadas y analizados utilizando el test estadístico t-Student (p < 0,05). 4.3 Microscopía electrónica de transmisión Para visualizar los filamentos por microscopía electrónica de transmisión, cultivos de Anabaena sp. PCC 7120 cepas silvestre y mutantes crecidos en BG11 (en presencia de Sm y Sp para all2320::C.S3) fueron colectados, lavados para realizar el cambio de medio y resuspendidos en BG11 o BG11o (en presencia de Sm y Sp para all2320::C.S3). Luego de 48 horas de crecimiento, 2 mL de cada cultivo fueron concentradas por centrifugación. Los pellets fueron fijados por 4 horas en glutaraldehído 3 % en un buffer de cacodilato de sodio 0,134 M (pH 7,2) a temperatura ambiente. Las células fueron lavadas por 16 horas con el buffer de cacodilato de sodio a 4 °C y teñidas durante 1 hora con tetróxido de osmio 1 % (p/v). Las muestras fueron lavadas 3 veces por 10 minutos con agua destilada, teñidas 1 hora con uranil acetato 1 % (p/v) y lavadas nuevamente. Las muestras fueron deshidratadas en una serie de acetona (30 % al 100 % (v/v)), pre-embebidas por 16 horas en una solución EPON™ resina-acetona 1:1 (London Resin), embebidas 6 horas en resina pura de EPON™, y finalmente polimerizada durante 24 horas a 60 °C. Las muestras fueron seccionadas por un ultramicrotomo (Sorvall MT5000) y montadas en grillas de cobre para 38 su observación en un microscopio electrónico de transmisión (Philips Tecnai 12) a un voltaje de aceleración de 80 kV. 39 RESULTADOS 1.- all1765 1.1 Analizar la presencia y abundancia del transcrito de all1765 e identificar la localización subcelular de la proteína All1765 en Anabaena sp. PCC 7120. Se analizó la expresión génica de all1765 mediante Northern Blot y RT-PCR en tiempo real en deprivación de nitrógeno disponible para investigar si este gen se encuentra regulado por el metabolismo del nitrógeno. Para llevar a cabo el análisis por Nothern Blot, se utilizó ARN aislado de cultivos de Anabaena sp. PCC 7120 silvestre y ARN aislado de cepas mutantes de Anabaena sp. PCC 7120 para los genes ntcA (Frías y cols., 1994) y hetR (Buikema & Haselkorn, 1991), todos crecidos en amonio e incubados en ausencia de nitrógeno disponible por distintos tiempos hasta las 24 horas (Figura 11). NtcA es un regulador transcripcional que controla el nitrógeno global en cianobacterias mediante la activación (y en algunos casos también la represión) de muchos genes involucrados en la asimilación de nitrógeno, siendo requerido para activar la diferenciación de heterocistos y para pasos posteriores en su desarrollo y funcionamiento (Herrero y cols., 2004). Por otra parte, HetR es una proteasa con actividad de unión al ADN y esencial regulador del desarrollo de heterocistos en cianobacterias (Buikema & Haselkorn, 1991). Al observar el contexto génico de all1765 (Figura 11a), este gen parece ser parte de un operón ya que sus genes aledaños se transcriben en su misma dirección (desde all1772 hasta all1766, y all1764). Un operón es una unidad funcional de ADN genómico que contiene un grupo de genes controlados por un mismo promotor, los cuales son generalmente transcritos en un ARNm policistrónico (ARNm que codifica para más de una proteína). Los resultados de la abundancia del transcrito en el Northern blot arrojaron un tamaño de transcrito completo de aproximadamente 3,8 kpb, el cual es un tamaño superior al de la región codificante de all1765 de 558 pb, indicando que el gen efectivamente se co-transcribe con otros genes del cluster génico al cual pertenece y, por lo tanto, forma parte de un operón (Figura 11b). En cuanto a la expresión génica de all1765, no se observaron cambios en los distintos tiempos en que la cepa silvestre se mantuvo en ausencia de nitrógeno disponible, y asimismo, no se detectó ni inducción ni represión en las cepas mutantes de los genes ntcA y hetR (Figura 11b). Se utilizó 40 el gen rnpB, el cual codifica la porción ARN de la ribonucleasa P, como normalizador de la expresión ya que tiene expresión constitutiva en cianobacterias (Vioque, 1997). Figura 11. Contexto génico y análisis Northern Blot de all1765. (a) Contexto génico de all1765 (cuadro rojo). Barra negra sobre el gen all2320 representa la región desde donde la cual se diseñó la sonda usada en el análisis Northern Blot. (b) ARN aislado de cultivos de Anabaena sp. PCC 7120 cepa silvestre, cepa mutante del gen ntcA (ΔntcA) y cepa mutante del gen hetR (ΔhetR) crecidos en presencia de amonio (0) e incubados en ausencia de nitrógeno disponible por 3, 6, 9, 12 y 24 horas (h), fue corrido en gel de electroforesis e hibridado con una sonda de aproximadamente el mismo tamaño del gen all1765. Un transcrito mayor de 3,8 kpb fue observado para all1765. La hibridación con una sonda del gen rnpB fue utilizada como control de carga y de transferencia. 41 Por otro lado, en el análisis por RT-PCR tiempo real, se utilizó ARN aislado de cultivos de Anabaena sp. PCC 7120 crecidos en presencia o en ausencia de nitrógeno disponible por distintos tiempos hasta las 48 horas. La amplificación de all1765 fue realizada utilizando partidores internos del gen (qfilD deltaNostoc F/qfilD deltaNostoc R, Tabla 1), mientras que el gen rnpB fue utilizado nuevamente como normalizador de la expresión (partidores qrnpB deltaNostoc F/qrnpB deltaNostoc R, Tabla 1). Como control experimental, bajo estas mismas condiciones, se analizó la expresión génica del gen hglD (GenBank AAA93154.1) (partidores qhglD deltaNostoc F/qhglD deltaNostoc R, Tabla 1), gen de la síntesis de la capa de glicolípido de heterocistos (Bauer y cols., 1997), y por lo tanto, marcador de diferenciación de heterocistos. 42 Tabla 1. Partidores usados en este trabajo diseñados utilizando el porgrama bioinformático Primer 3 Input versión 0.4.0. Sitios de restricción de endonucleasas incorporados en los partidores se muestran en negrita. 43 Como era de esperarse, hglD aumentó la abundancia relativa de su transcrito de forma significativa sólo en cultivos crecidos 24 y 48 horas en deprivación de nitrógeno disponible (Figura 12, panel izquierdo, barras verde claro), tiempos en los que se observó un considerable aumento en el número de heterocistos por filamento (Figura 12, panel izquierdo, círculos verde claro). En cuanto a la expresión de all1765, este gen presentó un comportamiento transcripcional similar, independiente de la fuente de nitrógeno en el medio (Figura 12, panel derecho), indicando que la expresión del gen no es regulada por el metabolismo del nitrógeno. Figura 12. Análisis de la abundancia relativa de los transcritos por RT-PCR en tiempo real de los genes hglD (panel izquierdo) y del gen all1765 (panel derecho), y medición del número de heterocistos por células totales (panel izquierdo). Muestras de filamentos y ARN fueron aislados de cultivos de Anabaena sp. PCC 7120 crecidos en presencia (N+) y en ausencia de nitrógeno disponible (N-) durante los tiempos indicados en horas. Las muestras de filamentos fueron utilizadas para contar el número de heterocistos del número total de células en los filamentos a cada hora en presencia (N+, panel izquierdo, círculos verde oscuro) y en ausencia de nitrógeno disponible (N-, panel izquierdo, círculos verde claro). ARN fue utilizado para realizar un RT-PCR en tiempo real para medir la expresión relativa de all1765 (panel derecho) y del gen control hglD (panel izquierdo) a cada hora en la presencia y ausencia de nitrógeno disponible. rnpB fue utilizado como gen normalizador en el análisis de expresión génica. Test estadístico utilizado ANOVA, Test Post Hoc utilizado Tuckey. Asteriscos (*) indican diferencias significativas en la expresión génica (p<0,05). 44 Para llevar a cabo la localización subcelular de All1765, dos metodologías fueron realizadas en paralelo: la generación de cepas con la proteína fusionada con GFP y la generación de un anticuerpo anti-All1765. All1765 es una proteína hipotética de Anabaena sp. PCC 7120 de 185 aminoácidos que presenta un dominio de transmembrana de 23 residuos con un sitio de metilación amino terminal (Figura 8). Proteínas con este sitio de metilación incluyen proteínas que forman los pili de las vías de secreción tipo II y IV, sistemas que permiten el transporte de proteínas (y en el caso del tipo IV también material genético) del citoplasma al periplasma. Los programas bioinformáticos que permiten predecir la localización subcelular y topología de proteínas de bacterias Gram-negativas (http://www.expasy.org/tools), no presentan consenso en la disposición de la proteína en la célula. Por ejemplo, el programa Sosui predice el extremo carboxilo terminal de la proteína en el espacio citoplasmático de la célula (Figura 13a), mientras que el programa bioinformático TopPred 0.01 predice el extremo carboxilo terminal de la proteína en el espacio extracitoplasmático de la célula (Figura 13b). Esta información es relevante al momento de generar la proteína de fusión ya que se recomienda fusionar GFP en el extremo carboxilo terminal de las proteínas, y de esta forma no tener inconvenientes con la funcionalidad de la proteína si ésta tuviera un péptido señal en su extremo amino terminal. Hasta el momento, sólo se han publicado estudios de localización subcelular en cianobacterias utilizando GFP-mut2, que es una de las versiones de la proteína GFP que se pliega de forma correcta únicamente en el citoplasma celular (Flores y cols., 2007; Merino-Puerto y cols., 2010). Sin embargo, como los programas bioinformáticos no generaron acuerdo en la disposición celular del extremo carboxilo terminal de All1765, se decidió generar dos tipos de cepas con proteínas All1765 fusionadas con GFP: una que tradujera proteínas fusionadas con GFP-mut2, y otra que tradujera proteínas fusionadas con “super folder GFP” (sf-GFP), la cual es otra versión de GFP que pliega de forma correcta también en el periplasma, pero que hasta ahora no había sido probada en cianobacterias sino sólo en Escherichia coli (Dinh & Bernhardt, 2011). 45 Figura 13. Predicción in silico de la disposición de All1765 en la célula. (a) Predicción de All1765 en la célula utilizando el programa bioinformático Sosui. EC: Espacio extracitoplasmático, MC: Membrana citoplasmática; CI: Espacio citoplasmático. Este programa predice el extremo carboxilo terminal de la proteína en el espacio citoplasmático de la célula. (b) Predicción de All1765 en la célula utilizando el programa bioinformático TopPred 0.01. Cytoplasm: Espacio citoplasmático, Franja gris: Membrana citoplasmática, Extracellular: Espacio extracitoplasmático. Este programa predice el extremo carboxilo terminal de la proteína en el espacio extracitoplasmático de la célula. 46 Después de obtener las cepas de Anabaena sp. PCC 7120 transformadas por conjugación triparental con las construcciones plasmidiales en las que all1765 presentaba en su extremo carboxilo terminal el gen que codifica para GFP-mut2 o sf-GFP (ver detalles de la generación de estas cepas en Materiales y Métodos), se verificó la inserción del plásmido con GFP en todas las copias del cromosoma de la cianobacteria, concepto que se conoce como “segregación”. Para corroborar la segregación, se realizó una reacción de PCR utilizando los partidores all1765-1 y all1765-2 (Tabla 1), los cuales debían amplificar una banda de tamaño de 1596 pb en la cepa silvestre, mientras que en las cepas con las fusiones de all1765 con gfp-mut2 o sf-gfp no debían amplificar ya que la inserción del plásmido en el genoma produce un amplicón de gran tamaño (aproximadamente 11.000 pb) que no es amplificable en la reacción. El resultado de la corroboración de la segregación mostró la presencia de la banda de 1596 pb en la cepa silvestre, sin embargo, también se observó esta banda en las cepas que contienen la fusión de all1765 con gfp-mut2 y sf-gfp (Figura 14). Esto indica que hay copias del cromosoma de la cianobacteria que se mantuvieron silvestres y no adquirieron el plásmido, por lo que son cepas no segregantes para la fusión de all1765 con gfp-mut2 o sf-gfp. Luego, para corroborar que efectivamente existieran cepas con copias del cromosoma con all1765 fusionado con gfp-mut2 o sf-gfp, se utilizaron los partidores all1765-1 y gfp4 (Tabla 1), siendo este último apto como partidor reverso en las amplificaciones de verificación de la inserción de GFP-mut2 y sf-GFP. No debía haber amplificación utilizando estos partidores en la cepa silvestre, mientras que en las cepas con GFPmut2 o sf-GFP, debían amplificar una banda de 1111 pb. En ambos casos, los resultados fueron los esperados (Figura 14). Por lo tanto, se pudo obtener cepas de Anabaena sp. PCC 7120 con la fusión de all1765 con gfp-mut2 o sf-gfp, pero no en todas las copias del cromosoma de la cianobacteria. Lamentablemente, al observar bajo microscopía confocal de estas cepas en donde All1765 contenía fusionada la proteína GFP-mut2 (Figura 15) o sf-GFP (Figura 16), el marcaje fluorescente fue imperceptible. 47 Figura 14. Corroboración de la segregación de las cepas de Anabaena sp. PCC 7120 con all1765 fusionado con gfp-mut y sf-gfp (all1765 + gfp). ADN de cultivos de Anabaena sp. PCC 7120 cepa silvestre (WT) y cepas con all1765 fusionado con gfp-mut2 (mut2) y sf-gfp (sf) fueron aislados y utilizados en reacciones de amplificación por PCR para corroborar la segregación de la inserción de gfp-mut2 y sf-gfp en las copias del cromosoma de la cianobacteria. I. Se observa el contexto génico de all1765, los partidores utilizados (flechas), los tamaños de los amplicones esperados y el gel de electroforesis realizado a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de la cepa WT, la cual fue utilizada como control negativo de la inserción de gfp-mut2 y sf-gfp. Los resultados fueron un amplicón de 1596 pb utilizando los partidores all1765-1 y all1765-2 (A) y ninguna amplificación utilizando los partidores all1765-1 y gfp4 (B). II. Se observa el contexto génico de all1765, los partidores utilizados (flechas), los tamaños de los amplicones esperados y los geles de electroforesis realizados a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de las cepas mut2 y de la cepa sf. Los resultados fueron un amplicón de 1596 pb utilizando los partidores all1765-1 y all1765-2 (cuadro rojo, C) y un amplicón de 1111 pb utilizando los partidores all1765-1 y gfp4 (cuadro rojo, D) tanto para las cepas mut2 como para la cepa sf, indicando la inserción de gfp-mut2 y sf-gfp respectivamente de forma contigua a all1765 en las cepas mut2 y sf, pero no en todas las copias del cromosoma de la cianobacteria. 48 Figura 15. Visualización de la fluorescencia de All1765-GFP-mut2 en Anabaena sp. PCC 7120. Cultivos de una cepa de Anabaena sp. PCC 7120, que contiene a all1765 fusionado con gfp-mut2 (All1765-GFPmut2), crecidos en nitrato y en ausencia de nitrógeno disponible, fueron visualizados por microscopía confocal. La autofluorescencia roja es dada por la clorofila de cianobacterias. El marcaje de All1765-GFP-mut2 fue imperceptible. Barra blanca representa escala de 10 µm. Figura 16. Visualización de la fluorescencia de All1765-sf-GFP en Anabaena sp. PCC 7120. Cultivos de una cepa de Anabaena sp. PCC 7120, que contiene a all1765 fusionado con sf-gfp (All1765-sf-GFP), crecidos en nitrato y en ausencia de nitrógeno disponible, fueron visualizados por microscopía confocal. La autofluorescencia roja es dada por la clorofila de cianobacterias. El marcaje de All1765-sf-GFP fue imperceptible. Barra blanca representa escala de 10 µm. 49 La segunda metodología utilizada en esta tesis para localizar All1765 en célula fue mediante la generación de un anticuerpo anti-All1765 (para más detalles de la generación de este anticuerpo ver Materiales y Métodos; anticuerpo realizado en colaboración con Dra. (c) Carla Trigo). Primero, se realizaron análisis Western Blot utilizando el anticuerpo generado en extractos celulares de Anabaena sp. PCC 7120 en la presencia y ausencia por 48 horas de nitrógeno disponible. Lamentablemente, no se detectó ninguna banda utilizando esta metodología. Se utilizaron cultivos de Anabaena sp. PCC 7120 crecidos en presencia de nitrógeno disponible o en su ausencia por 48 horas, los cuales fueron marcados con el anticuerpo antiAll1765 y analizados por microscopía confocal. En la Figura 17 podemos observar en el cuadro izquierdo la autofluorescencia generada por la clorofila presente en las células vegetativas de cianobacterias, en el cuadro central la señal dada por la fluorescencia del anticuerpo anti-All1765 y en el cuadro derecho la composición de ambas fluorescencias. Bajo ambas condiciones de crecimiento, All1765 se localiza sólo en algunas células del filamento, tanto en el citoplasma, en la membrana celular, en el septo (flechas blancas) e interesantemente en el polo sin contacto celular de células terminales del filamento. Es importante mencionar que el marcaje del anticuerpo no se observa en los heterocistos. 50 Figura 17. Localización de All1765 en filamentos de Anabaena sp. PCC 7120. Cultivos de Anabaena sp. PCC 7120 crecidos en nitrato fueron marcados con el anticuerpo anti-All1765 y analizados por microscopía confocal. La autofluorescencia roja es dada por la clorofila de cianobacterias. All1765 fue localizada en el citoplasma, en la membrana citoplasmática y en el septo de algunas células (flechas blancas en composición) y en el polo sin contacto celular de células terminales del filamento (flecha celeste en composición). Barra blanca representa escala de 5 µm. 51 1.2 Determinar el papel de All1765 en la estructura del filamento y en la diferenciación celular de Anabaena sp. PCC 7120. Para comprobar y analizar la potencial función de all1765 en cianobacterias filamentosas, cepas mutantes de este gen fueron generadas (Δall1765). Como el resultado del Northern Blot mostró que all1765 es parte de un operón de Anabaena sp. PCC 7120 (Figura 11), se decidió mutar el gen mediante la deleción de gran parte de éste y no mediante su interrupción con un cassette de resistencia, ya que esta última metodología podía generar un efecto polar en el resto del operón (para ver detalles de la generación de estas cepas, revisar Materiales y Métodos). Para corroborar la segregación de las cepas mutantes obtenidas (aproximadamente cien cepas), es decir, para verificar la deleción del gen all1765 en todas las copias del cromosoma de estas cepas, se realizaron dos reacciones de PCR utilizando los partidores all1765-1 y all1765-2 y los partidores all1765-1 y all1765-8 (Tabla 1). La primera pareja de partidores debían amplificar una banda de tamaño de 1596 pb en la cepa silvestre, mientras que en las cepas mutantes, debían amplificar una banda de 1080 pb producto de la deleción de 516 pb de all1765, que corresponde a la deleción del 92,5 % del gen. El resultado de la corroboración de la segregación utilizando estos partidores mostró la presencia de la banda de 1596 pb sólo en la cepa silvestre y de la banda de 1080 pb en las cepas mutantes (Figura 18). La segunda pareja de partidores que utilicé para corroborar la segregación debían amplificar una banda de tamaño de 1016 pb en la cepa silvestre, mientras que en las cepas mutantes, no debían amplificar ya que el partidor reverso all1765-8 es complementario a parte de la región delecionada de all1765 en estas cepas. En ambos casos, los resultados de las amplificaciones fueron los esperados (Figura 18). Por lo tanto, se pudieron obtener cepas de Anabaena sp. PCC 7120 con el gen al1765 delecionado en todas las copias del cromosoma de la cianobacteria, es decir, obtuve cepas segregantes para la mutación de all1765. Es importante mencionar que se secuenciaron las zonas de la deleción del gen all1765 en las algunas de las cepas mutantes, confirmando la mutación completa del fragmento deseado, como también se les realizó un análisis de expresión de all1765 para verificar la funcionalidad de la mutación, en donde en ninguna de las cepas mutantes analizadas hubo expresión del gen. 52 Figura 18. Corroboración de la segregación de las cepas de Anabaena sp. PCC 7120 mutante para all1765 (Δal1765). ADN de cultivos de Anabaena sp. PCC 7120 cepa silvestre (WT) y cepas mutantes para all1765 mediante la deleción del gen (Δal1765), fueron aislados y utilizados en reacciones de amplificación por PCR para corroborar la segregación de la deleción de all1765 en las copias del cromosoma de la cianobacteria. I. Se observa el contexto génico de all1765, los partidores utilizados (flechas), los tamaños de los amplicones esperados y el gel de electroforesis realizado a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de la cepa WT, la cual fue utilizada como control negativo de la inserción de C.S3. Los resultados fueron un amplicón de 1596 pb utilizando los partidores all1765-1 y all1765-2 (A) y un amplicón de 1016 pb utilizando los partidores all1765-1 y all1765-8 (B). II. Se observa el contexto génico de all1765, los partidores utilizados (flechas), los tamaños de los amplicones esperados y los geles de electroforesis realizados a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de las cepas mutantes en all1765. Los resultados fueron un amplicón de 1080 pb utilizando los partidores all1765-1 y all1765-2 (cuadro rojo, C) y ninguna amplificación utilizando los partidores all1765-1 y all1765-8 (D), indicando que el gen all1765 fue delecionado en todas las copias del cromosoma de las cepas mutantes. 53 En cuanto al análisis del fenotipo de estas mutantes (Δal1765 fenotipo 1 y 2), se pudo observar que ambas cepas crecieron normalmente en presencia de nitrógeno disponible en el medio de cultivo líquido (Figura 19a). Sin embargo, cuando fueron crecidas en ausencia de nitrógeno disponible en el medio, la cepa Δal1765 fenotipo 2 fue incapaz de sobrevivir más de 48 horas bajo esta condición, mientras que la cepa Δal1765 fenotipo 1 si creció en este medio (Figura 19a), de forma muy similar al crecimiento evidenciado en la cepa sielvestre. Interesantemente, cuando filamentos de la cepa Δal1765 fenotipo 2 previamente crecidos en deprivación de nitrógeno por menos de 48 horas fueron subcultivados en presencia de nitrógeno disponible, fueron capaces de recuperarse y crecer, indicando que el efecto de la mutación sería sólo frente a la ausencia de nitrógeno disponible en el medio, sin afectar aparentemente la estructura o funcionamiento del resto del filamento. Posteriormente, y para obtener un análisis acabado de la estructura de los filamentos, las dos cepas mutantes del gen all1765 y la cepa silvestre crecidas en presencia de nitrógeno disponible o durante 24 horas en ausencia de nitrógeno disponible, fueron observadas por microscopía óptica (Figura 19b, columnas primera y segunda). No se observaron diferencias en la estructura del filamento ni en el tamaño celular de las dos cepas mutantes al compararlas entre ellas y con la cepa silvestre. Cabe destacar la presencia de filamentos cortos en los cultivos de la cepa Δal1765 fenotipo 2 cuando fue crecida en deprivación de nitrógeno disponible por 24 horas (Figura 19b). Este resultado era esperable ya que, bajo ciertas condiciones desfavorables, los filamentos de Anabaena sp. PCC 7120 tienden a acortarse antes de la muerte de la cianobacteria, desenlace que ya se sabía tendría la cepa Δal1765 fenotipo 2 crecida bajo esta condición producto de los resultados obtenidos previamente. Los heterocistos indicados con flechas blancas de la Figura 19b se visualizaron de color azul porque fueron teñidos con Alcian Blue, que tiñe la capa de exopolisacáridos de heterocistos. Este resultado es muy interesante porque indica que, a pesar de no sobrevivir en deprivación de nitrógeno disponible, la cepa Δal1765 fenotipo 2 parece desarrollar heterocistos estructuralmente normales. Del mismo modo, las dos cepas mutantes y la cepa silvestre crecidas en presencia o durante 24 horas en ausencia de nitrógeno disponible, fueron observadas por microscopía electrónica de transmisión (Figura 19b, columnas tercera y cuarta). En ambas condiciones, no se observaron diferencias en la estructura ni en el tamaño celular de estas cepas al compararlas entre ellas y con la cepa silvestre. 54 En resumen, se observaron dos fenotipos en las cepas mutantes del gen all1765: el primero, observado en la mayoría de las cepas mutantes (Δal1765 fenotipo 1), señala una estructura celular semejante a la presente en la cepa silvestre, y el segundo, observado sólo en una de las más de cien cepas mutantes obtenidas, también establece una morfología celular similar a la observada en la cepa silvestre, pero un crecimiento inviable en ausencia de nitrógeno disponible (Δal1765 fenotipo 2). La explicación más probable sería la existencia de una o más mutaciones adicionales a la introducida mediante la mutación sitio dirigida del gen all1765 en la Δal1765 fenotipo 2. Esta o estas mutaciones estarían causando la muerte celular después de 48 horas en deprivación de nitrógeno disponible. Al ser el fenotipo de la cepa Δal1765 fenotipo 2 tan interesante, principalmente por su similitud al observado en la mutante del gen fraD (MerinoPuerto, 2010), el genoma de esta cepa se encuentra en proceso de secuenciación para identificar cual o cuales genes contiene adicionalmente alterados y así comenzar el estudio de ellos como potenciales genes asociados al metabolismo del nitrógeno. Al contener la cepa Δal1765 fenotipo 2 al menos una mutación adicional a la realizada al gen all1765, que es uno de los genes de interés de esta tesis, no se realizaron más análisis fenotípicos a esta cepa, sino que se continuó con la profundización y comparación del fenotipo de la cepa Δal1765 fenotipo 1 con respecto a la cepa silvestre. 55 Figura 19. Fenotipo de Anabaena sp. PCC 7120 cepa silvestre y cepa mutante del gen all1765 (Δal1765). (a) Cultivos de Anabaena sp. PCC 7120 cepas mutantes de all1765 (Δal1765), crecidos en medio líquido en presencia (N+) o durante 48 horas en ausencia de nitrógeno disponible (N-). Cepa Δal1765 fenotipo 2 no sobrevive bajo esta última condición. (b) Cultivos de Anabaena sp. PCC 7120 cepa silvestre (WT) y Δal1765 fenotipo 2 crecidos en presencia (N+) o durante 24 horas en ausencia de nitrógeno disponible (N-) fueron observados por microscopía óptica (primera y segunda columnas) y electrónica de transmisión (tercera y cuarta columnas). Δal1765 fenotipo 1 y 2 no presentan diferencias en la estructura del filamento ni en el tamaño celular al compararlas con la cepa WT. Crecida en N- durante 24 horas, las cepas Δal1765 fenotipo 1 y 2 desarrollan heterocistos estructuralmente normales. Heterocistos de Δal1765 fenotipos 1 y 2 se observan de color azul por tinción con Alcian Blue, que tiñe capa de exopolisacáridos de heterocistos. Flechas blancas apuntan a heterocistos. Barra blanca 10 µm, barra negra 1 µm. 56 Una característica que se advirtió al momento de observar por microscopía óptica los filamentos de la cepa Δal1765 fenotipo 1 crecidos en presencia de nitrógeno disponible, fue que eran de un largo superior al de los filamentos de la cepa silvestre (Figura 20a). Para alcanzar el dato preciso de esta observación, se realizó el conteo por microscopía óptica de las células contenidas en 150 filamentos de cultivos de Anabaena sp. PCC 7120 cepa silvestre y cepa Δal1765 fenotipo 1 crecidos en presencia de nitrógeno disponible (Figura 20b). El largo de los filamentos de la cepa 25 fue efectivamente significativamente superior (entre un 31 % y un 58 %) en todos los tiempos de medición al compararlo con la cepa silvestre (Figura 20b). Este fenotipo era muy interesante ya que si la ausencia de all1765 genera el alargamiento de los filamentos, entonces el gen tiene una participación en la regulación del largo de los filamentos de Anabaena sp. PCC 7120, pudiendo participar específicamente en la reproducción asexual por fragmentación del filamento. En este proceso de división celular asexual, segmentos del filamento parental se rompen y separan en dos o más fragmentos que crecen y forman nuevos filamentos. Se ha descrito que la fragmentación se produce cuando hay condiciones desfavorables en el ambiente, por lo que se considera una estrategia de dispersión hacia mejores condiciones ambientales (Cirés y cols., 2013; Alster y cols., 2010; Adamec, 2005). Sin embargo, cómo ocurre el proceso de fragmentación es un aspecto aún desconocido. También se realizó el análisis del fenotipo de la Δal1765 fenotipo 1 creciéndola en ausencia de nitrógeno disponible en el medio, y comparándola con la cepa silvestre Anabaena sp. PCC 7120. Se contaron las células presentes en 150 filamentos de cada cultivo para así analizar si la deprivación de nitrógeno produce algún efecto en el largo de los filamentos de estas cepas. En el caso de la cepa silvestre, la deprivación de nitrógeno efectivamente generó el acortamiento de sus filamentos de forma significativa a las 48 y 72 horas bajo esta condición al compararla con su condición óptima de crecimiento en presencia de nitrógeno disponible (Figura 20b). Este resultado demuestra que la ausencia de nitrógeno disponible es una condición que favorece la reproducción por fragmentación de Anabaena sp. PCC 7120. Interesantemente, al realizar el análisis de la medición del largo de los filamentos de la cepa Δal1765 fenotipo 1, se pudo observar que la cepa mutante mostró un acortamiento de sus filamentos, fenotipo contrario al observado cuando fue crecida en presencia de nitrógeno disponible. Bajo la condición 57 desfavorable, la cepa Δal1765 fenotipo 1 mantuvo un largo de filamentos similar al observado en la cepa silvestre en todos los tiempos de medición. Figura 20. Visualización y medición de filamentos de Anabaena sp. PCC 7120 cepa silvestre y cepa mutante del gen all1765 en cultivos crecidos en presencia de nitrógeno disponible. (a) Cultivos de Anabaena sp. PCC 7120 cepas silvestre (WT) y cepa Δal1765 fenotipo 1, fueron crecidos en medio líquido en presencia (N+) y en ausencia de nitrógeno disponible (N-), observados por microscopía óptica y fotografiados a las 48 horas de crecimiento. Escala 200 µm. (b) Cultivos de Anabaena sp. PCC 7120 cepas silvestre (WT) y cepa Δal1765 fenotipo 1, fueron crecidos en medio líquido en presencia (N+) y en ausencia de nitrógeno disponible (N-) por los tiempos indicados en horas. En estos tiempos, a ambos cultivos se les midió el largo de sus filamentos (contando el número de células que contenían 150 filamentos del cultivo) (barras rojas cepa WT, barras azules Δal1765 fenotipo 1) Test estadístico utilizado t-Student. Letras (a y b) indican diferencias significativas en el número de células por filamento (p<0,05). 58 2.- all2320 2.1 Analizar la presencia y abundancia del transcrito de all2320 e identificar la localización subcelular de la proteína All1765 en Anabaena sp. PCC 7120. Al igual que para el gen all1765, se analizó la expresión génica de all2320 mediante Northern Blot y RT-PCR en tiempo real en deprivación de nitrógeno disponible para investigar si este gen se encuentra regulado por el metabolismo del nitrógeno. Para el análisis por Nothern Blot, se utilizó ARN aislado de cultivos de Anabaena sp. PCC 7120 silvestre y ARN aislado de cepas mutantes de Anabaena sp. PCC 7120 para los genes ntcA (Frías y cols., 1994) y hetR (Buikema & Haselkorn, 1991), todos crecidos en amonio e incubados en ausencia de nitrógeno disponible por distintos tiempos hasta las 24 horas (Figura 22). Al analizar el contexto génico de all2320 (Figura 22a), este gen parece ser monocistrónico ya que el gen all2321, contiguo a all2320 en el genoma, se transcribe en dirección opuesta a all2320, y el gen glnB, también adyacente a all2320, ha sido descrito como monocistrónico en estudios previos (Paz-Yepes y cols., 2009). En cuanto al análisis experimental por Northern Blot, la sonda diseñada para all2320 hibridó con una molécula de ARN de 1 kpb, el cual corresponde a un tamaño de transcrito que no alcanza a cubrir el gen all2320 junto con all2321 y/o glnB, y por lo tanto, corresponde sólo al gen all2320, evidenciando que es efectivamente monocistrónico (Figura 21b). En cuanto a la expresión génica de all2320, no se observaron cambios en los distintos tiempos en que la cepa silvestre se mantuvo en ausencia de nitrógeno disponible, y asimismo, no se detectó ni inducción ni represión en las cepas mutantes de los genes ntcA y hetR (Figura 21b). Al igual que en el caso de all1765, se utilizó el gen rnpB como normalizador de la expresión. 59 Figura 21. Contexto génico y análisis Northern Blot de all2320. (a) Contexto génico de all2320. Barra negra sobre el gen all2320 representa la sonda diseñada y usada en el análisis Northern Blot. (b) ARN aislado de cultivos de Anabaena sp. PCC 7120 cepa silvestre, cepa mutante del gen ntcA (ΔntcA) y cepa mutante del gen hetR (ΔhetR) crecidos en presencia de amonio (0) e incubados en ausencia de nitrógeno combinado por 3, 6, 9, 12 y 24 horas (h), fue corrido en gel de electroforesis e hibridado con una sonda de aproximadamente el mismo tamaño del gen all2320. Un transcrito mayor de 1 kpb fue observado para all2320. La hibridación con una sonda del gen rnpB fue utilizada como control de carga y de transferencia. 60 Por otro lado, en el análisis por RT-PCR tiempo real, se utilizó ARN aislado de cultivos de Anabaena sp. PCC 7120 crecidos en amonio o en ausencia de nitrógeno disponible por distintos tiempos hasta las 48 horas. La amplificación de all2320 fue realizada utilizando partidores internos del gen (qfilB deltaNostoc F/qfilB deltaNostoc R, Tabla 1). rnpB fue utilizado nuevamente como normalizador de la expresión (partidores qrnpB deltaNostoc F/qrnpB deltaNostoc R, Tabla 1) y como control experimental, se utilizó el mismo gen analizado previamente en el estudio de all1765, el gen hglD (partidores qhglD deltaNostoc F/qhglD deltaNostoc R, Tabla 1). hglD aumentó la abundancia de su transcrito sólo en cultivos crecidos 24 y 48 horas en deprivación de nitrógeno disponible (Figura 22, panel izquierdo, barras verde claro), tiempos en los que se observó un considerable aumento en el número de heterocistos por filamento (Figura 22, panel izquierdo, círculos verde claro). En cuanto a la expresión de all2320, no hubo inducción ni represión del gen en los cultivos crecidos en presencia o ausencia de nitrógeno disponible. 61 Figura 22. Análisis de la abundancia relativa de los transcritos por RT-PCR en tiempo real de los genes hglD (panel izquierdo) y del gen all2320 (panel derecho), y medición del número de heterocistos por células totales (panel izquierdo). Muestras de filamentos y ARN fueron aislados de cultivos de Anabaena sp. PCC 7120 crecidos en presencia (N+) y en ausencia de nitrógeno disponible (N-) durante los tiempos indicados en horas. Las muestras de filamentos fueron utilizadas para contar el número de heterocistos del número total de células en los filamentos a cada hora en presencia (N+, panel izquierdo, círculos verde oscuro) y en ausencia de nitrógeno disponible (N-, panel izquierdo, círculos verde claro). ARN fue utilizado para realizar un RT-PCR en tiempo real para medir la expresión relativa de all2320 (panel derecho) y del gen control hglD (panel izquierdo) a cada hora en la presencia y ausencia de nitrógeno disponible. rnpB fue utilizado como gen normalizador en el análisis de expresión génica. Test estadístico utilizado ANOVA, Test Post Hoc utilizado Tuckey. Asteriscos (*) indican diferencias significativas en la expresión génica (p<0,05). Para llevar a cabo la localización subcelular de All2320, se realizaron las mismas dos metodologías utilizadas en para localizar la proteína Al1765: la generación de cepas con la proteína fusionada con GFP y la generación de un anticuerpo anti-All2320. All2320 es una proteína hipotética de Anabaena sp. PCC 7120 de 197 aminoácidos que incluye una región coiled-coil de 42 residuos cerca de su extremo amino terminal (aminoácidos 27 al 69), la cual permitiría interacción con otras proteínas, y un dominio de transmembrana de 22 residuos cercano a su extremo carboxilo terminal (aminoácidos 166 a 188) (Figura 8). Diversos programas bioinformáticos que permiten predecir la localización subcelular y topología de proteínas de bacterias Gram-negativas (http://www.expasy.org/tools), incluyendo los programas Sosui y TopPred 0.01, sugieren que la región amino terminal de 165 aminoácidos de All2320 es extracitoplasmática, y que 9 residuos de la región carboxilo terminal se encuentran en 62 el citoplasma (Figura 23). Con el resultado de estas predicciones, se decidió generar sólo cepas con proteínas All2320 fusionadas en su extremo carboxilo terminal con GFP-mut2, ya que como mencioné previamente en el diseño de las fusiones con GFP de la proteína All1765, esta versión de la proteína GFP pliega bien en el citoplasma celular (Flores y cols., 2007; Merino-Puerto y cols., 2010). Figura 23. Predicción in silico de la disposición de All2320 en la célula. (a) Predicción de All2320 en la célula utilizando el programa bioinformático Sosui. EC: Espacio extracitoplasmático, MC: Membrana citoplasmática; CI: Espacio citoplasmático. (b) Predicción de All2320 en la célula utilizando el programa bioinformático TopPred 0.01. Cytoplasm: Espacio citoplasmático, Franja gris: Membrana citoplasmática, Extracellular: Espacio extracitoplasmático. Ambos programas bioinformáticos predicen el extremo carboxilo terminal de la proteína en el espacio citoplasmático de la célula. 63 Después de obtener las cepas de Anabaena sp. PCC 7120 transformadas por conjugación triparental con las construcciones plasmidiales en las que all2320 presentaba en su extremo carboxilo terminal el gen que codifica para GFP-mut2 (ver detalles de la generación de estas cepas en Materiales y Métodos), se verificó si el plásmido con GFP-mut2 se encontraba inserto en todas las copias del cromosoma de la cianobacteria, es decir, se revisó si las cepas eran segregantes para la fusión de all2320 con gfp-mut2. Para corroborar la segregación, se realizó una reacción de PCR utilizando los partidores all2320-1 y all2320-9 (Tabla 1), los cuales debían amplificar una banda de tamaño de 912 pb en la cepa silvestre, mientras que en las cepas con las fusiones de all2320 con gfp-mut2 no debían amplificar ya que la inserción del plásmido en el genoma produce un amplicón de gran tamaño (aproximadamente 11.000 pb) que no es amplificable en la reacción. El resultado de la corroboración de la segregación mostró la presencia de la banda de 912 pb en la cepa silvestre, sin embargo, también se observó en las cepas que contienen la fusión de all2320 con gfp-mut2 (Figura 24). Esto indica que hay copias del cromosoma de la cianobacteria que se mantuvieron silvestres y no adquirieron el plásmido, por lo que son cepas no segregantes para la fusión de all2320 con gfp-mut2. Luego, para corroborar que efectivamente existían copias del cromosoma con all2320 fusionado con gfp-mut2, se utilizaron los partidores all2320-1 y gfp4 (Tabla 1), siendo este último complementario a una región de GFP-mut2, y por tanto, fue utilizado como partidor reverso en estas amplificaciones. No debía haber amplificación utilizando estos partidores en la cepa silvestre, mientras que en las cepas con GFP-mut2, debían amplificar una banda de 1071 pb. En ambos casos, los resultados fueron los esperados (Figura 24). Por lo tanto, se pudieron obtener cepas de Anabaena sp. PCC 7120 con la fusión de all2320 con gfp-mut2, pero no en todas las copias del cromosoma de la cianobacteria. Lamentablemente, al observar la fluorescencia bajo microscopía confocal de estas cepas en donde All2320 contiene fusionada la proteína GFP-mut2, el marcaje fue imperceptible (Figura 25). 64 Figura 24. Corroboración de la segregación de las cepas de Anabaena sp. PCC 7120 con all2320 fusionado con gfp-mut2 (all2320 + gfp-mut2). ADN de cultivos de Anabaena sp. PCC 7120 cepa silvestre (WT) y cepas con all2320 fusionado con gfp-mut2 (mut2) fueron aislados y utilizados en reacciones de amplificación por PCR para corroborar la segregación de la inserción de gfp-mut2 en las copias del cromosoma de la cianobacteria. I. Se observa el contexto génico de all2320, los partidores utilizados (flechas), los tamaños de los amplicones esperados y el gel de electroforesis realizado a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de la cepa WT, la cual fue utilizada como control negativo de la inserción de gfp-mut2. Los resultados fueron un amplicón de 912 pb utilizando los partidores all2320-1 y all2320-9 (A) y ninguna amplificación utilizando los partidores all2320-1 y gfp4 (B). II. Se observa el contexto génico de all2320, los partidores utilizados (flechas), los tamaños de los amplicones esperados y los geles de electroforesis realizados a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de las tres cepas mut2. Los resultados fueron un amplicón de 912 pb utilizando los partidores all2320-1 y all2320-9 (cuadro rojo, C) y un amplicón de 1071 pb utilizando los partidores all2320-1 y gfp4 (cuadro rojo, D), indicando la inserción de gfp-mut2 contiguo a all2320 en las cepas mut2, pero no en todas las copias del cromosoma de la cianobacteria. 65 Figura 25. Visualización de la fluorescencia de All2320-GFP-mut2 en Anabaena sp. PCC 7120. Cultivos de una cepa de Anabaena sp. PCC 7120, que contiene a all2320 fusionado con gfp-mut2 (All2320-GFPmut2), crecidos en nitrato y en ausencia de nitrógeno disponible, fueron visualizados por microscopía confocal. La autofluorescencia roja es dada por la clorofila de cianobacterias. El marcaje de All2320-GFP-mut2 fue imperceptible. Barra blanca representa escala de 10 µm. La segunda metodología utilizada en esta tesis para localizar All2320 en la célula fue mediante la generación de un anticuerpo anti-All2320 mediante la expresión de la proteína en Escherichia coli y su posterior purificación (para más detalles de la generación de este anticuerpo ver Materiales y Métodos; anticuerpo realizado en colaboración con Dra. (c) Carla Trigo). Primero, se realizó un análisis Western Blot utilizando el anticuerpo generado en extractos celulares de Anabaena sp. PCC 7120 en la presencia y ausencia por 48 horas de nitrógeno disponible (Figura 26). Bajo ambas condiciones, una banda única correspondiente al tamaño aproximado de la proteína All2320 fue detectada (All2320 tiene un tamaño estimado de 25,04 kDa por programa bionformático Expasy (http://web.expasy.org/compute_pi/), y la banda obtenida presentó un tamaño de 30 kDa) (Figura 26). 66 Figura 26. Análisis Western Blot utilizando un anticuerpo anti-All2320 en Anabaena sp. PCC 7120. Extractos celulares de cultivos de Anabaena sp. PCC 7120 fueron crecidos en presencia de nitrato (NO3-) y en ausencia de nitrógeno disponible por 48 horas (N2). Una banda correspondiente a una proteína de aproximadamente 30 kDa (All2320 tiene un tamaño estimado de 25,04 kDa) fue detectada. 67 Posteriormente, cultivos de Anabaena sp. PCC 7120 crecidos en presencia de nitrógeno disponible o en su ausencia por 48 horas, fueron marcados con el anticuerpo anti-All2320 y analizados por microscopía confocal, en donde la auto-fluorescencia roja que se observó es producto de la clorofila presente en las células vegetativas de cianobacterias (Figura 27I). Bajo ambas condiciones, All2320 fue localizada en la membrana citoplasmática de las células del filamento de Anabaena sp. PCC 7120, siendo observada en diferentes sitios dependiendo de la fase de división celular en la que se encontraba cada célula. Se pudo observar a la proteína All2320 en la membrana de células en crecimiento (Figura 27I B), polarizada en la membrana de células en crecimiento (Figura 27I C), en la membrana de sólo una de las células hijas formadas durante la división celular (Figura 27I D), y en el polo de una célula de tamaño normal (Figura 27I E). Es importante mencionar que el marcaje del anticuerpo no se observa en los heterocistos. Esta localización diferencial de All2320 en la membrana citoplasmática sólo de células vegetativas de Anabaena sp. PCC 7120 dependiente de la fase de división celular en la que se encuentra cada célula del filamento, permite suponer una función de esta proteína en la división celular de cianobacterias filamentosas, como también proponer dos modelos de la secuencia de localización de All2320 durante este proceso (Figura 27II) (si se hubiera logrado la observación en células vivas de All2320 fusionada con GFP, se tendría la certeza de cuál de los dos es el modelo de secuencia de localización correcto). El modelo de la izquierda de la Figura 27II localiza primero a All2320 en la membrana de una célula en crecimiento que entrará en división (B), luego re-localizándose hacia un polo celular (C), y finalmente estableciéndose en una sola de las células hijas (D y E), en dónde se degrada cuando el proceso de división celular finaliza (A). Por otra parte, el modelo de la derecha muestra a All2320 como una proteína selectora de la célula que va a someterse a división celular, concentrándose primero en un polo de la célula que va a dividirse (E), después localizándose en toda la membrana de la célula en crecimiento, antes del inicio de la división (B), y finalmente polarizando (C), estableciéndose en sólo una de las células hijas (D), en dónde después se degrada cuando termina el proceso de división celular (A). 68 Figura 27. Localización de All2320 en filamentos de Anabaena sp. PCC 7120. (I) Cultivos de Anabaena sp. PCC 7120 crecidos en nitrato o incubados 48 horas en ausencia de nitrógeno disponible fueron marcados con el anticuerpo anti-All2320 y analizados por microscopía confocal. La autofluorescencia roja es dada por la clorofila de cianobacterias. All2320 fue localizada siempre en la membrana citoplasmática, pero de forma diferencial dependiendo del estado de división de cada célula (A: célula de tamaño normal sin marca del anticuerpo anti-All2320, B: All2320 en la membrana de células en crecimiento, C: All2320 polarizada en la membrana de células en crecimiento, D: All2320 en la membrana de sólo una de las células hijas durante la división celular, E: All2320 en el polo de una célula de tamaño normal). Escala 1 µm. (II) Modelos de secuencia de localización de All2320 durante la división celular. (Modelo izquierdo) All2320 se localiza primero en la membrana de una célula en crecimiento que va a dividirse (B), luego se re-localiza hacia un polo celular (C), finalmente residiendo en sólo una de las células hijas (D y E), en dónde se degrada cuando se completa la división celular. (Modelo derecho) All2320 se localiza primero en un polo de la célula que se va a dividir (E), después se localiza en la membrana completa de la célula cuando está en crecimiento, antes de la división celular (B), y luego se polariza (C), residiendo en sólo una de las células hijas (D) en dónde se degrada cuando termina la división. 69 2.2 Determinar el papel de All2320 en la estructura del filamento y en la diferenciación celular de Anabaena sp. PCC 7120. Para comprobar y analizar la potencial función de all2320 en el proceso de división celular de cianobacterias filamentosas, cepas mutantes que portan copias inactivadas de este gen mediante su interrupción con el cassette de resistencia a los antibióticos Estreptomicina (Sm) y Espectinomicina (Sp) C.S3, fueron generadas (all2320::C.S3) (para ver detalles de la generación de estas cepas, revisar Materiales y Métodos). Se obtuvieron más de cien cepas mutantes para el gen all2320, a las cuales se les realizó el ensayo de corroboración de la segregación, es decir, se verificó la interrupción del gen all2320 en todas las copias del cromosoma de esta cianobacteria por el cassette C.S3. Para esto, se realizó una reacción de PCR utilizando los partidores all2320-1 y all2320-2 (Tabla 1), los cuales debían amplificar una banda de tamaño de 1039 pb en la cepa silvestre, mientras que en la cepa mutante no debían amplificar ya que la inserción del cassette C.S3 genera un amplicón de gran tamaño (aproximadamente 3.000 pb) que no es fácilmente amplificable en la reacción. El resultado de la corroboración de la segregación mostró la presencia de la banda de 1039 pb en la cepa silvestre. Sin embargo, también se observó esta banda en la cepa mutante (Figura 28), indicando que hay copias del cromosoma de la cianobacteria que se mantuvieron silvestres y no adquirieron el cassette de resistencia, siendo entonces la cepa all2320::C.S3, no segregante para la inactivación de all2320. Luego, para corroborar que efectivamente existían copias del cromosoma de la cepa all2320::C.S3 con all2320 interrumpido por C.S3, se utilizaron en una reacción de PCR los partidores all2320extCS3F y CS3 rev, en donde este último partidor es complementario a una región del cassette de resistencia C.S3, por lo cual no debía ampliflicar con estos partidores en la cepa silvestre, mientras que en la cepa mutante debían amplificar una banda de 535 pb. En ambos casos, los resultados de las amplificaciones utilizando estos partidores fueron los esperados (Figura 28). Por lo tanto, se pudo obtener una cepa de Anabaena sp. PCC 7120 con el gen all2320 inactivado, pero no en todas las copias del cromosoma de la cianobacteria, es decir, una cepa no segregante para la mutación de all2320 (cepa all2320::C.S3). 70 Figura 28. Corroboración de la segregación de las cepas de Anabaena sp. PCC 7120 mutante para all2320 (all2320::C.S3). ADN de cultivos de Anabaena sp. PCC 7120 cepa silvestre (WT) y cepa mutante para all2320 mediante la interrupción del gen con el cassette C.S3 de resistencia a los antibióticos Sm y Sp (cepa all2320::C.S3), fueron aislados y utilizados en reacciones de amplificación por PCR para corroborar la segregación de la inserción del cassette C.S3 en las copias del cromosoma de la cianobacteria. I. Se observa el contexto génico de all2320, los partidores utilizados (flechas), los tamaños de los amplicones esperados y el gel de electroforesis realizado a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de la cepa WT, la cual fue utilizada como control negativo de la inserción de C.S3. Los resultados fueron un amplicón de 1039 pb utilizando los partidores all2320-1 y all2320-2 (A) y ninguna amplificación utilizando los partidores all2320extCS3F y CS3rev (B). II. Se observa el contexto génico de all2320, los partidores utilizados (flechas), los tamaños de los amplicones esperados y los geles de electroforesis realizados a los productos de PCR obtenidos de utilizar como templado el ADN obtenido de la cepa all2320::C.S3. Los resultados fueron un amplicón de 1039 pb utilizando los partidores all2320-1 y all2320-2 (cuadro rojo, C) y un amplicón de 535 pb utilizando los partidores all2320extCS3F y CS3rev (cuadro rojo, D), indicando la inserción del cassette C.S3 interrumpiendo e inactivando el gen all2320 en la cepa all2320::C.S3, pero no en todas las copias del cromosoma de la cianobacteria. 71 Interesantemente, al analizar el fenotipo de la cepa all2320::C.S3, se distinguían dos tipos diferentes de filamentos cuando el cultivo era crecido tanto en presencia como en ausencia de nitrógeno disponible: la mayoría de los filamentos presentaban un fenotipo silvestre, sin mostrar diferencias en el largo de los filamentos o en el tamaño celular al compararlos con filamentos de Anabaena sp. PCC 7120, mientras que la minoría de los filamentos revelaban una estructuración en más de un eje al compararlos con la cepa silvestre (Figura 29a). Notablemente, cuando estos cultivos fueron crecidos en ausencia de nitrógeno disponible, los filamentos con divisiones celulares alteradas mostraron diferenciación y patrón normal de formación de heterocistos (Figura 29), comparable a lo observado en la cepa silvestre (Figura 29a), indicando nuevamente que este gen no tiene relación con este tipo de diferenciación de heterocistos. La cepa all2320::C.S3 también fue observada por microscopía electrónica de transmisión, en donde se pudo evidentemente registrar que el septo en formación en algunas de las células de los filamentos se encontraba en una disposición atípica, impidiendo el desarrollo y crecimiento de un filamento normal (Figura 29b). 72 Figura 29. Fenotipo de Anabaena sp. PCC 7120 cepa silvestre y cepa mutante del gen all2320 (all2320::C.S3). . Cultivos de Anabaena sp. PCC 7120 cepa silvestre (WT) y cepa mutante de all2320 (all2320::C.S3) crecidos en presencia (N+) o en ausencia de nitrógeno disponible (N-) (en presencia de Sm y Sp para la cepa mutante de all2320) fueron observados por microscopía óptica (a) y electrónica de transmisión (b). La cepa all2320::C.S3 presenta algunos filamentos estructurados en más de un eje y heterocistos normales. Flechas blancas apuntan a heterocistos. Barra blanca 10 µm, barra negra 1 µm. 73 Al crecer la cepa all2320::C.S3 en medio sólido y líquido en presencia y en ausencia de nitrógeno disponible, pude detectar que aproximadamente a las 48 horas de crecimiento, sólo los filamentos que presentaban estructuras en más de un eje empezaron a desaparecer de los cultivos (crecimiento en medio sólido, filamentos visualizados por microscopía óptica, Figura 30). Figura 30. Filamentos de Anabaena sp. PCC 7120 cepa silvestre y filamentos de Anabaena sp. PCC 7120 cepa mutante de all2320 con fenotipo de filamentos estructurados en más de un eje y fenotipo silvestre crecidos en medio sólido. Cultivos de Anabaena sp. PCC 7120 cepa silvestre y cepa mutante de all2320 fueron crecidos en medio sólido Agar con nitrógeno disponible (en presencia de Sm y Sp para la cepa mutante de all2320) y visualizados algunos de sus filamentos a las 2, 24 y 48 horas después de sembrados. Los filamentos de la cepa mutante de all2320 con fenotipo de filamentos estructurados en más de un eje no sobrevivían por más de 48 horas después de sembrados (fenotipo letal), no así los filamentos de la cepa mutante de all2320 con fenotipo silvestre (WT) (fenotipo no letal) o los filamentos de la cepa silvestre (WT), los cuales fueron sembrados como controles positivos de crecimiento. Barra 20 µm. 74 DISCUSIÓN Los dos genes estudiados en esta tesis fueron seleccionados a partir del trabajo realizado en nuestro laboratorio por Stucken y colaboradores (2010), en el cual se secuenciaron los genomas más pequeños hasta ahora descritos de cianobacterias filamentosas (Raphidiopsis brookii D9 de 3,2 Mpb y Cylindrospermopsis raciborskii CS-505 de 3,9 Mpb). Un análisis genómico comparativo entre los genomas de cianobacterias unicelulares y filamentosas, incluyendo a C. raciborskii y R. brookii, permitió reducir a sólo 10 el número de genes exclusivos de cianobacterias filamentosas. En esta tesis se caracterizaron dos de ellos: all1765 y all2320, ambos de Anabaena sp. PCC 7120. Las tres estrategias utilizadas en este estudio para determinar la función de las proteínas All1765 y All2320 en las estructuras y propiedades que determinan la generación de los filamentos de Anabaena sp. PCC 7120 fueron: la evaluación de la expresión de los genes all1765 y all2320 en condiciones que afectan la diferenciación de células del filamento; la identificación de la localización de las proteínas All1765 y All2320 en la célula; y por último, la evaluación del fenotipo generado a partir de cepas mutantes de los genes all1765 y all2320 en comparación con la cepa silvestre. 1.- all1765 El análisis de los dominios funcionales de All1765 indicó que esta proteína presenta un sitio de metilación amino terminal, que por lo que se conoce, proteínas con este sitio incluyen proteínas que forman los pili de las vías de secreción tipo II y IV. Estas proteínas que forman los pili, las pilinas, se ha descrito poseen un péptido señal que les permite ser transportadas a la membrana interna de bacterias Gram negativas (Filloux, 2004). Luego, este péptido señal es liberado de la proteína mediante proteólisis por peptidasas especializadas, las cuales normalmente se encuentran codificadas por genes que pertenecen al operón del cual son parte las pilinas (Filloux, 2004). Los péptidos señales de pilinas descritos en bacterias Gram-negativas, presentan una secuencia aminoacídica característica y un sitio de corte específico reconocido por las peptidasas, el cual es conformado por una glicina y una fenilalanina (Filloux, 2004). El análisis in silico realizado a la proteína All1765 arrojó la presencia del sitio de corte específico de péptidos señales de pilinas. Sin embargo, la secuencia aminoacídica del péptido señal no es homóloga a las 75 descritas para péptidos señales de pilinas. A su vez, el tamaño del supuesto péptido señal de All1765 es poco común según los reportes de pilinas descritos en diferentes bacterias Gramnegativas, el cual consiste generalmente en 6 o 7 residuos, con pocas excepciones que superan los 15 aminoácidos (el supuesto péptido señal de All1765 tiene un tamaño de 21 residuos) (Craig y cols., 2004). No obstante, se debe tomar en cuenta que la base de datos disponible de secuencias de péptidos señales de pilinas de bacterias Gram-negativas no incluye cianobacterias, por lo que no se tiene referencia de la secuencia ni del largo esperado de péptidos señales de pilinas de estos microorganismos. Cabe destacar también, que la proteína codificada por el gen all1770, el cual es uno de los genes que pertenece al cluster génico en el cual se encuentra all1765 y que se transcribe en su misma dirección, posee un 8 % de identidad aminoacídica con una proteasa alcalina de Pseudomonas TACII18. A pesar de no ser significativa, es la mayor homología encontrada para All1770, por lo que podría ser una proteasa encargada de reconocer y separar el supuesto péptido señal de All1765. Como ya fue mencionado previamente, la diferenciación de células vegetativas en heterocistos se genera cuando el medio en el que se encuentran los filamentos no contiene suficiente concentración de nitrógeno disponible, por lo que estas células especializadas se vuelven necesarias para llevar a cabo la fijación (Flores y Herrero, 2010). La expresión de all1765 (y también de all2320) fue evaluada por dos metodologías, Northern Blot y RT-PCR en tiempo real, ambas en deprivación de nitrógeno disponible para investigar si este gen tiene algún efecto en la diferenciación a heterocistos y/o si se encuentra involucrado en el intercambio de moléculas entre las células del filamento cuando hay fijación de nitrógeno. Se usaron ambas estrategias ya que permitían recuperar resultados diferentes que complementaban los análisis. Por ejemplo, el análisis Northern Blot entrega una cuantificación imprecisa de la expresión de los genes debido a la alta variabilidad en la medición de los pixeles de las bandas obtenidas en cada filtro, a diferencia de las cuantificaciones exactas entregadas por el análisis mediante RT-PCR en tiempo real. Sin embargo, la estrategia del Northern Blot permitía saber si el gen era parte de un operón, lo que era muy relevante al momento de diseñar las cepas mutantes de los genes para así intentar evitar un efecto polar, en el caso que el gen fuera efectivamente parte de un cluster génico. Fue así como, por el resultado del análisis Northern Blot, pudimos comprobar que all1765 es parte de un operón (Figura 11), lo que nos llevó a diseñar la cepa mutante de este gen mediante su deleción del genoma y no mediante la interrupción del gen con un cassette de 76 resistencia a antibióticos, que a pesar de ser una metodología que permite seleccionar de constante y eficiente las cepas mutantes, podía causar la terminación de la transcripción de algunos de los genes del operón al cual pertenece all1765. Además, el análisis Northern Blot realizado para analizar la expresión del gen en la cepa silvestre, permitió analizar también, y en el mismo filtro, la expresión del gen all1765 en cepas mutantes de genes que codifican los factores de transcripción asociados al metabolismo del nitrógeno NtcA (Frías y cols., 1994) y HetR (Buikema & Haselkorn, 1991), resultado que indicó que estos factores no controlan la expresión de all1765 al menos hasta las 24 horas en deprivación de nitrógeno (Figuras 11), información que habría sido más laboriosa de obtener por rt-PCR en tiempo real. En cuanto a los resultados obtenidos de los análisis de expresión génica del gen, se pudo concluir que el comportamiento transcripcional del gen all1765 es similar independiente de la fuente de nitrógeno en el medio de cultivo (Figura 12). Por otra parte, el aumento en la abundancia relativa del transcrito del gen estructural de heterocistos hglD (Bauer y cols., 1997) se inicia a las 24 horas en deprivación de nitrógeno disponible, lo cual es lógico ya que este gen es marcador de diferenciación tardía de heterocistos, y a las 24 horas bajo esta condición, se están terminando de desarrollar la mayoría de los heterocistos de los filamentos. Para llevar a cabo la localización subcelular de All1765 (y All2320), se realizaron dos metodologías en paralelo: la generación de cepas con la proteína fusionada con GFP y la generación de un anticuerpo contra la proteína. La ventaja de obtener cepas con proteínas fusionadas con GFP es que, de resultar funcionales estas proteínas fluorescentes, se podría visualizar la secuencia de movimiento de All1765 (y All2320) en la célula viva. Sin embargo, se ha descrito que proteínas fusionadas con GFP pueden plegarse de forma incorrecta (Waldo y cols., 1999) o exhibir un procesamiento alterado (Bertens y cols., 2003), lo que afecta la actividad y movimiento natural de las proteínas dentro de la célula. Por este motivo, se decidió realizar también la generación del anticuerpo. Al momento de generar las cepas con el gen all1765 fusionado con GFP, se pudieron obtener cepas de Anabaena sp. PCC 7120 con all1765 fusionado con gfp-mut2 y con sf-gfp, pero ninguna de ellas fue segregante para la fusión respectiva (Figura 14). Este resultado era esperable, ya que, como se demostró anteriormente, all1765 es parte de un operón, y la inserción de un plásmido (para generar las fusiones) interrumpiendo el operón pudo generar un efecto polar, produciendo la terminación de la transcripción de algunos de los genes que lo componen. En este caso se puede suponer que mutantes segregantes en este gen son 77 inviables. Otra explicación de la formación de cepas no segregantes para las fusiones con GFP es un plegamiento incorrecto o mal procesamiento de las proteínas fusionadas, como se mencionó anteriormente. Esto podría perturbar la actividad natural y movimiento de las proteínas en la célula, transformándolas en proteínas no funcionales, motivo por el cual cromosomas con las proteínas silvestres serían requeridos para el crecimiento de la cianobacteria. Al observar la fluorescencia bajo microscopía confocal de estas cepas en donde All1765 contiene fusionada la proteína GFP-mut2 o sf-GFP, el marcaje fluorescente fue imperceptible (Figuras 15 y 16). Este resultado no fue sorpresivo, ya que la obtención de cepas no segregantes para las fusiones hacía suponer una perturbación en las proteínas cuando se encontraban fusionadas con GFP, causando, posiblemente, la formación de proteínas no funcionales. La otra metodología utilizada en esta tesis para localizar la proteína en la célula fue mediante la generación de un anticuerpo. Para producir el anticuerpo anti-All1765, se seleccionó un minipéptido de 8 aminoácidos mediante análisis in silico de la proteína, el cual fue sintetizado por la empresa externa Genscript. Este análisis consistió en la selección de un péptido que se encontrara expuesto en la proteína y que no arrojara similitud con otras proteínas de Anabaena sp. PCC 7120. Los resultados de esta tesis mostraron mediante inmunodetecciones por fluorescencia y observación por microscopía confocal, que All1765 se localiza en el citoplasma, en la membrana celular, concentrada en el septo de algunas células del filamento cuando son crecidos en presencia y ausencia de nitrógeno disponible y en el polo sin contacto celular de células terminales del filamento (Figura 17). A pesar de localizarse concentrada en los septos, All1765 no se localiza en todas las conexiones intercelulares del filamento como las proteínas de los septosomas SepJ y FraD, las cuales se localizan en todos los septos celulares del filamento de forma simultánea (Flores y cols., 2007; Merino-Puerto y cols., 2010). Por otra parte, la localización en el polo sin contacto celular de células terminales del filamento sugiere que la función de All1765 no estaría relacionada con adhesión célula-célula o a comunicación intercelular, ya que esta no sería la localización lógica de proteínas con estas funciones. Por otra parte, si se considera la presencia de un péptido señal en la proteína All1765 por el previo análisis estructural realizado, esto podría explicar su localización en el citoplasma, en donde la proteína sería sintetizada, para luego ser translocada a la membrana celular, especialmente a los septos celulares. Es importante indicar que no se observó marcaje del 78 anticuerpo en los heterocistos, indicando que All1765 no tendría función en fijación de nitrógeno disponible ni en transporte de nitrógeno fijad desde los heterocistos hacia el resto de las células del filamento. En cuanto al resultado del análisis del fenotipo de las cepas mutantes en all1765, este análisis reveló que las cepas mutantes del gen all1765 presentaban dos fenotipos distintos: el de la cepa Δall1765 fenotipo 2, que fue advertido sólo en una cepa de un total de aproximadamente cien cepas analizadas, y el observado en las otras colonias, representados por las cepa Δall1765 fenotipo 1. La cepa Δall1765 fenotipo 2 se caracterizó por ser incapaz de sobrevivir más de 48 horas en ausencia de nitrógeno disponible, no así las cepas Δall1765 fenotipo 1 (Figura 19a). La hipótesis más probable para la aparición de este fenotipo parece ser la presencia de una mutación adicional a la generada intencionalmente al gen all1765, principalmente porque al verificar la deleción de all1765 en esta cepa, ésta no presentó diferencias con la generada en las otras cepas mutantes de este gen. Se descartó así, la deleción de una fracción mayor del genoma de la cianobacteria aledaño a all1765, escenario que podría haber generado el fenotipo observado. La o las mutaciones adicionales pudieron haberse generado por diversos motivos, como por ejemplo por errores generados en la replicación del ADN por la enzima ADN polimerasa, la cual podría adicionar o delecionar un nucleótido durante este proceso. Otra forma de generar mutaciones es mediante el movimiento de transposones o elementos de inserción en el genoma. Estos son elementos de transposición que pueden ser encontrados en genomas de organismos procariontes y eucariontes. En Anabaena sp. PCC 7120, se han caracterizado elementos de inserción, los cuales mediados por la enzima transposasa, son cortados y trasladados de un lugar a otro en el genoma, lo que podría generar distintos tipos de mutaciones, como cambios en el marco de lectura de uno o varios genes (Cai, 1991; Elhai y cols., 2008). Este tipo de transposiciones podrían estar relacionadas con los cambios ambientales a los cuales están expuestos los microorganismos. Por otro lado, es posible que una mutación adicional haya sido generada por virus de cianobacterias, también llamados cianofagos, ya que en algunos de estos virus, se han encontrado genes involucrados con la fotosíntesis de algunas cepas de cianobacterias (Suttle, 2000). Interesantemente, cuando filamentos de la cepa Δall1765 fenotipo 2 crecidos en deprivación de nitrógeno por menos de 48 horas fueron subcultivados en presencia de nitrógeno disponible, se recuperaron, lo que indica que el resultado letal de la mutación sería solo frente a la ausencia de nitrógeno disponible en el medio, sin afectar aparentemente la estructura o funcionamiento del 79 resto del filamento de forma irreversible. Esto apoya la idea de una mutación adicional que afectaría entonces la fijación de nitrógeno (o funcionalidad de los heterocistos) o el paso de nitrógeno disponible desde los heterocistos hacia el resto del filamento. No obstante, se observa normal la estructura de los heterocistos (Figura 19b), lo que indica que estas células serían funcionales y estarían cumpliendo su rol de fijar nitrógeno. De esta forma, la suposición más factible sobre la consecuencia de la o las mutaciones adicionales es que éstas están afectando el paso de nitrógeno desde los heterocistos hacia las células vegetativas. Para comprobar esta aseveración, se realizará prontamente la medición de la fijación de nitrógeno en los filamentos de la cepa Δall1765 fenotipo 2 mediante la técnica de reducción de acetileno (tesis de pregrado de Gabriela Gómez). Asimismo, al ser tan interesante el fenotipo observado en la cepa Δall1765 fenotipo 2 por su similitud al descrito en la cepa mutante del gen fraD (Merino-Puerto y cols., 2010), se secuenciará el genoma de esta cepa, para así compararlo con la cepa silvestre y averiguar el gen o los genes que fueron mutados adicionalmente y comenzar su estudio y descripción a cabalidad. Por otra parte, el otro fenotipo 1 observado en la mayoría de las cepas mutantes del gen all1765, se caracterizó por la presencia de filamentos entre un 31 % y un 58 % más largos que los observados para la cepa silvestre (Figura 20). Este fenómeno se asoció al proceso de fragmentación, el cual es un tipo de reproducción previamente observado en cianobacteria filamentosas que se considera una estrategia de dispersión hacia mejores condiciones ambientales y que aún no se ha descrito molecularmente (Cirés y cols., 2013; Alster y cols., 2010; Adamec y cols., 2005). Esta asociación se debió no sólo a la observación de este fenotipo en la mutante, que indica que la ausencia de all1765 está inhibiendo este proceso, sino también a la localización de la proteína All1765 concentrada en los septos celulares y en el polo si contacto celular de células terminales del filamento, que podría la ubicación lógica de proteínas que participan en este proceso. Al ser la deprivación de nitrógeno disponible una condición desfavorable para la vida de las cianobacterias, se probó si esta condición favorecía la fragmentación del filamento de Anabaena sp. PCC 7120. Se pudo observar que efectivamente los filamentos de la cepa silvestre disminuyen su largo a las 48 y 72 horas en ausencia de nitrógeno disponible en el medio con respecto a los filamentos crecidos en presencia de nitrógeno disponible. (Figura 20). Esta observación se convierte en la primera evidencia de una condición desfavorable que genera 80 fragmentación en Anabaena sp. PCC 7120. Por otra parte, el resultado esperado del análisis del tamaño de los filamentos de la cepa Δall1765 fenotipo 1 en deprivación de nitrógeno disponible era el crecimiento de filamentos largos, de similar tamaño a los observados en presencia de nitrógeno disponible, ya que el gen all1765, que se suponía estaba asociado a la fragmentación, se encontraba delecionado en esta cepa. Sin embargo, se pudo advertir que en ausencia de nitrógeno disponible, el cultivo de la cepa Δall1765 fenotipo 1 presentó fragmentación de sus filamentos, alcanzando tamaños equivalentes a los observados en la cepa silvestre crecida en esta misma condición (Figura 20). Este resultado puede explicarse atribuyéndole a All1765 una función en fragmentación como parte del ciclo reproductivo de la cianobacteria, mientras que en condiciones desfavorables, regiría otro mecanismo de fragmentación de los filamentos de Anabaena sp. PCC 7120 como estrategia de dispersión hacia condiciones más favorables, mecanismo que actuaría independiente de la proteína All1765. 2.- all2320 El análisis in silico realizado a All2320 señaló que la proteína contiene 197 aminoácidos y presenta una región coiled-coil de 42 residuos cerca de su extremo amino terminal (aminoácidos 27 al 69), y un dominio de transmembrana de 22 residuos cercano a su extremo carboxilo terminal (aminoácidos 166 a 188). El dominio coiled-coil consiste en el enrollamiento de alfa hélices formando una estructura tipo “cuerda”, la cual deja la región hidrofóbica de la estructura en el centro (Burkhard y cols., 2001). Las interacciones entre coiled-coils de diferentes proteínas juega un rol fundamental en la formación de complejos proteicos en procesos biológicos muy relevantes como lo son la transcripción y la división celular, entre otros (Rackham y cols., 2010). Toma interés este dominio en All2320 al observar que también se encuentra presente en las proteínas SepJ y FraD, que como ya fue mencionado previamente, participan en mantener la integridad del filamento formando parte del septosoma (Flores y cols., 2007; Merino-Puerto y cols., 2010). La expresión de all2320, al igual que la expresión del gen all1765, fue evaluada por Northern Blot y RT-PCR en tiempo real, ambas en deprivación de nitrógeno disponible para investigar si este gen tiene algún efecto en la diferenciación a heterocistos y/o si se encuentra involucrado en el intercambio de moléculas entre las células del filamento cuando hay fijación de nitrógeno. El análisis Northern Blot reveló que el gen all2320 es monocistrónico (Figura 21), 81 motivo por el cual la cepa mutante en este gen se diseñó mediante la interrupción del gen con un cassette de resistencia a antibióticos ya que no se generaría un efecto polar. A su vez, y al igual para all1765, el análisis Northern Blot permitió analizar también la expresión del gen all2320 en cepas mutantes de genes que codifican los factores de transcripción asociados al metabolismo del nitrógeno NtcA (Frías y cols., 1994) y HetR (Buikema & Haselkorn, 1991), resultado que indicó que estos factores no controlan la expresión de all2320 al menos hasta las 24 horas en deprivación de nitrógeno (Figura 21). Las dos estrategias utilizadas para realizar los análisis de expresión génica del all2320 revelan una expresión del gen mantenida en el tiempo, tanto en presencia como en ausencia de nitrógeno disponible (Figura 21 y 22), lo que indica que All2320 sería constantemente necesaria para un proceso biológico en cianobacterias filamentosas que aparentemente operaría independiente de la fuente de nitrógeno presente en el medio (nitrógeno molecular o disponible) y, por ende, no tendría relación con el metabolismo del nitrógeno. Para llevar a cabo la localización subcelular de All2320, se realizaron las mismas dos metodologías utilizadas para localizar All1765: la generación de cepas con la proteína fusionada con GFP y la generación de un anticuerpo anti-All2320. Se lograron obtener cepas de Anabaena sp. PCC 7120 con all2320 fusionado con gfp-mut2, pero ninguna de estas cepas fue segregante para las fusiones (Figuras 24). La explicación es la posible formación de cepas en donde la proteína fusionada con GFP haya presentado un plegamiento incorrecto o mal procesamiento, lo que habría generado proteínas All2320-GFPmut2 no funcionales. Al observar la fluorescencia bajo microscopía confocal de estas cepas, el marcaje fluorescente fue imperceptible (Figura 25), resultado que no fue sorpresivo, ya que la obtención de cepas no segregantes para las fusiones hacía suponer una perturbación en la funcionalidad de las proteínas fusionadas con GFP. La otra metodología utilizada para localizar la proteína en la célula fue mediante la generación de un anticuerpo anti-All2320. Este anticuerpo no se pudo generar mediante la síntesis de un minipéptido, ya que la proteína cristalizada de mayor identidad con All2320 no permitía una estimación confiable de los péptidos expuestos. Además, los que se pudieron obtener usando los modelos que más se acercaban, arrojaban similitud con otros péptidos de proteínas de la cianobacteria. Por estos motivos, se decidió producirlo mediante la expresión de la proteína en E. coli y su posterior purificación. 82 Los análisis Western Blot realizados utilizando el anticuerpo generado, fueron realizados en extractos celulares de Anabaena sp. PCC 7120 en la presencia y ausencia por 48 horas de nitrógeno disponible. De este análisis, se obtuvo una banda de 30 kDa, tamaño similar al estimado in silico por el programa bioinformático Expasy (25,04 kDa). En cuanto a la localización de esta proteína, ésta se observó en la membrana citoplasmática de las células del filamento de Anabaena sp. PCC 7120 en diferentes sitios celulares dependiendo de la fase de división celular en la que se encontraba cada célula (Figura 27). Con este antecedente, queda de manifiesto que All2320 es una proteína que participa en la división celular de cianobacterias filamentosas, y que se muestra en locaciones diferentes durante el proceso de división. Sin embargo, sin la secuencia del supuesto desplazamiento de la proteína en células vivas, no pudimos establecer sólo una alternativa de cómo sería el movimiento de la proteína, por lo que se formularon dos modelos de la posible secuencia de desplazamiento de All2320 (Figura 27). Los modelos se diferencian principalmente en la localización inicial de All2320 en la célula, ya que podría posicionarse en toda la membrana de la célula que va a dividirse, y después redistribuirse de modo que comande la generación de la nueva célula, manteniéndose en sólo una de las células hijas (Figura 27II, Modelo izquierda), o concentrándose primero en un polo de la célula que va a dividirse, actuando como una proteína selectora de la célula que va a someterse a división celular (Figura 27II, Modelo derecha). Una proteína que también posee un dominio coiled-coil y una función asociada a la división celular, es la proteína de la bacteria Gram-positiva Streptomyces coelicolor, Scy (“Streptomyces cytoskeletal element”) (Holmes y cols., 2013). S. coelicolor tiene una estructura filamentosa ramificada similar a la de los hongos. Posee un ciclo de vida y tipo de reproducción diferente al observado en la mayoría de las bacterias, iniciando su crecimiento con la formación de un micelio vegetativo desde una espora, seguido de un micelio aéreo que produce esporas. Cuando se dividen las células de S. coelicolor, no hay generación de dos células completamente separadas como en la división por fisión binaria, sino que cadenas de células permanecen unidas formando los micelios y sus ramificaciones (Chater, 2001). Scy es considerada una proteína organizadora de los “brotes” de las ramificaciones de los micelios de la bacteria, ya que se localiza polarizada en un sólo extremo celular, de manera muy similar a All2320 en Anabaena sp. PCC 7120, en los sitios de elongación celular en donde las ramificaciones se encuentran en crecimiento, como también se observa marcando puntos de futuras ramificaciones antes de la aparición de los brotes 83 (Holmes y cols., 2013). A su vez, la ausencia del gen scy en S. coelicolor genera severos defectos en la división celular afectando la geometría de los micelios, como también durante la esporulación, produciendo esporas con distintas cantidades de ADN (Holmes y cols., 2013). En resumen, All2320 y Scy son muy parecidas en muchos aspectos: ambas poseen un dominio coiled-coil, se localizan polarizadas en la célula y poseen funciones asociadas al proceso de división celular. Estas semejanzas podrían hacer que All2320 tuviera un comportamiento similar al de Scy en cuanto a la marcación de los sitios de divisiones futuros, pudiendo actuar como proteína selectora de la célula que va a someterse a división celular. Si este fuera el caso, el modelo de la derecha de la Figura 27II sería el más correcto de la secuencia de movimiento de All2320 en la célula. El antecedente más relevante observado de la localización de All2320 es su posicionamiento en una sola de las células hijas después de la división celular (Figura 27). En los procariontes, el método de división celular más común se denomina división o fisión binaria. En este proceso, la duplicación celular va precedida por la replicación del cromosoma bacteriano, para luego separarse el ADN “original” de su “réplica” en zonas diferentes de la membrana celular. De esta forma, cuando se comienzan a separar las células hijas en el proceso de citocinesis, también se separan los cromosomas idénticos. Finalmente, se obtienen como resultado dos células hijas idénticas. Se ha descrito que, tanto cianobacterias unicelulares como filamentosas, se dividen mediante fisión binaria (Nakanishi y cols., 2009). Sin embargo, al observar la localización de All2320 en una sola de las células hijas después de la división, vemos claramente que, al menos por la presencia de All2320, las células hijas obtenidas no son idénticas, observación que demuestra que la división celular en Anabaena sp. PCC 7120 no es un proceso de división binaria como el observado en otras bacterias. Es importante recalcar que el marcaje del anticuerpo no se observa en los heterocistos, lo que es lógico ya que éstas son células de diferenciación terminal, es decir, que no poseen la facultad de dividirse, por lo que All2320, siendo una proteína de división celular, no debiera expresarse en este tipo celular. En la búsqueda de otorgarle una función a la proteína All2320, que de acuerdo a su localización subcelular se infería estaba involucrada en la división celular de Anabaena sp. PCC 7120, se realizó un análisis fenotípico a la cepa all2320::C.S3, mutante del gen all2320. Es importante mencionar que se realizó la mutación del gen mediante su interrupción con un cassette 84 de resistencia a antibiótico (C.S3, SmR SpR) ya que siempre es conveniente generar una mutante que se pueda seleccionar fácilmente y así evitar contaminaciones. Aparte, por el análisis Northern blot, all2320 resultó ser monocistrónico, por lo que no había riesgo de producir efectos polares en el genoma de la cianobacteria. Este análisis reveló la presencia de dos tipos diferentes de filamentos cuando el cultivo era crecido tanto en presencia como en ausencia de nitrógeno disponible: la mayoría de los filamentos presentaban un fenotipo semejante al de la cepa silvestre, mientras que la minoría de los filamentos revelaban una estructuración en más de un eje (Figura 29). Este fenotipo de filamentos estructurados en más de un eje se debe a una disposición atípica de los septos de las células que conforman estos filamentos (Figura 29). Interesantemente, al crecer la cepa all2320::C.S3 en medio sólido y líquido en presencia y en ausencia de nitrógeno disponible, se pudo detectar que aproximadamente a las 48 horas de crecimiento, sólo los filamentos estructurados en más de un eje empezaron a desaparecer de los cultivos (Figura 30, crecimiento en medio sólido). Este resultado se explica suponiendo un fenotipo letal generado por la disrupción de all2320 en esta cepa mutante, en dónde en los filamentos estructurados en más de un eje, la mutación se encontraba presente en todas las copias del cromosoma de la cianobacteria. En los filamentos con fenotipo silvestre de la cepa all2320::C.S3, las copias inactivadas de all2320 se encontraban probablemente ausentes en la mayoría de las copias del cromosoma, lo que permitió su supervivencia. En consecuencia, all2320 parece ser un gen esencial de cianobacterias filamentosas que participa en la división celular, ya que mutantes en este gen, afectan la disposición del septo celular durante este proceso. La división celular es un proceso que ha sido pobremente descrito en cianobacterias, a diferencia de lo extensivamente que ha sido estudiado en procariontes unicelulares modelo como Escherichia coli (Gram-negativa) y Bacillus subtilis (Gram-positiva). En ambos microorganismos, varios componentes y pasos de la división celular han sido revelados, en dónde la primera etapa es la formación del anillo de división Z en el sitio de división celular estrictamente en la mitad de la célula, anillo que se encuentra compuesto por la proteína homóloga a tubulina, FtsZ (Errington y cols., 2003). Dónde y cuándo se lleva a cabo la división, depende de la localización y tiempo de ensamble del anillo Z, procesos que son controlados por mecanismos regulatorios. Entre ellos podemos encontrar el sistema Min, el cual está encargado de seleccionar el sitio de división. En bacterias Gram-negativas, el sistema Min impide la formación del anillo Z en cualquier zona excepto en el medio de la célula, actuando MinC y 85 MinD juntos oscilando de polo a polo celular como un regulador negativo del ensamblaje del anillo y MinE impartiendo una especificidad topológica al complejo MinCD, impidiendo su accionar en el medio de la célula (Errington y cols., 2003). Por otra parte, bacterias Grampositivas usan DivIVA, una proteína que actúa como punto de referencia en la localización de la formación del anillo Z reclutando las proteínas MinC y MinD cuando forma un anillo a cada lado del septo en formación, evitando el ensamblaje del anillo Z en la vecindad inmediata (Lutkenhaus, 2012). Otro mecanismo regulatorio que controla el ensamblaje del anillo Z es el logrado por varias proteínas de división asociadas a membrana como FtsA, FtsQ(DivIB), FtsL, FtsB(DivIC), FtsW, y FtsI (“Fts” nomenclatura que aplica para bacterias Gram-negativas y “Div” nomenclatura que aplica para sus homólogos en bacterias Gram-positivas). Estas proteínas son reclutadas al anillo Z, donde modulan la polimerización de FtsZ formando un complejo llamado “divisoma”, el cual causa la invaginación de las membranas celulares para formar el septo de división. FtsA parece mediar la interacción de FtsZ con la membrana, mientras FtsI, y posiblemente FtsW, participan en la síntesis del peptidoglicano del septo en formación. Las funciones de FtsQ(DivIB), FtsL y FtsB(DivIC) no han sido definidas de forma precisa aún (Errington y cols., 2003; Goehring & Beckwith, 2005; Harry y cols., 2006). A pesar que las cianobacterias son consideradas bacterias Gram-negativas en morfología considerando su membrana externa (Flores y cols., 2006), son filogenéticamente cercanas a bacterias Gram-positivas (Battistuzzi & Hedges, 2009). En cuanto al proceso de división celular, genes de ambos tipos bacterianos tienen homólogos en genomas cianobacterianos (Miyagishima y cols., 2005). Además, por análisis mutacionales, Miyagishima y colaboradores (2005) observaron que tanto MinE, como la proteína tipo DivIVA, Cdv3, participan en la división celular de cianobacterias unicelulares. Las mutantes en minE de Synechococcus elongatus PCC 7942 producen células muy alargadas, mientras que la mutante en cdv3 de esta misma cepa se divide asimétricamente, al igual que la mutante en divIVA de B. subtilis (Miyagishima y cols., 2005; Cha & Stewart, 1997). Aunque se demostró que MinC y MinD son proteínas involucradas en la división celular de cianobacterias unicelulares (Mazouni y cols., 2004), aún se desconoce si oscilan como en E. coli, se localizan en la vecindad del septo como en B. subtilis, o ninguna de las dos. 86 En cuanto a cianobacterias filamentosas, no existe mayor información sobre las proteínas o mecanismos involucrados en la división celular, principalmente porque no se han intentado o logrado mutar la mayoría de los genes involucrados en este proceso en estas cianobacterias. Muchas veces falla la generación de mutantes de genes asociados a la división celular, obteniéndose cepas inviables, porque muchos de ellos son genes que codifican proteínas esenciales en este vital proceso. Una de estas proteínas esenciales en la división celular es la proteína de B. subtilis DivIC. DivIC, como se mencionó previamente, es parte de las proteínas que forman el divisoma, el cual causa la invaginación de las membranas celulares para formar el septo (Errington y cols., 2003; Harry y cols., 2006). La función específica que cumple DivIC en el divisoma no ha sido dilucidada aún, justamente por no poder obtener mutantes viables del gen que codifica esta proteína. Interesantemente, el programa Pfam 27.0, indica que All2320 tiene la puntuación e-value más baja, aunque débil (0,0017), con DivIC, indicando que al menos en cuánto a sus dominios funcionales, es la proteína más parecida a All2320. Al igual que All2320, DivIC (así como su homólogo en E. coli FtsB) posee un único segmento de transmembrana, como también un dominio coiled-coil en su región extracitoplasmática, el cual al igual que lo observado para All2320, constituye la mayor parte de la proteína (aunque a diferencia de lo que es predicho para All2320, el extremo carboxilo terminal de DivIC es periplásmico) (Katis y cols., 1997). En consecuencia, los antecedentes mencionados y los resultados de esta tesis, permiten considerar a All2320 como una proteína esencial durante la división celular de cianobacterias filamentosas. El rol de All2320 no está definido, pero podría ser parte de las proteínas que seleccionan y definen el plano de división como las que pertenecen al sistema Min, debido a las divisiones celulares en más de un eje observadas en la cepa mutante de all2320. Más aún, el fenotipo de esta mutante en all2320 en cuanto a la estructuración del filamento, es muy similar a la mutante observada del gen scy en Streptomyces coelicolor, en donde las hifas aéreas observadas se estructuran en más de un eje de división (Holmes y cols, 2013). A su vez, cuando en la cepa mutante en scy fue localizado el anillo de división Z, esté se observó de forma irregular, con efectos en su orientación (Holmes y cols, 2013). Esto indica que el mecanismo de selección del sitio de división celular en la mutante en scy, que involucraría al sistema Min, está afectado en esta mutante, pudiendo estar pasando lo mismo en la mutante en all2320 en Anabaena sp. PCC 7120. Alternativamente, All2320 podría pertenecer al complejo del divisoma, 87 principalmente por su similitud estructural con la proteína esencial DivIC. Otra posibilidad es que All2320 sea parte de un mecanismo de división celular nuevo propio de cianobacterias filamentosas, lo que no sería extraño por tres motivos: primero, porque es una proteína exclusiva de cianobacterias filamentosas; segundo, por su particular y novedosa localización; y tercero, porque las células de estos microorganismos desarrollan una inusual e incompleta división celular que resulta en una membrana externa (y periplasma) continua que rodea todo el filamento (Ris & Singh, 1961; Flores et al, 2006), por lo tanto la existencia de una proteína con una función en este proceso excepcional de la división celular de cianobacterias filamentosas sería necesaria. Es por estos motivos que es crucial la identificación de proteínas que podrían estar interactuando con All2320 (dado su dominio coiled-coil) para entender mejor su función concreta en la división celular de cianobacterias filamentosas. 88 CONCLUSIONES La proteína All1765 es una proteína que se localiza en algunos septos celulares y en el polo sin contacto celular de células terminales del filamento, y tiene participación en la regulación del largo de los filamentos de Anabaena sp. PCC 7120. La proteína All2320 se localiza en distintas zonas celulares asociadas a estapas de la división celular, siendo una proteína de división celular esencial para que la formación del septo celular se genere en una disposición normal en los filamentos de Anabaena sp. PCC 7120. La división celular en Anabaena sp. PCC7120 no es binaria, como ocurre en la mayoría de las bacterias. 89 BIBLIOGRAFÍA ADAMEC, F. 2005. Stress-induced filament fragmentation of Calothrix elenkinii (cyanobacteria) is facilitated by death of high-fluorescence cells. J. Phycol. 41:835–839. ALSTER, A.; KAPLAN-LEVY, R.; SUKENIK, A.; ZOHARY, T. 2010. Morphology and phylogeny of a non-toxic invasive Cylindrospermopsis raciborskii from a Mediterranean Lake. Hydrobiologia 639:115–128. AUSUBEL, FM.; BRENT, R.; KINGSTON, R.; MOORE, D.; SEIDMAN, JG.; SMITH, JA.; STRUHL, K. 2002. In: Short protocols in Molecular Biology. Wiley (ed). 1-29. AUSUBEL, F.M.; BRENT, R.; KINGSTON, R.E.; MOORE, D.D.; SEIDMAN, J.G.; SMITH, J.A.; STRUHL, K. 2008. CurrentProtocols in Molecular Biology. New York: Greene Publishing & Wiley-Interscience. BATTISTUZZI, FU AND HEDGES, SB. 2009. A Major Clade of Prokaryotes with Ancient Adaptations to Life on Land Mol. Biol. Evol. 26(2):335–343. BAUER, C.; BUIKEMA, WJ.; BLACK, K.; HASELKORN, R. 1995. A short-filament mutant of Anabaena sp. strain PCC 7120 that fragments in nitrogen-deficient medium. J. Bacteriol. 177:1520–1526. BAUER, CC.; RAMASWAMY, KS.; ENDLEY, S.; SCAPPINO, LA.; GOLDEN, JW.; HASELKORN R. 1997. Suppression of heterocyst differentiation in Anabaena PCC 7120 by a cosmid carrying wild-type genes encoding enzymes for fatty acid synthesis. FEMS Microbiol Lett 151:23–30. BERGMAN B., GALLON JR., RAI AN., STAL LJ. 1997. N2 fixation by non-heterocystous cyanobacteria. FEMS Microbiol Rev 19:139–185. 90 BERTENS P., HEIJNE W., VAN DER WEL N., WELLINK J., VAN KAMMEN, A. 2003. Studies on the C-terminus of the Cowpea mosaic virus movement protein. Arch. Virol. 148:265–279. BLACK, TA.; CAI, Y.; WOLK, CP. 1993. Spatial expression and autoregulation of hetR, a gene involved in the control of heterocyst development in Anabaena. Molecular Microbiology 9:77-84. BUIKEMA, WJ.; HASELKORN, R. 1991. Characterization of a gene controlling heterocyst differentiation in the cyanobacterium Anabaena 7120. Genes Dev 5: 321–330. BUIKEMA, WJ.; HASELKORN, R. 2001. Expression of the Anabaena hetR gene from a copper-regulated promoter leads to heterocyst differentiation under repressing conditions. PNAS 98:2729–2734. BURKHARD P., STETEFELD J., STRELKOV, SV. 2001. Coiled coils: a highly versatile protein folding motif. Trends Cell Biol. 2:82–88. CAI, Y.; WOLK, CP. 1990. Use of a conditionally lethal gene in Anabaena sp. strain PCC 7120 to select for double recombinants and to entrap insertion sequences. J. Bacteriol. 172: 3138–3145. CAI, Y. 1991. Characterization of insertion sequence IS892 and related elements from the cyanobacterium Anabaena sp. strain PCC 7120. Journal of Bacteriology.173:5771–5777. CASTRO, D.; VERA, D.; LAGOS, N.; GARCIA, C.; VASQUEZ, M. 2004. The effect of temperature on growth and production of paralytic shellfish poisoning toxins by the cyanobacterium Cylindrospermopsis raciborskii C10. Toxicon 44:483-489. CHA JH., STEWART GC. 1997. The divIVA minicell locus of Bacillus subtilis. J Bacteriol. 179(5):1671-83. 91 CHATER KF. 2001. Regulation of sporulation in Streptomyces coelicolor. Curr. Opin. Microbiol. 4:667-673. CILIA, M.L.; JACKSON, D. 2004. Plasmodesmata form and function. Curr. Opin. Cell Biol. 16:500–506. CIRÉS, S.; WÖRMER, L.; WIEDNER, C.; QUESADA, A. 2013. Temperature-dependent dispersal strategies of Aphanizomenon ovalisporum (Nostocales, Cyanobacteria): implications for the annual life cycle. Microb Ecol. 65(1):12-21. CRAIG, L.; PIQUE, M.E.; TAINER, J.A. 2004. Type IV pilus structure and bacterial pathogenicity. Nat Rev Microbiol 2:363–378. DEL SAL, G.; MANFIOLETTI, G.; SCHNEIDER, C. 1989. The CTAB-DNA precipitation method: a common mini-scale preparation of template DNA from phagemids, phages or plasmids suitable for sequencing. Biotechiques 7:514-520. DINH, T.; BERNHARDT, TG. 2011. Using superfolder green fluorescent protein for periplasmic protein localization studies. J Bacteriol. 193(18):4984-7. DOYLE, JJ.; DOYLE, JL. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem Bull 19:11-15. ELHAI, J.; WOLK, C.P. 1988a. A versatile class of positiveselection vectors based on the nonviability of palindromecontaining plasmids that allows cloning into long polylinkers. Gene 68:119–138. ELHAI, J.; VEPRITSKIY, A.; MURO-PASTOR, A.; FLORES, E.; WOLK, P. 1997. Reduction of Conjugal Transfer Efficiency by Three Restriction Activities of Anabaena sp. Strain PCC 7120. J. Bacteriol. 179(6):1998–2005. 92 ELHAI, J.; KATO, M.; COUSINS, S.; LINDBLAD, P.; COSTA, J. 2008. Very small mobile repeated elements in cyanobacterial genomes. Genome Research. 18:1484–1499. ERRINGTON, J.; DANIEL, RA.; SCHEFFERS, DJ. 2003. Cytokinesis in bacteria. Microbiol Mol Biol Rev. 67(1):52-65. FAY, P. 1992. Oxygen relations of nitrogen fixation in cyanobacteria. Microbiol Rev. 56:340-73. FILLOUX, A. 2004. The underlying mechanisms of type II protein secretion. Molecular Cell Research. 1694:163–179. FLORES E.; HERRERO A.; WOLK CP.; MALDENER I. 2006. Is the periplasm continuous in filamentous multicellular cyanobacteria? Trends Microbiol. 14:439-43. FLORES, E.; PERNIL, R.; MURO-PASTOR, A.; MARISCAL, V.; MALDENER, I.; LECHNO-YOSSEF, S.; FAN, Q.; WOLK, C.P.; HERRERO, A. 2007. Septumlocalized protein required for filament integrity and diazotrophy in the heterocyst-forming cyanobacterium Anabaena sp. strain PCC 7120. J. Bacteriol 3884–3890. FLORES, E.; HERRERO, A. 2010. Compartmentalized function through cell differentiation in filamentous cyanobacteria. Nat Rev Microbiol. 8:39-50. FRÍAS, JE.; FLORES, E.; HERRERO, A. 1994. Requirement of the regulatory protein NtcA for the expression of nitrogen assimilation and heterocyst development genes in the cyanobacterium Anabaena sp. PCC 7120. Mol Microbiol 14:823–832. GOEHRING NW, BECKWITH J. 2005. Diverse paths to midcell: assembly of the bacterial cell division machinery. Curr Biol 15(13):R514-26. 93 GREEN, K.; VIAMONTES, G.; KIRK, D. 1981. Mechanism of formation, ultrastructure, and function of the cytoplasmic bridge system during morphogenesis in Volvox. J Cell Biol. 91:756-69. HARRY, E.; MONAHAN, L.; THOMPSON, L. 2006. Bacterial cell division: the mechanism and its precison. Int Rev Cytol. 253:27-94. HERRERO, A.; MURO-PASTOR, AM.; VALLADARES, A; FLORES, E. 2004. Cellular differentiation and the NtcA transcription factor in filamentous cyanobacteria. FEMS Microbiol Rev. 28(4):469-87. HOLMES NA., WALSHAW J., LEGGETT RM., THIBESSARDA A., DALTONA KA, GILLESPIE MD, HEMMINGSA AM., GUST B., KELEMEN GH. 2013. Coiled-coil protein Scy is a key component of a multiprotein assembly controlling polarized growth in Streptomyces. PNAS. 397-406. KATIS, VL.; HARRY, EJ.; WAKE, RG. 1997. The Bacillus subtilis division protein DivIC is a highly abundant membrane-bound protein that localizes to the division site. Mol Microbiol. 26(5):1047-55. KAISER, D.; ROBINSON, M.; KROOS, L. 2010. Myxobacteria, polarity, and multicellular morphogenesis. Cold Spring Harb Perspect Biol. 2:000380. KUMAR, N.M.; GILULA, N.B. 1996. The gap junction communication channel, Cell 84:381– 388. LUCAS, W.; HAM BK, K. 2009. Plasmodesmata - bridging the gap between neighboring plant cells. Trends Cell Biol. 19:495-503. LUTKENHAUS J. 2012. The ParA/MinD family puts things in their place. Trends Microbiol. 20(9):411-8. 94 MARISCAL, V.; HERRERO, A.; FLORES, E. 2007. Continuous periplasm in a filamentous, heterocyst-forming cyanobacterium. Mol Microbiol 65:1139–1145. MARISCAL, V.; FLORES, E. 2010. Multicellularity in a heterocyst-forming cyanobacterium: pathways for intercellular communication. Adv Exp Med Biol. 675:123-35. MAZOUNI K., DOMAIN F., CASSIER-CHAUVAT C., CHAUVAT F. 2004. Molecular analysis of the key cytokinetic components of cyanobacteria: FtsZ, ZipN and MinCDE. Mol Microbiol. 52(4):1145-58. MERINO-PUERTO, V.; MULLINEAUX, CW.; MARISCAL, V.; HERRERO, A.; FLORES, E. 2010. Fra proteins influencing filament integrity, diazotrophy and localization of septal protein SepJ in the heterocyst-forming cyanobacterium Anabaena sp. Molecular Microbiology 75:1159–1170. MULLINEAUX, C.; MARISCAL, V.; NENNINGER, A.; KHANUM, H.; HERRERO, A.; FLORES, E.; ADAMS, D.G. 2008. Mechanism of intercellular molecular exchange in heterocyst-forming cyanobacteria. The EMBO Journal 27:1299–1308. MIYAGISHIMA S., WOLK CP., OSTERYOUNG KW. 2005. Identification of cyanobacterial cell division genes by comparative and mutational analyses. Molecular Microbiology 56(1):126–143. NAKANISHI H., SUZUKI K., KABEYA Y., OKAZAKI K., MIYAGISHIMA SY. 2009. Conservation and differences of the Min system in the chloroplast and bacterial division site placement. Commun Integr Biol. 2(5):400-2. NAYAR, A.; YAMAURA, H.; RAJAGOPALAN, R.; RISSER, D.; CALLAHAN, S. 2007. FraG is necessary for filament Integrity and heterocyst maturation in the cyanobacterium Anabaena sp. strain PCC 7120. Microbiology 153:601–607. 95 PAZ-YEPES, J.; HERRERO A. 2007. The NtcA-regulated amtB gene is necessary for full methylammonium uptake activity in the cyanobacterium Synechococcus elongates. J. Bacteriol. 189:7791–7798. PAZ-YEPES, J.; FLORES, E.; HERRERO A. 2009. Expression and mutational analysis of the glnB genomic region in the heterocyst-forming Cyanobacterium Anabaena sp. strain PCC 7120. J Bacteriol. 191(7):2353-2361. PFAFFL, M.W. 2001. A new mathematical model for relative quantification in real-time RTPCR. Nucleic acid research. 29(9):2003-2007. RACKHAM OJ., MADERA M., ARMSTRONG CT., VINCENT T L., WOOLFSON D.N., GOUGH, J. 2010. The evolution and structure prediction of coiled coils across all genomes. J Mol Biol. 4(3):480-93. RIPPKA, R.; DERUELLES, J.; WATERBURY, J.; HERDMAN, M.; STANIER, R. 1979. Generic Assignments, Strain Histories and Properties of Pure Cultures of Cyanobacteria. Journal of General Microbiology 111:1-61 RIS, H.; SINGH, RN. 1961. Electron microscope studies on bluegreen algae. J. Biophys. Biochem. Cytol. 9:63–80. ROBINSON, C.; BOLHUIS, A. 2004. Tat-dependent protein targeting in prokaryotes and chloroplasts. Biochim Bipphys Acta 1694:135–147. ROKAS, A. 2008. The origins of multicellularity and the early history of the genetic toolkit for animal development. Annu. Rev. Genet. 42:235–51. SAKR, S.; JEANJEAN, R.; ZHANG, C.; ARCONDEGUY,T. 2006. Inhibition of cell division suppresses heterocyst development in Anabaena sp. Strain PCC 7120. J. Bacteriol 188:1396-1404. 96 SCHMITT, R. 2003. Differentiation of germinal and somatic cells in Volvox carteri. Curr Opin Microbiol. 6:608-13. SCHWARTZMAN, D.; CALDEIRA, K.; PAVLOV, A. 2008. Cyanobacterial emergence at 2.8 gya and greenhouse feedbacks. Astrobiology 8:187-203. STUCKEN, K.; JOHN, U.; CEMBELLA, A.; MURILLO, A.; SOTO-LIEBE, K.; FUENTES, J.; FRIEDEL, M.; PLOMINSKY, AM.; VÁSQUEZ, M.; GLOCKNER, G. 2010. The smallest known genomes of multicellular and toxic cyanobacteria: comparison, minimal gene sets for linked traits and the evolutionary implications. PLoS one 5(2):e9235. SUTTLE, C. 2000. Cyanophages and their role in the ecology of cyanobacteria. In: The ecology of the cyanobacteria. B. A. Whitton and M. Potts (ed.). 20:564-583. VIOQUE, A. 1997. The RNase P RNA from cyanobacteria: short tandemly repeated repetitive (STRR) sequences are present within the RNase P RNA gene in heterocyst-forming cyanobacteria. Nucleic Acids Res. 25:3471-3477. WALDO GS., STANDISH BM., BERENDZEN J., TERWILLIGER TC. 1999. Rapid proteinfolding assay using green fluorescent protein. Nat. Biotechnol. 17:691–695. WILK, L.; STRAUSS, M.; RUDOLF, M.; NICOLAISEN, K.; FLORES, E.; KÜHLBRANDT, W.; SCHLEIFF, E. 2011. Outer membrane continuity and septosome formation between vegetative cells in the filaments of Anabaena sp. PCC 7120. Cell Microbiol. 13(11):1744-54. WOLK, CP. 1973. Physiology and cytological chemistry of blue-green algae. Bacteriol. Rev. 37:32–101. 97 ZHANG, W.; GAO, H.; DU, Y.; KHUDYAKOV, I.; NING, D.; FAN, Q.; WOLK, P.; XU, X. 2007. A gene cluster that regulates both heterocyst differentiation and pattern formation in Anabaena sp.strain PCC 7120. Molecular Microbiology 66:1429–1443. ZHANG, L.; CHEN, Y.; CHEN, W. 2008. Existence of periplasmic barriers preventing green fluorescent protein diffusion from cell to cell in the cyanobacterium Anabaena sp. strain PCC 7120. Mol Microbiol 70:814–823. 98 ANEXOS Anexo 1. Medio BG11o: MgSO4∙7H2O 7,5 g/L, CaCl2∙2H2O 3,6 g/L, Ácido cítrico (C6H8O7) 0,6 g/L, citrato férrico amónico 0,6 g/L, Na2EDTA 0,093 g/L, Na2CO3 2 g/L, H3BO3 0,286 g/L, MnCl2∙4H2O 0,181 g/L, ZnSO4∙7H2O 0,022 g/L, NaMoO4∙2H2O 0,039 g/L, CuSO4∙5H2O 0,0079 g/L, CoCl2∙6H2O 0,0049 g/L Anexo 2. Medio MLAo: MgSO4∙7H2O 49,4 mg/L, NaCl 117 mg/L, K2HPO4 34,8 mg/L, H3BO3 2,47 mg/L, vitamina B12 50 ng/L, tiamina HCl 100 ng/L, biotina 50 ng/L, Na2EDTA 4,36 mg/L, FeCl3∙6H2O 1,58 mg/L, CuSO4∙5H2O 100 ng/L, ZnSO4∙7H2O 22 μg/L, CoCl2∙6H2O 100 ng/L, NaMoO4∙2H2O 6 ng/L, NaHCO3 169 mg/L, CaCl2∙2H2O 29,4 mg/L. Anexo 3. TAE 1X: 40 mM Tris, 20 mM ácido acético, 1 mM EDTA. Anexo 4. Medio Luria-Bertani (LB): 2 % (p/v) triptona, 1 % (p/v) extracto de levadura y 1 % (p/v) NaCl. 99