Acerca del sistema CRISPR Cas9 y su aplicación para editar genes
Anuncio

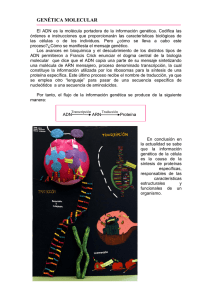
Acerca del sistema CRISPR Cas9 y su aplicación para editar genes Por Nicolás Jouve, Dr. en Biología, Catedrático Emérito de Genética. Presidente de CiViCa. Temas de formación sobre Bioética desarrollados en pocas palabras. Publicado en CiViCa el 3 de Febrero de 2016. Se adjunta en PDF. Antes de explicar en qué consiste esta nueva tecnología y sus potenciales aplicaciones conviene enmarcar su campo de actuación. En una primera aproximación diríamos que se trata de una metodología destinada a modificar el ADN. De este modo, se vincula a la “ingeniería genética”, una tecnología que se empezó a desarrollar en los años setenta del siglo pasado, tras los descubrimientos sucesivos de la estructura del ADN (1953), la expresión de los genes (1961), el conocimiento del código genético universal (1961-64) y el aislamiento del primer gen (1969). Esta serie de descubrimientos abrieron las puertas a la manipulación del ADN. En 1980, Paul Berg, Frederik Sanger y Walter Gilbert tras establecer las bases de la Ingeniería Genética recibieron el Premio Nobel de Química. La idea de modificar genes tiene que ver naturalmente con la posibilidad de cambiar las características genéticas de sus portadores y ahí, en la vertiente aplicada es donde se ha abierto un enorme abanico de beneficios, dependiendo de las especies en los que se realice con fines de mejoramiento agrícola o ganadero, industriales o clínicos. El sistema CRISPR Cas9, como veremos a continuación es un nuevo método que se añade a otras tecnologías para la obtención de “organismos modificados genéticamente” o “transgénicos”. El sistema CRISPR El sistema CRISPR forma parte de un mecanismo natural de inmunidad que actúa en las bacterias y Archaeas, para degradar y eliminar secuencias de ADN invasoras (virus, plásmidos…). Se trata de un sistema inmune adaptable que es utilizado por numerosas bacterias para defenderse degradando cualquier tipo de ADN extraño que se introduzca en su interior procedente del medio en el que viven. Esto es una necesidad pues en el medio natural de las bacterias hay restos de ADN que podrían introducirse en su interior y transformarlas o degradar su propio genoma, o como ocurre con los llamados bacteriófagos, apoderarse del aparato bioquímico de las bacterias para su propia reproducción. Para que esto no ocurra, las bacterias han desarrollado un 1 sistema de defensa que es el CRISPR Cas9 (Fig.1). La denominación CRISPR obedece a unas regiones del ADN del genoma de las bacterias consistentes en agrupaciones cortas de bases nucleotídicas repetidas palindrómicas regularmente interespaciadas. Es decir, pequeñas secuencias de ADN repetidas y “capicúas”, con unos espacios intercalares. Lo interesante es que estos espacios intercalares son ocupados por fragmentos del ADN extraño que, tras una invasión al interior de la bacteria, se troceó y se unió al ADN entre las secuencias CRSPR de la bacteria. Dicho de otro modo, la primera vez que un ADN extraño invade a una bacteria se trocea y los pequeños trocitos de ADN del agente invasor se integran en el genoma de la bacteria, justo entre las secuencias repetidas del sistema CRISPR. Estas pequeñas secuencias de ADN invasor se denominan "protoespaciadores" y serán utilizadas para defenderse de una ulterior entrada de ADN del mismo agente invasor. La denominación Cas 9 se refiere a una enzima, una nucleasa, es decir una proteína que puede cortar el ADN, que es la que se encarga de romper y degradar en ADN extraño. Para ejercer su tarea, esta enzima se asocia a una cadena de ARN en el momento en que se produce la nueva invasión de ADN extraño. La molécula de ARN a la que se asocia Cas9 es precisamente una molécula que se sintetiza utilizando los protoespaciadores intercalados entre las secuencias de la región CRISPR. De este modo, cuando se produce el segundo ataque o posteriores de ADN extraño, la bacteria expresa la región CRISPR dando lugar a la molécula de ARN que contiene las secuencias previamente integradas (protoespaciadores). Esta molécula será procesada para dar lugar a varios ARN más pequeños denominados crARN (CRISPR ARN) cada uno de los cuales llevará un protoespaciador distinto y una parte de las secuencias repetidas. Lo que va a ocurrir a continuación, es que cada crARN se unirá a otro ARN del sistema denominado transactivador y juntos forman un complejo con la nucleasa Cas9. El protoespaciador presente en cada complejo dirige al conjunto hacia las secuencias del ADN invasor, reconoce las bases homólogas presentes en él, hibrida con ellas y promueve su degradación por medio de la enzima Cas9 (Fig.2). Una condición más es que, para que se produzca el reconocimiento e hibridación del ARN unido al complejo con el ADN invasor, es necesario que la secuencia de éste se encuentre situada al lado de una secuencia corta denominada motivo adyacente al protoespaciador (PAM). Esto es fácil que ocurra pues el motivo PAM consiste en la secuencia de tres bases nucleotídicas, que habitualmente son NGG (donde N puede 2 ser cualquier nucleótido), lo que prácticamente permitiría encontrar dianas en todos los genes de un organismo. Aplicación para editar genes de otras especies Este sistema, siendo natural en las bacterias, ha sido adaptado para inducir roturas de ADN en sitios específicos con fines de ingeniería genética en otras especies. Es decir, se trataría de que en las condiciones que desee el investigador se produzca la invasión de la región del genoma o el gen que se desea "editar", con una secuencia de ADN homóloga, para que tras localizarlo sen el genoma se produzca el corte, para después restarurarla (“editar”). Naturalmente para hacerlo hay que conocer previamente las secuencias de los genes del organismo en el que se desea aplicar. Esto sería potencialmente posible en el genoma humano, por ejemplo, cuyo genoma completo se conoce desde 2003. Para utilizarlo con fines de editar un gen, se introducen en la célula u organismo cuyo ADN se quiere modificar únicamente dos de los componentes del sistema inmune para que se expresen, el gen de la nucleasa Cas9 y un ARN guía. Este consiste en un ARN portador del equivalente al protoespaciador, que sería una secuencia corta de unas 20 bases nucleotídicas, que se podrían sintetizar in vitro y que actuaría para reconocer la región homóloga del ADN diana. Este protoespaciador estaría situado en un extremo del ARN guía, que va a dirigir al complejo hacia la secuencia diana siguiendo las reglas de apareamiento estándar entre ARN y ADN. Finalmente, para que el sistema funcione, la secuencia diana debe de encontrarse justo en el extremo 5’ del motivo PAM (NGG). Por ello, con este sistema se pueden producir roturas en cualquier secuencia del genoma que presente la forma N 20-NGG simplemente cambiando los primeros 20 nucleótidos del ARN guía para que se correspondan con la secuencia diana. Debido a la facilidad de este sistema para inducir cortes en el genoma su uso se ha extendido rápidamente apareciendo en un corto espacio de tiempo un gran número de publicaciones que muestran la utilidad de la técnica para “editar”, es decir “cortar” y “restaurar” un gen específico en cualquier tipo de organismos. Naturalmente, siempre que se conozca bien la secuencia del gen a editar. 3