Editorial - Portal de Revistas Electrónicas
Anuncio

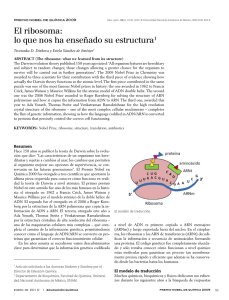
Vol. 10 Nº 2, Julio-Diciembre 2009 Revista Arbitrada e Indizada La fábrica celular de proteínas: Nuevas fronteras de la química biológica En el bienio 1.999-2.000 salió a la luz una serie de artículos revelando la estructura del ribosoma bacteriano con resolución atómica. Esta organela celular constituye tanto la base estructural como la fuente de los grupos químicos responsables de la catálisis de uno de los procesos más importantes para la vida tal como la conocemos: la síntesis de proteínas. Tres líderes de los grupos involucrados en este logro (considerado muy poco tiempo atrás como un virtual imposible): la Dra. Ada E. Yonath (Instituto Weizmann, Rehovot, Israel), el Dr. Thomas A. Steitz (Universidad de Yale, New Haven, USA) y Venkatraman Ramakrishnan (MRC Laboratory of Molecular Biology, Cambridge, Inglaterra), fueron reconocidos con el premio Nobel de Química 2009. La visión a alta resolución de la preservación, replicación y expresión de la información genética, mediante técnicas cristalográficas de rayos X, ha sido, con esta versión del premio, reconocida en tres ocasiones. La primera fue en 1962 cuando James Watson, Francis Crick y Maurice Wilkins fueron premiados por la elaboración del modelo atómico de la molécula de ADN de doble cadena, piedra fundacional de la Biología Molecular. La segunda, corresponde a las estructuras que muestran como la información contenida en el ADN es copiada en la molécula de ARN mensajero (Roger D. Kornberg en el 2006), primer paso en el mecanismo de expresión genética. El ribosoma bacteriano es un complejo macromolecular de ribonucleoproteína, con un coeficiente de sedimentación de 70S. Se ensambla por la unión de una subunidad mayor (50S, compuesta por dos moléculas de ARNr, 23S y 5S, y 34 proteínas) y una subunidad menor (30S, con una molécula de ARNr 16S y 21 proteínas), es con mucho, la estructura biológica más complicada resuelta hasta el presente. No resulta por tanto extraño, que una gran cantidad de trabajo previo debió ser acumulada para poder llegar a la feliz culminación, justo en el cambio de siglo. Toda la comunidad científica reconoce como pionera del campo a la Dra. Yonath. A finales de la década de 1.970, esta entonces joven cristalógrafa, fue contratada por el Prof. Heinz-Günther Wittmann, director del Instituto Max Planck para Genética Molecular en Berlín (Alemania Occidental), para explorar la posibilidad de cristalizar ribosomas bacterianos e iniciar estudios estructurales mediante cristalografía de rayos X. Como suele ocurrir en el trabajo científico, hubo un sorprendente éxito inicial y en poco tiempo se logró cristalizar la subunidad 50S de Bacillus stearothermophilus (publicado en 1980). Sin embargo, después del rápido avance inicial surgieron las dificultades propias del complejo trabajo acometido, y no fue sino hasta entrada la década de 1990 que se hizo posible la obtención consistente de cristales estables con buena difracción. Además, hubo que desarrollar métodos nuevos y seguros para la colección y procesamiento de la enorme cantidad de datos de los complicados patrones de difracción. Esta experiencia acumulada durante más de una década fue aprovechada, sin menoscabo de sus méritos propios, por los demás grupos trabajando en el tópico. Mientras el grupo de Yonath trabajaba arduamente para resolver el problema de fase en sus patrones de difracción usando diferentes enfoques, en el grupo de Steitz se lograron avances significativos usando información adicional obtenida mediante criomicroscopía electrónica por el grupo de Joachim 5 Vol. 10 Nº 2, Julio-Diciembre 2009 Frank en New York. El Dr. Ramakrishnan, por su parte, había acumulado una gran cantidad de información detallada de estructura de proteínas ribosomales aisladas y en complejo con secciones del ARN ribosomal donde estas se unen en la estructura de la partícula completa. Durante la década final todos los grupos incorporaron la potencia del modelaje computacional para organizar la información acumulada. En cierto modo se puede decir que la historia de la doble hélice del ADN fue repetida aunque con técnicas más avanzadas. Tal como Watson y Crick aprovecharon la información cristalográfica de Rosalind Franklin y Maurice Wilkins para construir un modelo del ADN, la combinación de métodos cristalográficos y la construcción de modelos computacionales compatibles con los resultados experimentales, permitió dar solución a la estructura del ribosoma. Como prueba de la confiabilidad de los resultados está el hecho de que los modelos publicados por los diferentes grupos, trabajando en completa independencia y confidencialidad, son de una compatibilidad casi absoluta (hay pequeñas diferencias solo en regiones particularmente móviles de la estructura). El mayor valor de la estructura no está precisamente en sí misma, sino en que esta da la base para interpretar la función de los ribosomas. Ahora es posible entender mejor las interacciones de las partículas ribosomales con los demás componentes involucrados en la biosíntesis de proteínas y entender el mecanismo molecular exacto del proceso. Un aspecto muy importante en los análisis funcionales que se hacen posibles es la comprensión molecular del mecanismo de acción de los antibióticos que afectan la función del ribosoma. La síntesis de proteínas es el blanco de acción de casi la mitad de los antibióticos naturales. Este hecho no debe ser sorprendente pues un antibiótico debe atacar procesos celulares fundamentales para ser efectivo como tal. El estudio de sitios y modos de unión de diferentes antibióticos que actúan en el ribosoma es ahora posible, y ha sido tema de trabajo de muchos grupos de investigación en los últimos 10 años. Sin embargo, la posibilidad más revolucionaria es que ahora es posible construir nuevos antibióticos a la medida. Conociendo la estructura y localización de los sitios funcionales del ribosoma se pueden diseñar moléculas que interfieran con la función, es decir antibióticos sintéticos para los cuales el fenómeno de resistencia no sea un problema en el uso terapéutico. Más aún, teniendo información de secuencia del ARN ribosomal y proteínas ribosomales de una bacteria particular, es posible el diseño de antibióticos sintéticos específicos dirigidos a patógenos difíciles de controlar con los medios convencionales. Estas perspectivas no han pasado desapercibidas para la industria farmacéutica que ha dedicado una considerable cantidad de recursos y personal a proyectos de investigación y desarrollo en el campo. Es previsible que durante la próxima década veamos nuevas generaciones de antibióticos sintéticos de gran potencia y efectividad. Dr. Francisco J. Triana-Alonso Director del Instituto de Investigaciones Biomédicas de la Universidad de Carabobo (BIOMED-UC, FCS - Sede Aragua) ftrianaalonso@yahoo.com, biomed@uc.edu.ve 6