Tema 5.6. Biosíntesis de proteínas
Anuncio

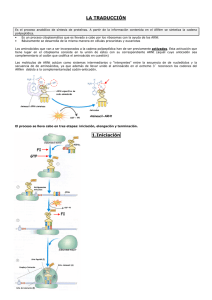
El flujo de la información genética: TRADUCCIÓN SÍNTESIS DE PROTEÍNAS • Dirigida por un molde de mRNA • Tres fases: iniciación, elongación de la cadena y terminación • Fase preliminar: formación de un precursor activado Aminoacil tRNA • Maquinaria de síntesis compleja: ribosomas y factores secundarios RIBOSOMAS RNA ribosomal Proteínas Estructura secundaria de un ARN ribosómico de 16S Estructura secundaria de RNAs ribosómicos de 23 y 5S Estructura tridimensional de un ARN ribosómico de 16S Composición del ribosoma procariótico L1, L2…L31 2904 nt 120 nt S1, S2…S21 1542 nt Importancia del RNA ribosomal • Se pueden omitir algunas proteínas durante la reconstitución in vitro de la subunidad 30S, lo que provoca disminución de la actividad pero no pérdida total de funcionalidad • Una secuencia del rRNA de 16S selecciona el lugar de iniciación en el mRNA • Una base del tRNA está apareada con una base del rRNA de 16S en una secuencia de 14 nucleótidos idéntica en todos los rRNAs secuenciados (mas de 1000) • El extremo aceptor 3’ del tRNA interacciona con una región conservada del rRNA de 23S • Ribosomas casi desprovistos de proteínas aun pueden catalizar la formación de enlaces peptídicos • La mayoría de antibióticos que interfieren en la síntesis de proteínas lo hacen por interacción específica con el rRNA y no con las proteínas Estructura del ribosoma de E. Coli La síntesis de proteínas transcurre desde el extremo amino al carboxilo H2N-CH-COOH + H2N-CH-COOH | | R1 R2 H2N-CH-CO-HN-CH-COOH | | R1 R2 H2N-aa1-aa2-aa3……….aan-COOH Proteína ya sintetizada antes de añadir el aa radiactivo Proteína sintetizada después de añadir el aa radiactivo El mRNA se traduce en la dirección 5’ a 3’ Formación del complejo de iniciación de la síntesis de proteínas procariótica Factor ¿Une GTP? Función IF1 No Promueve la disociación del ribosoma de 70S IF2 Si Ayuda a la unión del tRNA iniciador IF3 No Similar a IF1 Formación del complejo de iniciación de la síntesis de proteínas procariótica Secuencia de Shine-Dalgarno 5’ ...AGGAGGU...mRNA 3’... UCCUCCA... rRNA 16S Interacción entre el mRNA y el rRNA Estructura de la formil-metionina Síntesis del metionil-tRNAf Formilación del metionil-tRNAf El complejo de iniciación en procarioticos (Resumen) Comienzo de un ciclo de elongación Factores de elongación: EF-Tu EF-Tu transporta los aminoacil-tRNAs al sitio A del ribosoma EF-Tu protege de la hidrólisis al aminoacil-tRNA Sólo hidroliza su GTP si el apareamiento codonanticodon es correcto: fidelidad en la síntesis No reconoce fMet-tRNAf pero sí todos los demás Factores de elongación: EF-Ts EF-Ts participa en el reciclado de EF-Tu, cargándolo nuevamente con GTP Elongación de la cadena proteica: formación del enlace peptídico Centro peptidiltransferasa Proteínas L2 L3 L4 L15 L16 rRNA de 23S Elongación de la cadena proteica: translocación EF-G participa en la translocación Mimetismo Molecular Resumen de la elongación Sitios importantes en el ribosoma Terminación de la síntesis de proteínas procariótica Acoplamiento transcripción-traducción en procariotas Elementos de iniciación en el mRNA eucariótico A CCAUGG (Secuencia consenso de Kozac) G Formación del complejo de iniciación en eucarióticos Formación del complejo de iniciación en eucarióticos Formación del complejo de iniciación en eucarióticos Síndrome X frágil (trastorno mental hereditario) Cáncer de próstata y otros tumores Factores de iniciación eucarióticos Elongación y terminación en eucarióticos Nombre Equival. Procar. EF1α EF1β EF2 eRF1 Función EF-Tu EF-Ts EF-G Con GTP unido coloca los aa-tRNAs en el sitioA Recicla EF1a sustituyendo GDP por GTP Translocación RF1 RF2 Reconoce codones de terminación Antibióticos inhibidores de la síntesis de proteínas Estructuras de algunos antibióticos inhibidores de la síntesis de proteínas Tetraciclina: Inhibe la unión de los aminoacil tRNAs al ribosoma bloqueando así la traducción Estreptomicina: Interfiere con el apareamiento normal de los aminoacil tRNAs con los codones del mRNA, causando errores en la lectura y, por tanto proteínas aberrantes Eritromicina: Se une a un sitio específico del rRNA de 23S y bloquea la elongación interfiriendo en la fase de translocación Estructuras de algunos antibióticos inhibidores de la síntesis de proteínas Cloranfenicol: Bloquea la elongación, actuando como un inhibidor competitivo de la actividad peptidiltransferasa. El enlace amida (en azul) imita un enlace peptídico Semejanza entre la puromicina y un aminoacil-tRNA Puromicina: Causa terminación prematura de la síntesis, ya que se parece al extremo 3’ de los aminoacil-tRNAs. Por tanto, entrará en el sitio A y se le transferirá la cadena polipeptídica en crecimiento, pero su débil unión al ribosoma causará su liberación prematura Toxina diftérica Corynebacterium diphtheriae Diftamida eEF2 EF-G Translocación Glicosilación de proteínas El donante de azúcares a las glicoproteínas: dolicol fosfato Adición progresiva de azúcares al dolicol fosfato Tranferencia del oligosacárido desde el dolicolfosfato a la cadena lateral de asparagina Asn-X-Ser Asn-S-Thr Oligosacárido rico en manosas Oligosacárido complejo Glicoproteínas de la superficie celular como receptores víricos Procesamiento cotransduccional en el retículo endoplásmico SRP=1 RNA (300 nt) + 6 proteínas Péptidos señal de varias proteínas Acción de la partícula de reconocimiento de la señal Inserción de proteínas de membrana mediante secuencias de transferencia de bloqueo Señales de proteínas destino de algunas De retención en RE: Lis-Asp-GluLeu-COOH (de devolución de Golgi a RE) De tránsito a los cloroplastos: similares a las de las Mitocondrias. Varios compartimentos: varias señales Peroxisomas: Ser-Lis-Fen-COOH Nucleares: Lis-Lis-Lis-Arg-Lis (hay otras) Señales y translocación en las mitocondrias Chaperonas Familia Hsp60 GroEL (en el citosol bacteriano) Hsp60 (en la matriz mitocondrial) Proteína que se une a Rubisco (en cloroplastos) Proteínas Hsp70 Hsp70 (en el citosol de mamíferos) BiP (en el RE de eucariotas) Grp75 (en mitocondrias) DnaK (en el citosol bacteriano) Proteínas Hsp90 Hsp83 (en el citosol de eucariotas) Grp94 (en el RE de mamíferos) HtpG (en el citosol bacteriano) Otras Proteína isomerasa de puentes disulfuro Peptidil-prolil-isomerasa (PPI) Ciclo de acción de la chaperona BiP Complejo GroEL-GroES Ciclo de funcionamiento del complejo GroEL-GroES