area01-res138
Anuncio

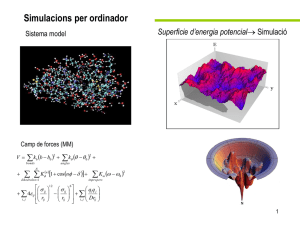
XIX Verano de la Investigación Científica y Tecnológica del Pacífico ESTRUCTURA Y ENERGÉTICA DE AGREGADOS MOLECULARES Y NANOESTRUCTURAS Eduardo Bernales Soto, Centro Universitario de Tonalá, Universidad de Guadalajara. laalo_01@hotmail.com . Asesora Aristea Prosmiti, Consejo Superior de Investigaciones Científicas, rita@iff.csic.es . PLANTEAMIENTO DEL PROBLEMA Recientemente clatratos sH han sido sintetizados con especies de gases raros. La estructura H de los hidratos clatratos de argón y metano han sido sintetizados bajo grandes presiones debido a que experimentos de difracción de neutrones, muestran que cinco átomos de argón logran un arreglo trigonal bipiramidal en el largo de las cajas, con dos átomos de argón localizados cerca de los anillos hexagonales polares de el largo de la caja y los otros tres localizados en alternativos lugares adyacentes a el hexágono ecuatorial.[1] De tal manera, se busca determinar clatratos de gases raros con diferentes ocupaciones en el largo de las cajas basándose en estudios teóricos. METODOLOGÍA Después de hacer una revisión bibliográfica en el marco de metodología, teoría y programación, se usa dinámica molecular, que es una técnica de simulación por computadora donde la evolución del tiempo de un grupo de partículas es seguido por la integración de su ecuación de movimiento.[2] El movimiento físico de los átomos es dado por numerosas soluciones con la ecuación de movimiento de Newton usando una descripción de la interacción interatómica. Dentro de varios códigos enfocados en simulación de dinámica molecular, se ha considerado el programa Moldy. [3] CONCLUSIONES Con los conocimientos, destrezas y habilidades adquiridas en este periodo de formación, debido a la revisión de literatura científica, presentación de resultados teóricos y algunas herramientas de programación, se tuvo una idea general de cómo abordar el problema planteado. Por otro lado, se da la introducción a cómo se maneja Moldy y la gran importancia que tiene el programa. En el cual se encuentran las herramientas necesarias para hacer estudios de dinámica molecular y al mismo tiempo generar datos que nos digan el comportamiento de dichas nanoestructuras. Cabe mencionar, que durante mi estancia he tenido la oportunidad de participar en un congreso científico internacional.[4] BIBLIOGRAFÍA [1] J.S. Loveday et al. PCCP,10, 937 (2008); V.I. Artyukhov et al., JCP,141,034503 (2014) [2] Göran Wahnström, Molecular Dynamics, (2013) [3] K. Refson, CPC, 126, 310 (2000) [4] IMAMPC 2014, 8-11 July (Salamanca) http://imampc2014.usal.es © Programa Interinstitucional para el Fortalecimiento de la Investigación y el Posgrado del Pacífico Agosto 2014