Un aminoácido, es una molécula orgánica con un grupo amino... ) y un grupo carboxilo (- alfa-aminoácidos AMINOÁCIDOS.
Anuncio

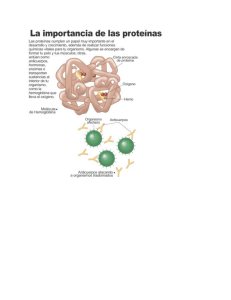
AMINOÁCIDOS. Un aminoácido, es una molécula orgánica con un grupo amino (NH2) y un grupo carboxilo (COOH). Todos los aminoácidos componentes de las proteínas son alfa-aminoácidos, lo que indica que el grupo amino está unido al carbono alfa (carbono adyacente al grupo carboxilo). Por lo tanto, están formados por un carbono alfa unido a un grupo carboxilo, a un grupo amino, a un hidrógeno y a una cadena (habitualmente denominada R) de estructura variable, que determina la identidad y las propiedades de los diferentes aminoácidos; existen cientos de cadenas R por lo que se conocen cientos de aminoácidos diferentes, pero sólo entre 21 forman parte de las proteínas y tienen codones específicos en el código genético. Hace pocos años, el AA 21 fue descubierto y se lo denomino selenocisteina. Es similar a la cisteína pero presenta un Se en vez de un S. La unión de varios aminoácidos da lugar a cadenas llamadas polipéptidos o simplemente péptidos, que se denominan proteínas cuando la cadena polipeptídica supera los 10.000 Dalton. Otra propiedad interesante de estos compuestos, es que pueden modificarse después de la síntesis de proteínas, para mejorar sus posibilidades produciendo AA no comunes como las fosfotreonina, fosfoserina, etc. Clasificación. Se han propuesto varios métodos para clasificar los aminoácidos sobre la base de sus grupos R. El más significativo se funda en la polaridad de los grupos R. Existen cuatro clases principales: 1. Aminoácidos con grupos R no polares o hidrofóbicos. Alanina, leucina, isoleucina, valina, prolina, fenilalanina, triptófano y metionina. Estos aminoácidos son menos solubles en el agua que los aminoácidos con grupos R polares. No presentan oxigeno ni nitrógeno en su cadena R. Estas pueden ser alifáticas (secuencias hidrocarbonadas), aromáticas (con estructuras cíclicas) y/o sulfurados. 2. Aminoácidos con grupos R polares sin carga. Estos aminoácidos son relativamente más solubles en el agua que los anteriores. Sus grupos R contienen grupos funcionales polares (cetonas o alcoholes) que pueden establecer enlaces de hidrógeno con el agua. La polaridad de la serina, la treonina y la tirosina se debe a sus grupos hidroxilos; la de la aspargina y la glutamina, a sus grupos amídicos (𝐻2 𝑁 − 𝐶 = 𝑂) y de la cistina a la presencia del grupo sulfhidrilo (-SH). 3. Aminoácidos básicos o cargados positivamente. Los aminoácidos en los que los grupos R poseen carga positiva neta a PH 7, por lo que sus PI son mayores a 7. Es el caso de la lisina, la arginina y la histidina. El hecho de ser básicos, corresponde a la presencia de un grupo amino en la cadena R, por lo que se conocen como ácidos diamino monocarboxilados. 4. Aminoácidos ácidos o cargados negativamente. Los dos miembros de esta clase son los ácidos aspártico y glutámico, cada uno de los cuales posee un segundo grupo carboxilo que se halla completamente ionizado y por tanto cargado negativamente a pH 6 y 7. Se denominan asimismo, ácidos monoamino dicarboxilo. Estereoquímica. Los AA presentan actividad óptica que se manifiesta por la capacidad de desviar el plano de luz polarizada que atraviesa una disolución de estos y es debida a la asimetría del carbono , ya que se halla unido (excepto en la glicina) a cuatro radicales diferentes. Esta propiedad 1 hace clasificar a los aminoácidos en Dextrógiros (+) si desvían el plano de luz polarizada hacia la derecha, y Levógiros (-) si lo desvían hacia la izquierda. Los enantiomeros en forma L se encuentran en las proteínas exclusivamente donde tienen el grupo amino a la izquierda. Propiedades acido-base. Los AA no siempre se comportan como compuestos orgánicos, por ejemplo tienen puntos de fusión superiores a 200C mientras que los compuestos orgánicos de peso molecular similar son líquidos a temperatura ambiente. Son solubles en solventes polares como el agua pero no en solventes no polares en general. Esto se debe a que un AA tiene un NH2 básico y un COOH acido en la misma molécula, esto origina una reacción interna acido-base para dar un ion dipolar llamado Zwitterión (hibrido). Por esto los AA a veces se comportan como una sal. + Un AA en una misma molécula con tienen el anión carboxilato (CO− 2 ) y el catión amonio (NH3 ), esto lo define como anfótero, sustancia que puede reaccionar con ácidos o bases dependiendo del pH del medio, originando un catión o anión respectivamente. Las soluciones acuosas de los AA neutros (en su PI) son ligeramente acidas debido a que porque el COO− es mas fuerte que el NH3+ , por lo que se disocia con mayor facilidad en medio acido y su titulacion termina antes de llegar a pH=7. El PH al cual un AA no lleva carga iónica neta se define como punto isoeléctrico (PI). Estos se determinan por electroforesis, se pone una disolución acuosa entre dos electrodos donde los aniones emigran al electrodo positivo y viceversa. En el PI un AA no migra por tener carga neutra. Para un AA neutro el PI es entre PH=5.5 y 6. Para un AA acido el Pi es alrededor de PH=3. Esto se debe a que como el COOH es un acido relativamente fuerte dentro de los débiles, con baja pK, ambos grupos se ionizan a niveles bajos de pH. Titulación de un AA hidrofóbicos. La titulación acido-base, implica la adición o eliminación gradual de protones. En este caso, se comienza con el AA totalmente protonado, diprótico por su acido carboxílico y su catión amonio. A medida que vamos agregando base, los H del acido carboxílico débil se combinan con el OH y forman agua. A medida que se sigue agregando base, los H disminuyen en cantidad, por lo que la reacción se desplaza hacia la derecha, el aminoácido se sigue disociando para cumplir con su constante de equilibrio. Se llega a un punto, donde la concentración del AA totalmente protonado y la concentración del AA conjugado, son las mismas. Este valor, se corresponde con el pK del acido carboxílico. Una unidad a ambos márgenes de este punto, corresponde a la zona tamponante. El hecho de que el COOH ceda primero su protón que el H3N, se debe a que el COOH es un acido mas fuerte que el segundo, por lo que se disocia mas fácilmente. Siguiendo agregando base, el AA protonado se va a disociar completamente hasta llegar a un punto donde solo existe el AA neutro, ion dipolar o Zwitterión en la solución. El punto donde el 100% de los AA en solución se encuentran como iones dipolares, se conoce como punto isoeléctrico. A partir de este momento, con agregados de base, se va a empezar a disociar el protón del catión amonio para satisfacer el equilibrio de la reacción y los OH de la base van a formar agua con este. Así, llegamos a un punto donde la mitad de los AA tienen disociados sus cationes amonios, por lo 2 que la concentración del AA 0 y la del AA -1 es igual. Este punto, es el pK del NH3, por lo que una unidad a ambos extremos de la pendiente corresponde a la zona tamponante de este AA. Siguiendo agregando base, se van a disociar el 100% de los AA y van a quedar con carga neta -1. Con la curva de titulación de un AA, podemos ver que excepto la histidina, ningún AA poseen un buen poder tamponante a pH fisiológico. Además, sirve para predecir la carga neta del AA a un pH determinado. Tanto los pK como los PI, constituyen puntos de inflexión en la grafica, dado que en ellos cambia la concavidad de la pendiente. Asimismo, dado que en el PI los AA se presentan con carga neta 0, se puede deducir a valores menores de pH el AA tendrá carga neta positiva (migra al cátodo o electrodo negativo) y viceversa (migrando hacia el ánodo o cátodo positivo). En los puntos de las pK, la carga neta es una fracción de ½ o -1/2, tal como vemos en la grafica. En este tipo de AA, el PI se calcula como el promedio de ambas pK, por lo que matemáticamente: PI = Pk1 +Pk2 2 3 Titulación de un AA polar con carga. Este tipo de AA son tripróticos, por lo que sus curvas de titulación presentan tres regiones correspondiente a la ionización de cada grupo. Los valores de pK de estos AA reflejan la naturaleza de su cadena R. Los PI de los AA ácidos son justamente, ácidos, dado que los dos COOH que presentan se disocian fácilmente. Mientras que los PI de los AA básicos son bases, dado que los grupos aminos son débiles y se disocian a pH más elevados. Como mencionábamos, la histidina es la única que tiene un poder tamponante a pH fisiológico, por lo que se la suele encontrar en fluidos intra y extracelulares. Generalmente, se ioniza primero el COOH, luego el grupo de la cadena R y por último el amonio. Las excepciones a esto sucede en: tirosina, donde el grupo OH se disocia por ultimo Lisina, el grupo amino de la cadena R se disocia por ultimo Arginina, sucede lo mismo que en la lisina. El cálculo del PI para estos AA se calcula como el promedio entre los pK más cercano. En los AA ácidos: PI = Pk1 +Pkr En los AA básicos: PI = 2 Pkr +Pk2 2 En definitiva; Si el PI es acido, el AA es acido Si el PI es neutro, el AA es hidrofóbico apolar o polar sin carga. Si el PI es básico, el AA es básico. Curva de titulación de un AA acido. 4 Curva de titulación de un AA básico. Estructuras Superiores de las proteínas. Antes de comenzar a hablar de las estructuras, es necesario precisar una serie de conceptos útiles para la descripción de las mismas. Se le llama conformación a la disposición espacial que ocupan los átomos en una proteína. Las posibles conformaciones se dan siempre y cuando no se rompan enlaces covalentes, sino seria una desnaturalización. Una proteína tiene muchas conformaciones a las que llega mediante la rotación de sus átomos, pero algunas más estables termodinámica y funcionalmente y se conoce como conformación nativa, la conformación en la cual una proteína desempeña su función. Cuando ésta rompe, se conoce como desnaturalización. La función de una proteína depende de su forma tridimensional, lo que le da su especificidad. Pero a su vez, su forma tridimensional depende de la estructura primaria. También, se define como estabilidad a la tendencia de mantener la conformación nativa, por medio de diferentes interacciones que se las puede clasificar en dos grandes categorías como explica el cuadrito: 5 COVALENTES CARACTERISTICAS Enlace disulfuro Se da entre dos grupos sulfhidrilos (-SH) presentes en las cisteínas. Responden a una reacción Redox Amida El enlace peptidíco general NO COVALENTE Efecto hidrofóbico Son efectos que estabiliza a moléculas hidrofóbicas cercanas en el interior de una proteína. Interacciones iónicas o salinas Se dan por atracción electrostática entre grupos R polares. Dependen de la distancia entre los grupos y de la cte. dieléctrica del medio. Puentes de hidrogeno Se dan generalmente entre el H unido al grupo amino y el O carboxílico. Dos átomos se unen compartiendo un mismo hidrogeno. Fuerzas de Van Der Waals Interacciones débiles que se dan por inducción polar entre moléculas. Las interacciones débiles son las que predominan como las fuerzas estabilizadoras de una proteína, pese a que son unas 10 o 15 veces menos fuertes que las covalentes, pero como dice en el cuadro, al ser muchas generan estabilidad y se ubican en el núcleo de las proteínas. También es importante que los grupos R cargados tengan grupos adecuados a su alrededor, para estableces uniones salínicas o puentes de hidrogeno. Estructura primaria: Al hablar de la estructura primaria, debemos entender que se nos habla de la cadena de aminoácidos extendida, dejando de lado la forma que puede adoptar en las estructuras superiores. La cadena de aminoácidos, determina la forma en la que se va a plegar, y ésta, determinará la función de la futura proteína. Cabe destacar que las proteínas no son sintetizadas por los clásicos mecanismos de síntesis, sino que vienen codificadas con un codón de ADN específico, el cual luego de varios procesos se convierte en una proteína. Cada tipo de proteína tiene una estructura tridimensional única que le confiere una función específica. Esto es algo que sucede en la mayoría de las proteínas, son casi todas específicas, todas actúan uniéndose a un receptor determinado, que no puede ser cualquiera, ej.: hormonas, inmunoglobulinas, enzimas, etc. Se sabe que proteínas con diferentes cadenas Aminoacídicas, tienen funciones diferentes. Además, muchas enfermedades genéticas humanas se deben a la producción defectuosa de algún gen. Por tanto, se puede concluir que al cambiar un aminoácido de la secuencia se altera la estructura primaria, cambiando la función de la proteína. No se sabe con exactitud el modo en que la secuencia de AA determina la estructura tridimensional ni tampoco puede predecirse, pero existen familias de proteínas que tienen características 6 funcionales y estructurales similares, por lo que comparten características. También se habla de dominios haciéndose referencia a subestructuras similares que se encuentran en proteínas diferentes. Sin embargo, se calcula que entre el 20 y 30% de las proteínas tienen diferencias en diferentes poblaciones humanas, pero función biológica se mantiene inalterada. Esto se puede deber a que estos polímeros de aminoácidos suelen tener regiones esenciales en su cadena para su función, la que se conserva siempre. Para analizar la estructura primaria de una proteína se utilizan diversos procedimientos. Se disponen de procedimientos que cortan el AA terminal, lo analizan e identifican, luego separan el segundo y así sucesivamente hasta tener la secuencia completa. Este procedimiento se conoce como degradación de Edman y se realiza en un instrumento conocido como secuenciador. Por último, vale la pena aclarar que el enlace peptidíco que une a los AA en la estructura primaria es una reacción de condensación, dado que libera una molécula de agua. El carbono carboxílico se une al hidrogeno del grupo amino del AA adyacente, formando un enlace amida. En un extremo (Nterminal) queda el grupo amino, mientras que en el otro queda el grupo carboxílico. Es un enlace plano, donde los seis átomos del grupo peptidíco (2 αC, 1 C, 1 N, 1 O y 1 H) son coplanarios. Pero por fuera de esto, el esqueleto polipeptídico es una serie de planos rígidos que puede girar sobre cada αC, ubicado en dos planos simultáneamente. Este hecho de la rigidez, limita el número de conformaciones que puede adquirir. En este enlace, tanto el C como el N unido al αC pueden girar con distintos ángulos. Los ángulos de giro son: φ (N- αC) y ψ (αC-C) Estructura secundaria: Esta estructura se refiere a la conformación loca de algunas partes del polipéptido. Se conocen muchas pero las mas comunes son las hélices α y las laminas u hojas plegadas β. Hélices α. En esta estructura, el esqueleto polipeptídico se encuentra arrollado en forma de hélice sobre un eje imaginario, donde los grupos R de los aminoácidos están hacia afuera de la hélice. Los residuos aminoacídicos giran con ángulos de φ= -60º y ψ= -45 a -50º, cada giro de la hélice tiene un promedio de 3.6 aminoácidos y con una dirección dextrógira, es decir, comenzando de abajo arranca a girar hacia la derecha. La estabilidad de esta estructura se debe a los puentes de hidrogeno, ya que se ubican la mayoría paralelos entre sí, determinando un vector suma muy intenso. Estos puentes de hidrogeno se dan entre el oxigeno carboxílico y el hidrogeno del grupo amino separados por 4 aminoácidos. Siendo los puentes de hidrogeno un tipo de enlace débil, juntos son suficientemente fuertes. Un ejemplo es la α queratina, presente en el cabello y estabilizada a su vez por puentes de disulfuro, resultado de la presencia de cisteína. Sin embargo, existen una serie de restricciones a la hora de formar una hélice α: 1. La cercanía entre grupos R cargados igualmente repelaría y así, desestabilizaría la hélice por fuerzas electrostáticas. 2. La presencia de grupos R muy voluminosos. 3. La presencia de pro o gly. La prolina porque su grupo amino forma parte de un anillo rígido el que le impediría el giro. Mientras que todo lo contrario con la gly, al tener un R tan pequeño (tan solo un H) es muy flexible y desestabiliza al polímero. 7 Hoja plegada β. En esta estructura, el esqueleto se extiende en zigzag y generalmente se disponen cadenas polipeptídicas adyacentes, que se estabilizan con puentes de hidrogeno, alrededor de uno en cada plano plegado. Nuevamente, las cadenas R sobresalen de la estructura en zigzag, generalmente para arriba o abajo. Orientando las cadenas por convención desde el N-terminal hacia el C-terminal, pueden ser paralelas cuando van en la misma dirección o anti paralelas cuando tienen direcciones inversas. También en estas estructuras, es favorable la presencia de grupos R pequeños, ya que al empaquetarse ocupan poco lugar y no desestabilizan la estructura. De hecho, la gly y ala, son los dos AA con cadenas mas pequeñas y por lo tanto, mas frecuentes en esta estructura. Existen los giros β, son elementos de conexión que unen tramos de otras estructuras. Se trata de un giro cerrado de 180º en el que se implican 4 AA formando un enlace de hidrogeno entre el AA 1 y el AA 4. Estructura terciaria: Es la forma tridimensional que adquiere la proteína en su conjunto, estabilizada por las fuerzas del cuadro inicial, donde predominan las no covalentes. Algunas proteínas se componen por dos o más cadenas polipeptídicas en complejos tridimensionales que forman parte de la estructura cuaternaria. Es decir, cuando una proteína se compone por más de una cadena polipeptídicas proteicas, pertenecen a esta estructura. Se le denomina dímero cuando son dos unidades, trímero cuando son tres y tetrámero cuando son 4, ej.: hemoglobina. Se denomina protómero o dominio a cada unidad componente. Al considerar estos niveles, se clasifican a las proteínas según su forma en fibrosas (largas hebras u hojas) o globulares (tienen forma de glóbulo). Las proteínas fibrosas constan generalmente de una sola estructura secundaria, mientras que las globulares tienen varios. También difieren en su función, mientras las fibrosas dan soporte, forma y protegen a los vertebrados, las proteínas globulares se encargan de las respuestas inmunes, transporte, catalizar, etc. Proteínas fibrosas. Son ejemplo de estas la α-queratina, la fibroina de la seda y el colágeno. Dentro de las propiedades que comparten todas son dar fuerza a otras estructuras y elasticidad. Son insolubles en agua debido a la presencia mayoritaria de grupos R hidrofóbicos. α-queratina. Constituyen casi la totalidad del peso seco del pelo, uñas, cuernos, lana, etc. Se localizan en el cito esqueleto de los organismos animales junto a otras, bajo el nombre de cito esqueleto. La resistencia que ofrecen al estiramiento se debe a que las hélices se enrollan de a dos formando una superhélice, Este enrollamiento es levógiro, en sentido opuesto al de la hélice α. En las queratinas más resistentes, como por ejemplo en los cuernos del rinoceronte, se observó que hasta un 18% de los residuos son cisteínas formando puentes de disulfuro. Colágeno. Se encuentra en el tejido conjuntivo de tendones, cartílago, matriz ósea y mas. Se forma de una hélice que no es α, dado que es levógira y tiene tres residuos aminoacídicos por cada vuelta. Además, también es una estructura superenrollada con tres cadenas polipeptídicas formando una 8 trenza. En general, la mayor parte de los AA componentes del colágeno son aquellos que tienen grupos R pequeños y se empaquetan todos sin ocasionar problemas, ej., gly y ala. Se lo puede encontrar en la gelatina, pero no tiene valor alimenticio porque carece de cantidades significativas de aminoácidos esenciales en la dieta humana. La estructura suele ser gly-x-y, donde x suele ser pro e y es 4-hidroxiprolina, un aminoácido no estándar. Proteínas globulares. En estas estructuras, los segmentos polipeptídicos se pliegan unos a otros formando una estructura totalmente compacta. Una de las primeras proteínas estudiadas fue la mioglobina, proteína presente en las fibras musculares, donde se encarga de transportar oxigeno de un lado a otro de la célula. Se forma por una única cadena polipeptídica de 153 residuos y por un grupo hemo, idéntico al de la hemoglobina, responsable del color rojo amarronado. Se esqueleto se forma por 8 segmentos α-hélice conectados por giros de los cuales algunos son giros β. La mayor parte de los grupos hidrofóbicos se encuentran empaquetados en el interior de la molécula y los grupos polares en la superficie, lo que hace que estas proteínas sean solubles en agua. Es una molécula tan compacta que en su interior solo entran 4 moléculas de agua. En este entorno tan empaquetado, las interacciones débiles estabilizan la estructura, especialmente por fuerzas de Van Der Waals. La comprensión de una estructura terciaria se realiza a partir de sus diferentes partes. Se define a motivos o plegamientos como las disposiciones estables que se dan en los segmentos junto a sus conexiones. Así, la complejidad de la estructuras terciaras disminuye al considerar las subestructura. Es posible que existan menos de 1000 dominios. También, se dice que dos proteínas pertenecen a la misma familia cuando presentan similitudes significativas de secuencia primaria, estructura y funciones. También, hay veces que estas familias se agrupan y forman superfamilia, como ene l caso de la hélice alfa que se juntan dos. Estructura cuaternaria: Cuando las proteínas presentan más de un dominio o subunidades peptídicas, se las asigna a la categoría de estructura cuaternaria. Cada cadena, pueden realizar funciones idénticas o complementaria, como regulación y catálisis. Se denomina generalmente multimero a estas proteínas, y cada subunidad o dominio es un protómero. La primer proteína en determinar la estructura cuaternaria fue la hemoglobina, formada por cuatro cadenas polipeptídicas y cuatro grupos hemo (uno en cada cadena) que cuentan con átomos de hierro. De las cuatro cadenas, hay dos cadenas α y dos cadenas β. Los multimeros pueden tener simetría rotacional (el eje de simetría es un eje rotacional) o helicoidal. El tamaño de estas proteínas tiene límites impuestos por la capacidad genética de codificarlas y el error a la hora de sintetizarlas. Por esto, a las células les es más sencillo sintetizar una proteína compuesta por varios polipéptidos, que sintetizar una proteína gigante. De hecho, las proteínas con masa molecular mayor a 100.000 Dalton, se componen por subunidades. 9 Desnaturalización y Renaturalización. Todas las proteínas empiezan siendo una secuencia de aminoácidos, para luego plegarse y alcanzar la conformación nativa. Las estructuras de las proteínas funcionan en entornos concreto, diversas situaciones pueden alteras la estructuras superiores y así, la función de la proteína, lo que se conoce como desnaturalización. El estado de desnaturalización no es necesariamente el desplegamiento completo de la proteína, de hecho, las proteínas desnaturalizadas tienen conformaciones de las que no se sabe mucho. Unos de los agentes desnaturalizantes mas comunes es el exceso de calor y cambios de pH del medio, los cuales afectan las interacciones débiles que estabilizan la conformación, principalmente los puentes de hidrogeno. Al aumentar la temperatura, la conformación se mantiene constante hasta que se rompe una interacción no covalente y se produce un efecto domino, en la que la ruptura de una propicia la ruptura de la otra y así sucesivamente. Como se ha mencionado anteriormente, la secuencia de aminoácidos de una proteína determina su estructura terciaria, lo que se ha demostrado con lo que se ha llamado renaturalización, proceso en el cual una proteína desnaturalizada adquiere su conformación nuevamente. Un ejemplo clásico sucede en la enzima ribonucleasa, la cual se estabiliza por cuatro puentes de disulfuro. Al exponerla en una solución concentrada de urea y un agente reductor, se rompen los cuatro puentes de disulfuro dando lugar a ocho residuos libres de cisteína. Una vez que se elimina la urea y el agente reductor, la ribonucleasa se renaturaliza adquiriendo nuevamente su estructura tridimensional, con los puentes de disulfuro ubicados exactamente en el mismo lugar que antes ocupaban. Otros de los agentes desnaturalizantes comunes son el detergente y los conocidos agentes caotrópicos (urea, guanidinio). Plegamiento. En las células vivas, las proteínas se forman a partir de los aminoácidos a una gran velocidad. Existe la famosa paradoja de Levinthal, en la que se supone que una cadena aminoacídica promedio consta de 100 residuos, los cuales por sus ángulos de giro pueden adoptar 10 formas diferentes, lo que en total existen 10100 conformaciones diferentes que pueden adoptar en general. Suponiendo que cada conformación se ensaya en un tiempo de 10−13 segundos (rapidaso), todas probar todas las conformaciones requeriría 10173 millones de años, tiempo mayor al de la vida del universo. Por lo tanto, una proteína toma atajos para el plegamiento y no sigue estas reglas. Pese a que no se conoce con detalle la forma del plegamiento de las proteínas, existen estudios que proponen diferentes modelos. Por ejemplo, un modelo considera que el proceso está jerarquizado, donde se forman las estructuras secundarias, luego se forman las estructuras supersecundarias (ej. se agrupan dos hélices alfa) hasta que se forma un dominio y se pliega completamente el polipéptido. Termodinámicamente, el plegamiento se puede ver como un embudo de energía libre, donde los estados no plegados tienen una alta energía libre y una alta entropía, debido a que no hay un orden establecido, ya que los AA pueden adoptar diferentes conformaciones. A medida que el plegamiento avanza, disminuye la entropía y la energía libre, se forman estados conformacionales intermedios que luego caducan por su inestabilidad. Al ir cayendo por el embudo cada vez hay menos entropía (el diámetro del cono es más pequeño) y por lo tanto son accesibles menos estados, 10 pero estos son más estables energéticamente. Al fin se llega a la forma plegada donde la energía será óptima y la entropía más pequeña. En lo que respecta a la cinética de la formación de las proteínas, estudios han propuesto que la síntesis ocurre a una velocidad de 5 residuos por segundo (promedio), por lo que resulta 300 residuos en un minuto. Por su parte, la velocidad en la que se pliegan es de 10 mili-segundos. También, se conoce la teoría del plegamiento cotraducional, la que plantea que las proteínas se pliegan a medida que van saliendo de los ribosomas. Esto se debe a que emergen del organelo por un canal estrecho, que no proporciona suficiente espacio para el plegado. Por eso, a medida que va saliendo hacia el citosol, se va plegando adoptando su forma determinada. Existen los llamados proteosomas, son moléculas que digieren proteínas mal plegadas, liberando sus AA correspondientes. Muchas veces, existen facilitadores celulares del plegamiento conocidos como proteínas accesorias del plegamiento. Estas se dividen en tres clases según su función: 1. PDI (Proteína Disulfuro Isomerasa). Es una enzima eucariota que se encarga de catalizar la ruptura y asociación de nuevos enlaces de disulfuro, para contribuir con la estabilidad de la proteína. 2. PPI (Peptidil Prolin cis-trans Isomerasa). Todos los enlaces peptídicos tienden a estar en una conformación trans, lo que los hace mas adecuados para la conformación de la proteína. A veces, la prolina, por tener su grupo amino en un anillo rígido, suele adoptar la conformación cis, lo que dificulta enormemente el establecimiento del enlace peptidíco. Esto sucede relativamente pocas veces en las células vivas, pero para cuando sucede es que están estas enzimas que llevan a la prolina nuevamente a la forma trans. 3. Chaperonas. Se encargan de reconocer zonas hidrofóbicas en la superficie y las separan, para evitar que se agreguen mucho en ese lugar. Se conocen dos clases de estas chaperonas moleculares. Por un lado, la familia Hsp70 (Heat-Shock protein), son abundantes en células sometidas a estrés por altas temperaturas. Actúan uniéndose a regiones hidrofóbicas de polipéptidos desplegados facilitando el plegamiento adecuado. También, evitan que determinadas proteínas se plieguen en momentos inadecuados, como por ejemplo, antes de salir de una membrana, y ayudan a empaquetar proteínas oligoméricas en sus estructuras cuaternarias. La segunda clase de las chaperonas moleculares se conocen como chaperoninas, son complejos proteicos útiles en proteínas que no se pliegan espontáneamente. Se conoce que actúan en el 10 o 15% de las proteínas de la Escherichia coli facilitando el plegamiento de éstas. 11 Enfermedades conformacionales. Una de las enfermedades de este tipo mas conocidas es la anemia falciforme. Cada proteína se traduce a partir de una secuencia de RNAm característica, donde cada codón (secuencia de 3 bases nitrogenadas) se traduce a una proteína. En las personas que sufren de esta enfermedad, muta el gen que codifica la creación del glu (con carga neta negativa), que se traduce a partir de la secuencia GA-G (guanina, adenina, guanina). La mutación ocurre en el lugar de la adenina, la cual se cambia por una timina, y resulta un codón GTG, el cual codifica para la valina, AA apolar. Por lo cual, en la hemoglobina (Hb) de estos pacientes encontramos una val en el lugar de un glu. Mediante estudios, se ha visto que la valina establece interacciones con otras moléculas de Hb, generando una agregación de moléculas de hemoglobina, apareciendo grandes hebras de hemoglobina, que generan una forma lunar característica. Esto, los hace perder la elasticidad y se destruyen con una velocidad mayor a la velocidad con la que se crean, debido a que los macrófagos se los fagocitan constantemente. Esto, produce anemia y trae múltiples consecuencias. Una característica importante de esta enfermedad, es que la valina actúa en la forma desoxihemoglobina, es decir, cuando la hemoglobina no contiene oxigeno unido a su grupo hemo. Por eso, a los pacientes enfermos se los protege contra enfermedades que reduzcan la cantidad de oxigeno en sangre como por ejemplo las enfermedades respiratorias, para no propiciar tanta cantidad de hemoglobina desoxigenada. Otro tipo de estas enfermedades deriva de los priones, proteínas capaces de causar una enfermedad de este tipo. Al principio, ocurría en tribus que practicaban canibalismo y se enfermaban comúnmente de un tipo de enfermedad que en ese entonces se creía que era de etiología viral o bacteriana. A su vez, sucedió con pacientes con déficit de la GH (growth hormone), a los cuales se les inyectaba esta hormona de cadáveres, ya que no se disponía del conocimiento para su síntesis artificial. Luego de esto, experimentaban una nueva enfermedad, en la que el común denominador era que el tejido nervioso adquiría una textura esponjosa y se creía que era causada por virus o bacterias. Luego de varios modelos etiológicos, un científico propuso que eran enfermedades causadas por cambios en la conformación de ciertas proteínas, por lo cual ganó hasta un Nobel. Actualmente, uno de los casos mas comunes es el mal de la vaca loca o BSE (Bovine Spongiform Encephalopathy), causa de muerte de muchas vacas en nuestro país, en donde las mismas experimentaban demencia y ataxia (falta de coordinación). El que gano el nobel sugirió que si eran realmente causadas por virus o bacterias, habría que quitar los ácidos nucleicos del tejido infectado (nervioso). Con otras investigaciones, se descubrió que todas estas eran enfermedades causadas por priones, proteínas que existen en la forma infectada y en la forma normal en nuestro cuerpo. El termino espongiforme corresponde a la textura en la que se muestra el tejido nervioso infectado. Sus funciones en la versión normal no están completamente comprobadas, aunque se conocen dos o tres funciones como que reducen algunos compuestos. 12