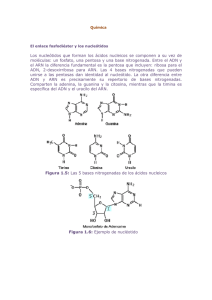
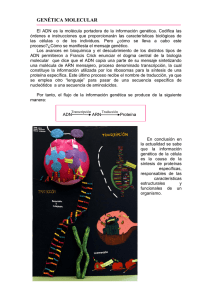
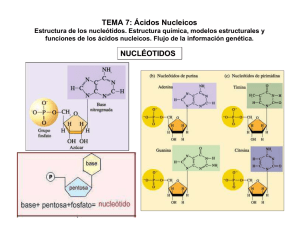
7.8.5 PCR en tiempo real Para detectar los genes encargados de la expresión de rGABAA y rAMPA se realizará una PCR en tiempo real con los siguientes pasos: 1. Extracción de ARN Se tomarán de 50 a 100 mg de tejido de amígdala o hipocampo y se lavará 2 veces con PBS 0.1M. Una vez lavado, el tejido se trasferirá a tubos ependorff de 2 ml y se agregará 1 ml de TRIzol® Reagent (Anexo 1) para formar un lisado, una vez obtenido se incubará 5 min y se añadirá 0.2 ml de cloroformo, para después mezclarse y dejar reposar durante 2-3 min. Después del reposo se centrifugará a 12000 g durante 15 min a 4oC. Luego se transferirá la fase acuosa a un tubo limpio ependorff de 2 ml dónde se procederá a la precipitación del RNA mediante la adición de 0.5 ml de isopropanol. Posteriormente se incubará nuevamente durante 10 min a temperatura ambiente y se centrifugará a 12000 g durante 10 min a 4oC, de nueva cuenta se transferirá la fase acuosa a un tubo limpio ependorff de 2 ml. Por último, se realizará un lavado con etanol al 70% y se centrifugará a 7500 g, 5 min a 4oC. El ARN obtenido se dejará secar hasta que se evaporé el etanol y el precipitado se observe incoloro. Posteriormente se resuspenderá en el volumen de 50 ul de H20 (Livak & Schmittgen, 2001). 2. Valoración del ARN El ARN se determinará midiendo la concentración obtenida en un cuantificador de ADN/ARN a 260 nm (GeneQuant Pro, RNA/DNA Calculator, ProMega) y comprobando la integridad del RNA. Para esto se cargará entre 0.5 a 1 g de ARN de cada muestra en un gel de agarosa 1%. 3. Retro Transcripasa -PCR Para generar una gran cantidad de copias de ADNc con ARN, se utilizará el kit SuperScript ® III First-Strand Synthesis System (Invitrogen) para realizar la reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR), la cual actúa como una retrotranscripta para convertir el ARN a ADN complementario (ADNc) siguiendo las indicaciones del manufacturado (Anexo 2) (Scientific, 2011). 4. PCR en tiempo real y expresión de Graba2 y Gria1 Para la cuantificación relativa se llevará a cabo una PCR en tiempo real, cuya reacción consistirá en un volumen total de 10 microlitros (μl), conteniendo: 1X de Master Mix (FastStarTM Taq DNA polimerasa, buffer de reacción, MgCl2 y una mezcla de los dNTPs), 0.5 μl de cada primer, 1 μl de cDNA y la cantidad de H 2O inyectable necesaria para completar 10 μl de mezcla total, donde se emplearán los primers (tabla 6): Gen Primer Tamaño del producto Gria1 “glutamate ionotropic receptor AMPA type subunit 1” Left: TCTGTCTTCTGTGGCGTGAC 145 pb Right: ATGCCCCTTGTAAGCAACAC Gene ID: 50592 Gabra2 “gamma-aminobutyric acid type A receptor rho 2 Left: GGCCTGTTCTTTCACATGGT Right: ATCCTGCAGACAGAGGCAGT 127 pb subunit” Gene ID: 29695 Gapdh Left:CACTGAGCATCTCCCTCACA glyceraldehyde-3phosphate dehydrogenase Right:AGAGGGTGCAGCGAACTTTA 108 pb Gene ID: 24383 Tabla 6. Primers. Primers a utilizar en la PCR en tiempo real (3). La amplificación y detección será desarrollada utilizando el termociclador CFX96 TouchTM Real-Time PCR BioRad con siguiente programación: preincubación 1 ciclo a 95 °C , 10 min; amplificación y cuantificación 35 ciclos a 95°C, 10 seg., 60 °C 20 seg., 72 °C, 1 seg.; Enfriamiento 1 ciclo 40 °C, 30 seg. El análisis de datos por real-time PCR, fue mediante el software CFX ManagerTM Software 1845000. Versión 3.1. Anexo 1. Reactivo TRIzolTM Protocolo experimental para la aislación de ADN. Procedimiento: 1. Realizar todos los procedimientos a temperatura ambiente (20°C-25°C). 2. Usar el reactivo TRIzolTM si el material contiene altos niveles de RNasas, como bazo y páncreas. 3. Usar material plástico desechable y estéril. 4. Usar guantes desechables mientras se manejan los reactivos y muestras de RNA para evitar la contaminación de las RNasas. Para aislamiento de ADN: 1. Después del lisado, remover cualquier residuo acuoso de la interfase. 2. Agregar 0.3ml de etanol al 100% por cada ml de TRIzolTM usado para lisis. 3. Cerrar el tubo mediante la mezcla invirtiendo el tubo varias veces. 4. Incubar de 2 a 3 min. 5. Centrifugar por 5min a 2000xga 4°C. 6. Transferir la suspensión etanol-fenol a un nuevo tubo. 7. La suspensión se utiliza para el aislamiento de la proteína (Puede almacenarse a -70°C por varios meses). Lavado del ADN: 1. Resuspender el pellet en 1ml de citrato de sodio a 0.1M en 10% de etanol, con un pH de 8.5, por cada ml de TRIzolTM usado para la lisis. 2. Incubar por 30min mezclando ocasionalmente por inversión. 3. Centrifugar por 5min a 200xg a 4°C 4. Descartar el sobrenadando con micropipeta. 5. Repetir los pasos 1 y 4 una vez (2 veces por pellets del doble de largo). 6. Resuspender el pellet en 1.5 a 2ml de etanol al 75% por cada ml de TRIzolTM usado para la lisis. 7. Incubar de 10 a 20 min mezclado gentilmente. 8. Centrifugar por 5min a 200xg a 4°C 9. Descartar el sobrenadando con micropipeta. 10. Secar con aire el pellet de ADN de 5 a 10min. (no usar centrífuga) Solubilizar el ADN: 1. Resuspender el pellet en 0.3-0.6 ml de 8mMNaOH pipeteando de arriba abajo. 2. Centrifugar por 10min a 12,000xg a 4°C para remover materiales insolubles. 3. Transferir el sobreflotante a un tubo nuevo y medir el pH. Anexo 2. Kit SuperScript ® III First-Strand Synthesis System (Invitrogen) Materiales necesarios: a. 2ml de agua desionizada libre de RNasas b. Vortex c. Termociclador con configuración a 65°C (CFX96- BioRad) d. Centrífuga e. Muestra: 5 µg de RNA (n µ/L) f. Primer (50µM oligo (dT) o 2 µM primer del gen específico (1 µ/L) g. Tapom Buffer (1µ/L) Procedimiento: 1. Mezclar y centrifugar de manera breve las sustancias antes de usarlas. 2. Precalentar el termociclador a 65°C 3. Combinar los reactivos e,f,g y a en un tubo de PCR de 2ml con paredes delgadas y conservar a 4°C. 4. Incubar en termociclador a 65 ° C durante 5 minutos y colocar sobre hielo al menos 1 minuto. 5. Agregar 10 µ/L de Reactivo Mix 2x First-Strand 6. Agregar 2 µ/L de mezcla de enzimas SuperScript II RNAasa OUT 7. Mezclar en el vortex de manera breve. 8. Incubar durante 50min a 50°C en Oligo (dT) 20 o GSP cebado 9. Incubar el primer durante 5 a 10 minutos a 25 ° C seguido de 50 minutos a 50 ° C 10. Terminar las reacciones a a 85 ° C durante 5 minutos. 11. Enfriar la reacción de síntesis de cDNA en hielo y almacenar a 20°C o proceder con la PCR.