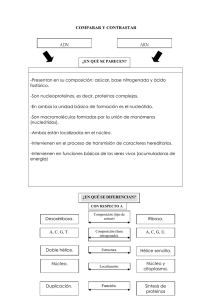
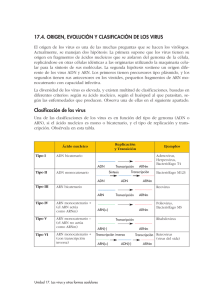
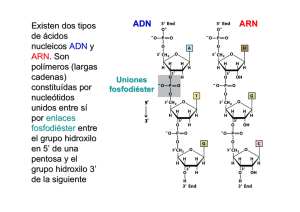
Proteína tiene 3 dominios 1. Dominio de reconocimiento 2. Dominio de acción 3. Dominio isomérico Transcripción mitocondrial ADN mt altamente conservado caracterizado por tener bajas concentraciones de regiones intronicas. Solo el 3% es de regiones codificantes. Vamos a tener 3 puntos de inicio: 2 ubicados en cadena pesada H1 y H2 1 punto en cadena ligera con L2 Permite que las dos cadenas se transcriban, no se transcriben al mismo tiempo las dos cadenas sino dependiendo del requerimiento. - Factor de iniciación A que inicia el complejo de iniciación y permite que se transcriban o la cadena pesada o la cadena ligera. Factor de transcripción de finalización que tiene afinidad por la unión y reconocimiento de ARNt de leucina. En cadena pesada se transcriben 28 genes que corresponden a: 2 de ARNr, 14 ARNt y 12 proteínas mitocondriales o polipéptidos. En cadena ligera se transcriben 9 genes polipéptido mitocondrial y 8ARN t. Tenemos que la región no codificante se la conoce como bluque D o Bluque de desplazamiento que estará junto al origen de replicación. ADN mt se transcribe como las procariotas de manera polisistrònica. Cadena pesada 1 o H1 el punto de inicio es delante del gen de ARN de transferencia fenilalanina y termina en extremo 3`de ARNr 16s. Permitiendo de esta manera que se sintetice ARNr 12s, 16s, ARNt fenilalanina y ARNt valina. Luego, se une el Factor de terminación MTRTF que se une a la secuencia de ARNt de leucina. Cadena pesada 2 o H2 se sobrepone sobre la cadena pesada H1 porque transcribe en su totalidad a partir del extremo 5` del gen 12s ARNr. Permite transcribir 12 ARNt y 12 polipéptidos. Cadena ligera se inicia cerca del bluque D se inicia transcripción en dirección contraria y sintetiza 1 polipéptido y resto de ARNt de manera polisistrònica. Cuando se transcribe todas las cadenas se obtiene la secuencia Cambridge que es la secuencia de todo el genoma mitocondrial. Permite identificar en que posiciones y puntos se completa en realidad las 16569 pb. - Puede haber diferentes incios. - Puede depender de que tipo de topoisomerasa entre para ver que cadena se trancribe. Cuando tenemos todo el ADN mt transcrito es evidente que se tiene lugar diferentes procesos de modificación: - - Transcritos primarios de cadenas pesadas y ligeras serán modificados por reacciones de endonucleasas que permiten identificar claramente donde inicia y finaliza c/u de los subproductos que se obtienen. Puede sufrir mutaciones (poco frecuentes) aunque es muy conservado. 1. Las mutaciones puntales: cambian la conformación de los polipéptidos mitocondriales. 2. Reorganizaciones: deleciones / duplicaciones suelen ser mas efectivas sobre material genético de ARNt. 3. Disminución del numero de copias de ADNmit: se ve afectado en polipéptidos ya que no completan todas las sub unidades. Objetivo: realizar proceso de transcripción con las mismas condiciones que transcripción nuclear, que solo se da dependiendo de la cadena que se necesite. Solo se lleva a cabo por ARN polimerasa que no tiene numero y tiene dos subunidades: 1. 2. Encargada de polimerización, es decir la síntesis de los diferentes tipos de ARNs. Encargada de reconocer adecuadamente la cadena molde, ya que se transcriben las dos y sirven de molde c/u dan transcriptos diferentes (no sucede en ADN nuclear). PROCESOS REPRESORES DE LA TRANCRIPCION Si inhibe la transcripción en algún punto las moléculas represoras se unen a cadena de ADN e inhiben paso de burbuja de transcripción. Solo con estas condiciones se habla de moléculas represoras, que existen tres tipos: 3 tipos: 1. Antibióticos tipo 1: inhiben transcripción en eucariotas y procariotas, depende de afinidad con ARN pol. Alfa amanitina alta afinidad por ARN pol 2, es decir la inhibe. Medianamente represible para ARN pol 3 y no tiene efecto con ARN pol1. Se une a domino CTD, de fosforila el dominio CTD y ARN pol pierde afinidad por cadena de ADN y se desacopla. DEPENDE DE CONCENTRACION: Si hay mayor concentración del medicamento también se inhibe a ARN pol 3. Si disminuyo la concentración disminuye también el control de inhibición. Inhibe transcripción de ARNm, parcialmente de ARNt y ARNr pequeño y no tiene inhibición sobre síntesis de ARNr grande. Rifampicina bloquea efectivamente a las 3 ARN polimerasas in vivo. Efecto tanto para procariotas y eucariotass. Se usa para eliminar infecciones. Inhibe transcripcion. 2. Antibióticos tipo 2: Inhiben transcripción y replicación ya que tienen estructura similar a nucleótidos. Molécula se intercala como nucleótido y entra en el puesto del mismo, provocando desacoplamiento de ADN pol porque ya no encuentra extremo 3` OH libre para seguir añadiendo los siguientes nucleótidos. Ya no avanza burbuja de transcripción. Utilizado para inhibir procesos de transcripción procariota. Cordicepina se intercala como si fuese un nucleótido que hace que no exponga extremo 3` Oh. Es un antitumoral, antifúngico y antiviral. Efecto sobre inhibición de la transcripción inversa. Actinomicina D se intercala y reconoce el complejo de iniciación permite que se forme el complejo de iniciación. Cuando se quiere empezar la elongación no permite que ARN polimerasa realice este procedimiento. Se une a sub unidad que sirve para la elongación. En procariotas: sub unidad beta y beta prima. En eucariotas: sub unidad RTB1. No se inhibe muchos procesos de transcripción mitocondrial. No funciona sub unidad catalítica que permite la elongación 3. Antibióticos tipo 3: No inhiben el proceso de transcripción, sino a las ADN girasas a topoisomerasas tipo 2. Esto permite que no se liberen tensiones generadas del ADN y no puede acceder maquinaria de transcripción MICRO ARN Se sintetizan normalmente en el organismo y tiene como efecto que mientras avanza la burbuja de transcripción, estos se meten ahí pegándose sobre ADN molde formando un bloqueo (hibrido ADN-ARN) y no permite el avance de burbuja. Tienen 20-25 nucleótidos de largo. Se forman micro ARN de doble cadena que provoca la inhibición de la transcripción. Si hay sobreexpresión se silencian los procesos transcripcionales. MODIFICACIONES POST TRANSCRIPCIONALES Permiten obtener ARNm maduro. Se edita sobre productos para ARNm, ARNt y ARNr. Las modificaciones son: - Colocación del casquete en extremo 5` de ARNm naciente. Proceso de corte y poliadenilación en extremo 3` ARNm naciente. Corte y empalme o splicing que depende del como identifiquemos a unidades de transcripción. Puede ser sucesivo o alternativo. 1. Colocación de una formación de casquete o cápside en extremo 5` de ARNm naciente. Se da cuando se transcribe. Importante: tiene efecto apenas sale el extremo 5` del ARN naciente. Activando la enzima colocadora de casquete se activa cuando se transcriben los primeros 25 nucleótidos de ARNm. Activados a través de dominio CTD de ARN pol. CTD fosforila a enzima haciendo que se una al ARNm. Enzima tiene 2 sub unidades: 1 de fosfatasa (retira grupos fosfatos en posición gamma deja el di nucleótido di fosfatado) y 2 hace transferencia del resto del GTP (enzima guanil fosfatasa) al dominio 5` di fosfatado (enlace inusual 5`5` guanosina fosfato); dominio de reconocimiento de ARN que capta el primer nucleótido trifosfatados de 5` Tienen RRM Metilacion se hace en posicion 7 del extremo 5` de la guanosina por 7 metil guanosina (formadora de casquete). 2. Cote y poliadenilación en 3` de ARNm naciente: ocurre cuando se llega al final de la transcripción, cuando se libera extremo 3` de ARN naciente. - En extremo 3` se identifican 3 señales: Se identifica el sitio de poliA Señal de poli A en dirección de 5` corriente arriba del sitio de Poli A. Con una secuencia invariable (AAUAAA) a veces solo en mamíferos (AUUAAA). Región rica de guaninas y uracilos en dirección 3` va a tener una secuencia formada por 20 pb. ¿Cómo se da? Siempre se realiza un corte y poliadenilación. Primero se corta parte del extremo 3` que forma parte de región UTR y luego se coloca la cola de poli A. Entra en juego el conjunto de proteínas formada por: CPSF, CStF, CF1 y CF2. CPSF= factor de especificidad de corte y poliadenilación. Se une a señal en dirección 5` CStF= Factor estimulante de corte. Se une sobre la secuencia rica en Adeninas y Uracilos. Cuando estas dos proteínas están unidas al ARNm, entre las dos hay atracción y doblan la cadena de ARN. Permite que se acople como complejo proteico el CF1 y CF2. CF1= factor de corte 1 CF2= factor de corte heterodimèrico Forman la plataforma de corte y poliadenilación. Se tiene como efecto que se exponga el sitio de poliadenilación y se une la PAP (Enzima poli A polimerasa) quien realiza el corte en el sitio de poliadenilación y libera en dirección 3` la secuencia de ARNm y además los factores de corte CStF, CF1 y CF2. Molécula de ARNm que se libero va a ser procesada por exosoma, que es una formación de 11 exonucleasas de varias proteínas unidas que tiene como actividad exoribonucleasas 5` y 3`. Que hidrolizan base por base la secuencia de ARNm para liberar ribonucleótidos que se reutiliza. La actividad exonucleasa o de hidrolisis que domina es 3` 5`. Importante: cuando se realiza la eliminación de regiones intronicas de ARNm también se logra procesar los pedazos que salen como regiones intronicas a través de los exosomas. Cuando ya se realiza el corte la PAP añade los 12 residuos de adenosina conocido como poliadenilación lenta. También, se añade múltiples copias de PABPII (proteína de unión a poli A tiene un motivo RRM para reconocer ARNm) se une y realiza la poliadenilación rápida que añade hasta 200 nucleótidos de adenosina, cubriendo el extremo 3` del ARNm. El objetivo es que el ARNm migre hacia el citoplasma y se inicie traducción por extremo 5`. El extremo 3` se ve afectado y es degenerando. 3. Corte y empalme: Sirve para proteger ARNm intermediario, por esto se unen a proteínas especializadas que se llaman ribonucleoproteínas heterogéneas nucleares o HNRNP (sintetizadas por ARN pol 2) tienen tres funciones: 1. Se asocia a cadena de ARN para protegerlo. 2. Permite que se dirija exclusivamente los lugares donde tiene efecto corte y empalme posterior. 3. Se de un transporte activo de la molécula del transcripto maduro hacia el citoplasma. ¿Como se unen estas HBRBP? Tienen dos dominios que permite unirse a cadena de ARN: - 1) Primer dominio de reconocimiento de ARN o RRM (también la tiene la enzima formadora de casquete y factores de corte y poliadenilación). Tiene una hoja beta que tiene 4 hebras flanqueadas por dos hélices alfa que permite que se ancle la proteína y permite la unión completa. - 2) RBD: dominio de unión a ARN. Caracterizado por tener dos clases de condiciones altamente conservados RNP 1 Y 2. Formados por 80 residuos de aminoácidos, se ubican a cada lado sobre las hojas beta centrales que tiene la proteína como tal, que exponga dominio de reconocimiento. Para que tenga contacto con cadena monocatenaria del ARNm y hace que interactúe la carga negativa del fosfato del ARNm con la hélice alfa de carga positiva en molécula de unión al ARN. Las HBRBP suelen unirse a lugares de unión entre regiones intronicas y regiones exónicos. Con el objetivo de proteger y marcar el lugar de corte y empalme y proteger la región exónica. - Otro dominio de unión es el Motivo KH (Dominio RBD) que está formado por proteínas k que tienen 45 residuos que permiten unión a ADN. Estructura similar a dominio de reconocimiento de ARN ósea RRM. Se caracteriza porque: las dos hélices alfa forman una interacción hidrófoba con el ADN y permite la interacción con 3 laminas beta flanqueadas. - Otro dominio de unión al ADN que exponen las HBRBP. Es la Caja RGG (dominio RBD) caracterizado por tener un dominio con 5 repeticiones seguidas de 3 aminoácidos constantes: Arginina, Glicina Glicina que dan carga positiva y añaden aa aromáticos que permiten interacción con ARN uniendo a monocatenaria de ARN. Para conocer moléculas intronicas que se deben eliminar se debe identificar ciertos puntos excepcionales para saber dónde cortar. ¿Qué delimita una región intrònica? Una región intronica siempre va a tener: 1. En extremo 5` de corte y empalme siempre va a tener una Guanina y un Uracilo que inicien. 2. En extremo 3` de corte y empalme siempre debe exponer una Adenina y una Guanina. 3. Región rica en pirimidinas (entre 10 a 12 pb) cerca del extremo 3`. Aquí se añaden las HBRBP. 4. Con una distancia de 20 - 50 pb del extremo 3` del intrón se tiene el punto de ramificación. Que es una Adenosina que permite que se elimine el intrón a manera de lazo. ¿Cómo se da la eliminacion del intron o corte y empalme? Se explica a traves de dos reacciones de transesterificacion: 1. Primera reaccion de transesterificacion: se necesita identificar el enlace ester entre el fosforo 5` del intron 1 y el oxigeno 3` del exon 1. Ademas, tambien, el oxigeno 2` dentro del punto de ramificacion que esta de 20 a 50 pb en direccion 3` del intron. Se rompe el enlace ester y se evidencia que el grupo fosfato del extremo 5` queda incompleto. Y se forma un enlace inusual 2` 5` fosfo ester. El hidrogeno pasa a formar parte del grupo OH que se expone en el exon 1 separado. Intron en forma de lazon aun se mantiene unido al exon 2. 2. Segunda reaccion de transesterificacion: Tiene efecto cuando aun esta unido el intron al exon 2. Se debe identificar: - El enlace ester entre oxigeno 3` del intron y el grupo fosfato en direccion 5` del exon 2. Se elimina el enlace ester y el fosforo se queda incompleto. Forma el enlace ester entre OH y grupo fosfato. Al final se tiene enclace fosfo ester y se libera un intron con un extremo 3` OH. Quedandose unidas solo las regiones exonicas. REACCIONES DE TRANSESTERIFICACION MEDIADAS POR ENCIMAS:. Son: 1. Las ribonucleoproteinas nucleares pequeñas o SnRNPs. Se unen y forman el Spliceosoma que es diferente del exosoma. El Spliceosoma conformado por un grupo proteico de aproximadamente de 170 proteinas que vienen a aprtir de ARNs nucleares pequeños que son secuencias pequeñas de ARN ricas en uracilos. Sub unidades requieren nombres de U. Que son: 1. 2. 3. 4. 5. Sub unidad 1 o U1: Sub unidad 2 o U2: Sub unidad 4 o U4: Sub unidad 5 o U5: Sub unidad 6 o U6: ¿Qué hace el spliceosoma? Cada sub unidad se integra en las dos reacciones de transesterificación. ¿Cómo se unen estas proteínas? Las mismas se unen en cierta forma: U1: se une para reconocer el enlace éster formado entre el fosforo 5` del intrón y el oxígeno 3` del exón 1. Se involucra en la primera reacción de transesterificación U2: Se une hacia el punto de ramificación y reconoce el oxígeno en posición 2` del punto de ramificación. También se une el factor asociado a U2 que permite que el factor U2 se mantenga unida en el punto de ramificiacion. Cuando se une U1 y U2 adicionalmente se une el factor 1 del spliceosoma el SF1 que facilita el reconocimiento de la U2 al punto de la ramificiacion. Cuando se libera SF1 se van a unir y van a empezar a interactuar las otras 3 sub unidades del spliceosoma Se une como dimero U4 y U5 y posteriormente U5. Cuando tenemos las 5 subunidades unidas se le denomina Spliceosoma o Empalmosoma. Primer reordenamiento: la primera rección de transesterificación, romper enlace éster entre oxigeno 3` de exón 1 y fosforo 5` del intrón 1. Hace que fosforo forme enlace fosfoester en dirección 5` 2` con punto de ramificación. Cuando se da la primera reacción de transesterificación se libera la sub unidad U1. Y también se libera U4 (permite que U5 le ancle a región exónica 1) U5: sub unidad que mantiene unida a la región exónica que se liberó. Segunda reacción de transesterificación: Cuando queda U2, U5 y U6. La responsable será la U6 de reconocer el fosforo 5` del exón 2 y el oxigeno 3` del intrón. U5 será la responsable de unir las regiones exónicas. Intrón es degradado por ribonucleasas que permitan que se dirigía la región intronica hacia el exosoma. Las dos reacciones de transesterificación con empalmosoma tienen lugar cada corte y empalme al querer realizar un corte de intrones. Existe la posibilidad del corte y empalme alternativo que elimina regiones exonicas que no se necesita. Con el mismo proceso.