Caracterización Estructural de Biomoléculas: Difracción y Espectroscopía
Anuncio

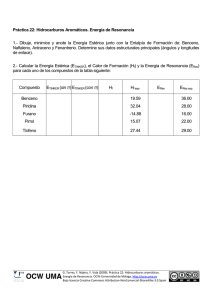
CARACTERIZACIÓN ESTRUCTURAL DE BIOMOLÉCULAS 9Interacción radiación-materia: 9Técnicas de dispersión (elástica): difracción de rayos X, electrones, neutrones... 9Técnicas inelásticas (espectroscopias): 9NMR 9EPR 9Espectroscopias ópticas 9Raman 9... 9Microscopias atómicas ¿Caracterización estructural? ¿Cómo es posible? 9Purificación de macromoléculas biológicas: Cromatografías DIFRACCIÓN DE RAYOS X EN MACROMOLÉCULAS BIOLÓGICAS Fundamento teórico: 9Ley de Bragg 9¿Pueden las macromoléculas biológicas organizarse en cristales? 9¿Puede obtenerse la estructura de un cristal de proteína? DIFRACCIÓN DE RAYOS X Maurice Wilkins Rosalind Franklin William Lawrence Bragg Max Perutz John Cowdery Kendrew DIFRACCIÓN DE RAYOS X Esquema general: DIFRACCIÓN DE RAYOS X CRECIMIENTO DE CRISTALES DE PROTEÍNAS Se crecen a partir de disoluciones (acuosas) DIFRACCIÓN DE RAYOS X Características de la muestra de proteína para cristalización: 9Alta concentración 9Alta pureza 9Alta homogeneidad 9Alta estabilidad Métodos para la obtención de la sobresaturación: 9Control de la Evaporación 9Control del pH 9Control de la temperatura 9Control de la concentración salina 9Uso de precipitantes 9Inclusión de sustratos DIFRACCIÓN DE RAYOS X Obtención de monocristales: Cristales inestables (pérdida de agua): DIFRACCIÓN DE RAYOS X Fuentes de rayos X y difractómetros Patrón de difracción obtenido DIFRACCIÓN DE RAYOS X El problema de la fase Función de Patterson: DIFRACCIÓN DE RAYOS X 9Métodos de resolución estructural (solución al problema de la fase): 9Métodos directos 9Método de reemplazamiento múltiple isomorfo (MIR) 9Método de dispersión anómala múltiple (MAD) 9Método de reemplazamiento molecular (MR) 9Refinamiento estructural DIFRACCIÓN DE RAYOS X Estructura obtenida: representaciones DIFRACCIÓN DE RAYOS X Estructura obtenida: representaciones DIFRACCIÓN DE RAYOS X Estructura obtenida: representaciones DIFRACCIÓN DE RAYOS X Estructura obtenida: representaciones DIFRACCIÓN DE RAYOS X Estructura obtenida: representaciones DIFRACCIÓN DE RAYOS X Estructura obtenida: representaciones “Protein Data Bank”: registro accesible de los varias decenas de miles de estructuras resueltas de proteínas: www.pdb.org DIFRACCIÓN DE RAYOS X EJEMPLO: EL COMPLEJO FNR-NADP La enzima “Ferredoxina FADP+ Reductasa” DIFRACCIÓN DE RAYOS X Ciclo catalítico de la FNR: DIFRACCIÓN DE RAYOS X Estructura obtenida: representaciones DIFRACCIÓN DE RAYOS X DIFRACCIÓN DE RAYOS X DIFRACCIÓN DE RAYOS X USO DE TÉCNICAS ESPECTROSCÓPICAS EN BIOFÍSICA ¿Para qué, la espectroscopia? 9Las estructuras mediante difracción de rayos X son muy útiles, pero... 9No siempre se dispone de cristales 9Debate: ¿estructura cristal-estructura disolución? 9Información sobre la proteína activa 9Información no contenida en la estructura molecular 9Las espectroscopias suministran métodos de caracterización inicial básica 9Las espectroscopias segurirán suministrando caracterización avanzada que no se puede sustituir mediante estructura por difracción. USO DE TÉCNICAS ESPECTROSCÓPICAS EN BIOFÍSICA Espectroscopia: estudio de la interacción radiación-materia con resolución espectral En general, son técnicas inelásticas (absorción, emisión, dispersión inelástica) Se clasifican por: 9Rango de frecuencias 9Grado de libertad energético del material que se excita 9Espectroscopias ópticas 9NMR 9EPR 9Raman 9Mössbauer 9... RADIACIÓN EM e INTERACIÓN INELÁSTICA Radiación EM Materia Longitud de onda: λ Frecuencia: ν = c/λ = ω/(2π) Niveles discretos de energía hν Energía del fotón: E = hν ∆E Condición de BOHR ∆E = hν PROCESOS Absorción Emisión espontánea hν Emisión estimulada hν hν Relaciones de Einstein P(Emis . espon.) ∝ν 3 P( Abs .) P(Emis . Est .) ∝ P( Abs .) hν INTRODUCIÓN AL FENÓMENO DE LA RESONANCIA (doblete) Sistema de dos niveles, |+⟩ y | - ⟩, separados en energía ∆E = gµBB0 en presencia de un campo estático B0. | + ⟩ ∆E = gµBB0 Z↑ n+ N = n+ + n - Z↓ (constante) | - ⟩ Estado estacionario Equilibrio a una temperatura T (Temperatura de espines) ⎛ ∆E n +ss ⎜⎜ − exp = n −ss ⎝ k BT ⎞ ⎟⎟ ⎠ n- Interacciones con “LA RED” y el CAMPO EM dn+ = − Z ↓ n+ + Z ↑ n− dt dn− = Z ↓ n+ − Z ↑ n− dt n ss = n −ss − n +ss = equilibrio n = n- - n+ dn = (Z ↓ − Z ↑ )N − (Z ↓ + Z ↑ ) n dt Z↓ − Z↑ N Z↓ + Z↑ 1 τ1 dn n − n ss =− τ1 dt = Z↓ + Z↑ TIEMPO DE RELAJACIÓN USO DE TÉCNICAS ESPECTROSCÓPICAS EN BIOFÍSICA ¿De qué color son las proteínas? 9 Espectrofotometría de absorción * IR * Visible * UV 9 Espectrofotometría de emisión 9 Dicroísmo Circular ESPECTROSCOPIA ÓPTICA Esquema de un espectrofotómetro de un solo haz y de doble haz ESPECTROSCOPIA ÓPTICA ESPECTROSCOPIA ÓPTICA Isomería óptica de aminoácidos ESPECTROSCOPIA ÓPTICA El enlace peptídico ESPECTROSCOPIA ÓPTICA Estructura de hélice α ESPECTROSCOPIA ÓPTICA Estructura de lámina β ESPECTROSCOPIA ÓPTICA Absorbancia visible-UV: Caracterización de aminoácidos ESPECTROSCOPIA ÓPTICA Fluorescencia de aminoácidos aromáticos ESPECTROSCOPIA ÓPTICA Estructura secundaria revelada mediante dicroísmo circular en el UV ESPECTROSCOPIA ÓPTICA Estructura secundaria revelada mediante absorción de infrarrojos ESPECTROSCOPIA ÓPTICA Espectro diferencial UV Detección de la compacidad de plegamiento Espectro de fluorescencia FUNDAMENTOS DE LA RESONANCIA MAGNÉTICA NUCLEAR Modelo “naïve” Momento magnético FUNDAMENTOS DE LA RESONANCIA MAGNÉTICA NUCLEAR Ondas de radio Acoplo de la oscilación B Detección de la frecuencia de giro FUNDAMENTOS DE LA RESONANCIA MAGNÉTICA NUCLEAR B FUNDAMENTOS DE LA RESONANCIA MAGNÉTICA NUCLEAR La interacción entre núcleos modifica la frecuencia de giro INFORMACIÓN ESTRUCTURAL FUNDAMENTOS DE LA RESONANCIA MAGNÉTICA NUCLEAR 9Los núcleos de algunos isótopos presentan degeneración de espín 9Al aplicar un campo magnético, la degeneración se resuelve, efecto Zeeman (nuclear) r r H = µ N g N B0 ⋅ I = µ N g N B0 I z E (M ) = E0 − µ N g N B0 M |M⟩ 2I+1 Transiciones dipolares magnéticas hν -|M⟩ Resonancia | ∆M | = ±1 hν = µN gN Bo De acuerdo con hν = µN gN Bo la resonancia se puede obtener experimentalmente variando la frecuencia, ν, o el campo estático, Bo. RESONANCIA MAGNÉTICA NUCLEAR Evolución de la magnetización bajo campo estático r B0 = B0 k̂ r B0 ZL r M (t) YL M⊥ (t) γ = µN gN r r M= I r r r dM = −γ M ∧ Bo dt ωL = γ Bo XL M x (t ) = M ⊥ (0 ) cos (ω L t ) dM x = ω L BM y dt dM y = −ω L BM x dt dM z =0 dt Frecuencia de Larmor (propia de cada núcleo) La magnetización realiza M y (t ) = M ⊥ (0 ) sin (ω L t ) una precesión en torno al M z (t ) = M z (0 ) campo magnético estático RESONANCIA MAGNÉTICA NUCLEAR M x (t ) = e − t / T2 La precesión de la magnetización M ⊥ (0 ) cos(ω L t ) relaja al estado de equilibrio a M y (t ) = e M ⊥ (0 ) sin (ω L t ) eq eq M z (t ) = M z − e −t / T1 M z − M z (0 ) − t / T2 [ ] través de la interacción con los modos de energía de la red Microondas: introducen un campo transversal, débil y variable [ r r r B1 (t ) = B1 cos(ϖ t ) i + sen(ϖ t ) j r B0 ZL r B1 (t) r M (t) YL ] Cuando ω = ωL, el campo de microondas se acopla a la magnetización, y se produce absorción de energía (resonancia) Eabs M⊥ (t) XL ωL ω DESCRIPCIÓN EN COORDENADAS ROTATORIAS (ejes rotando en torno a Z con una frecuencia ω) r El comportamiento de M se describe con rlas mismas r r −1 ecuaciones, considerando un campo efectivo r B0 XL ωt r ω $ k γ r b . resonancia ZL=Z − Bef =B−ωγ k Z r Bef Y r B1 YL X r ⎛ ω⎞ r b = ⎜ Bo − ⎟ k γ⎠ ⎝ r B1 9 Para ω = ω L , b = 0 : la magnetización precede en el plano Z-Y, pasando a estados de energía más alta v M Y X -Y Z ω´ r B1 Y X Descripción cuántica de la magnetización Mi = Ii = tr(Ii ρ ) = ∑ mi Ii ρ mi = ∑∑ mi Ii m'i ρm'i mi mi mi m'i Caso puro: el sistema contiene un conjunto de espines nucleares iguales MATRIZ DENSIDAD: sistema I = 1/2 (base de estados de Iz) Elementos diagonales (POBLACIONES) Elementos no diagonales (COHERENCIAS) α β +1 / 2 −1 / 2 ρ= α +1 / 2 a c* β −1 / 2 c b • En el equilibrio a o = −b0 ≠ 0 • Evolución temporal a ( t ) = a o − (a o − a (0)) e − t / T1 • En el equilibrio • Evolución temporal b ( t ) = bo − (bo − b (0)) e − t / T1 c=0 c( t ) = c( 0) e −i E j − Ei 2 πh t e − t / T2 Descripción cuántica de la magnetización Muestra inhomogénea: existen N distintos subsistemas (de espines) independientes N N n n ρ = ∑ ρn ⇒ Mi = ∑tr[(Ii )n ρn ] Si los sistemas no son independientes, se describen en el espacio ⊗(Iz)n Mi = tr(Ii ρ ) = ∑ m1 ,m2 ,...,mN ∑ m'1 ,m'2 ,...,m' N m1, m2 ,...,mN ⊗ Ii m'1 , m'2 ,...,m'N ρ(m'1,m'2 ,...,m'N ) (m1 ,m2 ,...,mN ) (Iz )1 Caso I = 1/2: +1/2 -1/2 +1/2 -1/2 (Iz )1 ρ = (Iz )2 (Iz )2 +1/2 -1/2 +1/2 -1/2 ... ... ... ... (Iz )N +1/2 -1/2 (Iz )N +1/2 -1/2 ... ... ... ... ... Descripción cuántica de la magnetización 9Efecto de las microondas: en la resonancia, se reducen los elementos diagonales y se aumentan los no diagonales (transferencia de poblaciones a coherencias) con absorción de energía RESONANCIA Descripción cuántica a0 0 0 b0 MW a c* c b Magnetización v M -Y Z ω´ r B1 Y X La magnetización transversal depende de los elementos no diagonales de ρ M y = I y = tr(I y ρ ) = ∑∑ mi I y m'i ρm'i mi mi I y m'i ≠ 0 ⇔ mi ≠ m'i mi m'i Aparece magnetización transversal con absorción de energía (equilibrio absorción-relajación) RESONANCIA MAGNÉTICA NUCLEAR NMR pulsado 9 Pulsos “duros”: modifican la magnetización (matriz densidad) en tiempos mucho más rápidos que la relajación, y con anchura de banda capaz de excitar todo el sistema de espines Z B1 θ r B0 tp X(π/2) Z X Y • θ = γB1tp pulso π/2: transfiere poblaciones a coherencias y coherencias a poblaciones a c* c b X Y M • tp << T1, T2 X(π/2) M Z X(π) X Y M pulso π: intercambia coherencias y poblaciones d f* a c* f e c b X(π) b -c -c* a RESONANCIA MAGNÉTICA NUCLEAR NMR pulsado: experimento general Medida de la MAGNETIZACIÓN TRANSVERSAL tras la aplicación de una secuencia de pulsos a un sistema que parte del equilibrio P1 θ1 P2 τ1 θ2 Pn-1 θn-1 Pn τn-1 θn DETECCIÓN 9Se detecta la magnetización transversal M y = I y = tr(I y ρ ) = ∑∑ mi I y m'i ρm'i mi mi m'i detección de coherencias entre estados de (Iz)n 9Las coherencias oscilan a frecuencias dadas por la diferencia de energía entre los estados: necesitamos conocer las energía (Hamiltoniano) RESONANCIA MAGNÉTICA NUCLEAR Jean Jeener Richard Ernst Kurt Wüthrich RESONANCIA MAGNÉTICA NUCLEAR Hamiltoniano (de espín) nuclear: Zemann nuclear Chemical Shift Acoplamiento escalar Interacción dipolar r r r r r r r r r r r r (I ) (I ) (I ) r (I ) r H = µN gN B ⋅ ∑(I )n − µN gN B ⋅ ∑σ n ⋅ (I )n + ∑ Jn,n' (I )n (I )n' + (µN gN )2 ∑ n 3 n' − n 5 n' r r n n n, n' n, n' [ Interacciones a través de orbitales (conectividad a uno dos o tres enlaces) Interacciones entre espines distintos (los espines que interaccionan dejan de ser independientes y pueden presentar coherencias no nulas fuera del equilibrio Da lugar al efecto Overhauser nuclear (NOE), dependiente de r-6, sin conectividad ][ ] RESONANCIA MAGNÉTICA NUCLEAR Experimento “Decaimiento libre de la inducción” 9Se pueblan las coherencias entre estados del mismo núcleo 9Oscilan con una frecuencia que es la diferencia entre las energías de los dos estados 9El espectro (transformada de Fourier del FID) muestra picos que asignan las energías del Zeeman nuclear y Chemical Shift RESONANCIA MAGNÉTICA NUCLEAR 9Espectros multidimensionales: una correcta elección de las secuencias de pulsos permite detectar coherencias relacionadas con el acoplamiento escalar o con el NOE Ejemplos: 9En la diagonal, picos del experimento FID (asignación del núcleo concreto) 9Fuera de la diagonal, correlaciones entre núcleos con interacción escalar exclusivamente 9La intensidad de los picos no diagonales depende de la conectividad RESONANCIA MAGNÉTICA NUCLEAR Ejemplos: 9En la diagonal, picos del experimento FID (asignación del núcleo concreto) 9Fuera de la diagonal, correlaciones entre núcleos con NOE exclusivamente 9La intensidad de los picos no diagonales depende de la distancia 9Se pueden ajustar los tiempos para excitar distintas coherencias NOE RESONANCIA MAGNÉTICA NUCLEAR Ejemplos: 9Experimentos heteronucleares: se detectan coherencias entre núcleos de naturaleza distinta 9Se hace uso de las abundancias (relativas o enriquecidas) de 13C, 17O... RESONANCIA MAGNÉTICA NUCLEAR Estrategias para la resolución de estructuras: Desplazamientos químicos en los 20 aminoácidos RESONANCIA MAGNÉTICA NUCLEAR Estrategias para la resolución de estructuras: Espectro COSY de la alanina RESONANCIA MAGNÉTICA NUCLEAR Hζ3 Estrategias para la resolución de estructuras: NOE4 Interacciones en el anillo aromático del residuo de triptófano H Cβ 1 Hε3 Cζ3 2 Hη2 Cε3 Cη2 Cδ2 Cζ2 Cγ Cε2 3 Hζ2 δ1 Nε1 C NOE1 H Hε1 NOE3 Hδ1 4 NOE2 1 2 3 COSY TOCSY ASIGNACIÓN SECUENCIAL DE PÉPTIDOS Y PROTEÍNAS Para la asignación secuencial se hace uso del efecto NOE entre diferentes núcleos cercanos en el espacio: Espectro 2D-NOESY. A principio de los 80, analizando las estructuras de un conjunto de proteínas, Kurt Wüthrich y cols. observaron que para la mayoría de los residuos las distacias entre protones de residuos adyacentes es en muchos casos menor de 5 Å. Por tanto, debe poder observarse casi siempre un NOE (NOE secuencial) entre dichos núcleos. El método de asignación secuencial propuesto por Wüthrich se basa en relacionar los sistemas de spin previamente identificados mediante los NOEs secuenciales en el espectro NOESY. Una alternativa a este protocolo, propuesta por el grupo de Walter Englander, se llama asignación dirigida por la cadena principal, que relaciona segmentos de la cadena principal mediante el uso combinado de NOEs secuenciales y de NOEs característicos de las diferentes estructuras secundarias. Generalmente se utiliza una combinación de ambos métodos. DISTANCIAS CORTAS CARACTERÍSTICAS DE LAS DIFERENTES ESTRUCTURAS SECUNDARIAS Lámina β paralela Lámina β antiparalela Hélice α Giro β tipo I Giro β tipo II DISTANCIAS CORTAS CARACTERÍSTICAS DE LAS DIFERENTES ESTRUCTURAS SECUNDARIAS Distancias cortas (< ≈2.5 Å) generalmente darán lugar a un NOE fuerte (pico de cruce intenso en el NOESY). Distancias intermedias (≈2.5-3.5 Å) dan un NOE intermedio. Distancias largas (≈ 3.5-5.0 Å) darán un NOE débil. ESQUEMAS DE NOEs SECUENCIALES Y CARACTERÍSTICOS DE ESTRUCTURAS SECUNDARIAS Se suelen realizar representaciones esquemáticas de los NOEs indicando su intensidad mediante el grosor de las líneas. IDENTIFICACIÓN DE LOS TIPOS DE NOEs Antes de proceder a la asignación secuencial, es necesario discernir entre NOEs intrarresiduo y NOEs interresiduo. Para ello se superponen los espectros NOESY y TOCSY y se marcan los NOEs intrarresiduo. Los NOEs restantes son interresiduo (secuenciales ó a larga distancia) J1 L5 E1 G18 V17 U1 E2 L1 A2 T1 W13 U3 A3 L4 A1 T1 L2 L3 U2 N11 T2 NOEs αN(i - i+1) Generalmente se comienza relacionando pares de sistema de spin que correspondan a pares de residuos únicos en la secuencia Ej. N11-A12 ó L19-K20(tipo U). Esto nos permite identificar A12, L19 y K20. J1 L5 E1 G18 V17 U1 E2 L1 A2 T1 W13 U3 A3 L4 A1 T1 L2 L3 U2 N11 T2 NOEs βN(i - i+1) Generalmente los NOEs αN(i-i+1) se acompañan de NOEs βN(i-i+1) entre los mismos residuos. N11 Hβ A12 HN NOEs NN(i - i+1) Se pueden observar también NOEs NN(i - i+1) L19 K20 N11 A12 A partir de los pares de residuos identificados podemos ir extendiendo la asignación a lo largo de la secuencia. Sistemas de spin: N11 A3 J1 L5 U2 E2 V17 G18 L2 U3 Secuencia: N11 A12 W13 L14 R15 E16 V17 G18 L19 K20 W13 L5 E1 G18 V17 U1 E2 L1 A2 T1 W13 U3 A3 L4 A1 T1 L2 L3 U2 N11 T2 Resto de la asignación secuencial: Sistemas de spin: U4 T1 L3 T2 L4 E1 A2 A1 L1 U1 Secuencia: K1 T2 L3 T4 L5 E6 A7 A8 L9 R10 Sistemas de spin: J2 A3 J1 L5 U2 E2 V17 G18 L2 U3 Secuencia: N11 A12 W13 L14 R15 E16 V17 G18 L19 K20 W13 L14 L9 E6 G18 R10 E16V17 A7 T2 W13 R15 L5 A8A12 L19 T2 L3 N11 T4 K20 NOEs NN(i - i+1) L19 G18 K20 E16 V17 R15 N11 A8 E6 A12 A7 W13 L9 A8 R10 R15T4 L5 L3 W13 Hε3 W13 HN W13 Hη3 ESQUEMA DE NOEs K1 T2 L3 T4 L5 E6 A7 A8 L9 R10 N11 A12 W13 L14 R15 E16 V17 G18 L19 K20 dαN(i,i+1) dβN(i,i+1) dNN(i,i+1) dαN(i,i+2) dαN(i,i+3) dNN(i,i+2) dαβ(i,i+3) A esta información se suelen añadir las constantes de acoplamiento JHN-Hα e información sobre la protección de cada HN frente al intercambio con deuterio. LISTA DE ASIGNACIÓN COMPLETA. Uniendo la información de la asignación secuencial con la identificación de las señales de cada sistema de spin, se obtiene la asignación completa que se suele expresar en forma de lista. Sec. K1 T2 L3 T4 L5 E6 A7 A8 L9 R10 N11 A12 W13 NH 8.67 8.57 7.78 8.43 8.38 7.92 8.27 8.56 8.16 8.26 8.14 8.54 αH 4.17 4.43 4.49 4.38 4.20 3.94 4.10 4.17 4.15 4.10 4.52 4.16 4.33 L14 R15 8.55 7.84 3.83 4.22 E16 V17 G18 L19 K20 8.11 7.89 7.69 7.89 7.62 4.12 4.02 3.86,4.00 4.40 4.23 βH 1.97,1.97 4.20 1.71,1.71 4.52 1.74,1.74 2.15,2.04 1.54 1.57 1.87,1.48 2.01,1.96 2.83,2.96 1.61 3.37,3.54 Otros γCH2 1.54,1.54 δCH2 1.77,1.77 εCH2 3.07,3.07 ζNH3 7.70 γCH3 1.28 γOH γCH 1.73 δCH3 0.96, 0.91 γCH3 1.33 γOH γCH 1.64 δCH3 0.97, 0.93 γCH2 2.46 γCH 1.80 δCH3 0.87, 0.87 γCH2 1,83,1.67 δCH2 3.23,3.23 εNH 7.24 δNH2 6.98, 7,72 ε1NH 9.83 δ1CH 7.15 ε3CH 7.41 η1CH 7.15 ζ2CH 7.03 ζ3CH 7.52 2.00,1.40 γCH 2.07 δCH3 0.98, 0.94 1.96,1.82 γCH2 1.65,1,65 δCH2 3.25,3.25 εNH 7.40 2.19,2.19 γCH2 2.46,2.52 1.89 γCH3 0.56,0.47 1.61,1.61 γCH 1.61 δCH3 0.89, 0.89 1.83,1.67 γCH2 1.42,1.42 δCH21.72,1.72 εCH2 3.02,3.02 ζNH3 7.65 El banco de datos de desplazamientos químicos de RMN BioMagResBank ( BMRB) http://www.bmrb.wisc.edu/index.html Contiene los desplazamientos químicos de unos 2900 péptidos, proteínas, DNA y RNA, además de parámetros relacionados con RMN, condiciones de estudio, datos de secuencias, datos cinéticos y termodinámicos obtenidos o relacionados con la RMN. ESTRUCTURAS DE PROTEÍNAS RMN Difracción de rayos X La estructura por RMN no requiere obtención de monocristales FENÓMENO DE LA RESONANCIA MAGNÉTICA (caso espín electrónico) Sistema asilado con momento angular J (degenración 2J+1) Razón giromagnética γ Campo magnético estático Momento magnético r B0 = B0 k̂ (Descenso de simetría O(3) → C∞) r r r r H = − B0 ⋅ µ = − γ B0 ⋅ J = −γ B0 J z -|M⟩ 2J+1 r µ =γ J r E (M ) = E0 − γ B0 M Transiciones dipolares magnéticas | ∆M | = ±1 hν = γBo Resonancia hν |M⟩ eh Electrones µB = Núcleos µn = µ B ⎜ 2mc = 13.996 GHz/T ⎛ m ⎜ mp ⎝ ⎞ 1 ⎟= µ ⎟ 1836.11 B ⎠ γ = -g µ B γ = -g N µ N De acuerdo con hν = γBo la resonancia se puede obtener experimentalmente variando la frecuencia, ν, o el campo estático, Bo. RESONANCIA PARAMAGNÉTICA ELECTRÓNICA Evolución de la magnetización bajo campo estático r B0 = B0 k̂ r B0 ZL r M (t) YL M⊥ (t) γ = µN gN r r M= I r r r dM = −γ M ∧ Bo dt ωL = γ Bo XL M x (t ) = M ⊥ (0 ) cos (ω L t ) dM x = ω L BM y dt dM y = −ω L BM x dt dM z =0 dt Frecuencia de Larmor (propia de cada núcleo) La magnetización realiza M y (t ) = M ⊥ (0 ) sin (ω L t ) una precesión en torno al M z (t ) = M z (0 ) campo magnético estático RESONANCIA PARAMAGNÉTICA ELECTRÓNICA M x (t ) = e − t / T2 La precesión de la magnetización M ⊥ (0 ) cos(ω L t ) relaja al estado de equilibrio a M y (t ) = e M ⊥ (0 ) sin (ω L t ) eq eq M z (t ) = M z − e −t / T1 M z − M z (0 ) − t / T2 [ ] través de la interacción con los modos de energía de la red Microondas: introducen un campo transversal, débil y variable [ r r r B1 (t ) = B1 cos(ϖ t ) i + sen(ϖ t ) j r B0 ZL r M (t) ] Cuando ω = ωL, el campo de microondas se acopla a la magnetización, y se produce absorción de energía (resonancia) YL M⊥ (t) Eabs XL ωL ω RESONANCIA PARAMAGNÉTICA ELECTRÓNICA DESCRIPCIÓN EN COORDENADAS ROTATORIAS (ejes rotando en torno a Z con una frecuencia ω) El comportamiento de r M se describe con rlas mismas r r −1 ecuaciones, considerando un campo efectivo r B0 XL ωt r ω $ k γ r b . resonancia ZL=Z − Bef =B−ωγ k Z r Bef Y r B1 r B1 YL X r ⎛ ω⎞ r b = ⎜ Bo − ⎟ k γ⎠ ⎝ 9 Para ω = ω L , b = 0 : la magnetización precede en el plano Z-Y, pasando a estados de energía más alta v M Z ω´ Y Y X -Y r B1 X RESONANCIA PARAMAGNÉTICA ELECTRÓNICA DESCRIPCIÓN CUÁNTICA r r La magnetización M ∝ tr ( ρ S ) es el la magnitud macroscópica asociada al observable espín α β +1 / 2 −1 / 2 MATRIZ DENSIDAD: sistema S = 1/2 Elementos diagonales (POBLACIONES) Elementos no diagonales (COHERENCIAS) ρ= α +1 / 2 a c* β −1 / 2 c b 1 I 2 + • En el equilibrio a o = −b0 ≠ 0 • Evolución temporal a ( t ) = a o − (a o − a (0)) e − t / T1 • En el equilibrio • Evolución temporal b ( t ) = bo − (bo − b (0)) e − t / T1 c=0 c( t ) = c( 0) e −i E j − Ei 2 πh t e − t / T2 RESONANCIA PARAMAGNÉTICA ELECTRÓNICA Hamiltoniano de espín: r ~ r r ~ r r ~ r r ~ r r r H = S ⋅ D ⋅ S + µB B ⋅ g ⋅ S + S ⋅ A ⋅ I + I ⋅ Q ⋅ I − µ N g N B ⋅ I ZFS Ze HF 9Interacción en general anisótropa 9Fuertemente influido por la configuración electrónica y el entorno (no caracteriza directamente en centro) 9D, tensor de traza nula 9Sólo se da para S > ½ (algunos metales, birradicales...) cuadrupolar Zn 9Interacción con el propio núcleo del átomo 9Interacción con núcleos próximos, por conectividad o dipolar RESONANCIA PARAMAGNÉTICA ELECTRÓNICA INFORMACIÓN OBTENIBLE DE UN ESPECTRO DE EPR IDENTIFICACIÓN DE ESPECIES Espín electrónico (Valencia) Espín nuclear (Elemento) PARÁMETROS ESPECTROSCÓPICOS Tensor g Mapa Tensor hiperfino distribución ZFS electrónica shf ..... DETERMINACIÓN ENTORNOS LOCALES Simetría local (anisotropías) INTERACCIONES SISTEMA - BAÑO Tiempos de relajación Procesos Reorientación dinámicos Difusión INTERACCIONES MAGNÉTICAS Interacciones entre especies magnéticas (pares, oligómeros) Sistemas concentrados (dipolar, canje) SONDAS PARAMAGNÉTICAS PARA Transiciones de fase estructurales Orden en CL Atrapamiento de productos metaestables .... ⇒ ⇒ …… RESONANCIA PARAMAGNÉTICA ELECTRÓNICA Caso S = 1/2, I = 1/2, g isótropo en interacción hiperfina: H = µ B g BS Z + a I z S z + 1 2 H0 M ,m (M = −S , S m = −I ,I ) a (S + I − + S − I + ) − g N µ N BI z H1 E (1) (M , m ) = gµ B BM + aMm − g N µ N Bm EPR regla de selección |∆M| = ±1, |∆m| = 0 B0 = hν gµ B a=0 B0 = hν = gµBB + am hν a Bm = − m gµ B gµ B 1 2I + 1 ∑ Bm = m a gµ B Espectro de EPR 2I+1 líneas B ∆B = Bm +1 − Bm = hν gµ B a gµ B RESONANCIA PARAMAGNÉTICA ELECTRÓNICA Caso S = 1/2, I = 1/2, g isótropo en interacción hiperfina: Iz +1/2 -1/2 -1/2 +1/2 α -1/2 +1/2 α -1/2 -1/2 β +1/2 -1/2 β +1/2 α : Sz = + 1/2 β : Sz = - 1/2 +1/2 En el experimento contínuo se detectan las transiciones permitidas (coherencias dentro de La misma variedad de espín electrónico) EPR regla de selección |∆M| = ±1, |∆m| = 0 RESONANCIA PARAMAGNÉTICA ELECTRÓNICA Interacción hiperfina con varios núcleos: Anión benceno H Anión butadieno H H H H CH2 C C CH2 H H - H 6 4 4 1 1 0.5 mT 1 mT RESONANCIA PARAMAGNÉTICA ELECTRÓNICA Ensanchamiento Inhomogéneo (interacción hiperfina, distintos centros, relajación inhomogénea...) Anchura de línea homogénea ωL ω0 f(ωL - ω0) 2T2−1 ω ω Solución: técnicas de pulsos permiten inducir coherencias entre transiciones ocultas en el ensanchamiento (en especial hiperfinas) y detectarlas P1 θ1 P2 τ1 θ2 Pn-1 θn-1 Pn τn-1 θn t DETECCIÓN Producción Fotosintética de NADPH NADP+ PSI FLD FNR NADPH PSI FLDrd + FNRox FLDsq + FNRsq FLDrd + FNRsq FLDsq + FNRrd FNRrd + NADP+ FNRox + NADPH + H+ Estados Redox del Anillo de Isoaloxazina en Flavoproteínas 2e- + 2H+ R CH3 8 1e- + 1H+ 7 9 10 -1 6 .5 4 CH3 N N O 2 3 N N H + R 1e- + 1H+ O R H N N H CH3 8 9 7 6 CH3 N 10 5 N N 1 4 Semiquinona neutra O 2 CH3 8 3N 7 6 1H+ H 9 CH3 10 5 4 N O O 1e- CH3 8 9 7 6 CH3 10 -1 .5 4 N Reducido N N 1e- O 2 3 N H O Semiquinona aniónica + 2H+ O 2 3 N H H R Oxidado 1 EPR DE FLAVOPROTEÍNAS ¿Por qué? 9Estado semiquinona, paramagnético (S = 1/2) 9Radical molecular: ¿distribución electrónica? 9Estructura hiperfina: Interacción entre el espín del núcleo y el del electrón Presencia del electrón en las Desdoblamiento hiperfino proximidades del núcleo “A knowledge of sites of high spin-density is of mechanistic importance in that it suggests potential sites where one-electron transfer could occur in flavoenzyme-catalysed reactions involving the semiquinone intermediate.” D. E. Edmondson, “Electron-spin resonance studies on flavoenzymes”, Biochem. Soc. Trans. 13, 593 (1985). CW-EPR de flavoproteínas •Semiquinona neutra •Semiquinona aniónica 1 mT g = 2.005 pequeñas diferencias en anchura: identificación de la forma de flavina 9Diferencias de anchura por intercambios: 1H 14N 2H (D) 15N 9Anchuras debidas a desdoblamiento hiperfino no resuelto 9Desdoblamientos resueltos el flavinas modelo Técnicas avanzadas Permiten la detección de desdoblamientos hiperfinos no resueltos ENDOR (doble resonancia electrón-núcleo) Metilo en C(8) R H en C(6) CH3 8 7 9 N 10 6 5 N CH3 H N 1 2 3 + 4 O H O N H Acoplamientos Hiperfinos de Algunos Protones de la Flavina Obtenidos Mediante ENDOR Flavoproteina semiquinona CH3(8) (+ 0.1 MHz) A⊥ A// Flavodoxina 7.91 Ferredoxina-NADP+ reductasa Colesterol oxidasa CH(6) a a 9.46 8.42 5.53 7.47 9.41 8.12 5.11 10.30 12.11 10.90 9.0 TÉCNICA DE EPR PULSADO ESEEM ESEEM bidimensional: HYSCORE F1:[MHz] 30 H en C(5) 25 20 15 N(10) 10 5 N(1), N(3) 0 -30 -20 -10 0 F2:[MHz] 10 20 30 R CH3 8 7 CH3 9 N 10 6 5 N + H N 1 4 O O 2 3 N H Parámetros de Interacción de los Núcleos 1H y de la Isoaloxazina Obtenidos por HYSCORE Flavoproteina semiquinona Posición atómica Colesterol Oxidasa H(5) N(A)a N(B)a N(10) FNR H(5) N(A)a N(B)a N(10) Flavodoxina H(5) N(A)a N(B)a N(10) a (MHz) 1.9 ± 0.2b 0.7 ± 0.2b 11.7 ± 1.0 T (MHz) K2(3+ η) (MHz2) 14N η < 0.3 2.3 ± 0.2 < 0.3 2.1 ± 0.1 9.0 ± 1.1 n.d. 0.5 ± 0.1 n.d. n.d. -18.7 1.2 0.7 11.7 ± ± ± ± 0.8 0.2b 0.3b 1.0 8.2 < < 9.0 ± 0.4 0.3 0.3 ± 1.1 2.2 ± 0.1 2.4 ± 0.2 n.d. 0.5 + 0.1c n.d. n.d. -18.8 1.3 0.8 11.7 ± ± ± ± 0.6 0.3b 0.2b 1.0 8.7 < < 9.0 ± 0.3 0.3 0.3 ± 1.1 2.2 ± 0.1 2.1 ± 0.1 n.d. 0.5 ± 0.1c n.d. n.d. a Estos nitrógenos corresponden a N(1) o N(3) del anillo de flavina. b Sólo se ha determinado el valor absoluto. c Estimado de los datos obtenidos para colesterol oxidasa. R CH3 Posiciones del Anillo de Isoaloxazina en Flavoproteínas Caracterizadas Mediante ESEEM e HYSCORE 8 7 9 N 10 6 . CH3 5 N 1 O 2 3 4 N N + H O H Semiquinona Neutra R CH3 9 N 10 6 . 8 7 CH3 5 N 1 O 2 3 4 N N H O Semiquinona Aniónica R CH3 Posiciones del Anillo de Isoaloxazina en Flavoproteínas Caracterizadas Mediante ENDOR, ESEEM e HYSCORE 8 7 9 N 10 6 .+ 5 N CH3 H N 1 O 2 3 4 N H O H Semiquinona Neutra R CH3 9 N 10 6 . 8 7 CH3 5 N 1 O 2 3 4 N N H H O Semiquinona Aniónica 9Mediante el uso combinado de las técnicas avanzadas de EPR ESEEM y ENDOR disponemos de un mapa del espín a través de las interacciones hiperfinas 9Rasgos del mapa: 9Elevada presencia del espín en el anillo central (“pirazinoide”), sobre todo en el N(5) 9Moderada presencia en el anillo de la izquierda (“bencenoide”), y muy baja en el de la derecha (“pirimidinoide”) R H3C H3C N N O N H N H O Adaptado de D. E. Edmondson, “Electron-spin resonance studies on flavoenzymes”, Biochem. Soc. Trans. 13, 593 (1985). 9Uso del mapa: Elucidación del mecanismo de reacción (¿por dónde salta el electrón?) 9Sensibilidad al entorno en la proteína 9Efecto de la entrada del sustrato CÁLCULOS CUÁNTICOS DE LA FLAVINA 9Programa Gaussian 9Cálculo de la distribución de espín en el estado semiquinona (capa abierta) 9Elección del funcional y de las bases de cálculo 9Elección del sistema a calcular Lumiflavina, neutra y aniónica CH3 CH3 8 7 9 6 CH3 H N 10 5 N CH3 N 1 O H 2 3 4 CH3 O 8 7 N H 9 N 10 N 1 6 5 N 4 CH3 H O 2 3 O N H Resultados de los cálculos para el orbital ocupado por el electrón desapareado Comparación cálculos-experimentos Calculated Data ATOM a (MHz) T (MHz) H(5) -21.4 H(6) Experimental Data η a (MHz) T (MHz) 11.0 -18.8 9.0 -6.4 2.1 -5.7 n.d. H(met8) 9.1 0.6 8.4 0.5 H(met10) 9.8 1.0 12.5 N(1) 0.1 0.3 0.7 0.8 0.8 e n.d. b N(3) -1.0 0.3 0.8 0.2 1.3 e n.d. b N(5) 13.6 14.6 0.7 0.3 22.1 N(10) 7.6 7.6 0.8 0.2 11.7 K (MHz) d g K (MHz) η b, c 1.15 d 0.8 e, f 0.5 0.7-0.9 - - 9.0 1.3 e e, f n.d. b h - Neutra Calculated Data Experimental Data η ATOM a (MHz) T (MHz) H(6) -11.3 2.7 -9.0 n.d. H(met8) 13.9 0.6 10.9 0.6 N(1) 0.7 1.1 0.7 0.8 0.7 d n.d. b 0.7-0.9 N(3) -1.3 0.1 0.8 0.2 1.9 d n.d. b 0.8 N(5) 16.0 17.4 0.7 0.3 20.4 N(10) 8.2 5.2 0.8 0.2 11.7 K (MHz) Aniónica a (MHz) e T (MHz) K (MHz) η b, c d - - 9.0 1.3 d n.d. 0.5 b d f n.d. b RESULTADOS DE LA COMPARACIÓN 9Los cálculos reflejan bastante bien (desviación < 20%) los valores de desdoblamiento hiperfino, tanto la parte isótropa como la anisótropa 9Los cálculos predicen correctamente las diferencias entre flavina neutra y aniónica RESULTADOS ADICIONALES DEL CÁLCULO 9El orbital ocupado por el electrón desapareado tiene un marcado carácter π. 9Además de en los átomos con densidad detectada (a través de la interacción hiperfina), hay una elevada densidad de espín en O(2), O(4) y C(4a) POSIBILIDADES DE LOS CÁLCULOS 9Disponer del mapa completo de densidades de espín 9Calcular el efecto del entorno del anillo dentro de la proteína Resultados de los cálculos para el orbital ocupado por el electrón desapareado considerando el entorno (R-X)