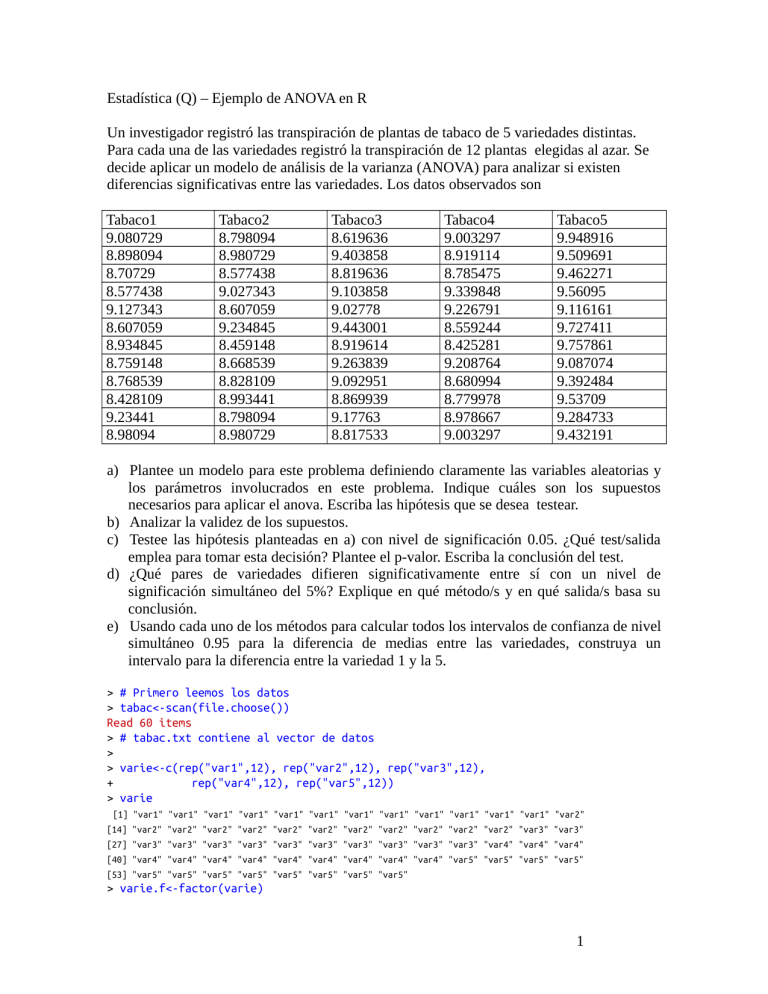
Estadística (Q) – Ejemplo de ANOVA en R Un investigador registró las transpiración de plantas de tabaco de 5 variedades distintas. Para cada una de las variedades registró la transpiración de 12 plantas elegidas al azar. Se decide aplicar un modelo de análisis de la varianza (ANOVA) para analizar si existen diferencias significativas entre las variedades. Los datos observados son Tabaco1 9.080729 8.898094 8.70729 8.577438 9.127343 8.607059 8.934845 8.759148 8.768539 8.428109 9.23441 8.98094 Tabaco2 8.798094 8.980729 8.577438 9.027343 8.607059 9.234845 8.459148 8.668539 8.828109 8.993441 8.798094 8.980729 Tabaco3 8.619636 9.403858 8.819636 9.103858 9.02778 9.443001 8.919614 9.263839 9.092951 8.869939 9.17763 8.817533 Tabaco4 9.003297 8.919114 8.785475 9.339848 9.226791 8.559244 8.425281 9.208764 8.680994 8.779978 8.978667 9.003297 Tabaco5 9.948916 9.509691 9.462271 9.56095 9.116161 9.727411 9.757861 9.087074 9.392484 9.53709 9.284733 9.432191 a) Plantee un modelo para este problema definiendo claramente las variables aleatorias y los parámetros involucrados en este problema. Indique cuáles son los supuestos necesarios para aplicar el anova. Escriba las hipótesis que se desea testear. b) Analizar la validez de los supuestos. c) Testee las hipótesis planteadas en a) con nivel de significación 0.05. ¿Qué test/salida emplea para tomar esta decisión? Plantee el p-valor. Escriba la conclusión del test. d) ¿Qué pares de variedades difieren significativamente entre sí con un nivel de significación simultáneo del 5%? Explique en qué método/s y en qué salida/s basa su conclusión. e) Usando cada uno de los métodos para calcular todos los intervalos de confianza de nivel simultáneo 0.95 para la diferencia de medias entre las variedades, construya un intervalo para la diferencia entre la variedad 1 y la 5. > # Primero leemos los datos > tabac<-scan(file.choose()) Read 60 items > # tabac.txt contiene al vector de datos > > varie<-c(rep("var1",12), rep("var2",12), rep("var3",12), + rep("var4",12), rep("var5",12)) > varie [1] [14] [27] [40] [53] "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var5" "var1" "var2" "var3" "var4" "var1" "var2" "var3" "var5" "var1" "var2" "var4" "var5" "var1" "var3" "var4" "var5" "var2" "var3" "var4" "var5" > varie.f<-factor(varie) 1 > levels(varie.f) [1] "var1" "var2" "var3" "var4" "var5" > # El vector "varie" contiene las variedades (o grupos) a las que corresponden > # las observaciones. La instruccion "factor" le dice al R que guarde en varie.f > # las palabras (o numeros) del vector varie y que las utilice como codigos > # que indican la pertenencia a distintas variedades (o grupos). > > ####################################################################### > # otra manera de leer los datos > ####################################################################### > # cuando los datos se guardan en un archivo con dos columnas, una > # con los datos y la otra con el grupo al que pertenecen, se pueden > # cargar al R de la siguiente forma, ejemplificamos con los datos > # de un ejercicio de la practica > # cereales<-read.table(file.choose(),header=T) #y buscar el archivo "cereales.txt" en la computadora > # attach(cereales) #para convertir a las dos columnas del archivo en los vectores "tiamina" y "cereal" > # cereal.f<-factor(cereal) #convierte al vector "cereal" en un factor > # Seguimos con el ejercicio # En papel, escriba las variables, el modelo, los supuestos y defina # los parametros y las hipotesis que testea anova. En R, > salida<-aov(tabac~varie.f) > summary(salida) Df Sum Sq Mean Sq F value Pr(>F) varie.f 4 3.563 0.8908 14.26 4.78e-08 *** Residuals 55 3.436 0.0625 --Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 > names(salida) [1] "coefficients" "residuals" "effects" "rank" "fitted.values" [6] "assign" "qr" "df.residual" "contrasts" "xlevels" [11] "call" "terms" "model" > # vemos que el objeto producido por la instruccion "aov" es una lista > # que tiene muchos componentes adentro. La instruccion summary(aov) muestra > # en pantalla los que son relevantes para testear la hipotesis de igualdad > # de medias > qf(0.95,df1=4,df2=55) #cuantil para hacer el test a mano [1] 2.539689 > # El p-valor del test de F para la igualdad de medias resulta ser > # 4.78*10^(-8) < 0.05, luego rechazamos la hipotesis nula de > # igualdad de medias y concluimos que tenemos evidencia suficiente > # en la muestra para afirmar, a nivel 0.05, que las transpiraciones > # medias de al menos dos variedades de tabaco son distintas entre si. > # En un item posterior veremos cuales son esas variedades. > > boxplot(tabac~varie.f,main ="boxplot de la transpiracion para distintos tabacos") > boxplot(residuals(salida),main="boxplot de los residuos") > qqnorm(residuals(salida)) # otra instrucción que proporciona graficos, hay que apretar 2 # el <Enter> para ver los siguientes graficos > plot(salida) <Enter> para ver el próximo gráfico: # Al mirar los boxplots de las transpiraciones de las 12 plantas de # tabaco de cada variedad EN LA MISMA ESCALA vemos que las variabilidades # de los cinco grupos de observaciones no parecen diferir entre si. Con # respecto al centro de cada conjunto de datos, vemos que la mediana de # la variedad 5 es mas alta que el resto, seguida por la de la variedad 3. # Para ver si esta diferencia observada en la muestra es suficiente como # para implicar una diferencia entre las transpiraciones medias poblacionales # es que hicimos el test F, en el que vimos que al menos dos medias poblacionales # son diferentes. Por otro lado, los cinco conjuntos de datos no muestran # severos apartamientos de la normalidad. Sin embargo, como cada boxplot esta # basado en solo 12 datos, para tener una vision mas clara de la validez de los # supuestos, estudiamos los residuos, que son la resta entre cada observacion y # la media muestral del grupo al que pertenece. Los residuos se guardan en el # vector residuals(salida). Tanto del qqplot como del boxplot # de los 60 residuos concluimos que no pareciera haber razones para sospechar # de la normalidad de los datos. Otro grafico que permite ver la homoscedasticidad # (igualdad de varianzas) y un poco la normalidad (sobre todo la asimetria o la # existencia de observaciones atipicas) es el grafico de residuos (o residuos 3 # estandarizados), es decir, divididos por el valor de sigma estimado versus el # grupo al que pertenecen las observaciones. En este caso tampoco se ven # alejamientos de la normalidad. # De paso, una pregunta, cuales son los estimadores de los 6 parametros # del modelo de anova? (Primero: cuales son los 6 parametros del modelo?) # # # # # # Las 5 medias poblacionales son estimadas por las medias muestrales de cada variedad (o grupo). Observemos que esta informacion no aparece directamente en la salida del comando aov del R (ni en la tabla de anova clasica). Por otro lado, la varianza poblacional se estima con un promedio ponderado de las varianzas muestrales de cada grupo. Este dato en que lugar de la tabla anova se puede leer? # Para calcular las medias y varianzas muestrales por variedad "a mano" > medias<-tapply(tabac,varie.f,mean) > medias var1 var2 var3 var4 var5 8.841995 8.829464 9.046606 8.909229 9.484736 > # vemos que las medias se comportan de igual modo que las medianas que analizamos > # en los boxplots > varianzas<-tapply(tabac,varie.f,var) > varianzas var1 var2 var3 var4 var5 0.05912479 0.05037476 0.06259336 0.07631125 0.06398942 > # las varianzas muestrales no parecen ser muy diferentes, mas abajo > # lo testeamos > # reconstruimos el S_p al cuadrado, o sea la estimacion de sigmacuadrado bajo el > # modelo a partir de las varianzas muestrales. > sum(varianzas)*11/55 [1] 0.06247872 > # En que parte de la tabla anova esta esta informacion? > # Tambien podemos calcular las medias usando la instruccion aov > model.tables(salida,"means") Tables of means Grand mean 9.022406 varie.f varie.f var1 var2 var3 var4 var5 8.842 8.829 9.047 8.909 9.485 # primero testeamos la igualdad de varianzas # de las observaciones de cada grupo con el test de levene que no es 4 # # # # muy potente pero es resistente a apartamientos de la normalidad (es decir, funciona bien aun cuando las observaciones no son normales) Una pregunta, que hipotesis testea este test? Otra mas: a que nivel conviene llevarlo a cabo? Por que no es 0.05? > install.packages("lawstat") > library("lawstat") > levene.test(tabac,varie.f) modified robust Brown-Forsythe Levene-type test based on the absolute deviations from the median data: tabac Test Statistic = 0.1292, p-value = 0.9711 # como el p-valor da 0.9711 > 0.20, no tenemos evidencia para rechazar # la hipotesis de homoscedasticidad, es decir, no tenemos razones para # dudar de la igualdad de varianzas # # # # # habiendo pasado el test de igualdad de varianzas, testeamos la hipotesis de normalidad de las observaciones a traves del test de shapiro-wilks aplicado a los residuos. Una pregunta, que hipotesis testea este test? Otra mas: a que nivel conviene llevarlo a cabo? Por que no es 0.05? > shapiro.test(salida$res) Shapiro-Wilk normality test data: salida$res W = 0.9811, p-value = 0.4761 # el p-valor del test de shapiro da 0.4761 > 0.20, luego no rechazamos # H0 en base a la muestra dada, es decir, no tenemos razones para dudar # de la normalidad de las observaciones # # # # # # # para quedarnos tranquilos, una vez que nuestros datos pasaron el test de normalidad, podemos aplicar el test de bartlett para la igualdad de varianzas, que necesita que los datos sean normales para funcionar. Cuando los datos son normales, el test de bartlett es mas potente que el de levene Una pregunta, que hipotesis testea este test? Otra mas: a que nivel conviene llevarlo a cabo? Por que no es 0.05? > bartlett.test(tabac,varie.f) Bartlett test of homogeneity of variances data: tabac and varie.f Bartlett's K-squared = 0.4754, df = 4, p-value = 0.9758 # El p-valor del test es 0.9758 > 0.20 por lo que confirmamos la conclusion # de levene: no tenemos razones para dudar de la igualdad de varianza de # las poblaciones de las que provienen los datos. 5 # # # # Ya vimos que al menos dos medias poblacionales difieren entre sí. Las instrucciones que siguen permiten realizar las comparaciones entre pares de medias, a posteriori de haber detectado que existen diferencias estadísticamente significativas. > install.packages("asbio") > library("asbio") # Cuantas comparaciones hacemos? > choose(5,2) #numero de comparaciones a hacer, choose calcula el numero combinatorio [1] 10 ############################################### # Intervalos de confianza de bonferroni ############################################### # Para que parametros se calculan los intervalos? > int.bonf<-pairw.anova(y = tabac, x = varie.f, method = "bonf") > plot(int.bonf,type="2",las=1) > int.bonf 95% Bonferroni confidence intervals muvar1-muvar2 muvar1-muvar3 muvar2-muvar3 muvar1-muvar4 muvar2-muvar4 muvar3-muvar4 muvar1-muvar5 muvar2-muvar5 muvar3-muvar5 muvar4-muvar5 # # # # # # # # # # Diff Lower Upper Decision Adj. p-value 0.01253 -0.28592 0.31098 FTR H0 1 -0.20461 -0.50306 0.09384 FTR H0 0.498824 -0.21714 -0.51559 0.08131 FTR H0 0.378412 -0.06723 -0.36568 0.23122 FTR H0 1 -0.07977 -0.37822 0.21869 FTR H0 1 0.13738 -0.16107 0.43583 FTR H0 1 -0.64274 -0.94119 -0.34429 Reject H0 1e-06 -0.65527 -0.95372 -0.35682 Reject H0 0 -0.43813 -0.73658 -0.13968 Reject H0 0.00072 -0.57551 -0.87396 -0.27706 Reject H0 6e-06 las=1 es una opcion grafica para que los rotulos del eje y aparezcan horizontales y sean mas faciles de leer FTR H0: fail to reject H0 si el pvalor ajustado es menor que el alfa = 0.05, rechazamos la hipotesis nula de dicha comparacion de pares de medias Una pregunta, que hipotesis testearia cada uno de estos tests? Recordar que al utilizar este procedimiento (o el de tukey, o algunos otros disponibles) las conclusiones sobre las comparaciones de a pares se se extraen a nivel conjunto, es decir con nivel de confianza sobre TODOS los intervalos igual a 0.05. # Que pares de medias difieren entre si a nivel conjunto 0.05, si utilizamos el # metodo de Bonferroni para sacar las conclusiones? Asegurese de detectar los 4 pares. > choose(5,2) #numero de comparaciones a hacer [1] 10 6 > 1-0.05/20 [1] 0.9975 > qt(0.9975,df=55) #percentil para armarlo a mano [1] 2.924701 # Calcule a mano el IC para mu1 - mu5 segun bonferroni # y chequeelo con el calculado por R. ############################################### # Intervalos de confianza de tukey ############################################### > int.tuk<-pairw.anova(y = tabac, x = varie.f, method = "tukey") > plot(int.tuk,type="2",las=1) > int.tuk 95% Tukey-Kramer confidence intervals muvar1-muvar2 muvar1-muvar3 muvar2-muvar3 muvar1-muvar4 muvar2-muvar4 Diff Lower Upper 0.01253 -0.27527 0.30033 -0.20461 -0.49241 0.08319 -0.21714 -0.50494 0.07066 -0.06723 -0.35503 0.22057 -0.07977 -0.36756 0.20803 Decision Adj. p-value FTR H0 0.999947 FTR H0 0.27725 FTR H0 0.223187 FTR H0 0.964232 FTR H0 0.934857 7 muvar3-muvar4 0.13738 -0.15042 muvar1-muvar5 -0.64274 -0.93054 muvar2-muvar5 -0.65527 -0.94307 muvar3-muvar5 -0.43813 -0.72593 muvar4-muvar5 -0.57551 -0.86331 > qtukey(0.95,5,55) [1] 3.988545 > qtukey(0.95,5,55)/sqrt(2) #es [1] 2.820328 0.42518 -0.35494 -0.36747 -0.15033 -0.28771 FTR Reject Reject Reject Reject H0 H0 H0 H0 H0 0.663893 1e-06 0 0.000665 6e-06 lo que usamos para los ic de tukey # Calcule a mano el IC para mu1 - mu5 segun tukey # y chequeelo con el calculado por R. # Que pares de medias difieren entre si a nivel conjunto 0.05, si utilizamos el # metodo de Tukey para sacar las conclusiones? Asegurese de detectar los 4 pares. # Cual de los dos metodos estudiados (Bonferroni o Tukey) conviene elegir en # este caso? 8