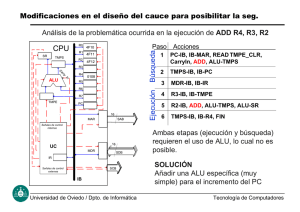
Detección de la secuencia Alu mediante la técnica PCR
Anuncio

Universidad de Puerto Rico Recinto de Río Piedras Departamento de Biología Laboratorio de Genética Biol 3350 Detección de la secuencia Alu mediante la técnica PCR En este experimento analizaremos y amplificaremos una región del cromosoma 8 para detectar si tiene o no la inserción de una secuencia conocida como Alu que se encuentra dentro del gen TPA (tissue plasminogen activator). En humanos, la familia de elementos Alu consisten de pequeñas secuencias de DNA, conocidas como “SINEs-short interspersed repeated DNA elements”-que se repiten a lo largo del genoma y representan el 3% del genoma total. Los elementos Alu son de aproximadamente 300pb y derivan su nombre porque en el medio de la secuencia tiene un lugar de reconocimiento para la endonucleasa Alu I. Aunque el DNA entre individuos es muy parecido, existen regiones en el cromosoma humano que exhiben una gran diversidad. Estas regiones se conocen como secuencias polimórficas (“varias formas”). Se estima que entre 500-2,000 elementos Alu, en su mayoría, están restringidos al genoma humano. Uno de estos elementos Alu, TPA-25, se encuentra en un intrón del gen TPA (tissue plasminogen activator). Esta inserción es dimórfica lo que quiere decir que está presente en unos individuos y en otros no. La reacción en cadena de la polimerasa, PCR, permite la amplificación in vitro de pequeñas cantidades de DNA en poco tiempo. Consiste en el uso de varias temperaturas, formando un ciclo, para desnaturar el DNA templado, enlazar los “primers” y luego extender la cadena complementaria de DNA. En este experimento utilizaremos “primers” que flanquean el lugar de la inserción para amplificar el fragmento de 400pb cuando TPA-25 está presente y un fragmento de 100pb cuando está ausente. Por lo tanto, existen tres posibles genotipos: homocigóticos para la presencia de TPA-25 (un fragmento de 400pb), homocigótico para la ausencia de TPA-25 (un fragmento de 100pb) y heterocigótico (2 fragmentos de 400 y 100pb). El DNA templado se obtiene mediante el procedimiento para aislar células epiteliales bucales. Después de enjuagarse la boca con la solución salina, las células se centrifugan y resuspenden en una resina conocida como Chelex (la cual enlaza iones de metales que pueden inhibir la reacción de PCR). Las células son lisadas al calentar en agua hirviendo y luego se centrifugan para bajar el chelex junto a los demás contaminantes. Una muestra del DNA genómico que se encuentra en el sobrenadante se mezcla con la Taq polimerasa, “primers”, dNTPs, MgCl2 y agua. Las muestras son colocadas en la máquina (thermocycler) con el programa que nos permitirá amplificar la secuencia Alu. Al finalizar este proceso, se analizarán y compararán los diferentes genotipos de los miembros de la clase haciendo una electroforesis en un gel de agarosa al 2%. Procedimiento para aislar células de epitelio bucal 1. Obtenga un tubo de centrífuga de 15mL que contenga 10mL de solución salina y rotule con su nombre. 2. Enjuague su boca con la solución salina y por 10 segundos (como si fuera un enjuagador bucal). 3. Devuelva el enjuague a un vaso o cono de papel y transfiera al tubo original y cierre bien la tapa. 4. Centrifugue a 2,000-3,000 rpm (revoluciones por minuto) por 10 minutos hasta formar un precipitado o “pellet”. 5. Descarte el sobrenadante cuidadosamente para evitar perder el “pellet”. Si el “pellet” no se ha adherido a la pared del tubo, entonces elimine lo más que pueda del sobrenadante y luego utilice una micropipeta de 100-1000uL para eliminar el exceso. 6. Con la micropipeta de 100-1000uL, resuspenda varias veces el Chelex1 10% y luego transfiera 500uL al tubo de centrífuga con el “pellet”. 7. Resuspenda el “pellet” con la resina varias veces hasta que la solución quede homogénea. 8. Transfiera 500uL a un microtubo de 1.5mL e identifique. 9. Incube la muestra por 10 minutos en agua hirviendo. 10. Luego de incubar la muestra, retire del calor y enfríe en hielo por 1 minuto. 11. Centrifugue a 10,000rpm por 30 segundos para que el Chelex quede en el fondo. 12. Transfiera 200uL del sobrenadante a otro microtubo de 1.5mL y mantenga en hielo hasta que pueda llevar a cabo la reacción. 1 El Chelex es una resina con carga negativa que remueve iones que pueden interferir con la reacción de PCR. Reacción PCR La reacción se llevará a cabo en un microtubo de 0.2mL que contiene en una pequeña esfera blanca (bead) lo siguiente: Taq polimerasa: Es la enzima encargada de replicar el DNA. Amortiguador para la Taq polimerasa Nucleótidos MgCl2: Co-factor Al “bead” se le añade: DNA: Templado que se utilizará para determinar la presencia o ausencia de TPA25. “primers”: Secuencias, de 25 pares de bases, complementarias al terminal 3’del DNA que se quiere amplificar. Son necesarios porque la polimerasa sóla no comienza la replicación; extiende o continúa el proceso. H2O estéril: Completar la reacción hasta 25uL. Programa PCR (thermocycler) 95 ºC 5 minutos 95 ºC 58 ºC 72 ºC 1 minuto 30 segundos 30 segundos 72 ºC 4 ºC 5 minutos ∞ 30 ciclos