Descargar pdf
Anuncio

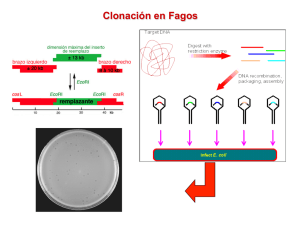
¿Qué es una librería o genoteca? Nuestro genoma •Genoteca genómica – moléculas de vector que contienen insertos de DNA genómico •Genoteca de cDNA – moléculas de vector que contiene insertos de cDNA Recuerda: RNA cDNA (específico de tejido) ¿Por qué necesitamos una genoteca? Encontrar lo que buscamos es como buscar una aguja (gen) en un pajar (genoma/mRNA) r ma for LIBRERÍA La pregunta es: ¿qué plasmico contiene nuestro gen de interés y como lo aislamos de la genoteca? ns LIBRERÍA genoma tra Li ve gar a cto u n r dig eri r Nuestro gen! BÚSQUEDA DE COLONIAS USANDO UNA “SONDA” ¿Qué podemos usar como sonda? La lógica de cribar una genoteca Normalmente: El cribado es un compromiso entre: Un oligonucleótido Una cadena corta de ácidos nucleicos (sintetizada por nosotros) que hibrida con nuestro DNA objetivo Una sonda de cDNA Un trozo de DNA mayor Un anticuerpo El tamaño del genoma A genomas más grandes mayor número de colonias hay que cribar Tamaño del inserto A medida que el tamaño del inserto es mayor, menor número de colonias que cribar El vector debe producir la expresión de proteínas! 1 Para P = 99% y F = 20,000 bp….. ESPECIES TAMAÑO DEL N GENOMA N = LOG 10( 1 – P ) LOG 10( 1 – F ) E coli 4.6 x 10 6bp 1.1 x 10 3 Human o 3.0 x 10 9bp 6.9 x 10 5 N = no de recombinantes necesarios de cribar P = probabilidad de obtener el clon F = tamaño medio de inserto en vector Para placas con 500 colonias, hay que cribar > 1000 plates para tener un 99% de probabilidad para tener una colonia positiva TIPO LIBRERÍA cDNA Genomic VECTOR INSERTO MÁXIMO plásmido < 10kb fago 23kb cósmido 45kb BAC 350kb YAC 1000kb Figure 9.2 Propiedades que un vector bacteriano debe tener 1. Capaz de replicarse: replicarse: • replicarse de manera autónoma (su propio origen de replicación) • extracromosómico al cromosoma del huesped 2. Marcadores de selecció selección: • normalmente resistencia a antibiótico en E. coli • actividad B-galoctosidasa 3. Sitios de restricció restricción únicos: nicos: • sitio presente sólo una vez en el plásmido • al linearizar el fragmento puede ser clonado 4. Alto nú número de copias si es posible: posible: • 1-50 copias (mientras más mejor) Vectores: Vectores: • llevan el DNA extraño (normalmente plásmidos • origin of replicación, sitios de E.R. únicos Vectores plasmí plasmídicos: dicos: • ocurren de manera natural, moléculas dsDNA • Insertos de hasta 15kb de RruI longitud PvuI • se replica en células del PstI huesped • 1,000 bp a 100 kb pBR322 (generado (generado por ingenierí ingeniería gené genética) tica) • 2 genes antibióticosR • sitios de restricción únicos • solo 10-50 copias/ copias/c élula 2 Digiere DNA extraño con el mismo enzima Plásmidos bacterianos se han manipulado para que sirvan de vehículos (vectores) para la replicación de nuestro DNA • Si el sitio de reconocimiento es de 6 bp está al azar en el genoma corta cada 4096 bp (4 6) Linearizar plásmidos con enzimas de restricción Propiedades: • genoma humano 3 x 10 9 bp…>7 x 10 5 fragmentos Una población completa de transfomantes representa una librería genómica • origen de replicación • MCS (sitios de restricción para introducir el DNA •Fragmentos individuales se ligan a plásmidos individuales de DNA • métodos de selección del plásmido en la bacteria • transformación introduce plásmidos individuales (clones) en bacteriasf Pasos implicados en clonaje molecular Selection 1. Digerir DNA purificado para clonar es decir obtener el inserto y el vector con el MISMO E.R 2. Tomar los fragmentos de DNA digerido con un tamañ tamañ o adecuado para insertarlo en el vector 3. Ligar con DNA ligasa para formar DNA recombinante Pasos implicados en clonaje molecular 4. Transfiere el vector con inserto a cé cé lulas vivas • Se llama Transformació Transformación • Se generan copias por replicació replicación, y por tanto se conocen como clones de DNA • Cada clon es gené genéticamente idé idéntico 5. Seleccionar células que llevan plámidos recombinantes • resistencia a antibiótico, producto coloreado ¿Cómo sabemos que un plá plámido contiene un inserto? inserto? Selecció Selección y cribado de molé moléculas recombinantes: recombinantes Posibilidades en una bacteria transformada: • La bacteria sola (no transformación) - 1x107 posib. • bacteria + plá plásmido religado • Bacteria + inserto DNA religado (sin sitio ori) • Bacteria + plá plásmido + inserto (molé moléculas recombinantes) recombinantes) Necesitamos un modo de seleccionar fácil y rápido por recombinantes • ¿Cómo usamos los marcadores de selección? pBR322 Uso de marcadores de selecció selección pBR322 tiene 2 genes AntibioticR cada uno con un sitio único E.R. • Inserto en sitio Sal1 se inactiva el gen tet R • se llama inactivació inactivación por inserció inserción • convierte el fenotipo de la bacteria: TetR a Tet S 3 Colonias con E. coli AmpR Colonies missing on Tet plate insert Master Plate Ampicillin in media Replica Plate Tetracycline in media Volver a la placa original y picar colonias que no crecen en placas Tet. Estas tendrán el inserto de interés. Seleccionar E. coli con plásmidos 1) crecer en medio LB con Amp (las E. coli con plásmidos serán AmpR) -permite eliminar todas las E. coli sin transformar (no tendrán plásmido y serán AmpS) 2) Pero estamos interesados en plásmidos con inserto – ¿como podemos identificar estos clones usando marcadores de selección? 3) Hacer una placa réplica de una placa original Amp y transferir a placas con Tet. Plásmidos con inerto no crecerán en Tet (al ser Tet S) Selección de transformantes – el plásmido confiere resistencia a un antibiótico Amp r Amp r recombinante transformantes Ampicillina Selección de transformantes recombinantes – p.e. selección blanco/azul. El operón de la lactosa (Lac) Lactose B-galactosidase INSERTO – inactiva el gen lacZ’: colonias blancas galactose + glucose (+allolactose) Blue mRNA policistrónico X-gal lacI Alolactosa se une al represor lac e inhibe represión El gen lacI está controlado aparte (situado aguas arriba) Expresión de lac Z, posibilita a E. coli metabolizar glucosa Expresión de lac Y permite a E. coli importar lactosa lac A es una tiogalactosidasa transacetilasa Cter m lacZ’ -ve plasmid IPTG Genoma de la bacteria 4 Vectores pUC: pUC: β- galactosidasa (Lac Z) convierte lactosa en galactosa + glucosa Amp r recombinantes transformantes El cribado de colonias azules y blancas es lo más usado en procesos de clonación Identificar Transformantes Como identicar clones de interés en este plásmido • Usar un producto sintético llamado X-gal • β-galactosidasa brompe X-gal a galactosa + colorante azul Select for White colonies on Ampicillin (AmpR) and X-gal plates (LacZ (LacZ-) pUC Vector Blue white Ampicilina + medio X-Gal Medio con Ampicilina Permite seleccción y cribado en una placa. No hace falta réplica. Picar colonias blancas Clonaje direccional Evita autoligación y a menudos se quiere introducir en una orientación determinada • Se puede hacer usando dos enzimas de restricción distintos en el vector • Generar el DNA a clonar con los dos mismos enzimas de restricción • DNA sólo se puede insertar en una dirección 5 Los virus también se pueden usar para clonar moléculas de DNA exógenos debido a su capacidad de propagarse en bacterias Sub-clonación plásmido A plásmido B • digerir DNA de fago lambda • eliminar sección intermedia (contiene genes para lisogenia) • ligar DNA extraño a los brazos del fago lambda •infect E. coli • mezclar con proteínas de la cápsida para empaquetar DNA recombinante Crear una librería genómica usando fago lambda... •Infectar una célula bacteriana con un fago recombinante • mezclar con bacteria sin infrectar y plaquear Todas las placas de fago juntas es una genoteca genómica Cósmidos smidos (CLON) • Es un plá plásmido pequeñ pequeño que contiene el sitio cos, un origen de replicació replicació n de un plá plásmido y genes de resistencia a antibió antibió tico • Se empaqueta como un bacterió bacteriófago, fago, pero el plá plásmido se replica como un plá plá smido cuando el DNA está está en la bacteria • Pueden insertarse fragments de DNA 35 y 45 kilobases 6 Cósmidos • Vectores que son híbridos entre plásmidos y fago lambda • Se pueden replicar en la bacteria como un plásmido y empaquetar como un virus • Pueden contener fragmentos de DNA más grandes que los plásmidos Vectores en levaduras Cromosomas artificiales en levaduras (YACs) YACs) 1. 2. Cromosomas artificiales de bacterias (BACs) BACs) • Vectores sinté sint éticos que se han usado para clonar fragmentos grandes de cromosomas de eucariotas ((entre entre 100 y 300 kilobases kilobases)) • Usado en el an aná álisis de grandes fragmentos de genomas complejos, complejos , genes completos y construcci construcció ó n de mapas fí f ísicos de genomas • Generado usando un pl plá ásmido pequeñ peque ño, llamado factor F (fertilidad ( fertilidad), ), permite la clonaci clonació ón y marcadores de selecció selecció n • El factor F permite al vector integrar grandes fragmentos de DNA, siendo hasta un 25% del tama tamañ ño del cromosoma bacteriano Contiene los componentes siguientes: siguientes : Un centr centró ómero que permite al cromosoma que se transfiera a ccé é lulas hijas durante la divisió división celular • Un tel teló ó mero al final del cromosoma de levadura que permite replicació replicaci ón correcta y evita degradació degradaci ón • Una secuencia de replicaci replicació ón autó aut ónoma (ARS) que consiste en secuencias de DNA espec especíífica que permite la replicació replicació n de la mol molé écula • Un gen que permite un modo de detectar la detecció detecci ón del fragmento insertado Util para clonar grandes fragmentos de DNA (entre 200 y 1500 kilobases) • Estudios genéticos clásicos identifican un gen con un fenotipo interesante…como lo clonamos La genoteca genómica contiene >10 6 clones individuales Resumen de la construcción de una genoteca genómica ¿Cómo identificamos un clon que contiene un gen de interés en una genoteca? Nuestro genoma ¿Cómo identificamos un clon que contiene un gen de interés en una genoteca? Nuestro gen! 7 r ma for La pregunta es: ¿qué plasmico contiene nuestro gen de interés y como lo aislamos de la genoteca? ¿Qué podemos usar como sonda? ns LIBRERÍA tra dig eri r Li ve gar a cto u n r LIBRERÍA genoma BÚSQUEDA DE COLONIAS USANDO UNA “SONDA” Información sobre la secuencia de proteína da información sobre la síntesis de sondas de oligonucleótidos Normalmente: Un oligonucleótido Una cadena corta de ácidos nucleicos (sintetizada por nosotros) que hibrida con nuestro DNA objetivo Una sonda de cDNA Un trozo de DNA mayor Un anticuerpo El vector debe producir la expresión de proteínas! Cribado usando secuencias similares • Si un tejido está enriquecido en un mRNA determinado, éste se puede usar como sonda – p.e. secuencias de genes de globina • A veces es más fácil aislar una secuencia de un organismo que de otro, y este gen relacionado se puede usar para pescar el mismo gen de otro genoma – p.e. secuencias de genes de actina • replica plate plaques or colonies onto filter Librería genómica • denaturedeDNA fago lambda on filter plaqueadas en placas • incubate with petri radiolabeled probe Cada clone ocupa un that • detect cells lugar específico contain en correct una placagene by autoradiography • grow up appropriate clone 8 • replica de placas o colonias en un filtro • desnaturalizar DNA en el filtro • incubar con sonda radioactiva • detectar las células que contienen el gen por autoradiografía • crecer el clon apropiado Ejemplo: Estamos interesado una proteína (P450) que metaboliza drogas anti-HIV Hemos purificado una proteína de 50kDa y reconstituimos una actividad que metaboliza la droga (estamos bastante seguros que hemos purificado la proteína correcta) Secuenciamos los aminoácidos del amino N La secuencia que obteniemos es: MALIPDLAM????. ilegible…. Queremos saber la secuencia de aminoácidos completa y tener el clon para manipularlo Usamos el código genético para designar oligonucleótidos para el cribado No secuenciamos proteínas para tener su secuencia de aas completa: técnicamente dificil Deducimos la secuencia de aas a patir de su secuencia de cDNA Necesitamos clonar el cDNA para el enzima P450 ¿Qué hacemos? 1. Hacemos una genoteca de cDNA de un tejido que exprese altos niveles de la proteína (si cantidad de proteína es alta, es lógico que cantidad de mRNA sea alta y por tanto enriquecemos el cDNA) 2. Cribamos la genoteca con una sonda: - ¿oligonucleotido? - ¿anticuerpo? UN AU U/C/ CCN GA U/C U/ C A Por ej. UUN AUG CCN GAC 5’ GTC NGG GAT NAA ci ó ma for GENOTECA mRNA consenso Reverso complementario Secuencia de la sonda AAAAAAAAAA 5’ n Codones de mRNA posibles ns 5’ UUA AUU CCU GAU G C C C CUU A A C G A G Secuencia de proteína tra M A L I P D L A M 3’ 5’ 9 Hacer una copia en nilon de la placa de bacterias OBJETIVOS Cribar nuestra copia en nilon con nuestro oligo marcado 1. Qué es clonar El oligo hibridará con nuestro plásmido recombinante que contengo el reverso complementario de la secuencia del oligo 2. Enzimas que se usan en tecnología del DNA recombinante 3. PCR 3 Generación y cribado de genotecas * * * Picar y crecer (clon) * NILON AUTORA DIOGRAFÍA 4. ¿PORQUÉ? – Expresión del gen / mutagénesis. 5. Secuenciación del DNA PLACA DE AGAR ¿Por qué el peñazo de clonar? Razones posibles- •Secuenciación de cDNA sequence …. Determinar la secuencia de aminoácidos de una proteína •Expresar el cDNA – proteína, producir grandes cantidades y proteína pura Purificar por cromatografía de afinidad bacteria Ni •Crear una proteína con etiqueta: p.e. Generar una etiqueta de His HI S ligate Ni Ni VECTOR Eluir HIS Eg plasmid Otras proteínas Si podemos obtener nuestro vector recombinante: Determinados resíduos de Aas se pueden mutar: M A L I P D L A M 5’ UUA AUU CCU GAU Secuencia de proteína Posibles codones de mRNA Dentro de células humanas y si permaneciera estable y expresada la proteína lo podríamos usar como terapia génica Si cambiamos UUA AUU CCU GAU a UUA AGG CCU GAU o UUA CCU GAU o UUA UAG Mutación I/R Delección Sin embargo, no funciona – actualmente se están intentando vectores virales Truncar stop 10 OBJETIVOS Secuenciación del DNA 1. Qué es clonar Nos informa de la secuencia del gen / cDNA / aminoácido 2. Enzimas que se usan en tecnología del DNA recombinante ¿Cómo se hace? Método enzimático de Sanger- recordar DNA polimerasas: Necesitamos un cebador con un 3’OH libre 3. PCR 3 Generación y cribado de genotecas DNA se sintetiza en una nueva cadena SIEMPRE en dirección 5’ 3’ 4. ¿PORQUÉ? – Expresión del gen / mutagénesis. Se basa en le parada de polimerización en los dideoxinucleótidos de la nueva cadena 5. Secuenciación del DNA 5’ 3’ 3’OH 5’ Dideoxinucleotidos: N se usan para parar la síntesis en una base específica 5’ N N 5’ P + P N P N P P DNApol N N P P P OH OH P Análogo Dideoxy NO TIENE 3’ OH N P OH Dideoxynucleótidos N 5’ N N 5’ P + P N P N P DNApol N P P N P P OH P N H P H PARADA!! • Se preparan 4 reacciones de síntesis de DNA diferentes • comienza la síntesis de un lugar donde se ceba específicamente • se añaden los componentes para síntesis de DNA + ddNTP específico para for cada una de las 4 reacciones No 3’OH... 11 Correr las 4 reacciones diferentes en un gel de electroforesis… los fragmentos más pequeños (más cercanos al cebador) migra más lejos A A T C T G G G C T A T T C G G G C G T Para la cadena sintetizada ... Leer 5’>3’ Secuenciación automática de DNA: • cada reacción se marca específicamente con una molécula fluorescente distinta 5’ a 3’ para la cadena sintetizada... •Puesto que cada fragmento se marca con un color distinto se pueden cargar en la misma calle •Un laser incorporado en el aparato puede iluminar las bandas y grabar la señal fluorescente cuando el DNA pasa el detector… computador La salida del detector se envía directamente a un computador y la secuencia de DNA se graba (en tiempo real) a medida que se corre el gel… Ejemplo de salida: Señal azul fluorescente detectada A A T C T G G G C T A T T C G G G C G T Resumen 1. ¿Qué es clonar? Hacer múltiples copias de la misma cosa 2. Enzimas empleados en tecnología de DNA recombinante DNA polimerasa RNA polimerasa Transcriptasa reversa Endonucleasas de restricción DNA ligasa Fosfatasa Quinasa 12 3. Hacer genotecas 4. ¿Por qué? – expresión del gen / mutagénesis DIGERIR / LIGAR / VECTOR / TRANSFORMACIÓN / HUESPED Estudios de función génica / purificación / mutagénesis • Genoteca genómica – moléculas de vector que contienen insertos de DNA genómico • Genoteca de cDNA – moléculas de vector que contienen insertos de cDNA – recuerda RNA cDNA (específico de tejido) • Tamaño del inserto / tipo de vector • Tipos de sonda 5. Secuenciación del DNA DNA polimerasa / cebador / ddNTPs MOLECULAR BIOLOGY OF THE GENE 4. PCR. 13