Traducción - Traduccion
Anuncio

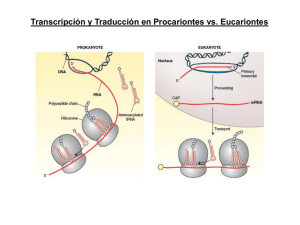
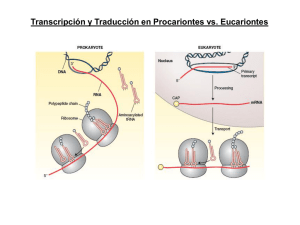
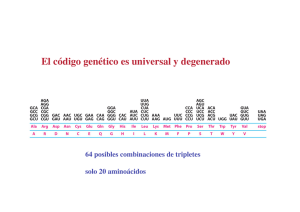
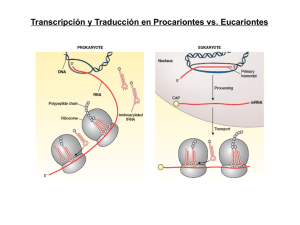
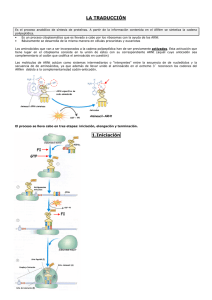
Traducción Los elementos tRNA iniciador aminoacilado tRNAs elongadores aminoacilados 3’ 5’ mRNA Ribosomas El tRNAi iniciador de la traducción (bacteria) THF = tertra hidro folato FMT = formil metionil transferasa El tRNAi iniciador de la traducción (bacteria) El ARNt iniciador tiene características especiales Necesario para la formilación Necesario para la entrar al sitio P El Ribosoma Subunidad grande Subunidad grande Sitio E Sitio P Sitio A Subunidad pequeña Unión del mRNA Subunidad pequeña El ribosoma realiza 3 funciones: Aunque está formado por proteínas y RNA las funciones son realizadas Principalmente por el RNA RIBOZIMA a) Función genética (decodificar la secuencia de nucleótidos en aminoácidos) b) Función enzimática (catalizar la formación del enlace peptídico) c) Función de translocación (maquina que se mueve a lo largo del mRNA, por la que van pasando los tRNAs y se elonga la cadena peptídica) Función genética --------------- subunidad pequeña Función enzimática ----------- subunidad grande Función de translocación --- ambas subunidades 16S o 18S 23S o 25/28S Organización de los mRNA bacterianos En procariontes los mRNAs son POLICISTRÓNICOS: llevan la información para VARIAS proteínas que forman parte de una ruta metabólica. Cada CISTRÓN se traduce de manera independiente Alberts et al., 3rd ed., p.237 Selección del Codón de inicio AUG (bacteria) El marco abierto de lectura lo define el AUG de inicio Bacteria SD: Shine-Dalgarno Shine-Dalgarno es una secuencia en el mRNA que se encuentra alrededor de 10 nt ANTES (hacia el 5’) del AUG y es complementaria a una secuencia en el extremo 3’ del RNA ribosomal 16S Selección del Codón de inicio AUG (bacteria) SD = sitio de unión al ribosoma AUG queda colocado en el sitio P de 30S Etapas de la traducción v Inicio v Elongación v Terminación Para el inicio de la traducción bacteriana: v Las subunidades ribosomales deben estar separadas v La subunidad pequeña debe reconocer al mRNA v El tRNA aminoacilado (fmet-tRNAmet) debe colocarse en la posición P de la subunidad ribosomal pequeña v Debe ocurrir el reconocimiento codón-anticodón de inicio v Se hidroliza una molécula de GTP v El proceso es asistido por: Factores de iniciación de la traducción (IF1, IF2, IF3) Complejos de Inicio (IC) de la Traducción fMet 4. Se hidroliza GTP y salen los IFs. GTP IF2 IF3 5’ 50S IF3 mRNA S-DA UG IF1 70S-IC 3. Se une la subunidad grande 50S 30S 5’ S-D AUG 3’ fMet 5’ 50S IF3 3’ 3’ IF3 5’ fMet IF1 S-D AUG 1. Interacción entre ShineDalgarno (S-D) y el extremo 3’ del rRNA 16S coloca el AUG en el sitio P del ribosoma S-D AUG fMet IF2 3’ 30S-Pre-IC GTP GTP IF2 IF3 5’ S-D AUG 2. Se une el complejo ternario IF2-tRNAi-GTP IF1 3’ 30S-IC Listo!!! FACTORES DE INICIO DE LA TRADUCCIÓN (PROCARIONTES) Factor Función IF1 Previene la unión de tRNAs en el sitio A de la subunidad 30S IF2 GTPasa que interacciona con 3 componentes claves durante iniciación: la subunidad 30S, IF1, y fMet-tRNAif-Met) IF3 Se une a 30S y evita re-asociación con 50S. Participa en el reconocimiento codon-anticodon f-met 5’ aa-tRNA IF3 IF1 E P A 3’ Organización de los mRNA en Eucariontes: 5’ CAP 1. NO hay algo similar a Shine-­‐ Dalgarno (S-­‐D) 2. La subunidad 40S se recluta al 5’ CAP: lejos del codón de inicio AUG 3. Hay solo UN CISTRÓN 4. Hay cola de poli A 5. Las regiones no traducidas 5’UTR y 3’UTR son mucho mas largas Factores de iniciación de la traducción (eIF) Los factores eIF4 forman un complejo circularizado con los extremos 5’ y 3’ del mRNA met aa-tRNA eIF5 eIF3 4A 4G AUG 5’ mGpppG 4E eIF1 eIF4E: reconoce el 5’ CAP eIF4G: une a eIF4E eIF4A (helicasa) eIF3 (unido a 40S) PABP (unido a poli A) PABP AAAAAAA 7 3’ 50-300 nt ¡Los factores eIF4 NO existen en bacteria! Búsqueda desde 5’ Cap hasta encontrar el primer AUG en contexto apropiado + FACTORES + met-tRNAi + GTP Secuencia AUG en contexto apropiado en Eucariontes Secuencia Kozak: Purina en posición -3 G en posición +4 El mRNA está “circularizado” durante traducción eucarionte El tRNA iniciador-Met Cuando se completa el inicio, sale eIF2GDP El factor eIF2B debe intercambiar GDP por GTP 1. Tiene características particulares (entra por el sitio P) 2. La metionina NO está formilada 3. Es unido por eIF2 y GTP (complejo ternario Este proceso es inhibido por diversas condiciones de estrés (FOSFORILACIÓN de eIF2) para DISMINUIR la síntesis de proteínas 242 7 Initation of Protein Synthesis 3 IFs 11 eIFs Separación de subunidades ribosomales Unión de tRNA iniciador (sitio P) Reconocimiento del mRNA: • Shine-Dalgarno • rRNA16S • AUG en sitio P • • • Regeneración de complejo eIF2-GTP REGULADA Reconocimiento del mRNA: • 5’Cap • Factores eIF4 • Secuencia Kozac Hidrólisis de GTP Salida de factores Unión Subunidad grande Figure 7.2-1 Pathways of translation prokaryotes Protein initiation Synthesis in and Ribosome structure, 2004 v INICIO v ELONGACIÓN Salida de tRNAs desacilados Cadena polipeptídica creciente Centro PeptidilTransferasa (PTC) Centro decodificador Movimiento del Ribosoma Para la elongación de la traducción: ribosomas mRNA tRNAs-aa ENERGÍA (GTP) FACTORES DE ELONGACIÓN DE LA TRADUCCIÓN GTP GTP x aminoácido Los tRNA-aminoacilados entran por el sitio A y salen sin aminoácido por el sitio E FACTORES DE ELONGACIÓN DE LA TRADUCCIÓN Factor procarionte Factor eucarionte Función EF-Tu eEF-1A Lleva el aa-tRNA al sitio A disponible; hidrolisis GTP EF-Ts eEF-1B Factor que intercambia GDP por GTP para EF-Tu/eEF-1A EF-G eEF-2 Ayuda a la translocación del ribosoma exactamente 1 codón; hidroliza GTP En cada ciclo de Elongación: 1. Entrada de un aa-tRNA correcto (sitio A) • Interacción codón-anticodón • Cambio conformacional • Hidrólisis de GTP EF-Tu - GTP 2. Formación del enlace peptídico (sitio P) Ribosoma 3. Translocación (sitio A vacío) • Cambio conformacional • Hidrólisis de GTP • Recorrido 1 codón (3 bases) EF-G - GTP Ciclo de recuperación para EF-Tu – GTP Unión al ribosoma tRNA activo EF-Ts a. Unión a EF-Ts b. Recambio GDP por GTP c. Llevar a otro tRNA-aa inactivo a. Lleva al tRNA-aa por el sitio A b. Reconocimiento codón/anticodón c. Hidrólisis de GTP El ribosoma (rRNA 23S/28S) realiza la catálisis RIBOZIMA Formación del enlace peptídico Translocación del ribosoma Estado PRE-translocación a. b. c. d. La cadena peptídica se encuentra unida al tRNA del sitio A El tRNA en el sitio P se encuentra libre de aminoácido Entra el factor EF-G – GTP por el sitio A Se hidroliza GTP y los tRNAs junto con el mRNA se recorren 3 nt Estado POST-translocación a. El tRNA libre de aminoácido sale por el sitio E b. La cadena peptídica se encuentra sobre el tRNA en el sitio P c. El sitio A está libre para la entrada de un nuevo tRNA aminoacilado Terminación ribosoma mRNA factores de terminación GTP proteína subunidades ribosoma mRNA tRNA libre Factores de terminación Bacteria Eucariontes RF1 RF2 RF3 RRF eRF1 eRF3 El gasto energético del proceso de traducción Se hidrolizan 2 GTP´s por cada aminoácido incorporado La hidrólisis promueve cambios conformacionales Cargado de tRNA con su aminoácido 1 ATP /aa TRADUCCION Iniciación 1 GTP (1er aminoácido) Elongación 2 GTPs /aa Terminación 1 GTP POLIRIBOSOMAS Múltiples ribosomas que se encuentran traduciendo el mismo mRNA Antibióticos inhibidores de traduccion Antibiótico/Toxina Organismo Función Tetraciclina Procarionte Sitio A subunidad 30S Cloramfenicol Procarionte Centro PTC subunidad 50S Puromicina Procarionte/Euca- Centro PTC subunidad 50S rionte Eritromicina Procarionte Tunel de salida del péptido naciente Acido fusídico Procarionte EF-G Ricina Procarionte Modifica el RNA en el centro activador de GTPasa Toxina de difteria Eucarionte Modifica eEF-1A Cicloheximida Eucarionte Translocación del ribosoma durante elongación Conociendo la estructura del ribosoma se pueden diseñar nuevos antibióticos... Llenar la siguiente tabla como se indica, observar diferencias y similitudes entre procariontes y eucariontes INICIO ELONGACIÓN TERMINACIÓN Transcripción Promotor y reconocimiento Escape promotor procarionte Reacción catalizada ¿Cómo se determina? Transcripción RNA polimerasas eucarionte Promotores y reconocimiento Escape promotor Procesamiento RNA ¿Cómo se determina? Traducción procarionte tRNA-iniciador elementos en mRNA Factores de inicio Pasos en cada ciclo y factores Hidrólisis GTP Codones de paro ¿Cómo se libera la proteína? Traducción eucarionte tRNA-iniciador elementos mRNA Factores de inicio exclusivos eucariontes Reacciones catalizadas RIBOZIMAS mRNA, tRNA, rRNA (características principales en procariontes y eucariontes)