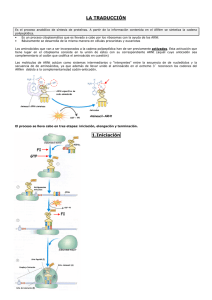
El código genético es universal y degenerado
Anuncio

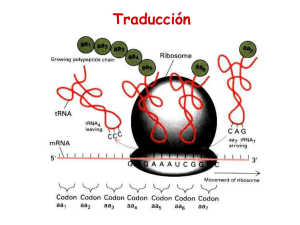
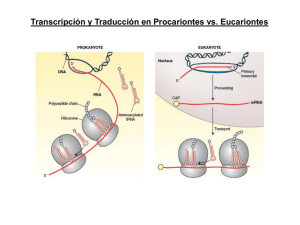
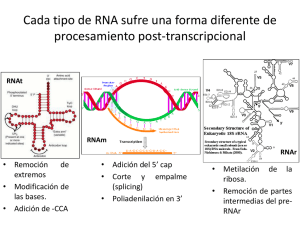
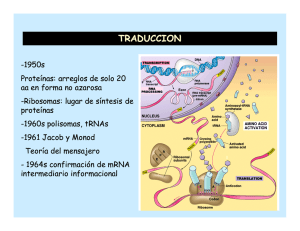
El código genético es universal y degenerado 64 posibles combinaciones de tripletes solo 20 aminoácidos Las secuencias se pueden traducir en tres diferentes marcos de lectura Los intermediarios entre los codones y los aminoácidos son los tRNA tienen una longitud de aproximadamente 80 nucleótidos Interacción codón-anticodón la 3ª posición puede no aparear (hipótesis del “bamboleo”) las bacterias tienen 31 diferentes tRNA los eucariontes tienen 48 El apareamiento en la tercera posición es laxo Bases modificadas que se encuentran en los ARNt • Pseudouracilos (ψ) • 4 tiouridina • 2 metilguanina • 2 isopententenil adenina • Dihidrouridina (D) • Inosina Las aminoacil tRNA sintetasas acoplan el aminoácido con la molécula de tRNA Las aminoacil ARNt sintetasas tienen varios sitios importantes Aminoacil-tRNA sintetasas Existen diferentes interacciones entre las enzimas con sus tRNA´s Existe un mecanismo de corrección tanto para el tRNA como para el aminoácido Aminoácidos similares pasan por un doble filtro El crecimiento de la cadena polipeptídica es por adición del amino al grupo carboxilo La traducción se realiza en los ribosomas Ribosomas procariontes y eucariontes Sitios de unión del tRNA al ribosoma El ARNr juega un papel muy importante en la traducción Interacciones con ARN de la subunidad chica del ribosoma permite un reconocimiento correcto La síntesis de proteínas tiene 3 etapas Factores de iniciación IF3 The mRNA binds to the 30S subunit. The Shine-Dalgarno sequence is complementary to a portion of the 16S rRNA. Portion of 16S rRNA IF3 5′ 30S subunit IF1 IF1 Start Shine- codon Dalgarno sequence (actually 9 nucleotides long) 3′ IF2, which uses GTP, promotes the binding of the initiator tRNA to the start codon in the P site. tRNAfMet Initiator tRNA GTP IF2 IF1 IF3 3′ 5′ IF1 and IF3 are released. IF2 hydrolyzes its GTP and is released. The 50S subunit associates. tRNAfMet 70S initiation complex This marks the end of the initiation stage E P A 70S initiation complex 3′ Interacción del ribosoma con el RNAm Interacción con el ARNr 16S En bacterias los mensajes policistrónicos tienen varios sitios de reconocimiento del ribosoma Shine-Dalgarno 5´AGGAGGU3´ En bacterias existen dos aminoacil- tRNA para met El fMet-tRNA iniciador tiene características especiales En la mitad de los casos, la metionina es removida de la proteína Reconoce los codones AUG ó GUG Solo el tRNA de inicio entra al sitio P Factores de iniciación forman un complejo con los extremos del mensaje Complejo de la subunidad pequeña y el tRNA de inicio Complejo mensaje-ribosoma Diferentes factores de iniciación están involucrados en eucariontes En eucariontes el reconocimiento del codón de inicio es diferente Regla de Kozak Proteínas adicionales requeridas para la traducción (factores de elongación) EF-Tu, EF-G bacterias EF1, EF2 eucariontes El polipéptido se trasloca al aminoacil-tRNA La actividad de peptidil transferasa está asociada al ARNr 23S La traslocación ocurre en dos pasos Los dos factores de elongación tienen funciones distintas El final de la traducción se marca con tres codones específicos (UAA, UAG, UGA) que no son reconocidos por ningún tRNA UAA ocre UAG ambar UGA UAA RF1 RF2 Factores de terminación La peptidil transferasa cataliza la transferencia del péptido a una molécula de agua y el péptido se libera El factor de liberación eucarionte semeja a un tRNA eRF1 utiliza agua para hidrolizar el péptido Los ribosomas se reciclan al final de la traducción El papel de la hidrólisis de GTP Se hidrolizan 2 GTP´s por cada aminoácido incorporado La hidrólisis promueve cambios conformacionales Cargado de tRNA con su aminoácido 1 ATP (2 enlaces) /Aa TRADUCCION Iniciación 1 GTP Elongación 2 GTP/ Aa Terminación 1 GTP Comparación entre la traducción procarionte y eucarionte Síntesis en polisomas Regulación a nivel de la traducción Inhibidores en la síntesis de proteínas se usan como antibióticos Inhibidores de la síntesis de proteínas en eucariontes y en procariontes Selenocisteína es introducida en el codón de paro UGA