Diapositiva 1 - Genética y Biología Molecular
Anuncio

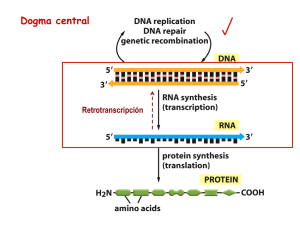
Universidad Nacional Autónoma de México Facultad de Química Curso Genética y Biología Molecular (1630) Licenciatura Químico Farmacéutico Biológico Dra. Herminia Loza Tavera Profesora Titular de Carrera Departamento de Bioquímica Lab 105, Edif E 5622-5280 hlozat@unam.mx VI. TRANSCRIPCIÓN Y PROCESAMIENTO DEL RNA • Objetivo general – El alumno revisará como se expresa la información genética, identificando los factores involucrados en este proceso: DNA y proteínas. Reconocerá los mecanismos por los que se lleva a cabo la maduración de los mensajeros. Objetivos particulares El alumno... 1. Tipos de genes. 2. Promotores procariontes. 3. Síntesis de RNA en procariontes. 4. Las fases de la transcripción en procariontes. 1.1. Conocerá los diferentes tipos de genes: los que codifican proteínas, los que codifican RNA ribosomal y RNA de transferencia. 1.2. Identificará los diferentes tipos de RNA: RNA mensajeros (mRNA), RNA de transferencia (tRNA) y RNA ribosomales (rRNA), sus características, abundancia en la célula y sus funciones. 2.1. Conocerá las características y funciones de las secuencias promotoras que definen el sitio de iniciación de la transcripción (+1) de mRNA en procariontes: caja TATA (-10). 2.2. Conocerá algunas otras secuencias involucradas en la regulación de la transcripción de mRNA en procariontes. 3.1. Analizará que la reacción de síntesis de RNA es en dirección 5’-3’, que requiere de ribonucleótidos, de un molde de DNA y de una RNA polimerasa. 3.2. Distinguirá la cadena codificante (con sentido +) de la cadena molde (sin sentido). 3.3. Conocerá la composición de la RNA polimerasas de procariontes y la función de sus subunidades (factores sigma). 3.4. Conocerá que los mRNA tienen regiones no traducibles (5’UTR y 3’UTR) que son importantes en la regulación de su vida funcional. 3.5. Será capaz de transcribir una secuencia de mRNA a partir de una secuencia de DNA en la que se indique cual es la hebra molde, la caja TATA y el sitio de inicio de la transcripción. 4.1. Conocerá los requerimientos para el inicio de la transcripción, identificando el complejo cerrado y el complejo abierto. 4.2. Comprenderá como ocurre la polimerización de ribonucleótidos durante el alargamiento de la hebra de mRNA. 4.3. Distinguirá entre el tipo de terminación Rho dependiente y el tipo Rho independiente. Conoci- Compren- Aplicamiento sión ción X X X X X X X X X X X X Objetivos particulares El alumno... 5. Transcripción en eucariontes. 6. Inhibición de la transcripción. 7. Procesamiento post-transcripcional de RNAm de eucariontes. 8. Procesamiento post-transcripcional de RNAr y RNAt de procariontes y eucariontes. Conoci- Comprenmiento sión 5.1. Conocerá las señales que controlan la transcripción en eucariontes: secuencias consenso y secuencias intensificadoras. 5.2. Reconocerá que las RNA polimerasas I, II y III sintetizan diferentes tipos de RNAs en eucariontes. 5.3. Reconocerá que para la síntesis de mRNAs se requieren diferentes factores proteicos (factores de transcripción). 5.4. Comparará los tres pasos de la transcripción de eucariontes con los mismos pasos de procariontes. 6.1. Conocerá algunos inhibidores de la transcripción en procariontes (actinomicina D, acridina, rifampicina) y en eucariontes (-amanitina). 7.1. Conocerá que los RNAs se transcriben como un transcrito primario que debe ser madurado para ser funcional (procesamiento post-transcripcional). 7.2. Reconocerá que existen secuencias en el RNA y factores proteicos que participan en el procesamiento post-transcripcional. 7.3. Comprenderá que los mRNAs de eucariontes se procesan por tres mecanismos: splicing (empalme), capping y poliadenilación. 7.4. Conocerá que el RNA puede ser catalítico. X 7.5. Conocerá que el mecanismo de "splicing" alternativo genera diferentes mRNAs que dan origen a diferentes proteínas (un gen codifica varias proteínas relacionadas). 8.1. Comprenderá como son procesados los RNAs ribosomales: splicing, modificación de bases. 8.2. Reconocerá como son procesados los RNAs de transferencia: splicing, modificación de bases, adición de CCA en el 3’. 8.3. Conocerá que los rRNA y tRNAs de procariontes también son procesados post-transcripcionalmente. X X X X X X X X X X X X Aplicación DOGMA CENTRAL Síntesis de RNA: TRANSCRIPCIÓN 1. Tipos de genes Un gene es una secuencia de DNA que codifica: a) un polipéptido (RNA mensajero) a) un RNA (RNA ribosomal y RNA de transferencia) Estructura del RNA cadena sencilla En el RNA… …el uracilo está en lugar de la timina …el uracilo se aparea con la adenina Dúplex El RNA puede presentar diferentes tipos de estructura secundaria Región de cadena sencilla Tallo - asa Burbujas internas Protuberancia Juntas Tres tallos, cuatro tallos Desapareamiento, simétrica, asimétrica La transcripción es el proceso por el cual se sintetiza RNA Los RNAs más abundantes y más estudiados son: rRNA RNA ribosomal, componente estructural y catalítico del ribosoma tRNA RNA de transferencia, adaptador entre mRNA y proteína mRNA RNA mensajero, codifica la secuencia primaria de una proteína Otros tipos de RNA snRNA RNAs pequeños nucleares, funcionan en corte y empalme de intrones snoRNA RNAs pequeños nucleolares, procesamiento y modificación de rRNA scaRNA RNAs pequeños de cajal, modifican snoRNAs, snRNAs, miRNAs, regulan traducción específica miRNA siRNA Otros no codificantes microRNAs, regulan la expresión genética RNAs pequeños interferentes, apagan la expresión de genes mediante degradación de mRNAs específicos Otros RNAs no codificantes que funcionan en procesos diversos como síntesis de telómeros, inactivación del cromosoma X, transporte de proteínas, etc. RNA ribosomal PROCARIONTES: RNAr 23S: 2,904 nts. RNAr 16S: 1,542 nts. EUCARIONTES: RNAr 28S: 4,718 nts. RNAr 18S: 1,874 nts. Estructura secundaria del rRNA RNA de transferencia Región aceptora Asa anticodón Tamaño: 75 – 80 nucleótidos RNA mensajero El tamaño de los RNAs mensajeros es variable y depende del tamaño del gen que se transcribe. La estructura de los mRNAs es variable y depende de la secuencia. Autoradiografía de mRNA marcado radioactivamente con 32CTP. Se observan los diferentes tamaños del mRNA. La transcripción es la síntesis de RNA a partir de un molde de DNA Micrografía electrónica de la síntesis de RNA ribosomal Dirección de la síntesis: 5’ 3’ Solamente un fragmento de DNA, que corresponde a un gen, es copiado a RNA y sólo una de las dos cadenas de DNA es copiada a RNA • ¿Cómo sabe la RNA polimerasa cuál de las dos cadenas usará como molde? • ¿Cómo sabe la RNA polimerasa dónde comenzar a sintetizar? • ¿Cómo sabe la RNA polimerasa donde terminar de sintetizar? • ¿Quién abre la doble hélice de DNA? ¿En qué sitio del gen inicia la transcripción? El sitio donde se inicia la transcripción es el +1 • ¿Cómo reconoce la RNA pol el sitio para iniciar la transcripción? • Es reconocido porque antes de él, hacia el 5’, hay una región llamada promotor • Esta secuencia de DNA no se transcribe, es decir no se sintetiza RNA correspondiente a esa región del DNA. • El promotor sirve de señal de reconocimiento para que la RNA pol se una a esa región y lleve a cabo la transcripción 2. Promotores procariontes Secuencias que se encuentran “corriente arriba” del sitio de inicio de la transcripción. Hay secuencias muy conservadas en los promotores procariontes. 5 –8 pb • Secuencia -10 o caja Pribnow TATAAT Apertura de la cadena. • Secuencia –35 TTGTCA Es la región de reconocimiento e interacción con el factor de la RNA polimerasa. Secuencias consenso en promotores procariónticos Caja TATA Las secuencia del promotor está referida a la cadena codificadora, no a la cadena molde El promotor Lac controla la expresión del gen lacZ, pero no del gen lacI. Observa que la posición de la caja TATA en el sitio -10 se representa sobre la cadena codificadora, es decir la que va de 5’ a 3’. Los promotores tienen orientación La dirección en la que se transcriben los genes puede variar sobre el cromosoma Las flechas azules indican la dirección en la que se transcribirá el mRNA (siempre se sintetiza en dirección 5’ – 3’), por lo que se puede deducir cual es la cadena que sirve de molde. El promotor contiene señales para iniciar la transcripción pero no se transcribe La enzima que sintetiza RNA es la RNA polimerasa. Solo hay una RNA pol en E. coli y está formada por 4 subunidades : ensamblaje de las unidades y unión al promotor : sitio catalítico ’: se une al DNA y parte de la subunidad catalítica : reconoce el promotor específico RNA polimerasa RNAP crystal structure with modeled DNA (Adapted from Korzheva et al., Science 2000) La estequiometría de subunidades en la holoenzima es 2' La subunidad es disociable Núcleo (core) de la enzima Cuando la subunidad se asocia al núcleo se forma la holoenzima Modelo de la RNA polimerasa de E. coli a partir de los datos cristalográficos Núcleo de la enzima 2 Holoenzima 2 La subunidad sirve como nodo para ensamblar la RNA polimerasa holoenzima y esta función reside en el dominio N- terminal de la proteína. El domino C-terminal de la subunidad interactúa con la región UP de los promotores que la tengan. ¿Cómo reconoce la RNA pol cuáles genes debe transcribir? Porque existen diferentes factores que reconocen promotores específicos: Factores sigma Funciones de la subunidad El factor sigma selecciona los genes a transcribirse al facilitar la unión entre la RNA polimerasa y el promotor. Esta unión depende de la denaturalización local del DNA que permite la formación de un complejo de promotor abierto El factor se recicla, i.e. cuando se disocia puede ser usado por otra RNA polimerasa. Al unirse al promotor, la RNA polimerasa causa la apertura de al menos 10 - 17 pb de la doble cadena de DNA. Esta “burbuja” de transcripción se mueve con la polimerasa exponiendo la cadena molde, de tal manera que puede ser transcrita. Factores sigma (s) Sigma 38: Regulates gene expression against external stresses. 3. Síntesis de RNA en procariontes Para la reacción química de la transcripción se requiere: •La enzima RNA polimerasa •DNA molde •Ribonucleótidos (trifosfatados) ATP, GTP, CTP, UTP •Proteínas o factores de transcripción A diferencia de la replicación, en la transcripción... • Solamente un fragmento de DNA, que corresponde a un gen, es copiado a RNA • Sólo una de las dos cadenas de DNA es copiada a RNA DNA Cadena codificadora: 5’-ATTCCGATGTACGAGG-3’ DNA Cadena molde: 3’-TAAGGCTACATGCTCC-5’ RNA 5’-AUUCCGAUGUACGAGG-3’ La secuencia de la molécula de RNA que se sintetiza a) es complementaria y antiparalela a la cadena molde b) tiene la misma dirección y secuencia (U -> T) que la cadena codificadora La polimerización se lleva a cabo en dirección 5’ 3’ La cadena molde se lee en dirección 3’ 5’ Reacción de polimerización Mecanismo: ataque nucleofílico del 3’OH de la ribosa del nucleótido n, al -P del nucleótido n+1 4. Las fases de la transcripción en procariontes •Inicio •Alargamiento •Terminación RNA pol Holoenzima Complejo cerrado Complejo abierto Sigma se disocia Complejo de elongación Inicio de la transcripción El factor sigma determina la iniciación de la transcripción permitiendo que la RNA polimerasa se una fuertemente al promotor (COMPLEJO CERRADO) La doble hélice en esa región debe abrirse para permitir la lectura de la secuencia formando el COMPLEJO ABIERTO. El factor sigma se disocia de este complejo. La RNA polimerasa tiene un canal abierto al cual se une el DNA. Una vez que se unen al DNA, los “dedos” de la enzima se cierran alrededor del DNA. Alargamiento 1.- Se disocia la subunidad de la RNA polimerasa. 2.- Comienza la adición de ppp G o ppp A, en el extremo 5’ del RNA naciente. ATP GTP UTP CTP Sustratos + DNA (como molde) Polimerasa de RNA dirigida por DNA (Mg++) nPPi 5’ 3’ pppApUpCpCpCpGpU… RNA (tipo, longitud y secuencia de este RNA dependen del gen de DNA que se está transcribiendo) El RNA que se va copiando del DNA se llama transcrito Estructura de una cadena de RNA Para la síntesis de RNA se van añadiendo ribonucleótidos al 3’ OH, polimerizándose la cadena a través de enlaces fosfodiéster (la cadena va creciendo en la dirección 5’ –> 3’) H Alargamiento RNA polimerasa Rebobinado Desenrollado Hebra codificadora Hebra molde 3 5 3 5 Hélice híbrida RNA - DNA 5’ ppp RNA naciente 3 Punto de elongación Desplazamiento de la polimerasa La “burbuja de transcripción” va avanzando, por un lado se abre el DNA duplex y por el otro se re-bobina. El RNA sintetizado va formando un híbrido (transitorio), con la cadena 3’ – 5’ del DNA. BURBUJA DE TRANSCRIPCIÓN Alargamiento La subunidad contiene el sitio activo de la RNA polimerasa donde se forman los enlaces fosfodiéster Hay dos sitios en el DNA que interactúan con la RNA polimerasa: 1. Un sitio de unión débil que involucra la zona del DNA desnaturalizada y el sitio 2. Un sitio de unión fuerte que involucra al DNA río abajo del sitio activo y lo activo en la subunidad de la polimerasa. La interacciones son principalmente electrostáticas. conforman las subunidades y ‘ de la enzima. La subunidad ‘ une dos átomos de Zn2+ que participan en la catálisis. Esta subunidad se une fuertemente al DNA. Terminación Hay dos mecanismos de terminación de la transcripción Dependiente de la proteína Rho Independiente de la proteína Rho Terminación: dependiente de la proteína Rho (trans) La proteína rho es un hexámero que hidroliza ATP en presencia de RNA. Se une al RNA que se está sintetizando y se mueve en dirección al sitio de síntesis. Desestabiliza al híbrido DNA – RNA, facilitando así la terminación de la transcripción. Terminación independiente de la proteína Rho (cis) La secuencia al final del gen contiene repeticiones invertidas que permiten la formación de una estructura de horquilla en el RNA. Hay una región rica en Adeninas, de tal forma que el híbrido DNA-RNA que se forma es débil y se disocia. En un mRNA maduro existen tres regiones: el 5’UTR, la región codificadora de la proteína y la región 3’UTR UTR: untranslated region 5’UTR o líder: contiene sitios de unión a ribosoma En bacteria esas secuencias son llamadas Shine-Dalgarno (AGGAGG) En vertebrados se conoce como caja Kozak 5. Transcripción en eucariontes http://www.stromastudios.com/portfolio/transcription.html La transcripción en eucariontes es mucho más compleja que en procariontes En eucariontes hay 3 tipos de RNA polimerasas 1. RNA Polimerasa I sintetiza RNA ribosomal (rRNA) 5.8S, 18S y 28S. 2. RNA Polimerasa II sintetiza RNAs mensajeros (mRNA), RNAs pequeños nucleolares (snoRNA), micro RNAs (miRNA), RNAs pequeños interferentes (siRNA) y la mayoría de RNAs pequeños nucleares (snRNA). 3. RNA polimerasa III sintetiza RNAs de transferencia (tRNAs), rRNA 5S y algunos RNAs pequeños nucleares (snRNAs). Los promotores reconocidos por la RNA pol II se llaman Promotores de clase II Los promotores clase II tienen diferentes secuencias reguladoras dependiendo de la combinación de factores de transcripción requeridos para formar el complejo transcripcional funcional en cada promotor. Algunos de los elementos que han sido descritos en los promotores de clase II de eucariotes son los siguientes: • La TATA Box. Su secuencia consenso es TATAAAA. Está localizada 25 bp upstream de el punto de inicio de la transcripción. • El Iniciador es una secuencia que es encontrada en muchos promotores y define el sitio de inicio de la transcripción. • La GC box. Su secuencia consenso es GGGCGG. Puede haber una o más copias localizadas entre 40 y 100 bp upstream del inicio de la transcripción. • La CAAT box – secuencia consenso CCAAT - es también frecuentemente encontrada entre 40 y 100 bp upstream del inicio de la transcripción. El factor de transcripción CTF o NF1 se une a la CAAT box. TATA Box Localizada aproximadamente 25 bp upstream de el punto de inicio de la transcripción, es encotnrada en muchos promotores. La secuencia consenso es TATAAAA (semejante a la caja TATA de procariontes localizada en la región 10). La caja TATA parece ser más importante para seleccionar el punto de inicio de la transcripción (i.e. posicionar la enzima) que para definir el promotor. GC box y CAAT box La GC box es un elemento común en promotores eucariónticos de clase II. Su secuencia consenso es GGGCGG. Puede estar presente en una o más copias las cuales pueden estar localizadas entre 40 y 100 bp upstream del inicio de la transcripción. El factor de transcripción factor Sp1 une la GC box. La CAAT box – secuencia consenso CCAAT - es también frecuentemente encontrada entre 40 y 100 bp upstream del inicio de la transcripción. El factor de transcripción CTF o NF1 se une a la CAAT box. Los promotores eucariontes son muy complejos y tienen diversas secuencias regulatorias Diagram based on and adapted from Figure 28.26 of Mathews & van Holde, Biochemistry, 2nd ed. Sitios consenso en promotores eucariontes RNA polimerasa II de levadura Arthur Kornberg, premio Nobel Medicina 1959 por el descubrimiento de la DNA polimerasa III bacteriana Transcripción por la RNA polimerasa II - 12 subunidades formando un complejo de mas de 500 kDa - múltiples factores accesorios (factores de transcripción) RNA pol II cola que se fosforila RNA pol bacteriana Promotor tipo II basal de eucariontes Caja TATA 25 pb río arriba del Inr (+1) D Factores de transcripción generales: TFIID A TBP: proteína de unión a caja TATA TAFs B TFIIA TFIIB E Ayudan a posicionar al complejo de factores sobre el promotor F TFIIF Proporciona la ocupación de +30 bp Promotor tipo II basal de eucariontes TFIIE H TFIIJ TFIIH Une a RNA pol II al complejo montado sobre el promotor Promueve el escape del promotor y fosforila el CTD (dominio carboxilo terminal) de RNA pol II brindando procesividad a la transcripción Diversos factores de transcripción participan en el inicio de la transcripción por la RNA pol II en eucariontes La unión de la TATA binding protein (TBP) al sitio TATA distorsiona la cadena Proteínas activadoras de la transcripción Activadores (enhancers) Además de los elementos anteriormente mencionados, Enhancers pueden ser requeridos para la expresión adecuada de un gen. Estos elementos no son parte del promotor per se. Pueden estar localizados upstream o downstream del promoter e incluso estar muy lejos de él. El mecanismo por el cual funcionan no se conoce. Podrían proporcionar un punto de entrada para la RNA polimerasa o podría asistir en la unión de otras proteínas a la región promotora Activadores: se unen a secuencias intensificadoras en el promotor del gen. Incrementan los niveles de transcripción Coactivadores: integran señales de activadores y/o represores a la maquinaria de transcripción con los factores de transcripción basal Represores: se unen a secuencias silenciadoras en el promotor del gen. Disminuyen los niveles de transcripción Factores de transcripción basal: Estos factores posicionan a la ARN polimerasa sobre el gen y comienzan la transcripción en respuesta a la señal de activadores y/o represores Diferentes componentes en la transcripción en eucariontes http://www.biochem.umd.edu/biochem/kahn/molmachines/newpolII/home.html Diferencias en la transcripción entre procariontes y eucariontes Procariontes Eucariontes 1. Todas las especies de RNA son sintetizadas por la misma especie de RNA polimerasa. 1. Hay 3 diferentes RNA polimerasas responsables de la transcripción de diferentes moléculas de RNA 2. El mRNA se traduce durante la transcripción. 2. El mRNA es procesado antes de ser transportado a citoplasma (adición de CAP, cola de poliA, remoción de intrones) 3. Los genes son segmentos contiguos de DNA alineados ininterrumpidamente con el RNA traducido a proteína. 3. Los genes frecuentemente se interrumpen por intrones. 4. Los mRNAs son frecuentemente policistrónicos 4. Los mRNAs son monocistrónicos Comparación de las estructuras de los mRNAs maduros de procariontes y eucariontes Alberts et al., 3rd ed., p.237 6. Inhibición de la transcripción en procariontes Las rifampicinas son producidas por Streptomyces mediterranei, con buena actividad contra bacterias Gram-positivas y contra Mycobacterium tuberculosis . Bloquea la transición de iniciaciónelongación. Se une de manera no covalente a la subunidad en el complejo RNA pol-promotor una vez que se han incorporado dos o tres nucleótidos a la cadena de RNA. La estreptolidigina inhibe a la RNA polimerasa durante el alargamiento. Inhibición de la transcripción en eucariontes -amanitina Actinomicina D Sar L-Pro Sar L-meVal D-Val O L-Pro D-Val L-Thr O L-meVal O L-Thr C C O N NH2 O O CH3 CH3 Se intercala entre bases G y C Acridina + N H Se intercala entre bases Octapéptido bicíclico que se obtiene del hongo Amanita phalloides. Inhibe la translocación de la RNA polimerasa II eucarionte durante la transcripción. Amanita phalloides El RNA después de ser transcrito debe ser procesado para madurar: procesamiento post-transcripcional mRNA: sólo en eucariontes tRNA y rRNA: tanto en eucariontes como en procariontes Otros RNAs no codificantes: tanto en eucariontes como en procariontes 7. Procesamiento del mRNA en eucariontes 1. Adición del CAP en el extremo 5’ 2. Splicing. Exclusión de intrones 3. Poliadenilación en el extremo 3’ a. Adición del CAP 1) La fosfatasa remueve un fosfato del 5´ 2) Una guanilil transferasa agrega GMP 3) Una metil transferasa agrega el grupo metilo b. Reacción de corte en la unión exón-intrón (splicing) Hay un tipo de splicing en el que el RNA es autocatalítico Existen secuencias específicas en los límites exón-intrón que determinan el sitio de corte preciso R= purinas Y= pirimidinas En otros tipos de splicing participan proteínas y también moléculas de RNA las cuales son las responsables del corte El apareamiento de las moléculas de snRNA requiere complementación de bases con el pre-mRNA snRNPs (small nuclear RNA binding proteins): U1, U2, U4, U5, U6 BBP U2AF El splicing (corte y empalme de intrones) lo realiza el snRNA de U6 snRNP: actividad de ribozima El RNA puede tener actividad catalítica: ribozimas La actividad catalítica del RNA fue descubierta en 1981 por Thomas Cech y Sidney Altman en Tetrahymena, al observar que un intrón de rRNA puede escindirse sola, sin la intervención de proteínas. Muchas ribozimas son intrones con capacidad de autoprocesamiento, es decir, se autoeliminan uniendo los dos exones adyacentes. Los intrones autoprocesadores normalmente solo actúan una vez y se destruye su actividad, con lo cual no cumplen todos los requisitos que definen un catalizador. Hay ribozimas implicadas en diversas e importantes reacciones celulares: procesamiento o maduración del mRNA en la síntesis de proteínas el rRNA 28S cataliza la reacción de transpeptidación o unión de los aminoácidos). Procesamiento alternativo de intrones en el pre-mRNA Un pre-mRNA 5 mRNAs maduros 5 proteínas diferentes c. Poliadenilación Proteínas específicas reconocen las secuencias de poliadenilación en el mRNA El mRNA es cortado y la enzima Poli-A polimerasa (PAP) agrega adeninas Proteínas de unión a la cola de poli-A se unen al extremo 3’ hasta que el mensaje sale del núcleo En eucariontes, la etapa de alargamiento durante la síntesis del mRNA está acoplada a su procesamiento La salida del mensaje del núcleo es a través de un poro nuclear El receptor de exporte nuclear dirige la salida del mRNA del núcleo En eucariontes, cada paso en la regulación de la expresión genética (de transcripción a traducción), es una subdivisión de un proceso continuo. 8. El RNA ribosomal y el RNA de transferencia también deben ser procesados post-transcripcionalmente para ser funcionales RNA ribosomal Es el RNA más abundante de la célula (80%). Tiene función tanto estructural como catalítica en la síntesis de proteínas. Existen diferentes tipos de acuerdo a su coeficiente de sedimentación asociados a las subunidades ribosomales: En procariontes Subunidad ribosómica grande 23S 5S Subunidad ribosómica pequeña 16S En eucariontes 28.5S 5.8S 5S 18S En eucariontes, el nucléolo es el sitio de síntesis de rRNA y pre-ribosomas Diversos pasos están involucrados en la síntesis de ribosomas 2: 514-520 El ribosoma Alberts et al., 3rd ed., p 232 Ribosomas procariontes y eucariontes Cumbre cabeza Protuberancia central Tallo plataforma base El rRNA juega un papel muy importante en la traducción rRNA 16S rRNA 23S RNA de transferencia Es el RNA más pequeño, con unos 75 nt de longitud, en promedio. Su función es transportar los aminoácidos en forma activada hasta el ribosoma donde ocurre la síntesis de proteínas. Existe al menos un tipo de tRNA para cada uno de los 20 aminoácidos. Sin embargo, algunos aminoácidos tienen varios tRNAs. Todos los tRNAs presentan en su 3’ la secuencia CCA, independientemente del aminoácido que transporta. El extremo 3’ constituye el sito aceptor del aminoácido. Procesamiento del rRNA en procariontes Procesamiento del rRNA en eucariontes Procesamiento de rRNA en eucariontes http://www.nobelprize.org/educational/medicine/dna/a/translation/rrna_synthesis.html Modificación de bases en los rRNA y en los tRNA • Pseudouracilos (y) • 4 tiouridina • 2 metilguanina • 2 isopententenil adenina • Dihidrouridina (D) • Inosina tRNA Acarrean los aminoácidos a los ribosomas para la síntesis de proteínas tienen una longitud de aproximadamente 80 nucleótidos Procesamiento de los tRNAs Modificación de bases Procesamiento del extremo 5’: Rnasa P (Ribonucleoproteína compuesta por RNA 377 nt y proteína 20 kDa) Acción catalítica: RNA (ribozima) Procesamiento: co-transcripcional Procesamiento del extremo 3’: a) endonucleasa elimina un grupo de bases b) exonucleasa, RNAsa D elimina los restantes nucleótidos c) El triplete CCA característico de estas moléculas es añadido por una tRNA nucleotidil transferasa Eliminación del intrón Procesamiento del intrón en los tRNAs de eucariontes Una endonucleasa corta en los sitios donde hay protuberancias y una DNA ligasa sella esos cortes