DNA - Traduccion
Anuncio

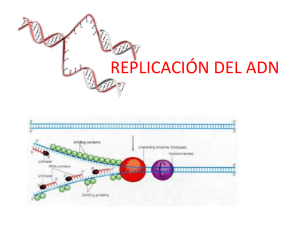
La complementariedad entre bases es escencial para la replicación MECANISMO SEMI-CONSERVATIVO DE LA REPLICACIÓN Experimento de Messelson-Stahl ¿Qué debe ocurrir para que el DNA pueda replicarse? La doble cadena se separa y cada una de las hebras es copiada para formar dos nueva doble hélices que llevarán una cadena PARENTAL y una cadena NUEVA ¿Dónde comienza la replicación? ¿En qué sentido ocurre la replicación? ¿Qué moléculas están involucradas en replicación? Origen de Replicación bacteriano OriC Determinado por una SECUENCIA específica en el ADN Cajas ricas en AT 1. Para activar el ORIGEN de replicación la enzima Dam metilasa metila las adeninas en las cajas ricas en AT. Cajas ricas en AT Cajas de unión a DnaA 2. DnaA-ATP reconoce sus cajas en el ORIGEN activado y mediante unión cooperativa ABRE la doble hélice en este sitio. La helicasa DnaB 3. La HELICASA es responsable de seguir abriendo la doble hélice, mediante ruptura de puentes de hidrógeno y gasto de ATP. La apertura es BIDIRECCIONAL: hay DOS hexámeros de DnaB abriendo la doble hélice tanto hacia la IZQUIERDA como hacia la DERECHA Proteínas de unión a cadena sencilla (SSB) mantienen las hebras separadas 4. La unión de SSB es cooperativa y ayuda a la polimerasa facilitando su actividad 5. Síntesis de cebador de ARN: Primasa Sin cebador NO hay síntesis de ADN CEBADOR de ARN DNA RNA 5’ 3’ molde La ADN polimerasa puede actuar 6. Síntesis de ADN por la POLIMERASA La síntesis se realiza en el sentido 5’ è 3’ ADN polimerasa (DNA)n+1 + PPi DNAn + dNTP Mg2+ • Cada cadena de DNA original sirve de molde para una cadena nueva (replicación semi-conservativa) • Los precursores son desoxiribonucleósidos 5’ TRI-fosfato (dNTPs: dATP, dTTP, dGTP, dCTP) (DNA)n+1 DNAn γ β α α γ dNTP β PPi ADN polimerasa III: REPLICASA La estructura de las polimerasas asemeja un mano que agarra el ADN y lo sujeta firmemente mientras va sintetizando la nueva cadena La fidelidad de la ADN polimerasa III es de 109 POLIMERIZANDO EDITANDO Actividad de polimerasa 5’ 3’ Actividad de exonucleasa 3’ 5’ Replicación bidireccional El cromosoma bacteriano tiene DOS horquillas de replicación activas Cada horquilla tiene una cadena que crece de manera continua y otra discontinua Ambas cadenas crecen en dirección 5’ 3’ La cadena continua se sintetiza en el mismo sentido de la apertura de la doble hélice Los fragmentos de OKAZAKI corresponden a la cadena cuyo crecimiento es opuesto al sentido de apertura Cadena GUIA (continua) Cadena RETRASADA (discontinua) Replisoma En cada horquilla de replicación, la ADN pol III es la que replica las dos hebras al mismo tiempo en sentido de 5’ 3’. La replicación de la hebra retrasada se interrumpe cada 1000 nt aproximadamente, por lo que se forman los fragmentos de OKAZAKI Subunidades de la ADN polimerasa III de E. coli Sub # por holoenzima Mr Función α 2 132,000 Actividad polimerasa ε 2 27,000 Exonucleasa 3’→5’ θ 2 10,000 Se requiere para la union de DnaB τ 2 71,000 γ 1 52,000 δ 1 35,000 δ’ 1 33,000 χ 1 15,000 ψ 1 12,000 β 4 37,000 Unión estable al molde, dimerización del núcleo Complejo que carga las subunidades β al DNA Pinzas que forman una rueda (abrazadera) sobre el DNA y aseguran óptima procesividad Núcleo de la polimerasa Fijalkowska et al., 2012 Cadena GUIA (continua) Cadena RETRASADA (discontinua) La ADN pol III es altamente procesiva gracias a dos subunidades β que forman una abrazadera para cada subunidad catalítica El complejo γ ayuda a posicionar una abrazadera sobre cada hebra de ADN complejo γ proteínas β (abrazadera) 1108 ADN polimerasas en E. coli Fig. 3. The five DNA polymerases of Escherichia coli and some of their relevant properties. I.J. Fijalkowska et al. Fijalkowska et al., 2012 7. ADN polimerasa I La DNA pol I rellena los espacios entre fragmentos de Okazaki Utiliza actividad exonucleasa 5’→3’ para eliminar cebador de RNA 8. ADN ligasa La DNA ligasa sella un enlace fosfodiester cuando ya no falta ningún nulceótido por añadir 9. Acción de la Girasa (Topoisomerasa tipo II) La helicasa causa superenrollamiento Enfrente de cada horquilla de replicación La Girasa actúa delante de la helicasa para aliviar tensión La Girasa corta una doble hélice, pasa la otra doble hélice por el corte (desenrolla) y sella Utiliza ATP Posición de la Girasa en cada horquilla 10. Terminación de la replicación bacteriana Opuesto al Origen hay secuencias de terminación (Ter) para cada horquilla que son reconocidas por proteínas Tus Nature, 2015 11. Resolución de concatémeros por una Topoisomerasa Topo I ADN polimerasa Topo I Topo IV Topo IV Replicación de ADN en EUCARIONTES ² Inicio ² Elongación ² Terminación Semi-conservativa Bi-direccional Cebadores de ARN DNA polimerasas Los cromosomas eucariontes tienen múltiples origenes de replicación La activación de los orígenes depende de proteínas del ciclo celular Las horquillas de replicación son similares a las bacterianas, pero …. Hebra Retrasada Topoisomerasa DNA ligasa I Rnasa HEN 1 DNA pol α Primasa Cebador ARN CDC 45 MCM 2 Helicasa RFC PCNA RFC Hebra Guía RPA (SSB) Topoisomerasa DNA pol δ PCNA ¿Qué pasa con los nucleosomas? Es MUY DIFERENTE a bacterias en complejidad estructural CROMATINA PARENTAL Se necesita el doble de Histonas para las cadenas hijas Chaperonas se encargan de posicionar los nucleosomas en las cadenas hijas Las modificaciones de histonas parentales se heredan a las nuevas (herencia epigenética) Modificación de Histonas en los nucleosomas parentales Solo la mitad de Histonas lleva la marca de modificación parental El patrón de modificaciones parentales se restablece mediante un complejo de lectura-escritura Solo así una célula epitelial dará lugar a más células epiteliales Replicación del ADN: en E. coli vs. células humanas Cantidad de DNA, pb/ cél. E. coli 3.9 106 Cél humanas 109 Velocidad avance horquilla µm/min 30 3 Velocidad de replicación, nt / seg 750 60-90 Número de orígenes de replicación / célula 1 103-104 Tiempo 1 replicación genómica (hs) 0.27 8 Tiempo 1 división celular (hs) 0.33 24 La terminación de la replicación eucarionte es un problema Cada uno de los dos extremos 5’ en cada cromosoma queda incompleto al remover el último cebador de ARN A mayor número de divisiones celulares, telómeros más cortos Solución: Extensión del Telómero (TELOMERASA) Extremo 5’ incompleto La Telomerasa es una ADN polimerasa que utiliza ARN como molde y extiende la misma secuencia varias veces El ARN es parte de la Telomerasa y es complementario a las últimas bases del ADN desapareado La ADN polimerasa sintetiza la cadena complementaria del telómero extendido. Telomerasa Ribonucleoproteína: RNA proteína Los telómeros indican la edad celular de células somáticas