downloading - genoma . unsam . edu . ar
Anuncio

GENETICA BACTERIANA
PROCARIOTAS
Fáciles y rápidos de crecer
Rápida expansión clonal
PROCARIOTAS
PROCARIOTAS
PROCARIOTAS
Genética mas simple que eucariotas
Genomas haploides compactos.
La información genética puede encontrarse en el genoma o plásmidos
No hay intrones
No hay poliadenilación de mensajeros
Muchas genes que pertenecen a una misma vía están organizados en
operones
Gran plasticidad para “mover” y/o adquirir material genético
ALGUNOS TERMINOS
Genotipo
Fenotipo
Mutante
Resistencia antibiótica
Clon
Auxótrofa
UN POCO DE HISTORIA…….
EXPERIMENTO DE LURIA-DELBRUCK
Origen de las mutaciones: previo a la adición del agente
inactivante o posterior a el (adaptativa)?
PLASMIDOS
Unidades genéticas autónomas que se encuentran presentes
en casi todas las bacterias
Escenciales en la capacidad adaptativa o evolutiva de las
bacterias
PLASMIDOS
Replicación
Theta: primer de ARN (Uni o
Bidireccional)
Círculo rodante: Nick en el ADN que
funciona como primer
EN TODOS LOS CASOS LA REPLICACION SE
INICIA EN EL ORI (ORIGEN DE REPLICACION)
PLASMIDOS: replicación
PLASMIDOS
Propiedades: Rango de huésped
Número de copias
Capacidad de movilización
Rango de huésped
Amplio
Acotado
PLASMIDOS
Número de copias: es el número de unidades de
plásmido por célula
Altamente regulado
Regulación por ARN antisentido
Regulación por ARN antisentido
y proteína
Regulación por iterones
PLASMIDOS
Regulación por ARN antisentido
RNAII: inicia la síntesis
del ADN
RNAI: es complemenario
a RNAII
Duplex RNAI-RNAII es
degradado
PLASMIDOS
Regulación por ARN antisentido y proteína
RepA: proteína de replicación
CopB: reprime a PrebA. Ocurre
inicialmente luego de lo cual RepA
se sintetiza desde el transcripto
copB-repA
CopA: ARN antisentido que afecta
la estabilidad del transcripto
copA-repA
RNAseIII cliva el duplex copA-repA
Incompatibilidad de plásmidos
Dos plásmidos pertenecen al mismo grupo de incompatibilidad (Inc)
cuando uno interfiere con la replicación o la partición del otro
Si coexiste
Si no pueden
coexistir
Diferentes grupos de incompatibilidad
Mismo grupo de incompatibilidad
Incompatibilidad de plásmidos
Diferente grupo Inc
Mismo grupo Inc
INTRODUCCION DE MATERIAL GENETICO EN
BACTERIAS
CONJUGACION
TRANSFORMACION
TRANSDUCCION
CONJUGACION
Capacidad para intercambiar material genético
(en general plásmidos) entre bacterias
SEXO
DADOR Y ACEPTOR
TRANSCONJUGANTE
CONJUGACION
Pilus: producido por
el dador
CONJUGACION
Plásmidos
Movilizables: no codifican todas las
funciones
Transmisibles per ser: codifican
para todas las funciones
CONJUGACION
oriT: origen de
transferencia
Región de clivaje
del ADN
El ADN que se
transfiere es simple
cadena
CONJUGACION
Espacio extracelular
}
citoplasma
pilus: polímero de la
pilina que esta
codificada en un único
gen
CONJUGACION
Genes de transferencia: encargados del transporte de ADN
Helicasas
Primasas
Endonucleasas
}
Encargadas de cortar,
desenrrollar y transportar el
ADN
CONJUGACION
Plásmidos transferibles: codifican para la sintesis del pilus y de
todas las proteínas necesarias para la transferencia
Plásmidos movilizables: no codifican para la sintesis del pilus y por
lo tanto deben usar el pilus sintetizado por otro plásmido. En
principio solo necesitan el oriT. Naturalemente no existen plásmidos
que solo contengan el oriT, los plasmidos movilizables contienen los
genes mob encargados de la transferencia y similares a los genes tra
CONJUGACION
Plásmido F: transferible
Capaz de integrarse al
cromosoma por
recombinación homóloga
en las regiones IS
Que ocurre si un plásmido F integrado al cromosoma
intenta transferirse?
CEPA Hfr
Cepas Hfr
Movilizan el cromosoma
Si no hay
recombinación la
información
genética no se
hereda
UTILIZACION DE CEPAS HFR EN LA DETERMINACION
DEL MAPA DEL GENOMA DE E. COLI
Conjugación de:
Hfr a+ b+ c+ d+ e+ Strs
X
F-
a- b- c- d- e- Strr
Producto de conjugación
(Se corta la conjugacion a distintos tiempos
Por agitación)
Plaqueo en medio con estreptomicina
y todos los aa menos:
a
b
c
d
e
CONJUGACION
Biparental: en la misma intervienen dos cepas
Que ocurre si, ni la cepa dadora del plásmido ni el plásmido,
poseen las funciones tra?
Triparental: en la misma intervienen tres cepas
CONJUGACION TRIPARENTAL
Una cepa actúa como
“helper” o ayudante
TRANSFORMACION
Proceso por el cual el ADN libre se incorpora en una célula
receptora y lleva a cabo un cambio genético heredable
Para ello las bacterias deben ser competentes
Naturalmente transformable: bacterias naturalmente
competentes capaces de tomar ADN del ambiente (Bacillus
subtilis, Haemophilus influenza, Streptococcus pneumoniae)
En el proceso de transformación natural intervienen proteínas de
membrana que asocian ADN, autolisinas de pared celular y
nucleasas
TRANSFORMACION
Experimento de Griffith: descubrimiento de la transformación
Presencia de la cápsula para patogenicidad: R vs S
TRANSFORMACION
Si la bacteria no es naturalmente competente se puede inducir
el estado de competencia
Competencia por cloruro de calcio o electrocompetencia por
ejemplo
Herramientas de la biología molecular
Transformación con
plásmidos
Replicativo
Complementación
No replicativo (suicida)
Eficiencia de
= número de colonias
Transformación
µg ADN
Recombinación
Complementación
Mutación
TRANSFORMACION
Aunque el evento sea de
baja eficiencia la selección
es muy poderosa
(antibióticos)
BACTERIOFAGOS
Virus de bacterias
(especie específicos)
Parásitos que explotan la
maquinaria replicativa del
huésped
Placas de lisis
BACTERIOFAGOS
Vienen en muchos colores y tamaños
BACTERIOFAGO T4
Ciclo de vida: lítico vs lisogénico
lítico
lisogénico
Equilibrio que se altera por
factores ambientales
Determinado por la
competencia entre el
activador CII y la proteína Cro
Activa
CII
Cro
Activa
CI (represor
de Cro)
Genes
cascada lítica
Ciclo de multiplicación
Genes tempranos:
sintetizados por la ARN pol
bacteriana (ARN
polimerasa, primasas,
ligasas y helicasas)
Acumulación de genomas
del fago
Genes tardíos: proteínas
estructurales del fago
Empaquetamiento del
genoma, ensamble del fago
y lisis
Transducción
Movimiento de material genético através de un fago
Transducción generalizada: se transfiere cualquier región del
cromosoma
Transducción especializada: se transfieren las regiones cercanas al
sitio de integración en los fagos lisogénicos
Transducción generalizada
Una baja proporción de los
fagos “se llevan” información
de la bacteria
SELECCION
Transducción especializada
Profagos lisogénicos
Transducción especializada
Utilización de fagos en estudios de genética bacteriana
Estudio sobre la función de un determinado
gen en virulencia
Cepa no motil
Cepa motil
Avirulenta
Virulenta
Obtenida en un
estudio sobre
metabolismo
Cepa utilizada
en estudios de
virulencia
Resistencia a Km
Como se hace en el laboratorio?
Cepa A: dadora
Cepa B: receptora
infección con
el fago
centrífuga
A
Infección del
cultivo aceptor
Aislamiento
de la mutante
B
Plaqueo en
medio con
kanamicina
Regulación de la expresión génica
COMO Y BAJO QUE CONDICIONES LOS GENES SE
EXPRESAN O NO
Bacterias: sistemas simples que han servido de marco para el
estudio de regulación en eucariotas superiores
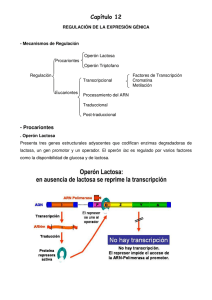
Regulación transcripcional
A nivel del inicio de la
transcripción
Regulación post-transcripcional
Estabilidad del mensajero o
regulación de la traducción
Estructura canónica de un promotor bacteriano
Sitio de union a
ribosoma o shinedalgarno
Factores sigma
Son factores de iniciación de la transcripción de procariotas
Le aportan la especificidad de promotor a la ARN polimerasa
Los diferentes factores sigma se activan en respuesta a
diferentes condiciones ambientales
Factores sigma
Escherichia coli
σ70: housekeeping
σ54: limitación de nitrógeno
σ38: fase estacionaria
σ32: heat shock
σ28: sistema flagelar
σ24: extracitoplasmática, temperaturas extremas
σ19: transporte de hierro
Operon
Genes de una misma vía metabólica que se encuentran en un
mismo policistrón
Regulación positiva y negativa
Regulación positiva y negativa
Operón catabólico: degradación de componentes
Operón anabólico: síntesis de compuestos escenciales
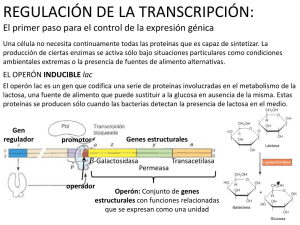
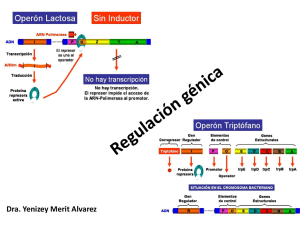
Operón lactosa
Regulación negativa
Jacob y Monod (1950): estudio sobre la utilización de
lactosa por Escherichia coli
glucosa lactosa
Jerarquía de
utilización
Curva de crecimiento con
glucosa y lactosa
Operón lactosa
lacZ: β-galactosidase
lacY: permease
lacA: acetylase
lacI: represor
Represión catabólica
AMPc: AMP cíclico es
una señal de “hambre”
En presencia de glucosa
el operón está
“apagado”
Operón biosintético
Regulación negativa
Operón triptofano
Otros niveles de regulación
Autoregulación del represor
Atenuación
Autoregulación negativa del represor
El represor TrpR regula negativamante la expresión de su
propio transcripto
Mayor velocidad de represión cuando aparece el
triptofano
Atenuación
Mutantes en trpR aún regulan la transcripción
del operón en respuesta a triptofano
Nivel extra de regulación
Atenuación
Operón arabinosa
Operón de degradación de la arabinosa
Regulación positiva por un activador
Arabinosa
Activador
AraC
Xilulosa-5-fosfato
Operón arabinosa
AraC funciona como un activador y un anti-activador
dependiendo si tiene arabinosa unida o no
arabinosa
AraC
AraC
P1
Anti-activador
AraC AraC
P2
Activador
Operón arabinosa
El sistema le otorga un alto nivel de regulación. Hay
muy baja expresión basal
Posee, al igual que el operón lac, represión catabólica.
La ausencia de glucosa potencia la expresión
Operón galactosa
Operón que se reprime en ausencia de galactosa
En presencia de galactosa se desreprime el operón
Las zonas regulatorias se encuentran duplicadas
Otros operones
Operón maltosa: adquisicion de maltodextrinas y maltosa y su
utilización
Operón histidina: síntesis de histidina
Operón tol: degradación de compuestos hidrocarbonados
cíclicos
Riboswitches
Mecanismo de regulación de la expresión génica
evolutivamente ancestral
Utiliza la estructura secundaria del ARN
Mecanismo post-transcripcional
Riboswitches
Dos tipos
De terminación de la transcripción
De inicio de la traducción
Existen diferentes y muy variados tipos de riboswitches
Como hace una bacteria para responder a una señal
ambiental?
{
Falta de nutrientes
Señal ambiental
Cambios de temperatura
Presencia de un posible huésped
bacteria
Respuesta adaptativa
Sistemas de dos componentes
Sistemas ampliamente distribuidos en bacterias
que median la respuesta transcripcional a factores
o cambios ambientales
Componente de detección: proteína de membrana que detecta
el estímulo y fosforila al componente de respuesta
Componente de respuesta: proteína citoplamática que activa o
reprime la actividad transcripcional de los genes de respuesta
Sistemas de dos componentes