Ingeniería de proteínas - Departamento de Biotecnología
Anuncio
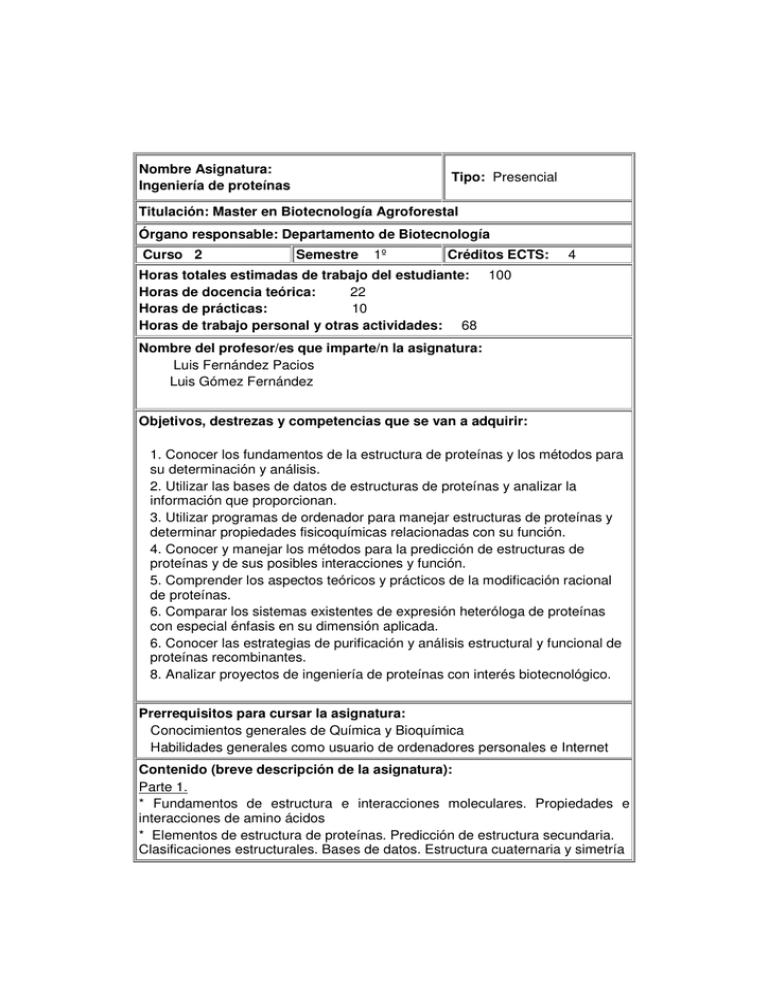
Nombre Asignatura: Ingeniería de proteínas Tipo: Presencial Titulación: Master en Biotecnología Agroforestal Órgano responsable: Departamento de Biotecnología Curso 2 Semestre 1º Créditos ECTS: 4 Horas totales estimadas de trabajo del estudiante: 100 Horas de docencia teórica: 22 Horas de prácticas: 10 Horas de trabajo personal y otras actividades: 68 Nombre del profesor/es que imparte/n la asignatura: Luis Fernández Pacios Luis Gómez Fernández Objetivos, destrezas y competencias que se van a adquirir: 1. Conocer los fundamentos de la estructura de proteínas y los métodos para su determinación y análisis. 2. Utilizar las bases de datos de estructuras de proteínas y analizar la información que proporcionan. 3. Utilizar programas de ordenador para manejar estructuras de proteínas y determinar propiedades fisicoquímicas relacionadas con su función. 4. Conocer y manejar los métodos para la predicción de estructuras de proteínas y de sus posibles interacciones y función. 5. Comprender los aspectos teóricos y prácticos de la modificación racional de proteínas. 6. Comparar los sistemas existentes de expresión heteróloga de proteínas con especial énfasis en su dimensión aplicada. 6. Conocer las estrategias de purificación y análisis estructural y funcional de proteínas recombinantes. 8. Analizar proyectos de ingeniería de proteínas con interés biotecnológico. Prerrequisitos para cursar la asignatura: Conocimientos generales de Química y Bioquímica Habilidades generales como usuario de ordenadores personales e Internet Contenido (breve descripción de la asignatura): Parte 1. * Fundamentos de estructura e interacciones moleculares. Propiedades e interacciones de amino ácidos * Elementos de estructura de proteínas. Predicción de estructura secundaria. Clasificaciones estructurales. Bases de datos. Estructura cuaternaria y simetría * Determinación experimental de estructuras de proteínas. PDB (Protein Data Bank). Manejo por ordenador de estructuras PDB * Superficies moleculares de proteínas. Potencial electrostático. * Predicción y diseño de estructuras de proteínas. Optimización. Mecánica Molecular. Predicción ab initio de estructura terciaria. Modelado por homología. Simulación: Dinámica Molecular. Parte 2. * Expresión heteróloga de proteínas. Parámetros relevantes y su optimización. El problema de la agregación. * Modificación racional de proteínas. Mutagénesis dirigida y métodos combinatorios. Modificación química. Proteínas quiméricas y multifuncionales. * Purificación de proteínas recombinantes. Métodos high-throughput. Análisis de propiedades estructurales y funcionales. * Aspectos de interés biotecnológico. Mejora de propiedades estructurales Mejora de propiedades catalíticas. Análisis de casos prácticos. Metodología docente: Clases teóricas con apoyo de medios informáticos y visuales Clases prácticas en aula de informática: manejo de los programas necesarios Exposición y discusión en grupo de publicaciones científicas de interés Elaboración de un proyecto de ingeniería de proteínas Tipo de evaluación: (exámenes/ trabajos/ evaluación continua) Evaluación continua en discusiones en grupo y clases prácticas con ordenador Trabajo final individual sobre un problema específico para cada alumno Idioma en que se imparte: Español e inglés Observaciones: Bibliografía 1. Carl Branden, John Tooze, Introduction to Protein Structure, 2nd. Edition, Garland Publishing, New York, 1999 2. Anna Tramontano, Protein Structure Prediction. Concepts and Applications, Wiley-VCH, Weinheim, 2006 3. Torsten Schwede, Manuel Peitsch, Computational Structural Biology. Methods and Applications, World Scientific Publishing Co., Singapore, 2008 4. Protein Engineering Protocols, Methods in Molecular Biology, Vol. 352, Humana Press, USA, 2006