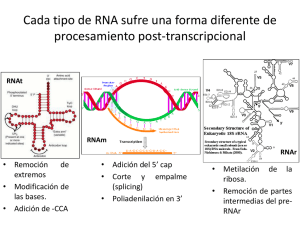
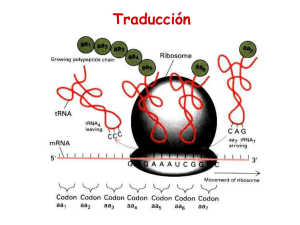
Transcripción y Procesamiento de rRNA y tRNA
Anuncio

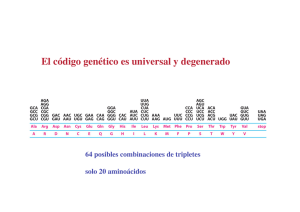
Transcripción y Procesamiento de rRNA y tRNA Organización de los genes de rRNA Los rRNAs se encuentran altamente conservados ITS rRNA Procarionte 3´ ETS 5´ ETS 16S tRNA 5S 23S rRNA Eucarionte NRPA! ITS1 5´ ETS 18S ITS2 5.8S 3´ ETS 25S / 28S 3´ ETS NRPC! 5S ETS: External transcribed spacer ITS: Internal Transcribed spacer Nat Rev. Mol. Cell Biol. 2:7:514-20 Transcripción por RNA polimerasa I Organización de repetidas en tandem (DNA) Unidad transcripcional (DNA) Transcrito precursor (45S) rRNA maduros Procesamiento POST-transcripcional derRNA rRNAProcariontes Procesamiento post-transcripcional de PROCARIONTES 16S 30S tRNA 23S 5S Exonucleasa= Endonucleasa= 16S tRNA 23S 5S Nat Rev. Mol. Cell Biol. 2:7:514-20; WIREs RNA 2015, 6:225–24 Procesamiento POST-transcripcional derRNA rRNA Eucariontes Procesamiento post-transcripcional de S. cerevisiae 18S 35S 5.8S Mamíferos EUCARIONTES 25S 5.8S 18S 47S 28S 45S 33S 41S 20S 27S 32.5S 21S 32S 26S 12S NÚCLEO CITOPLASMA 18S 6S 5.8S 25.5S 25S 28.5S 18S 18S 6S 5.8S 28S En eucariontes ribonucleoproteínas nucleolares Procesamiento POST-transcripcional depequeñas rRNA (snoRNPs) establecen modificaciones químicas en el rRNA (45S) durante su Participación de RNAs pequeños nucleolares (snoRNAs) para las modificaciones químicas de bases procesamiento. Box D´ Box C´ C/D box snoRNA Box C H/ACA snoRNA Box D 5´ 3´ ψ ψ 3´ rRNA 5´ 3´ CH3 CH3 3´ rRNA rRNA 5´ 3´ 5´ 5´ Box H Box ACA ANANNA ACA 3´ La Biogénesisel procesamiento de los Ribosomas un proceso En eucariontes de rRNA es y ensamble de sistemático y complejo ribosomas ocurre en el núcleo Pol I NUCLEOLO Cluster de rDNA Transcripción Pol III Pol II 5S snoRNA Pre-rRNA 47S Procesamiento 18S 5.8S 28S 18S 5.8S 28S m 7Gcap rRNA 5S snoRNA Modificación Proteínas ribosomales (RPs) y Factores de ensamblaje (AFs) AAn Ensamblado CITOPLASMA Pre-40S Pre-60S Transporte RanGTP Pre-40S NUCLEOPLASMA RanGTP Pre-60S Modificado de doi:10.1038/nsmb.2939 Muy pocos genes se encuentran tan altamente conservados como el rRNA16S (18S). Conservación de la secuencia y esructura secundaria de Aun cuando no se conoce con exactitud la tasa de cambios (tasa de mutación) en la secuenciaa del gen. Esde ampliamente utilizado como marcador de las distancias y rRNA lo largo la evolución relaciones evolutivas de los organismos rRNA%16S rRNA%16S Eubacteria Arqueobacteria rRNA%18S Eucarionte rRNA%23S rRNA%25S Composición deRibosoma los ribosomas procarionte y eucarionte Composición Eucarionte y Procarionte En el ribosoma ocurre la síntesis de proteínas Los procariontes tienen un ribosoma 70S Formado por 2 subunidades: - Grande (mayor) de 50S - Pequeña (menor) de 30S 70S Los Eucariontes tienen un ribosoma 80S Formado por 2 subunidades: - Grande (mayor) de 60S - Pequeña (menor) de 40S 80S 50S 30S 60S 40S - rRNA 23S - rRNA 5S - 34 proteínas - rRNA 16S - 21 proteínas - rRNA 25/28S - rRNA 5.8S - rRNA 5S - ~49 proteínas - rRNA 18S - ~33 proteínas La estructura de los ribosomas es muy parecida entre procariontes y eucariontes. El tamaño de los rRNAs y el número de proteínas en las subunidades ribosomales varía entre procariontes y eucariontes. ¿Por qué? Transcripción de tRNA por RNA pol III Los promotores de Clase III (RNA pol III) se distinguen por ubicarse dentro de la región que se transcribe (entre +41 y +87) Unidades transcripcionales de tRNAs y rRNA 5S, otros snRNAs El complejo de RNA pol III es el mas grande y pesa 700 kDa Procesamiento POST-transcripcional del tRNA 1. 2. 3. 4. Extremo 5’: Corte por RNasa P (RIBOZIMA) Extremo 3’: Corte por RNasa D (proteína) y adición de CCA Remoción de intrón Modificación de bases tRNA maduro: estructura de trébol • 75-­‐90 ribonucleoPdos. • ConPene bases modificadas. Brazo aceptor Brazo TψC Brazo D Brazo Variable Brazo an(codón Plegamiento espacial del ARNt • El ARNt se pliega sobre sí mismo para adoptar una estructura 3° con forma de L inverPda. • En un extremo queda el brazo aceptor y en otro el brazo anPcodón. Bases modificadas se encuentran los tRNA Bases modificadas queque se encuentran en losen ARNt Bases normales Bases modificadas Uridina Ribotimidina (T) Citidina 3-metilcitidina 5-metilcitidina Adenosina Guanosina Inosina Dihidrohuridina (D) Pseudouridina (⍦) Metiladenosina 7-metilguanosina Queuosina (Q) 4-Tiouridina Isopentiladenosina Wyosina (Y) Bases modificadas que se encuentran en los tRNA Alrededor del 12% de los residuos de tRNAs sufren modificaciones (aprox 8 modificaciones por tRNA. Se han identificado más de 100 modificaciones diferentes Posición Modificación en tRNA Función 9 m1G Plegamiento de tRNA 54 m5U y rT Estabilidad de tRNA 1, 29, 30, 35, 36 Ψ Desconocida 64 2ʹ-O-ribosyladenosine Discriminación entre tRNAMet iniciador y elongador 16, 17 y 47 D Desconocida Modificado de doi:10.1038/nrg3861 Función del tRNA en traducción: intérprete AA específico (Aminoácido) mRNA XYZ El AA debe ser adaptado para reconocer el codón XYZ Solución: El RNA de transferencia como Adaptador a) Apareamiento de bases con el codón Interacción Codón | Anticodón b) Reconocimiento específico El Código Genético El código genético Es una serie de reglas que definen como los 4 nucleótidos en el mRNA se traducen en en un código de 20 aminoácidos, los cuales conforman las proteínas. Tres letras: un aminoácido Consiste en la combinación de tres letras (nucleótidos) llamados codones; cada uno Cuatro aletras en total, ordenadas en tripletes: corresponde un aminoácido específico o una señal de paro. 64 posibilidades A U El Código Genético No es sobrelapado C G U C U U A C A U Tres bases codifican Un aminoácido El permite la traducción fidedigna de RNA a Proteína identificando El Código CódigoGenético Genético permite la traducción fidedigna de ARN a las bases por tripletes para cada aminoácido Proteína identificando lasincorporar bases por tripletes para incorporar No polar cada aminoácido Aromáticos G A C U G A C U G A C U G A C U U C A G UC A UC G G UC A C U G A G A C AG U C G U A C C UG A U U G A C U GA C Polar sin carga A G U Polar carga + C A G U C G A C U G A G U C A G U C A G U C A Polar carga - Modificado;de;doi:10.1038/nchembio847 El código genético es degenerado: Hay 64 posibles combinaciones de tripletes, pero solo 20 aminoácidos; Más de un triplete por aminoácido El código genético NO es ambiguo: Un triplete solo puede significar un aminoácido Interacción codón-anticodón Interacción codón-anticodón tRNA Las cadenas que interaccionan son ANTIPARALELAS La 3ª posición del CODON puede no aparear (hipótesis del “bamboleo”) Las bacterias tienen 31 diferentes tRNA Los eucariontes tienen 48 Base en codón en Posibles bases posición de en anticodón bamboleo U A, G o I C Anticodón Base en codón en Posibles bases posición de en anticodón bamboleo U A, G o I GoI C GoI A UoI A U G CoU G G 3´ 5´ Posición de bamboleo 5´ 3´ Codón mRNA El carboxilo del aminoácido forma un enlace ester con la ribosa 3’ del tARN Aminoacilación del tRNA por AARS 1ATP 1. Formación de aminoacil-adenilato 2. Síntesis de aminoacil-tRNA Activación del aminoácido antes de su unión al tRNA. Esta activación se realiza por la aminoacil tRNA sintetasa Aminoacil Adenilato (aminoacil AMP) (AARS) y ATP para dar lugar a un Las AARS son específicas para cada aminoácido Fidelidad del código genético! Las AARS tienen dos mecanismos de corrección: para el aminoácido y para el ARNt Este mecanismo de edición reduce los errores a 1 por cada 40,000 aminoacilaciones ¿Cómo dsicriminar entreentre aminoácidos similares? ¿Cómo discriminar aa muy parecidos? Val Ile Leu = VIL Aminóacidos de cadena ramificada (Branched Chain Aminoacids; BCAA) Son muy frecuentes en todas las proteínas, se utilizan como suplemento alimenticio Aminoácidos similares pasan por un doble filtro en las aa-tRNA Aminoácidos similares pasan por un doble filtro sintetasas El sitio de edición es más pequeño que el catalítico Sitio Catalítico Sitio de edición Isoleucil – tRNA sintetasa Leu Leu es demasiado grande para el sitio activo de síntesis Ile Ile cabe en el sitio de síntesis pero no en el de edición Val Val cabe en el sitio de síntesis y en el de edición por lo que es eliminado Garantiza la fidelidad del código genético Hay dos clases de AARS I II Clase I: aminoacila primero el 2’OH y luego transesterifica al 3’OH de la ribosa Clase II: aminoacila directamente el 3’OH de la ribosa Si la secuencia del mRNA se lee por tripletes existen tres posibles marcos de lectura. ¿Cómo saber cuál de los tres es el correcto? ¡ Se define por el codón de inicio AUG ! Marco abierto de lectura (ORF; open reading frame) Policistrónico (varios ORFs) Monocistrónico (1 ORF) Tenemos la siguiente secuencia de DNA en la que se señala el sitio de inicio de la transcripción (Inr) y la dirección (de izquierda a derecha). Inr 5’ CAACGATGCCATCAGAGCCCAGGACGTGATTTAA 3’! 3’ GTTGCTACGGTAGTCTCGGGTCCTGCACTAAATT 5’! C a) Escribe la secuencia de mRNA transcrita indicando los extremos 5’ y 3’. b) Ubica el sitio donde comienzan a leerse los tripletes de codones y deduce la secuencia de aminoácidos de la proteína que se traduce. Señala los extremos amino (NH2) y carboxilo (COOH) de la proteína. c) Si se produce una mutación por inserción de C en el sitio indicado con la flecha roja, ¿Cuál será la secuencia de aminoácidos?