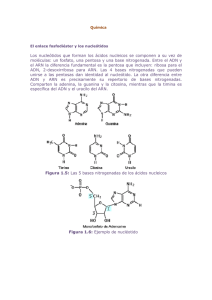
plantas resistentes a los virus que tiene un arn inverso.
Anuncio

k OFICINA ESPAÑOLA DE PATENTES Y MARCAS 19 k ES 2 076 143 kInt. Cl. : C12N 15/00 11 N.◦ de publicación: 6 51 ESPAÑA A01H 1/00 C12N 5/00 C12N 15/11 C12N 15/82 k TRADUCCION DE PATENTE EUROPEA 12 kNúmero de solicitud europea: 87302827.8 kFecha de presentación : 01.04.87 kNúmero de publicación de la solicitud: 0 240 332 kFecha de publicación de la solicitud: 07.10.87 T3 86 86 87 87 k 54 Tı́tulo: Plantas resistentes a virus que tienen ARN antisentido. k 73 Titular/es: Pioneer Hi-Bred International, Inc. k 72 Inventor/es: Loesch-Fries, Sue L.; k 74 Agente: Carpintero López, Francisco 30 Prioridad: 02.04.86 US 847425 700 Capital Square 400 Locust Street Des Moines Iowa 50309, US 45 Fecha de la publicación de la mención BOPI: 01.11.95 45 Fecha de la publicación del folleto de patente: 01.11.95 Aviso: k k Merlo, Donald J. y Jarvis, Nancy P. k En el plazo de nueve meses a contar desde la fecha de publicación en el Boletı́n europeo de patentes, de la mención de concesión de la patente europea, cualquier persona podrá oponerse ante la Oficina Europea de Patentes a la patente concedida. La oposición deberá formularse por escrito y estar motivada; sólo se considerará como formulada una vez que se haya realizado el pago de la tasa de oposición (art◦ 99.1 del Convenio sobre concesión de Patentes Europeas). Venta de fascı́culos: Oficina Española de Patentes y Marcas. C/Panamá, 1 – 28036 Madrid 1 ES 2 076 143 T3 DESCRIPCION Campo La presente invención pertenece al campo de la ingenierı́a genética y la agricultura, y proporciona especialmente unos medios para producir una planta resistente a virus por transformación de una planta para contener un gen extraño expresable en plantas que dirige la sı́ntesis de ARN complementariamente a un ARNm viral. También se proporcionan unos vectores plásmidos procarióticos que transforman plantas que llevan tal ARN expresable en plantas y las células de planta transformadas por tal vector. Antecedentes Visión de conjunto de la Agrobacterium Las cepas virulentas del género gram-negativo de Agrobacterium hospedan grandes plásmidos conocidos como plásmidos Ti (inductores de tumor o transformación) (pTi) en A. tumefaciens y plásmidos Ri (inductores de raı́z) (pRi) en A. rhizogenes, clasificados frecuentemente por la opina que catabolizan o cuya sı́ntesis causan. Tanto el plásmido Ti y como el Ri contienen secuencias ADN conocidas como ADN-T (ADN-transferido), las cuales, en tumores se encuentran integradas en el genoma de la planta huésped. Varios genes de ADN-T están bajo el control de promotores ADNT que se asemejan en estructura a promotores eucarióticos canónicos. Estos plásmidos también llevan genes fuera de la región de ADN-T. Los plásmidos Ti y Ri son funcionalmente equivalentes para muchos propósitos. Análisis de enfermedades causadas por Agrobacterium, transformación de plantas, ingenierı́a genética, y expresión de gen, incluyen aquellos producidos por, o encontrados en, Merlo DJ (1982) Avd. Plant Pathol. 1:139-178; Ream LW y Gordon MP (1982) Science 218:854-859; Bevan MW y Chilton M-D (1982) Ann. Rev. Genet. 16:357-384; Kahl G y Schell J (1982) Molecular Biology of Plant Tumors; Barton KA y Chilton M-D (1983) Meth. Enzymol. 101:527-539; Depicker A y col. (1983) en Genetic Engineering of Plants: an Agricultural Perspective, eds.: Kosuge T y col., pp. 143-176; Caplan A y col. (1983) Science 222:815-821; Hall TC y col., solicitud de Patente Europea 126.546; Binns AN (1984) Oxford Surveys Plant Mol. Cell Biol. 1:130160; Hall TC (1985) Oxford Surveys Plant Mol. Biol. 2:329-338; Hooykaas PJJ y Schilperoort RA (1985) Trends Biochem. Sci. 10:307-309; Thomas TL y Hall TC (1985) Bioassays 3:149-153; Weissbach A y Weissbach H, eds. (1986) Meth. Enzymol. 118 (ver especialmente Rogers SG y col., pp. 627-640); Pühler A, ed. (1983) Molecular Genetics of the Bacteria-Plant Interaction; y Schilperoort RA (1984) en Efficiency in Plant Breeding (Proc. 10th Congr. Eur. Assoc. Res. Plant Breeding), eds.: Lange W y col., pp. 251-285. Transformación de Plantas por Agrobacterium Las células de Plantas pueden ser transformadas por Agrobacterium por varios procedimientos conocidos en la técnica. Para un análisis del trabajo reciente, véase Syono K (1984) Oxford Surveys Plant Mol. Cell Biol. 1:217-219. La infección de tejido de planta por Agrobac2 5 10 15 20 25 30 35 40 45 50 55 60 65 2 terium es una técnica simple bien conocida por aquellos expertos en la técnica. Tı́picamente, después de hacerle una herida, una planta se inocula con una suspensión de la bacteria. Alternativamente, se inoculan trozos de tejido, por ejemplo discos de hoja (Horsch RB y col. (1985) Science 227:1229-1231). Después de la inducción con Agrobacterium de tipo salvaje, los tumores son capaces de crecimiento independiente de fitohormonas. La inoculación tradicional y las técnicas de cultivo, pueden ser modificadas para utilizar vectores ADN-T desactivados incapaces de crecimiento independiente de hormonas. (por ej. véase Zambyski P y col.(1984) en Genetic Engineering, Principles, and Methods, 6, eds.: Hollaender A y Setlow J, pp. 253-278). La Agrobacterium es además capaz de infectar células aisladas, células cultivadas en cultivos, células de tejido leñoso cicatrizal, y protoplastos aislados (por ej. Fraley RT y col. (1984) Plant Mol. Biol. 3:371-378: Fraley RT y Horsch RB (1983) en Genetic Engineering of Plants: an Agricultural Perspective, eds.: Kosuge T y col., pp. 177-194; Muller A y col. (1983) Biochem. Biophys. Res. Comm. 123:458-462). La frecuencia de transformación de trozos de tejido leñoso inoculados puede incrementarse por la adición de una opina o de precursores de opina (Cello LM y Olsen WL, Patente U.S. 4.459.355). El intervalo de huésped de patogenesis de agalla de copa puede ser influido por funciones codificadas ADN-T tales como genes onc (Hoekema A y col. (1984) J. Bacteriol. 158:383385: Hoekema A y col. (1984) EMBO J. 3:30433047: Buchholz WC y Thomasshow MF (1984) 160:327-332; Yanofsky M (1985) Mol. Gen. Genet. 201:237-246). Los genes vir también afectan el intervalo huésped (Yanofsky, véase más arriba). Ausich RL, Solicitud de Patente Europea 108.580, informa la transferencia de ADN-T a partir de A. tumefaciens a células de algal verde, y la expresión en ellas de genes de resistencia a la kanamicina Tn5 y ocs. Hooykaas-van Slogteren GMS y col. (1984) Nature 311:763-764, y Hernalsteens J-P y col. (1984) EMBO J. 3:3039-3041, han demostrado la transformación de células monocotiledóneas por Agrobacterium sin la tumorigenesis acostumbrada. El ADN-T, el ADN-T desactivado, y los genes extraños funcionales de plantas transformadas, son normalmente transmitidos a la progenie a través de meiosis aparentemente inalterados de forma Mendeliana dominante, unidos próximamente, (por ej. véanse Horsh RB y col. (1984) Science 223:496-498; Tepfer D (1984) Cell 37:959-967; DeBlock M y col. (1984) EMBO J. 3:1681-1689; Wöstemeyer A y col. (1984) Mol. Gen. Genet. 194:500-507; Wallroth M y col. (1986) Mol. Gen. Genet. 202:6-15). Dos ADN-T no unidos pueden transformar una célula simple y, después de la regeneración de la planta, segregar en la generación F1 (de Framond AJ y col. (1986) Mol. Gen. Genet. 202:125-131) ADN plásmido Ti El ADN-T está integrado frecuentemente (es 3 ES 2 076 143 T3 decir insertado) en el ADN huésped en múltiples posiciones en el núcleo. El ADN de planta lindante puede o bien estar repetido o en bajo número de secuencia de copias. El ADN-T integrado puede encontrarse en disposiciones tándem directas o invertidas, y puede separarse por espaciadores. El ADN-T puede transformar también cloroplastos (De Block M y col. (1985) EMBO J. 4:1367-1372; véase análisis por Flavell RB (1985) Bioessays 3:177-178). Ha sido descrita la secuencia completa del ADN-T de un plásmido tipo octopina encontrado en ATCC 15955, pTi15955, (Barker RF y col. (1983) Plant Mol. Biol. 2:335-350) como que tiene la región TL de pTiAch5 (Gielen J y col. (1984) EMBO J. 3:835-846). Los genes de ADN-T publicados, no contienen intrones. Pueden reconocerse secuencias que parecen elementos promotores eucarióticos canónicos y posiciones de poliadenilación. Los plásmidos Ti octopina llevan un gen ocs que codifica la octopina sintasa (lisopina deshidrogenasa). Koncz C y col. (1983) EMBO J. 2:1597-1603 proporciona un análisis funcional de ocs. Dhaese P y col. (1983) EMBO J. 2:419-426, informa de la utilización de varias posiciones de poliadenilación por “transcrito 7” (ORF3 Barker R y col., véase más arriba) y ocs. La presencia de la enzima octopina sintasa dentro de un tejido puede proteger ese tejido del efecto tóxico de varios análogos de aminoácido, por ej. aminoetil cisteı́na (Dahl GA y Tempe J (1983) Theor. Appl. Genet. 66:233-239; Koziel MG y col. (1984) J. Mol. Appl. Genet. 2:549-562). Los plásmidos Ti nopalina codifican el gen de la nopalina sintasa (nos) (secuenciado por Depicker A y col. (1982) J. Mol. Appl. Genet. 1:561573). Shaw CH y col. (1984) Nucl. Acids Res. 12:7831-7846, proporciona un análisis funcional de nos. Se han identificado genes equivalentes a tms y tmr sobre un plásmido tipo nopalina (Willmitzer L y col. (1983) Cell 32:1045-1056). Los genes de plásmido Ri y Ti fuera de la región ADN-T incluyen los genes vir, los cuales cuando mutan dan como resultado un plásmido Ti avirulento. Los genes vir funcionan en trans, siendo capaces de causar la transformación de células de planta con ADN-T de un tipo de plásmido diferente y localizado fı́sicamente en otro plásmido. Tales disposiciones se conocen como sistemas binarios y los plásmidos que soportan ADN-T se conocen generalmente como plásmidos micro-Ti. Descripciones de sistemas binarios y de plásmidos micro-Ti incluyen las siguientes: Hoekema A y col. (1985) Plant Mol. Biol. 5:85-89; Deblaere R y col. (1985) Nucl. Acids Res. 13:4777-4788; van den Elzen P y col. (1985) Plant Mol. Biol. 5:149-154; Anderson DM, Patente U.S. 4.536.475; de Framond AJ y col. (1983) Biotechnol. 1:262-269; Hoekema A y col. (1983) Nature 303:179-180; Hille J y col. (1984) J. Bacteriol. 158:754-756; Hoekema A y col. (1984) J. Bacteriol. 158:383-385; An G y col. (1985) EMBO J. 4:277-284; Anderson DM, 5 10 15 20 25 30 35 40 45 50 55 60 65 4 Patente U.S. 4.536.475; Klee HJ y col. (1985) Biotechnol. 3:637-642); de Framond AJ y col. (1986) Mol. Gen. Genet. 202:125-131; Dahl GA y col., solicitud de Patente Europea 140.556; y Bevan M (1984) Nucl. Acids Res. 12:8711-8721. El ADN-T no necesita estar sobre un plásmido para transformar una célula de planta; el ADN-T localizado cromosómicamente es funcional (Hoekema A y col. (1984) EMBO J. 3:2485-2490). El ADN-T tiene repeticiones directas de alrededor de 25 pares de bases asociados con los bordes izquierdo y derecho, es decir con las uniones ANDT/ADN de planta, las cuales pueden estar involucradas o bien en la transferencia a partir de Agrobacterium o bien en la integración en el genoma del huésped. Las caracterı́sticas determinadas de plásmido Ti han sido analizadas por Merlo, véase más arriba (véase especialmente la Tabla II), y por Ream y Gordon, véase más arriba. Expresión de Gen Externo Un gen que codifica la faseolina de alubia se ha transferido y se ha expresado en tumores de girasol (Murai N y col. (1983) Science 222:476482). El gen de faseolina se expresó a un alto nivel en semillas de tabaco en desarrollo (SenguptaGopalan C y col. (1985) Proc. Natl. Acad. Sci. USA 82:3320-3324). Se han observado resultados similares con un gen homólogo, betaconglicinina de haba de soja (Beachy RN y col. (1985) EMBO J. 4:3047-3053). Se ha transcrito en tejido leñoso de girasol un gen para zeı́na de proteı́na de endosperma, a partir de la monocotiledónea Zea mays (Matzke MA y col. (1984) EMBO J. 3:1525-1531). La expresión de un gen de subunidad pequeña RuBP-Case de guisante se regula por la luz en células de petunia transformadas; la proteı́na de subunidad pequeña de guisante producida es procesada correctamente y aislada dentro de cloroplastos (Broglie R y col. (1984) Science 224:838-843). Se han identificado las secuencias involucradas en esta inducibilidad por la luz y las necesarias para expresión máxima, (Morelli G y col. (1985) Nature 315:200204; Nagy F y col. (1985) EMBO J. 4:3063-3068; Timko MP y col. (1985) Nature 318:579-582). La expresión de un gen de proteı́na de unión a/b de clorofila de trigo es regulada por luz y organo-especı́fica en plantas de tabaco transformadas (Lamppa G y col. (1985) Nature 316:750752). Un gen de choque por calor de haba de soja es termoinducible en tejido de girasol (Schöffl F y Baumann G (1985) EMBO J. 4:1119-1124). (Un promotor de choque por calor de Drosophila melanogaster es funcional de manera similar en tejido de tabaco (Spena A y col. (1985) EMBO J. 4:2739-2743).) Genes Quiméricos que tienen Promotores ADN-T El promotor nos puede conducir la expresión de genes estructurales de resistencia a drogas útiles para la selección de células de plantas transformadas. Se han creado genes de resistencia para kanamicina (Bevan MW y col. (1983) Nature 304:184-187; Fraley RT y col. (1983) Proc. Natl. Acad. Sci. USA 80:4803-4807; Herrera-Estrella L 3 5 ES 2 076 143 T3 y col. (1983) EMBO J. 2:987-995), metotrexato (Herrera-Estrella y col., véase más arriba), cloramfenicol (Herrera-Estrella L y col. (1983) Nature 303:209-213), higromicina B (van den Elzen PJM y col. (1985) Plant Mol. Biol. 5:299-302). Helmer G y col. (1984) Biotechnol. 2:520-527, han creado un gen de fusión que tiene el promotor y el extremo 5’ del gen estructural de nos fusionado a secuencias beta-galactosidasa (lacZ) de E. coli. Los tejidos de plantas transformados con este marcador seleccionable, pueden reconocerse por un color caracterı́stico cuando crecen en el sustrato cromogénico apropiado. Se han creado y son funcionales los genes de proteı́na de fusión entre el gen estructural ocs, que también proporciona promotores, y los genes estructurales para faseolina y resistencia a B higromicina (Waldron C y col. (1985) Plant. Mol. Biol. 5:103-108; Murai N y col. (1983) Science 222:476-482). Se ha construido un gen ocs glifosato conducido (Comai L y col. (1985) Nature 317:741-744). Los promotores para genes de octopina TL ORF24 y ORF25 podı́an también conducir expresión de gen estructural (Velten J y col. (1984) EMBO J. 3:2723-2730; Velten J y Schell J (1985) Nucl. Acids Res. 13:6981-6998; Gelvin SB y col. (1985) Mol. Gen. Genet. 199:240-248; Comai L y col. (1985) Nature 317:741-744). Genes Quiméricos que tienen Promotores de Planta Una proteı́na quimérica de subunidad pequeña RuBP-Case/ de resistencia a kanamicina, se traslocó en el cloroplasto (Van den Broeck G y col. (1985) Nature 313:358-363). Ese promotor de gen confiere expresión fotoinducible en tejido leñoso a genes de resistencia a kanamicina y cloramfenicol (Herrera-Estrella L y col. (1984) Nature 310:115-120; Facciotti D y al (1985) Biotechnol. 3:241-246). Un promotor de sintasa chalcona también condujo expresión fotoinducible de un gen de resistencia a kanamicina (Kaulen H y col. (1986) EMBO J. 5:1-8). Se han utilizado promotores de proteı́na de unión a/b de clorofila para dirigir la expresión del gen ocs y del gen estructural de resistencia a la kanamicina (Jones JDG y col. (1985) EMBO J. 4:2411-2418; Simpson J y col. (1985) EMBO J. 4:2723-2729). Genes Quiméricos que tienen Promotores Virales Se expresó un gen de resistencia a la kanamicina bajo el control de un promotor de virus de mosaico de coliflor (CaMV) en células de plantas transformadas por ADN-T (Koziel MG y col. (1984) J. Mol. Appl. Genet. 2:549-562). Un gen de resistencia a metotrexato detrás del promotor 35S CaMV confirió resistencia a metotrexato (Brisson N y col. (1984) Nature 310:511-514). Se ha expresado la proteı́na de la cubierta del virus del mosaico del tabaco en tejido de tabaco transformado bajo el control de un promotor CaMV (Bevan MW y col. (1985) EMBO J. 4:1921-1926). Odell JT y col. (1985) Nature 313:810-812, han mapeado secuencias del promotor CaMV 35S ne4 5 10 15 20 25 30 35 40 45 50 55 60 65 6 cesarias para la transcripción. Transformación de Plantas sin Agrobacterium Se ha analizado recientemente la transferencia directa de ADN en células de planta (Jones MGK (1985) Nature 317:579-580; Potrykus I y col. (1985) Plant. Mol. Biol. Reptr. 3:117128; Howe C (1985) Trends Genet. 1:38-39; Paszkowski J y Saul MW (1986) Meth. Enzymol. 118:659-668; Power JB y col. (1986) Meth. Enzymol. 118:578-594). Ambas células, las monocotiledóneas y las dicotiledóneas, pueden ser transformadas directamente por marcadores seleccionables por kanamicina bajo el control de un promotor nos o CaMV (Paszkowski J y col. (1984) EMBO J. 3:2717-2722; Gardner RC y col. (1984) Plant. Mol. Biol. Reporter 2:3-8; Hain R y col. (1985) Mol. Gen. Genet. 199:161168; Potrykus I y col. (1985) Mol. Gen. Genet. 199:183-188; Lörz H y col. (1985) Mol. Gen. Genet. 199:178-182; Shillito RD y col. (1985) Biotechnol. 3:1099-1103; Meyer P. y col. (1985) Mol. Gen. genet. 201:513-518). Se pueden cotransformar moléculas de ADN distintas en una célula de planta; es ventajoso que un ADN en la mezcla lleve un marcador seleccionable (Peerbolte R y col. (1985) Plant. Mol. Biol. 5:235246). Los descendientes de plantas regeneradas a partir de dichas células transformadas heredan el gen hı́brido transformado como un rasgo Mendeliano, dominante único (Potrykus y col., Mol. Gen. Genet., véase más arriba). El CaMV ha probado ser útil como un vector de transformación de planta (Brisson N y col. (1984) Nature 310:511-514; Brisson N y Hohn T (1986) Meth. Enzymol. 118:659-668). El virus de mosaico de grama (BMV), un virus ARN, puede utilizarse también en plantas para expresar genes estructurales extraños (French R y col. (1986) Science 231:1294-1297). Se ha probado que la electroforación es útil para la introducción de genes quiméricos dentro de células de plantas en un sistema de expresión transitorio (Fromm M y col. (1985) Proc. Natl. Acad. Sci. USA 82:5824-5828) y para la transformación estable de células de maı́z (Fromm ME y col. (1986) Nature 319:791-793). Las células pueden absorber ADN rodeado por membranas. El ADN, incluyendo el ADN pTi, puede introducirse a través de liposomas (por ej. Deshayes A y col. (1985) EMBO J. 4:27312737) o por fusión de células de planta y bacteria después de eliminar sus paredes celulares respectivas (por ej. Hain R y col. (1984) Plant Cell Rept. 3:60-64). Los protoplastos de planta pueden absorber células Agrobacterium delimitadas por paredes celulares. El ADN-T puede ser transmitido a tejido regenerado a partir de protoplastos fusionados. El ADN puede estar integrado de forma estable en un genoma de planta después de microinyección (Crossway A y col. (1986) Mol. Gen. Genet. 202:179-185). Ohta Y (1986) Proc. Natl. Acad. Sci. USA 83:715-719, y Zhou G-Y y col. (1983) Meth. En- 7 ES 2 076 143 T3 zymol. 101:433-418, han informado respectivamente de la introducción de ADN durante la fertilización o polinización en células de plantas de maı́z y algodón. Visión de conjunto del AMV El virus de mosaico de la alfalfa (AMV), pertenece a una clase de virus de planta que tiene un genoma ARN multi cadena, de cadena simple, tripartito. El genoma (excluyendo las moléculas de ARN subgenómicas) está segmentado en tres moléculas de ARN. Esta clase incluye: el grupo de virus del mosaico de la alfalfa (AMV), los ilarvirus, los bromovirus, los cucumovirus y los hordeivirus (van Vloten-Doting L y col. (1981) Intervirol. 15:198-203; Matthews REF (1982) Classification and Nomenclature of Viruses). Los segmentos de genoma se encapsulan separadamente en partı́culas baciliformes de diferentes longitudes. Al lado de los tres componentes ARN genómicos (ARN1, ARN2 y ARN3), se encuentra un cuarto ARN subgenómico (ARN4) en las preparaciones de virus. Para iniciar la infección, se requiere una mezcla de los tres ARN de genoma junto con una pequeña cantidad de proteı́na de la cubierta o su mensajero, ARN4 (Bol JF y col. (1971) Virol. 46:73-85). Se ha descrito la secuencia completa de ARN4 de AMV (Brederode FT y col. (1980) Nucl. Acids Res. 8:2213-2223). El ARN4 tiene 881 nucleótidos de longitud. La región codificante tiene 660 nucleótidos (sin incluir el codón de iniciación y terminación) flanqueado por una región 5’ no traducida de 39 nucleótidos y una región 3’ no traducida de 182 nucleótidos. La secuencia de ARN4 está presente y localizada en el extremo 3’ de ARN3 (Gould AR Symons RH (1978) Eur. J. Biochem. 91:269-278). Se ha descrito la secuencia nucleótida completa de ARN3 de AMV (Barker RF y col. (1983) Nucl. Acids Res. 11:2881-2891). Una región 5’ no codificante de 240 bases precede a una secuencia de lectura abierta (ORF) de 903 nucleótidos. Esta ORF está seguida por una región intercistrónica de 49 bases y una ORF de 666 nucleótidos, esta última ORF codifica la proteı́na de la cubierta de AMV. El gen de la proteı́na de la cubierta está seguido por una secuencia 3’ no traducida de 179 bases. El ARN4 de AMV es idéntico y está determinado por el extremo 3’ de ARN3, que tiene 36 nucleótidos de la región intercistrónica, el gen estructural de proteı́na de la cubierta, y la secuencia 3’ no traducida. Se ha obtenido la secuencia nucleótida completa de un ARN1 de AMV (Cornelissen BJC y col. (1983) Nucl. Acids Res. 11:1253-1265). El ARN1 tiene 3645 nucleótidos de longitud y contienen una ORF larga para una proteı́na de Mr 125.684 flanqueada por una secuencia 5’ no traducida de 99 nucleótidos y una región 3’ no traducida de 163 nucleótidos. La comparación de las secuencias 3’ terminales de los cuatro ARNs del AMV revela una extensa homologı́a entre los 140 a 150 nucleótidos terminales de 3’ (Houwing CJ y Jaspars EMJ (19878) Biochem. 17:2927-2933). Hay alrededor de 20 sustituciones de base en los 145 nucleótidos del extremo 3’ de los ARN del AMV; estos están o 5 10 15 20 25 30 35 40 45 50 55 60 65 8 bien localizados en los bucles de las estructuras de bases apareadas o convierten pares de bases A-U a pares de bases G-C en los troncos de las horquillas de la estructura secundaria (Koper-Zwarthoff EC y col. (1979) Nucl. Acids Res. 7:1887-1900). ARN antisentido Sequeira L (1984) Trends Biotechnol. 2:2529, analiza la protección cruzada y la resistencia inducida. Stebbing N (1979) Pharmac. Ther. 6:291-332 analiza el diseño de agentes antivirales basados en las secuencias de ARN viral y el efecto antiviral de los ácidos nucleicos de cadena simple. Weintraub H y col. (1985) Trends Genet. 1:22-25, analiza resultados publicados y no publicados utilizando ARN antisentido para análisis genético. Travers A (1984) Nature 311:410, y Laporte DC (1984) Trends Biochem. Sci. 9:463, analizan papeles reguladores de ARN antisentido (ARNa). En la naturaleza, el ARN complementario o antisentido, puede funcionar de una forma reguladora. En E. coli, un ARN ompC de 174 b regula la expresión ompF. En ARN antisentido, que probablemente se unirá antes a regiones de ARNm antes de ponerse en contacto con ribosomas, pareció mas eficaz descendiendo la expresión de gen (Mizun T y col. (1984) Proc. Natl. Acad. Sci. USA 81:1966-1970; Coleman J y col. (1984) Cell 37:429-436). El ARN complementario puede inhibir la replicación de ADN (Tomizawa J y col. (1981) Proc. Natl. Acad. Sci, USA 78:1421-1425; Tomizawa J y Iton T (1982) Cell 31:575-583. In vitro, un oligonucleótido complementario a tan poco como cinco nucleótidos de la región terminal 3’ de ARN ribosomal 16S de E. coli puede inhibir la iniciación de traducción de los ARNm de virus bacterianos (Eckhardt H y Luhrmann R (1979) J. Biol. Chem. 254:11185-11188; Jayaraman K y col. (1981) Proc. Natl. Acad. Sci. USA 78:1537-1541; Taniguchi T y Weissmann C (1978) Nature 275:770-772). La traducción in vitro de ARNm puede inhibirse si el ARNm está mezclado con un ADN complementario (ADNc) y se somete a condiciones de reconocimiento de ácido nucleico; se puede traducir una mezcla no reconocida de ARNm y ADNc (Paterson BM y col. (1977) Proc. Natl. Acad. Sci. USA 74:4370-4374). La replicación in vitro de fragmentos de ARN viral de planta puede inhibirse si los fragmentos de ARN se mezclan con un ADNc y se someten a condiciones de reconocimiento; se puede replicar una mezcla no reconocida de los fragmentos de ARN y de ADNc (Ahlquist P y col. (1984) Plant Mol. Biol. 3:3744). Se inhibe la replicación del virus de sarcoma de Rous y la transformación de la célula por la adición de un oligonucleótido complementario a 13 nucleótidos de una repetición 5’ y 3’ terminal (Zamecnik PC y Stephenson ML (1978) Proc. Natl. Acad. Sci. USA 75:280-284). El ARNa de globina inhibió la traducción de ARNm de globina cuando ambos se inyectaron en el citoplasma de oocitos de rana. El ARNa inhibió la traducción cuando se inyectaba con o antes que el ARNm. Los hı́bridos ARNa:ARNm 5 9 ES 2 076 143 T3 parecı́an formarse dentro de los oocitos, aunque los hı́bridos eran mucho mas cortos que los esperados para duplex de longitud completa (Melton DA (1985) Proc. Natl. Acad. Sci. USA 82:144148. Los resultados sugieren que la estructura secundaria podı́a limitar las regiones de ácidos nucleicos que formaban hı́bridos. Se ha mostrado que la producción de secuencias de ARN viral antisentido, protege parcialmente las células de E. coli contra la infección por el bacteriófago correspondiente. Los sitios de unión de secuencia complementaria al ribosoma, fueron inhibidores mas eficaces que una secuencia complementaria al extremo 3’ de un gen estructural y la región 3’ no traducida (Coleman J y col. (1985) Nature 315:601-603). El ARN antisentido puede reducir la expresión de gen en sistema de expresión transitorio en células de ratón. Cuando un gen de timidina kinasa (TK) de un virus de herpes simple (HSV) y un exceso de 100 veces de una modificación de ese gen, que tiene la secuencia codificante de proteı́na basculada de tal forma que se codificó una ARNa de TK, se coinyectaron en células TK− , se observó que la actividad TK disminuı́a 4 veces (Izant JG y Weintraub H (1984) Cell 36:1007-1015). Se observó que la expresión del gen Lac en células de ratón se reducı́a diez veces cuando se cotransformaba con un gen ARNa a una proporción de 1:1 o mayor de gen:gen ARNa (Rubenstein JLR y col. (1984) C. R. Acad. Sci., Parı́s 299:271-274). Resumen de la invención Esta invención se refiere a la ocurrencia de infecciones virales en plantas y a los esfuerzos de horticultores y agrónomos para combatir estas infecciones en especies de plantas económicamente significativas. Las infecciones de virus ocurren en todas las especies de plantas conocidas y provocan reducciones significantivas en el rendimiento y calidad de todas las especies de plantas agrı́colas y hortı́colas. Ninguna industria de plantas en ningún paı́s del mundo está exenta de tales daños causados por virus y no se conoce un tratamiento consistente para tratar o prevenir tales infecciones virales. Por ejemplo, el 90% de las plantas de tapioca en Kenya están infectadas por el virus del mosaico de la tapioca, dando como resultado una reducción del rendimiento estimada en el 75%. Como otro ejemplo, en una reciente epidemia viral en Ghana, se perdieron más de cien mil árboles de cacao por infección con virus de inchazón de brotes. Se podrı́an dar muchos otros ejemplos que hacen evidente que las epidemias e infecciones de virus tienen un gran significado económico. La reducción en el rendimiento de las cosechas de alimentos es también relevante con el continuo crecimiento de la población humana y con la malnutrición crónica que ya existe. Por lo tanto, está claro que tanto los medios para producir genotipos de plantas resistentes a virus como las propias plantas resultantes serı́an muy útiles para incrementar la capacidad del mundo para auto alimentarse. En particular, el virus del mosaico de la alfalfa ha demostrado causar serias disminuciones 6 5 10 15 20 25 30 35 40 45 50 55 60 65 10 en el rendimiento de las cosechas. El AMV infecta la alfalfa y otros cultivos anuales de legumbres. Esto es importante económicamente; sólo en los Estados Unidos se plantan aproximadamente 30 millones de acres de alfalfa. El virus del mosaico de la alfalfa también provoca daños económicamente importantes en cultivos tales como pimientos, patatas, apios, guisantes y habas. La alfalfa puede ser un huésped venteado a partir del cual los áfidos transportan el virus. La enfermedad también se difunde a partir de la alfalfa a otras especies de cultivos siguiendo un crecimiento de infección de áfidos. En muchos casos, las plantas infectadas por AMV no muestran sı́ntomas, haciendo difı́cil detectar la ocurrencia y difusión de la enfermedad. En otros casos, la enfermedad del mosaico es evidente pero en ese momento seguramente el virus que se ha difundido a través de una gran superficie de plantas de un campo. Aparte de retirar las plantas infectadas y de la utilización de insecticidas para eliminar los áfidos que transmiten el virus, no hay desarrollados procedimientos prácticos para prevenir la difusión del AMV. Por esta razón, está claro que tanto los medios para la creación de genotipos resistentes a AMV como las plantas resultantes, serı́an muy útiles para incrementar la productividad agrı́cola de un número de cultivos. Por lo tanto, es un objeto de la presente invención proporcionar plantas que tienen unos nuevos genes de resistencia a virus y, en particular, proporcionar plantas que resisten la infección por AMV. Encaminados hacia este objetivo, se proporcionan procedimientos para crear genes de resistencia viral, en particular, genes de ARN antisentido inhibidores de la replicación viral. También se proporcionan plantas, células de plantas, tejidos de plantas y semillas de plantas que contienen genes ARNa que son responsables de que esos materiales de planta tengan un fenotipo de resistencia viral. Adicionalmente, se describen también moléculas de ADN útiles para la creación de tales plantas resistentes a virus. Se dan ejemplos de la presente invención poniendo en tabaco un gen ARNa complementario al ARN mensajero de la proteı́na de la cubierta del virus del mosaico de la alfalfa. El ARN antisentido no ha mostrado nunca afectar a la expresión de gen en plantas, aunque se ha mostrado que lo hace en sistemas no vegetales. Existen razones por las que la presencia de un ARNa viral puede no proteger a una planta de una infección viral. El ARNa viral es un ARN que no se encuentra de forma natural en el núcleo; el ARNa viral puede no ser estable en ese entorno. El nivel de acumulación de ARNa puede ser demasiado bajo para inhibir la infección. El ARNa puede no acumularse en una parte de una célula donde se inician o se mantienen las infecciones virales. Aunque el ARNa viral puede ser estable en citoplasma de planta, el ARNa viral puede no ser estable en un citoplasma de planta en la ausencia de una infección viral. Una infección viral puede establecerse mas rápidamente que el ARNa pueda reconocer a un ARN viral. La acumulación de moléculas ARNa viral a niveles suficientes para interferir con un ciclo de vida de virus puede ser 11 ES 2 076 143 T3 tóxica para las células de la planta. En efecto la presencia de un gen ARNa puede ser negativo para las plantas. Descripción detallada de la invención Se proporcionan las siguientes definiciones para eliminar ambigüedades en la intención o el alcance de su utilización en la especificación y reivindicaciones. Promotor: Se refiere a secuencias en el extremo 5’ de un gen estructural involucradas en la iniciación de la transcripción. Un promotor expresable en plantas es cualquier promotor capaz de dirigir la transcripción en al menos untipo de células de plantas en al menos una etapa de desarrollo. Las secuencias de promotor eucariótico se conocen comúnmente por la presencia de homólogos de secuencias de ADN a la forma canónica 5’...TATAA...3’ alrededor de 10-30 pares de bases (pb) 5’-a la posición del extremo 5’ del ARNm (posición de caperuza). Alrededor de 30 pb 5’- al TATAA, se encuentra a menudo otra secuencia de promotor que reconoce por la presencia de homólogos de secuencias DNA a la forma canónica 5’...CCAAT...3’. Terminador de Transcripción: Se refiere en este documento a cualquier secuencia de ácido nucleico capaz de determinar la posición del extremo 3’ de un transcrito. El segmento de ADN terminador del transcrito puede ser él mismo un material compuesto por segmentos derivados de una pluralidad de fuentes, de ocurrencia natural o sintética, procariótica o eucariótica, y puede estar formado a partir de un ADN genómico o un ADNc derivado de ARNm (ARNm: ARN mensajero). Los sitios de terminación de transcrito, incluyen los sitios de poliadenilación y los sitios que determinan el extremo 3’ de ARN ribosómicos (ARNr), ARN de transferencia (ARNt), y ARNm no poliadenilados, por ej. ARNm de histona: Krieg PA y Melton DA (1984) Nature 308:203-206). Un sitio de poliadenilación es una secuencia de ácidos nucleicos correlaccionada con poliadenilación de ARNm en eucariotas, es decir después de la terminación transcripcional, se añaden “colas” de ácido poliadenı́lico al extremo 3’ de los precursores de ARNm. Los sitios de poliadenilación se reconocen comúnmente por la presencia de homologı́a a la forma canónica 5’...AATAAA...3’, aunque no son extrañas variaciones de distancia 5’ al extremo 3’ del transcrito, “lectura” parcial, y secuencias canónicas tándem múltiple. Parecen ser necesarias las secuencias de ADN entre 20 y 35 pb corriente abajo a partir del extremo 3’ del transcrito (McDevitt MA y col. (1984) Cell 37:993-999). Se deberı́a reconocer que un “sitio de poliadenilación” canónico puede realmente determinar la posición del extremo 3’ del ARNm y no la poliadenilación per se (Proudfoot N (1984) Nature 307:412-413; Birnstiel ML y col. (1985) Cell 41:349-359). Secuencias de control de la Transcripción: Se refiere a una combinación de un promotor/sitio de terminador de transcripción que flanquea un gen estructural. Las secuencias de ADN del promotor y terminador que flanquean un gen estructural extraño particular no necesitan derivarse de la misma fuente de genes (por ej. emparejando dos 5 10 15 20 25 30 35 40 45 50 55 60 65 12 transcritos de ADN-T diferentes) o de la misma fuente taxonómica (por ej. emparejando secuencias procedentes de ADN-T con secuencias procedentes de fuentes no ADN-T tales como secuencias de plantas, animales, hongos, levaduras, virus eucarióticos, bacterias y secuencias sintéticas). ARN antisentido (ARNa): se refiere en este documento a un ARN que es complementario completamente o en parte a al menos una secuencia de ARNm viral diana (de sentido). Un virus diana es un virus contra el que se desea lograr una resistencia. La secuencia viral diana comprendida en el complemento por el ARNa puede consistir en un ARNm viral diana de longitud completa o en una parte del mismo, siendo dicha parte una región codificante, una posición de iniciación de traducción y/o una región 5’, 3’ o una región no traducida interna. Un ARNa puede incluir secuencias complementarias a una pluralidad de virus distintos, confiriendo de ese modo resistencia a cada uno de la pluralidad de virus. El ARNa por si mismo puede funcionar o no como un ARNm. Gen ARN antisentido (gen ARNa): Se refiere en este documento a un promotor, una secuencia que determina un ARNa (ADNc viral), y un terminador de transcrito, el promotor, el ADNc viral, y el terminador, teniendo tal posición y orientación unos respecto a otros que, cuando están en la célula de la planta, se puede transcribir un ARNa bajo el control del promotor. En otras palabras, un gen ARNa expresa un ARN que comprende un ARN complementario a un ARNm viral. Un gen ARNa puede ser un material compuesto de segmentos derivados de una pluralidad de fuentes, de ocurrencia natural o sintética. Un transcrito de gen ARNa puede incluir secuencias derivadas totalmente o en parte a aprtir de ADN procariótico, ADN eucariótico, ADN episomial, ADN de plásmido, ADN de plástido, ADN genómico, ADNc, ADN viral, ADNc viral, ADN sintetizado quı́micamente, o los similares. Se contempla adicionalmente que un gen ARNa puede contener una o mas modificaciones en cualquiera de las secuencias de control de transcripción, las secuencias transcritas, o ADNc viral, que podrı́an afectar la actividad biológica o la estructura quı́mica del ARNa, la proporción de expresión o la forma de control de expresión. Tales modificaciones incluyen, pero no están limitadas a, mutaciones, inserciones, eliminaciones, y sustituciones de uno o mas nucleótidos, y modificaciones que no alteran la función ARNa pero que afectan la localización intercelular, el transporte, o la estabilidad del ARNa. El ADN que codifica un ARNa puede determinar una secuencia ARNa ininterrumpida o puede incluir uno o mas intrones, unidos por las uniones funcionales apropiadas de plantas, que pueden obtenerse a partir de una fuente de ocurrencia sintética o natural. ADNc (ADN complementario): Aunque este término es bien entendido en la técnica, tiene dos significados. (1) Un ADNc puede ser un ADN de cadena simple complementario a un ARN (por ej. un ARNm viral). (2) Un ADNc puede ser también un segmento de ADN de doble cadena, una cadena de las cuales es complementaria a un ARN, la otra cadena tiene una secuencia equivalente a ese ARN (sustituyendo T por U). Gene7 13 ES 2 076 143 T3 ralmente, un ADNc de doble cadena se deriva de un ADNc de cadena simple. Sin embargo, según se define en este documento, un ADN de doble cadena que codifica secuencias de ARNm, por ej. el ADN de un gen estructural, está incluido dentro del término ADNc. En las reivindicaciones, el ADNc se refiere siempre a la forma de doble cadena (significado (2)). En otra parte en esta especificación, el significado de ADNc se define por el contexto y se entenderá bien por los técnicos en la materia. Multi-cadena: se refiere a virus que tienen genomas que tienen secuencias equivalentes a aquellas de los ARNm de los virus; es decir, el ARN encontrado en partı́culas de virus no es complementario al ARNm viral. Frecuentemente el ARN viral de virus multi cadena puede utilizarse como un ARNm. El AMV es un ejemplo de un virus milti cadena; cada uno de los cuatro ARNs encontrados en los viriones de AMV es capaz de proporcionar un ARNm. Genoma de ARN Tripartito: Se refiere a la organización de un material genético de virus. “Genoma” se refiere al material genético total de los virus. “Genoma ARN” indica que como se presenta en viriones (partı́culas de virus), el genoma está en forma ARN. “Tripartito” indica que el genoma está dividido entre tres moléculas de ARN separadas. Un ejemplo de un virus con un genoma de ARN tripartito es el AMV. El genoma de AMV es portado por los ARN 1, 2, y 3 de AMV. La secuencia de ARN4 está contenida completamente por el ARN3 y el ARN4 no se replica; por esta razón, el ARN4 se refiere como un ARN subgenómico y no se cuenta como uno de los ARN genómico. Posición de Iniciación de Traducción: Se refiere en este documento al codón de comienzo de traducción 5’AUG3’ en el extremo 5’ de un gen estructural, el nucleótido siguiente al AUG, y los 3 nucleótidos qee preceden al AUG (véase Kozak M (1983) Mocrobiol. Rev. 47:1-45, y Kozak M (1984) Nucl. Acids Res. 12:857-872). Secuencia no traducida 5’: Se refiere en este documento a la parte de un ARNm entre su extremo 5’ o posición caperuza, y el codón de inicio de traducción. Secuencia conservada 3’: Se refiere en este documento a una secuencia en el extremo 3’ de un genoma ARN multipartito no poliadenilado que es la misma para todos los componentes del genoma. La secuencia conservada 3’ del AMV comprende alrededor de 145 nucleótidos a partir del extremo 3’ de todos los 4 ARN del AMV. ADNc de longitud esencialmente completa: Se refiere en este documento a un ADNc que es complementario a un ARNm completo, posiblemente exceptuando unos pocos (por ej. cinco) nucleótidos en cada extremo de esa secuencia de ARNm. Marcador seleccionable o identificable expresable en plantas: Se refiere en este documento a un marcador genético funcional en una célula de planta. Un marcador seleccionable (por ej. un gen de resistencia a la kanamicina) permite a células que contienen y expresan ese marcador crecer bajo condiciones desfavorables al crecimiento de células que no expresan ese marcador. 8 5 10 15 20 25 30 35 40 45 50 55 60 65 14 Un marcador identificable (por ej. un gen betagalactosidasa) facilita la identificación de células que expresan ese marcador. Transformación: Se refiere al acto de provocar que una célula contenga una molécula o una secuencia nucleica que no es parte original de esa célula. Tejido de planta: Incluye tejidos de planta diferenciados y no diferenciados que incluyen pero no se limitan a raı́ces, brotes, polen, semillas, tejido tumoral, tales como agallas de copa, y varias formas de agregaciones de células de plantas en cultivo, tales como embriones y tejidos leñosos. El tejido de planta puede estar in planta, o en un órgano, tejido o en cultivo celular. Célula de planta: Según se utiliza en este documento incluye células de planta in planta y células de planta y protoplastos en cultivo. Los siguientes términos son bien conocidos en la técnica y no van a ser especificados o especialmente definidos en este documento: de cadena simple, genoma, grupo de virus del mosaico de la alfalfa (véase Mattheus REF (1982) Classification and Nomenclature of Viruses, p. 177), Tobamovirus (véase Mattheus, véase más arriba, pp. 158-159), promotor de CaMV 19S (véase Hohn T y col. (1982) Curr. Top. Microbiol. Immunol. 96:193-236), ADN-T tipo octopina (posiciones, orientaciones, y secuencias de lectura abierta (ORFs) se definen como se designan en Barker RF y col. (1983) Plant Mol. Biol. 2:335-350), repetición de borde ADN-T, transcripción bajo el control de un gen promotor, ligando, descendido y estructural. La producción de una célula modificada genéticamente que expresa un gen ARNa combina las enseñanzas especı́ficas de la presente descripción con una variedad de técnicas y expedientes conocidos en la técnica. En la mayorı́a de los casos, existen expedientes alternativos para cada etapa del proceso total. La elección de expedientes depende de variables tales como la elección del virus particular contra el que se quiere obtener una resistencia, el sistema de vector básico para la introducción y mantenimiento estable de un gen ARNa, las especies de plantas que se van a modificar, y la estrategia de regeneración deseada, las secuencias de control de transcripción utilizadas, las secuencias virales particulares comprendidas por el transcrito del gen de ARNa, y las similares, todas las cuales presentan etapas de procedimiento alternativas que aquellos expertos en la técnica son capaces de seleccionar y utilizar para conseguir un resultado deseado. Como se han desarrollado nuevos procedimientos para la inserción estable y la transcripción de ADN extraño en células de plantas, aquellos de conocimiento ordinario en la técnica, serán capaces de seleccionar entre aquellas etapas alternativas del proceso para conseguir el resultado deseado. Los aspectos fundamentales de la invención son la naturaleza del gen de ARNa y su utilización para conferir resistencia a las infecciones virales de plantas transformadas con él. Otros aspectos incluyen la naturaleza y estructura de la secuencia del ARNa y sus medios de inserción y expresión en un genoma de planta. Las etapas 15 ES 2 076 143 T3 restantes de la realización preferida para obtener una planta modificada genéticamente incluyen la inserción del gen ARNa en ADN-T, la transferencia del ADN-T modificado a una célula de planta en donde el ADN-T modificado llega a estar integrado de forma estable como parte del genoma de la célula de planta, técnicas para cultivo in vitro y regeneración eventual en plantas completas, que puede incluir etapas para selección y detección de células de plantas transformadas y etapas de transferencia del gen ARNa introducido a partir de la cepa transformada original a cultivos comercialmente aceptables, y observación de la expresión en plantas transformadas. Un punto de comienzo para la construcción de un gen ARNa es la obtención de clones ADN de secuencias virales. Si el virus es un virus ADN, los clones ADN son obtenibles utilizando procedimientos bien conocidos en la técnica de ADN recombinante. Si el virus es un virus ARN, puede obtenerse un clon ADNc a partir de la secuencia viral deseada. Se conocen en la técnica del ADN recombinante varios procedimientos para realizar clones ADNc; la elección de procedimientos dependerá de variables tales como poliadenilación, longitud de ARN, conocimiento anterior de la secuencia de ARN, preparaciones anteriores en el laboratorio particular para otros experimentos de clonado ADNc, y factores similares. Las secuencias virales clonadas son un componente necesario de un gen ARNa. Una caracterı́stica principal de la presente invención en su realización preferida es la construcción de un derivado de ADN-T que tiene un gen insertado bajo el control de secuencias que controlan la transcripción expresables en planta, es decir, entre un promotor y un terminador de transcripción, según se han definido estos términos, véase más arriba. El ARNa que codifica ADN debe insertarse en la posición y orientación correcta con respecto al promotor. La posición se refiere a qué lado del promotor del ARNa que codifica ADN está insertado. Se sabe que la mayorı́a de los promotores controlan la iniciación de la transcripción y traducción solo en una dirección a lo largo del ADN. La región de ADN que cae bajo el control del promotor se dice que cae “corriente abajo” o alternativamente “debajo” o “3’ a” el promotor. Por lo tanto, para ser controlado por el promotor, la posición correcta de una inserción de ARNa que codifica ADN debe ser “corriente abajo” a partir del promotor. La orientación se refiere a la direccionalidad del gen estructural. Esa parte de un ARNa que codifica ADN que es complementaria al ARNm viral que codifica el término amino de una proteı́na viral se denomina el extremo 3’ del ADN que codifica ARNa, mientras que el extremo que es complementario al ARNm viral que codifica aminoácidos próximos al extremo carboxilo de la proteı́na se denomina el extremo 5’ del ADN que codifica ARNa. En otras palabras, el extremo 5’ y el extremo 3’ de un ADN que codifica ARNa, codifican respectivamente el extremo 3’ y el extremo 5’ de ARNm viral. La orientación correcta del ADN que codifica ARNa es con el extremo 5’ del mismo próximo al promotor. De forma similar a la región del promotor, el terminador de transcrito debe estar localizado 5 10 15 20 25 30 35 40 45 50 55 60 65 16 en la posición y orientación correcta relativa al ARNa, estando próximo al extremo 3’ del ARNa. Se pueden observar diferencias en proporciones de expresión de gen ARNa o de control de evolución dependiendo de las particulares componentes ARNa, promotores, terminadores de transcripción, secuencias de ADN flanqueantes, o posiciones de inserción en los genomas de las plantas transformadas. La acumulación de ARNa puede estar también fuertemente influida por los detalles de la estructura secundaria de ARNa, especialmente las estructuras de bucle de tronco. Cuando los componentes de gen ARNa se varı́an, se pueden observar diferentes propiedades, incluyendo, pero no limitándose a, tales propiedades como la estabilidad, la localización intracelular, el procesamiento posttranscripcional y otras propiedades funcionales del propio ARNa expresado. Todas estas variaciones presentan numerosas oportunidades para manipular y controlar las propiedades funcionales del fenotipo de resistencia viral, dependiendo de las propiedades fisiológicas deseadas dentro de la célula de la planta, el tejido de la planta y la planta completa. El principio fundamental de la presente invención es que la presencia de secuencia ARN antisentido en una célula de planta es capaz de conferir a esa célula al menos algún nivel de resistencia viral. Los requisitos por los que segmentos de secuencia viral son incluidos en un ARNa son mejor expresados en términos funcionales. La presencia de ARNa en una célula confiere resistencia viral por interferencia con una o mas funciones virales. Tales funciones pueden incluir traducción de proteı́nas virales, replicación de ARN virales, encapsulación de ácido nucleico viral, etc. El ARNa interfiere presumiblemente por reconocimiento de un segmento multi cadena del virus o por unión con al menos una proteı́na involucrada en el ciclo de vida viral. La presente invención no está limitada por el modo o mecanismo de interferencia. El ARNa no necesita incluir una secuencia viral de longitud completa; basta con cualquier segmento de secuencia complementaria a un segmento multi cadena capaz de interferir con una función viral. En otras palabras, se requiere en un ARNa la presencia de al menos una secuencia viral capaz de interferir con funciones virales, y puede ser preparado con cualquier segmento de secuencia viral incapaz de tal interferencia, en base a consideraciones de estabilidad de ARN, localización celular, y las similares. Pueden combinarse secuencias virales de algunos virus distintos, siendo cada secuencia capaz de funcionar como un ARNa, para formar un ARNa que tiene actividad antiviral contra cada uno de los distintos virus. Según se ha definido, un ARNa puede incluir secuencias no virales. Incluso en casos en los que se incluye una secuencia viral de longitud completa, un ARNa puede incluir secuencias no virales. Usualmente, estas secuencias no virales estarán en los extremos 5’ y 3’ del ARNa. Frecuentemente estos componentes no virales derivarán de segmentos de ADN del terminador del transcrito o del promotor. La inclusión de varias secuencias no virales puede afectar a la estabilidad del ARN, a la localización celular del ARNa, al procesamiento posttranscripcional y a los si9 17 ES 2 076 143 T3 milares. Se sabe en la técnica que la estabilidad del ARN está afectada por estructuras terminales tales como la caperuza 5’ y la poliadenilación 3’ y por el alargamiento de la estructura interna, es decir, emparejamiento intramolecular de bases. Un ARNa puede estar localizado dentro de una célula, por ej. un ARNa que tiene una secuencia traducible que codifica una señal polipéptido que contiene péptido tenderá a estar unido a retı́culo endoplasmático rugoso. Un intrón puede estar incluido en un ARNa, siempre que, si las posiciones de unión se derivan de dos genes diferentes, las posiciones de unión de intrón sean compatibles. Un ARNa debe designarse teniendo en cuenta algunos factores biológicos. La estructura secundaria de ARN puede afectar a la actividad antiviral, ası́ como a la estabilidad y procesamiento. En particular, el emparejamiento intramolecular de bases que comprende secuencias que interfieren con funciones virales no debe ser tan fuerte que impida el reconocimiento del ARNm viral. Un transcrito ARNa deberı́a se incapaz de ser convertido por el mecanismo de replicación viral en un componente viral en sentido funcional capaz de incrementar la severidad de la enfermedad viral. La producción de cadenas de sentido puede bloquearse por inclusión de secuencias no virales en el ARNa, las cuales interfieren con la función de replicado, especialmente en uno o ambos extremos de ARNa. Si la producción multi cadena no estuviera bloqueada, se pueden realizar otras etapas. La eliminación de un segmento de secuencia que codifica proteı́na o la introducción de una mutación alternancia de cadena posicionada apropiadamente puede provocar que la proteı́na viral copiada por la cadena de sentido, no sea funcional. Las mutaciones de alternancia de cadena se introducen convenientemente abriendo un ADNc viral que transporta un plásmido en un único, extremo adhesivo que genera posición de restricción, quitando los extremos adhesivos y volviendo a ligarlo. Dependiendo de la enzima y los procedimientos utilizados, esto eliminará o insertará dos o cuatro pares de bases, generando por ello una alternancia de cadena (y también eliminando esa posición de restricción). Una realización importante de la presente invención es la inhibición de la propagación viral provocada por la unión a un ARNm viral de una longitud esencialmente completa de ADNc (por ej. a uno de los cinco nucleótidos de cada extremo de la secuencia ARNm). Un ADNc de longitud completa tiene cuatro regiones importantes: una secuencia 5’ no traducida, una posición de comienzo de traducción, un gen estructural y una secuencia 3’ conservada. La unión de un ARNa a una secuencia 5’ no traducida puede interferir con la iniciación de la traducción por interferencia con la exploración de preiniciación del ARNm para posiciones de iniciación de la traducción por subunidades ribosomales 40S (véase Kozak M (1983) Mocrobiol. Rev. 47:1-45). La unión a una posición de iniciación de la traducción puede interferir con la iniciación de la traducción. La unión a la secuencia del gen estructural, es decir a la secuencia que codifica la proteı́na, puede disminuir la eficacia de la traducción. La unión a la secuencia 3’ conservada puede interferir con el comienzo 10 5 10 15 20 25 30 35 40 45 50 55 60 65 18 de la replicación. La combinación de segmentos de ADN, incluyendo secuencias virales, no virales, promotoras y terminadoras de transcripción, para formar un gen ARNa se consigue por medios conocidos por aquellos de conocimiento normal en la materia de tecnologı́a de ADN recombinante. La elección del promotor depende de la regulación de desarrollo deseada. La utilización de promotores regulados en desarrollo para la expresión de gen en plantas está descrita en los antecedentes. Los promotores de ADN-T o CaMV son ventajosos por ser constitutivos. El promotor de subunidad pequeña RuBP-Case puede ser útil para la expresión en los tejidos verdes de una planta de ARNa transformada genéticamente. En las realizaciones preferidas el terminador de transcripción es un sitio de poliadenilación. La fuente de genes de planta del sitio de poliadenilación no es crucial siempre que el sitio de poliadenilación, el promotor y el ARNa sean compatibles para el proceso de transcripción y posttranscripcional. Como será aparente para aquellos de conocimiento normal en la técnica, la combinación del gen ARNa puede estar colocada entre cualesquiera sitios de restricción convenientes para que se pueda extraer la combinación del plásmido y convenientes para la inserción en el vector de transformación de la planta elegida. Por ejemplo, la localización de la posición de inserción de gen ARNa dnetro del ADN-T no es crı́tica, puesto que la función de transferencia de secuencias que rodean inmediatamente a los bordes del ADNT no está interrumpida, ya que en estudios de la técnica anterior estas regiones aparecen como esenciales para la inserción del ADN-T modificado en el genoma de la planta. La combinación se inserta en el vector de transformación de la planta por técnicas estándar bien conocidas por aquellos expertos en la técnica. La orientación del gen de ARNa ensamblado, con respecto a la dirección de la transcripción y la traducción de los genes de vector endógenos no es normalmente crı́tica; generalmente, son funcionales cualquiera de las dos posibles orientaciones. Como se analizó en los Antecedentes (ADN plásmido Ti), el ADN-T de los plásmidos microTi puede transferirse desde una cepa de Agrobacterium a una célula de planta siempre que la cepa de Agrobacterium contenga ciertos genes que actúan en trans cuya función es promover la transferencia de ADN-T a una célula de planta. Los plásmidos micro-Ti son ventajosos puesto que son pequeños y relativamente fáciles de manipular directamente eliminando la necesidad de transferir el gen a ADN-T a partir de un vector lanzadera por recombinación homóloga. Después de que el ARNa deseado se ha insertado, ellos pueden introducirse fácilmente directamente en una célula de Agrobacterium que contiene los genes vir que actúan en trans, estando usualmente los genes vir sobre un “plásmido auxiliar”, que promueve la transferencia de ADN-T. La introducción en una cepa de Agrobacterium se produce convenientemente o bien por transformación de la cepa de Agrobacterium o por transferencia conjugada a partir de una célula bacteriana donante, cuyas técnicas son bien conocidas por los de conoci- 19 ES 2 076 143 T3 miento normal en la materia. Para propósitos de introducción de secuencias de ADN nuevas en un genoma de planta, deberı́an considerarse funcionalmente equivalentes, plásmidos Ti, plásmidos Ri, plásmidos micro-Ti, y ADN-T integrado en cromosomas. El ADN-T que tiene un gen ARNa puede transferirse a células de planta por cualquier técnica conocida en la materia. Por ejemplo, esta transferencia es conseguida de la manera más conveniente por cocultivo de la cepa de Agrobacterium con células de planta o con tejidos de planta. Utilizando estos procedimientos, se transforma una cierta proporción de las células de planta, lo que quiere decir tener dentro un ADN-T transferido e insertado en el genoma de la célula de la planta. En cualquier caso, las células transformadas deben ser seleccionadas o identificadas para distinguirlas de las células no transformadas. La selección se lleva a cabo de la manera más fácil proporcionando un marcador seleccionable incorporado en el ADN-T en adición al gen ARNa. Ejemplos de marcadores artificiales incluyen aquellos analizados en los Antecedentes (véase las secciones acerca de Genes Quiméricos). Además, el ADN-T proporciona marcadores endógenos tales como gen(es) que controla(n) la morfologı́a anormal de las raı́ces de tumor inducido por Ri y gen(es) que controla(n) la resistencia a compuestos tóxicos tales como análogos de aminoácidos, estando proporcionada tal resistencia por una enzima que sintetiza la opina (por ej. ocs). Procedimientos de identificación bien conocidos por aquellos expertos en la técnica incluyen, pero no se limitan a, ensayos para la producción de opina, hibridación especı́fica a secuencias caracterı́sticas de ácido nucleico (por ej. ARNa o ADN-T) o ensayos inmunológicos para proteı́nas especı́ficas. Adicionalmente, un fenotipo de un gen ARNa expresado (por ejemplo resistencia a un virus huésped) puede ser utilizado para identificar tejido transformado. El ensayo de resistencia viral puede realizarse por uno de los varios procedimientos bien conocidos en la técnica de virologı́a de plantas, incluyendo la detección de la disminución de la producción de un componente de un virus (por ej. ARN viral o una proteı́na viral), disminución de la capacidad de infección según se prueba por ensayos de infección de protoplasto o lesión local, disminución de producción del número de virus y los similares. Aunque las realizaciones preferidas comprenden la utilización de plásmidos micro-Ti, pueden ser fácilmente sustituidos otros sistemas de vector basados en ADNT conocidos en la técnica. Adicionalmente, aunque la realización preferida de esta invención incorpora un sistema mediado por Agrobacterium basado en ADN-T para la incorporación del gen ARNa en el genoma de la planta que se va a transformar, se incluyen también en el alcance de esta invención otros medios para transferir e incorporar el gen ARNa. Otros medios para la incorporación estable del gen ARNa en un genoma de planta incluyen adicionalmente, pero no están limitados a, la utilización de vectores basados en genomas virales, minicromosomas, transposones, y recombinación homóloga o no homóloga en cromosomas de planta. Formas alternativas de su- 5 10 15 20 25 30 35 40 45 50 55 60 65 20 ministro de estos vectores a una célula de planta, incluyen pero no se limitadan a, fusión con liposomas que contienen vector o esferoplastos bacterianos, microinyección, encapsulación en proteı́na de cubierta viral seguido por un procedimiento tipo infección, y admisión directa de ADN, posiblemente después de inducción de permeabilidad a plasmalema por un pulso eléctrico, un láser o un agente quı́mico. También se incluyen dentro del alcance de esta invención los medios para la incorporación transitoria y/o expresión. Los sistemas basados en células de Agrobacterium y ADN-T pueden utilizarse para transformar angiospermas, incluyendo dicotiledóneas y monocotiledóneas, por transferencia de ADN a partir de una bacteria a una célula de planta; pueden utilizarse sistemas basados en vectores alternos o medios para el transporte del vector para transformar gimnospermas y angiospermas. La regeneración de células y tejidos transformados se consigue recurriendo a técnicas conocidas. Un objeto de la etapa de regeneración es obtener una planta completa que crece y se reproduce normalmente pero que retiene el ADN-T integrado. Las técnicas de regeneración varı́an algo de acuerdo con los principios conocidos en la técnica y puede depender del vector de transformación de la planta y de las especies de planta transformada. Es bien conocida en la técnica la regeneración de plantas de tabaco transformadas, de plantas de petunia, y de plantas de especies relacionadas. Según se vayan descubriendo medios para la regeneración de otras especies de plantas, la técnica entenderá, sin experimentación indebida, como adaptar estos medios de nuevo descubrimiento a la regeneración de plantas a partir de células de plantas transformadas y de tejidos de plantas transformadas. El genotipo del tejido de planta transformado se elige frecuentemente por su facilidad para que sus células puedan crecer y regenerarse en cultivo in vitro y por su susceptibilidad al agente selectivo que se va a utilizar. Cuanto mas inadecuado para estas manipulaciones es un cultivo de interés agrario, antes se transforma una variedad tratable. Después de la regeneración, el gen ARNa introducido nuevamente puede ser transferido fácilmente al cultivo agrario deseado por técnicas bien conocidas por aquellos expertos en la materia de cultivo de plantas y genética de plantas. Los cruces sexuales de plantas transformadas con los cultivos agronómicos de plantas producen hı́bridos iniciales. Estos hı́bridos pueden después retrocruzarse con plantas de los antecedentes genéticos deseados. La progenie se identifica o selecciona continuamente para la presencia continua de ADN de gen ARNa integrado, ADN-T, o para un nuevo fenotipo que resulta de la expresión del gen ARNa u otros genes portados por el ADN insertado. De esta manera, después de un número de vueltas de retrocruzamiento y selección, pueden producirse plantas que tienen un genotipo idéntico a los progenitores deseados agronómicamente con la adición de secuencias insertadas de ADN. Una alternativa para la incorporación de un gen ARNa en un genoma de planta para producir una célula de planta que contiene ARNa es infec11 21 ES 2 076 143 T3 tar una planta con un ARN viral devector capaz de ser mantenido en la planta, el ARN viral que tiene secuencias de ARNa para un virus diana distinto (es decir un virus contra el que se desea protección). Tı́picamente, secuencias de ADNc de doble cadena del virus vector y del virus diana se manipulan utilizando tecnologı́a de ADN recombinante. Después del ensamblado de una secuencia de ADN correspondiente a la combinación deseada de vector ARN/ARNa de diana, la combinación ADNc de vector viral de planta/ADNc de ARNa puede situarse detrás de un promotor que puede conducir la transcripción in vitro. Descripciones de tales vectores y condiciones para su utilización incluyen Melton DA y col. (1984) Nucl. Acids Res. 12:7035-7056, Krieg PA y Melton DA (1984) Nucl. Acids Res. 12:7057-7070, Ahlquist P y Janda M (1984) Mol. Cell. Biol. 4:28762882, y French R y col. (1986) Science 231:12941297. Después que se ha ensamblado tal combinación de vector viral/ARNa/vector de transcripción in vitro, se puede producir una combinación ARN de vector viral/ARNa diana por transcripción in vitro y mezcla con cualquier otro componente ARN viral necesario para el mantenimiento del vector viral en una célula de planta. La infección por una combinación de vector/ARNa de una célula de planta puede efectuarse entonces por procedimientos conocidos y, después de la inoculación con el virus diana o con el ARN de virus diana, se puede probar la inhibición a la infección o la producción de un componente viral por procedimientos bien conocidos en la técnica. De manera similar, los vectores de ADN virales de planta, pueden utilizarse para introducir un gen ARNa en una célula de planta. La utilidad de tales vectores ha sido demostrada por Brisson N y col. (1984) Nature 310:511-514. Los medios para la creación de genes funcionales ARNa según se muestra en la presente invención combinarse por aquellos de conocimiento común en la materia con la utilización de vectores basados en virus de ADN de plantas. Después de la infección de una célula huésped de planta apropiada, la inhibición de la infección del virus diana puede ensayarse según se ha descrito anteriormente. Ejemplos Los siguientes ejemplos se presentan con el propósito de ilustrar realizaciones especı́ficas dentro del alcance de la presente invención sin limitar el alcance, estando dicho alcance definido por las Reivindicaciones. Aparecerán inmediatamente numerosas variaciones para aquellos de conocimiento común en la materia. Los Ejemplos utilizan muchas técnicas bien conocidas y accesibles por aquellos expertos en la materia de biologı́a molecular y manipulación de ADN-T y Agrobacterium; tales procedimientos están completamente descritos en una o mas de las referencias citadas si no se describen en detalle en este documento. Todas las referencias citadas en esta Especificación se incorporan aquı́ por referencia. Las enzimas se han obtenido a partir de fuentes comerciales y se han utilizado de acuerdo con las recomendaciones de los vendedores y otras variaciones conocidas en la técnica. Los reactivos, los tampones, y las condiciones de 12 5 10 15 20 25 30 35 40 45 50 55 60 65 22 cultivo son también conocidos por aquellos expertos en la técnica. Los trabajos de referencia que contienen tales técnicas estándar incluyen los siguientes: Wu R, ed. (1979) Meth. Enzymol. 68; Wu R y col., eds. (1983) Meth. Enzymol. 100 y 101; Grossman L y Moldaveeee K, eds. (1980) Meth. Enzymol. 65; Weissbach A y Weissbach H, eds. (1986) Meth. Enzymol. 118 (véase especialmente Rogers SG y col., pp. 627-640); Miller JH (1972) Experiments in Molecular Genetics; Davis R y col. (1980) Advanced Bacterial Genetics; Schleif RF y Wensink PC (1982) Practical Methods in Molecular Biology; Walker JM y Gaastra W, eds. (1983) Techniques in Molecular Biology; y Maniatis T y col. (1982) Molecular Cloning. Adicionalmente, Lathe RF y col. (1983) Genet. Engin. 4:1-53, hacen comentarios útiles sobre manipulación de ADN. La utilización textual del nombre de una endonucleasa de restricción aisladamente, por ej. “BclI”, se refiere a la utilización de esa enzima en una digestión enzimática, excepto en un diagrama en donde puede referirse a la posición de una secuencia susceptible a la acción de tal enzima, por ej. una posición de restricción. En el texto, las posiciones de restricción se indican por la utilización adicional de la “posición” de trabajo, por ej. “posición BclI”. La utilización adicional del “fragmento” de trabajo, por ej. el “fragmento BclI”, indica una molécula de ADN de doble cadena lineal que tiene los extremos generados por la acción de la citada enzima (por ej. un fragmento de restricción). Una frase tal como un “fragmento BclI/SmaI” indica que el fragmento de restricción fue generado por la acción de dos enzimas diferentes, en este caso BclI y SmaI, resultando los dos extremos de la acción de dos enzimas diferentes. Los plásmidos, y sólo los plásmidos, se preceden con una “p”, por ej., pTi15955 o pH400, y unas designaciones de cepa entre paréntesis incidan un plásmido albergado dentro, por ej. A. tumefaciens (pTi15955) o E. coli H802 (pH400). Las siguientes cepas están en depósitado y se ponen a disposición de la técnica en base a las denominaciones patentes: E. coli MC1061 (pH400A4I) NRRL B-18062 NRRL B-18009 E. coli K802 (pH4-1) A. tumefaciens (pTi15955) ATCC 15955 E. coli CSH52 (pSUP106) NRRL B-15486 NRRL B-15481 E. coli SM10 NRRL B-15483 E. coli S17-1 (ATCC: American Type Culture Collection, 12301 Parklawn Dr., Rockville, MD 20852 USA; NRRL: ARS Patent Collection, Northern Regional Research Center, 1815 N. University St., Peoria, IL 61614 USA.) Otros plásmidos y cepas están ampliamante disponibles y son accesibles por los técnicos en la materia. Ejemplo 1 Preparación de ADNc de ARN4 de AMV El pSP65A4 (Loesch-Fries LS y col. (1985) Virol. 146:177-187) transporta una copia de ADNc de longitud completa de ARN4 de AMV. El ADN de pSP65A4 digerido con EcoRI y SmaI 23 ES 2 076 143 T3 se sometió a la electroforesis en gel de agarosa y se diluyó del gel un fragmento de 0,89 kpb. Este fragmento se mezcló y se ligó con el ADN de pSP64 linealizado y se transformó en MC1061. Los transformantes resistentes a la ampicilina se identificaron por hibridación con una sonda de ARN4 de AMV. Se identificó una colonia que hospedaba un plásmido, llamado pSP64A4I, que portaba un ADNc de ARN4 de AMV de longitud completa. Los pSP64 y pSP65 son designados para la transcripción in vitro bajo el control de un promotor SP6 de un bacteriófago (Melton DA y col. (1984) Nucl. Acids Res. 12:7035-7056; Krieg PA y Melton DA (1984) Nucl. Acids Res. 12:70577070;. El pSP65A4 y el pSP64A4I dirigen respectivamente la sı́ntesis de secuencias multi-cadena de ARN4 de AMV y secuencias de ARNa de ARN4 de AMV. Cuando los dos plásmidos se cortan respectivamente por SmaI y EcoRI in vitro los transcritos resultantes son esencialmente secuencias de ARNm de proteı́na de la cubierta de longitud completa y el complemento a las mismas. Los fragmentos EcoRI/SmaI de ADNc de AMV de pSP65A4 y pSP64A4I son idénticos. Ejemplo 2 Pruebas in vitro de inhibición ARNa de la infección por AMV Se infectaron in vitro protoplastos de tabaco y alfalfa con ARN esencialmente según se describe por Samac DA y col. (1983) Virol. 131:455462; siendo la mayor modificación que el ARN se añadió en un pequeño volumen (por ej. 10 µl) al sedimento de protoplasto, que fue resuspendida después en sobrenadante residual antes de la adición de polientilengliol (PEG). Esta modificación al procedimiento de Samac y col. consistente en añadir ARN al PEG antes de la combinación con los protoplastos llevó a una reducción de aproximadamente cincuenta veces en la cantidad de ARN necesario para una infección. Se cree que las condiciones de inoculación son desfavorables para la formación de duplex cadena de sentido:cadena de sentido inverso fuera de una célula. Los ARN sintéticos fueron realizados esencialmente según se describe por Melton y col., véase más arriba y por protocolo del vendedor de la SP6 polimerasa. Se sintetizaron los ARN con caperuza fueron sintetizados por inclusión de 0,5 mM 0 0 7 mG5 ppp5 G y reducción de la concentración de GTP a partir de 0,5 mM a 0,025 mM. el ARN4 de AMV sintético se templó por pSP65A4 linelizado con SmaI y el ARNa de ARN4 de AMV se templó por pSP64A4I linealizado con EcoRI. En un experimento tı́pico utilizando o protopolastos de alfalfa o de tabaco, ARN1, ARN2 y ARN3 de AMV naturales mezclados con ARN4 con caperuza sintético, dieron como resultado infecciones de alrededor de dos tercios de los protoplastos inoculados. La inclusión de una cantidad equivalente, relativa al ARN4 sintético de ARNa de ARN4 con caperuza llevó a al menos una reducción de uno a cien en los niveles de infección, y frecuentemente a una infección no detectable. 5 10 15 20 25 30 35 40 45 50 55 60 65 24 Ejemplo 3 Preparación de secuencias de control de transcripción de CaMV El pDOB512, que lleva secuencias de control de transcripción del virus del mosaico de la coliflor (CaMV) (obtenido de Dr. Ken Richards, Centre National de la Recherche Scientifique, Institute de Biologie Moleculaire y Cellulaire, 15, rue Descartes, F-67084 Strasbourg, France) se construyó como sigue: (Para un análisis de CaMV, véase Hohn T y col. (1982) Curr. Top. Microbiol. Inmunol. 96:193-236.) Un fragmento de HindIII que lleva la región promotora de ARN 19S de CaMV (nucleótidos 5376-5851 de CaMV) se insertó en pBR322 y se recortó dentro de un par de bases de la posición de caperuza del transcrito de 19S. Un fragmento de HincII que porta el terminador de transcripción de CaMv 19S (nucleótidos 7018-7794 de CaMV) al que han sido añadidos los ligandos BamHI, se insertó después detrás del promotor 19S; el plásmido resultante se designó como pDOB412. El ADN de pDOB412 se digirió con Bg1II y Sa1I, se rellenó con el fragmento de Klenow del ADN polimerasa I de E. coli, y se religó, eliminando por ello el ADN, el cual incluye posiciones BamHI y HindIII, entre el sitio Bg1II en la posición 7644 y el sitio Sa1I en la posición 650 y regenerando un sitio Bg1II. El plásmido resultante se designó como pDOB512. Los extremos pegajosos del ADN de pDOB512 linealizado con HindIII se rellenaron con el fragmento de Klenow (o alternativamente por la ADN polimerasa T4). El ADN de pDOB512 de extremos romos se mezcló y ligó con ligandos Bg1II disponibles comercialmente que se recortaron después por digestión con Bg1II y se religaron. La mezcla de ligado se transformó en E. coli K802 y se aisló un transformante resistente a ampicilina que albergaba un plásmido, designado pDOB513, el pDOB513 tiene secuencias de control de transcripción 19S de CaMV en un fragmento Bg1II. Las posiciones SmaI y BamHI se encuentran entre los segmentos de ADN que tienen el promotor y la posición de poliadenilación en pDOB412, pDOB512 y pDOB513, proporcionando por ello una localización conveniente para la inserción de ADN extraño que va a ser un templado para un transcrito. Ejemplo 4 Colocación de ADNc de AMV detrás del promotor de CaMV Se digirió el ADN de pSP65A4 con EcoRI y SmaI y el ADNc de AMV de 0,89 kb se purificó por dilución a partir de gel de agarosa después de separación por electroforesis. El extremo adhesivo de EcoRI se convirtió en un extremo romo por incubación con el fragmento de Klenow de la ADN polimerasa I de E. coli. Después el ADN de pDOB513 linelizado con SmaI se mezcló y ligó con ADNc de extremo romo, y los productos de ligado se transformaron en MC1061. Los ADNs de plásmidos aislados a partir de tansformantes resistentes a la ampicilina, se identificaron por hibridación con colonias de tinción a 13 25 ES 2 076 143 T3 una sonda de ARN4 de AMV. Se identificó una colonia que albergaba un plásmido, denominado pDOBA4I, que tiene ADNc de AMV insertado entre las secuencias controladoras de transcripción 19S de CaMV orientadas de tal forma que al transcribirse, se sintetizarı́a un ARNa; es decir de tal forma que el extremo EcoRI y una posición terminal de BamHI interno están distantes del promotor y próximos al terminador de transcripción. Una combinación de secuencias de control de transcripción de CaMV/ARNa de AMV podrı́a extraerse a partir de pDOBA4I sobre un fragmento Bg1II de 1,92 kpb. Ejemplo 5 Construcción de pH400, un plásmido micro-Ti El pH4-1 es un plásmido micro-Ti albergado por E. coli K802 (pH4-1), que está depositado como NRRL B-18009. El pH4-1 ha sido descrito por Sutton DW y col., solicitud de Patente U.S. no. de serie 778.384∗, la cual se incorpora aquı́ por referencia, y por Merlo D y col. (1985) Abstracts, 1st Int. Cong. Plant Mol. Biol., ed.: Galau GA. El pH4-1 contiene dos fragmentos de ADN-T, un fragmento HindIII que se extiende entre las posiciones 602 y 2.290 (según ha sido definido por Barker RF y col. (1983) Plant Mol. Biol. 2:335-350) que lleva el borde izquierdo de TL y secuencias de promotor asociadas con ORF1, y un fragmento SmaI/BclI que se extiende entre las posiciones 11.207 y 14.711, teniendo un tml 3’ suprimido, un ocs intacto y el borde derecho de TL . Entre la posición del sitio HindIII 3.390 y la posición del sitio SmaI 11.207 (habiéndose convertido el sitio SmaI a un sitio BglII de inserción de ligandos de BglII) de estos ∗ Véase también Gray, L.E. Plant Science (1986) 47:45-55. dos fragmentos hay un marcador seleccionable expresable en plantas. Este marcador tiene un promotor de CaMV 19S, un gen estructural de resistencia a la kanamicina Tn5 que codifica neomicina fosfotransferasa II, y dos sitios de poliadenilación, uno a partir de CaMV y otro a partir de ORF26 de ADN-T, donado por un fragmento de ADN-T que se extiende en sitios HincII en las posiciones 21.727 y 22.440. El gen de resistencia a la kanamicina se orienta paralelo a ocs y tml y antiparalelo al promotor ORF1. La combinación ADN-T/marcador seleccionable se inserta en la posición HindIII de pSUP106, un plásmido de amplia gama de huéspedes de 11 kpb capaz de mantenerse tanto en E. coli como en Agrobacterium (Priefer UB y col. (1985) J. Bacteriol. 163:324-330; E. coli CSH52 (pSUP106) está depositado como NRRL B-15486). El ADN-T está orientado dentro del pSUP106 de tal forma que el borde derecho de TL está próximo al sitio EcoRI pSUP106, que está presente dentro del gen de resistencia al cloramfenicol de pSUP106. El pH4-1 tiene dos sitios BglII, flanqueando ambos el marcador seleccionable kan. Uno de los sitios BglII se extrajo como sigue, dejando por ello un único sitio BglII útil para la inserción de ADN codificante extraño. Se linealizó el ADN de pH41 por digestión parcial con BglII y se aisló ADN lineal de longitud completa por electroforesis. Se eliminaron entonces los extremos pegajosos del 14 5 10 15 20 25 30 35 40 45 50 55 60 65 26 BglII por incubación con el fragmento de Klenow de la ADN polimerasa I de E. coli. El ADN resultante de extremos romos se religó a si mismo y se transformó en E. coli. Los ADN de plásmido aislados a partir de transformantes resistentes a cloramfenicol se identificaron por análisis de restricción y se identificó una colonia que albergaba un plásmido denominado pH400. El pH400 era idéntico al pH4-1 excepto en la ausencia del sitio BglII entre el gen kan y el promotor ORF1, el único sitio BglII del pH400 estaba localizado entre el gen kan y el gen ocs. Ejemplo 6 Inserción de un gen ARNa de AMV4 en pH400 El ADN de pDOBA4I se digirió con BglII y después se mezcló y ligó con ADN de pH400 linealizado con BglII. Después de la transformación en MC1061 y de la selección para resistencia al cloramfenicol, las colonias se tintaron e hibridaron con una sonda de ARN4 de AMV. Se identificó una colonia que albergaba un plásmido, que se denominó pH400A4I, que tenı́a un gen ARNa insertado en el sitio BglII de pH400. El pH400A4I tiene un gen ARNa que tiene secuencias de ARN4 de AMV de longitud completa, estando orientado el gen paralelamente al gen tml de 3’ eliminado, ocs, y el marcador seleccionable para kanamicina. Ejemplo 7 Transformación de la planta Se transfirió por la técnica de emparejamiento triparental pH400A4I a A. tumefaciens LBA4404 (Ooms G y col. (1981) Gene 14:33-50), una cepa movilizante de micro-Ti, que alberga el gen vir (Ruvkun GB y Ausbel FM (1981) Nature 289:8588), que es bien conocida en la técnica, o por emparejamiento a partir de una cepa de movilización de E. coli, por ej. SM10 (NRRL B15481) o S17-1 (NRRL B-15483) (Simon R y col. (1982) Biotechnol. 1:784-791 se obtuvo tejido de hojas de tabaco de plantas Xanthimc de 4 o 5 semanas cultivadas axenicamente en cajas Magenta. La inoculación se realizó mediante una modificación del procedimiento Horsch RB y col. (1985) Science 227:1229-1231. Los inóculos se prepararon colocando dos espirales de agrobacteria en 10 ml de caldo-L. Después de suspensión por pipeteo enérgico con una pipeta Pasteur, los inóculos se podı́an utilizar inmediatamente. Las hojas se escindieron y se extrajeron las partes intercostales, las superficies cortadas se humedecieron con caldo-L para ayudar a mantener las hojas húmedas. Los trozos de hoja eran de alrededor de 2-4 mm de ancho y de alrededor de 7-10 mm de longitud. Los trozos de hoja se sumergieron en el inóculo durante 5-10 min, aunque, en algunos experimentos, los trozos de hoja fueron solamente sumergidos en el inóculo o se infiltraron con el inóculo en un matraz de vacı́o. Los trozos fueron secados sobre papel de filtro estéril y se colocaron con el anverso hacia abajo sobre placas de alimentación preparadas a partir de un cultivo de suspensión Xanthi. Las placas de alimentación tenı́an un medio SMPi (SMPi: MX− suplementado con 0,1 mg/l de ácido p-clorofenoxiacético (pCPA) y 7,5 mg/l de 6-(8,8-dimetilalilamino)purina (2iP); 27 ES 2 076 143 T3 MX− : 1,65 g/l NH4 NO3 , 1,9 g/l KNO3 , 440 mg/l CaCl2·H2 O, 370 mg/l MgSO4 ·7H2 O, 170 mg/l KH2 PO4 , 0,83 mg/l KI, 6,2 mg/l H3 BO3 , 22,3 mg/l MnSO4 ·4H2 O, 8,6 mg/l ZnSO4 ·7H2 O, 0,25 mg/l Na2 MoO4 ·2H2 O, 0,025 mg/l CuSO4 ·5H2 O, 0,025 mg/l CoCl2 ·6H2 O, 1 g/l inositol, 50 mg/l ácido nicotı́nico, 50 mg/l piroxidina·HCl, 50 mg/l tiamina·HCl, 30 g/l sacarosa, pH 5,8, solidificada con 8 g/l de agar). Los trozos de hoja se retiraron de las placas de alimentación después de 4-6 dı́as y se colocaron en medio SMPi suplementado con 500 mg/l de carbenicilina, 50 mg/l de cloxacilina, y 100-300 mg/l de kanamicina (siendo óptimo 200 mg/l). Los brotes resultantes se escindieron y se colocaron en medio MX− suplementado con 100-300 mg/l de kanamicina (siendo óptimo 200 mg/l). Ejemplo 8 Expresión en plantas Se autofertilizaron plantas de tabaco regeneradas descendientes de células transformadas por 5 10 15 20 28 A. tumefaciens LBA4404 (pH400A4I). Las semillas resultantes se germinaron en MX− suplementado con 100-300 mg/l de kanamicina (siendo óptimo 200 mg/l) para seleccionar las plantas que contenı́an el ADN-T de comportamiento de gen ARNa. La presencia de ARNa se confirmó por análisis de tinción Northern. Las plantas de tabaco no transformadas de control y las plantas que contienen gen ARNa se probaron siendo inoculadas con los cuatro ARN de AMV después de abrasión de las hojas con carborundo, un procedimiento bien conocido en la técnica. Bajo estas condiciones, es necesaria la traducción de moléculas ARN4 de AMV presentes en el inóculo para el establecimiento de una infección de AMV. Cuando se comparan con las plantas de control, se observa que las plantas que tienen un gen ARNa son resistentes a la infección por AMV o que tienen sı́ntomas retrasados o reducidos, dependiendo del grado de expresión de ARNa en tejidos de una planta particular. 25 30 35 40 45 50 55 60 65 15 29 ES 2 076 143 T3 REIVINDICACIONES 1. Una célula de planta, tejido de planta, semilla de planta o planta resistente a virus que no es infectada por un virus y que comprende una molécula de ADN integrada de forma estable que comprende un promotor, un ADNc que codifica ARN antisentido a al menos una parte de un genoma ARN de un virus de planta que tiene un genoma ARN tripartito de cadena simple, de cadena múltiple, y un terminador de transcrito, en donde dicho ADNc codifica ARN antisentido capaz de conferir resistencia viral a una planta, en donde el promotor, el ADNc, y el terminador de transcrito tienen tal posición y orientación unos con respecto a otros que el ARN antisentido puede transcribirse en una célula de planta bajo control del promotor y del terminador. 2. Una célula de planta, tejido de planta, semilla de planta o planta resistente a virus que no es infectada por un virus y que comprende una molécula de ADN integrada de forma estable que comprende un promotor, un ADNc que codifica ARN antisentido a ARN4 de un virus de planta que tiene un genoma ARN tripartito de cadena simple, de cadena múltiple, y un terminador de transcrito, en donde dicho ADNc codifica ARN antisentido capaz de conferir resistencia viral a una planta, en donde el promotor, el ADNc, y el terminador de transcrito tienen tal posición y orientación unos con respecto a otros que el ARN antisentido puede transcribirse en una célula de planta bajo control del promotor y del terminador. 3. Una célula de planta, tejido de planta, semilla de planta o planta resistente a virus que no es infectada por un virus y que comprende una molécula de ADN integrada de forma estable que comprende un promotor, un ADNc que codifica ARN antisentido a ARN1, ARN2 o ARN3 de un virus de planta que tiene un genoma ARN tripartito de cadena simple, de cadena múltiple, y un terminador de transcrito, en donde dicho ADNc codifica ARN antisentido capaz de conferir resistencia viral a una planta, en donde el promotor, el ADNc, y el terminador de transcrito tienen tal posición y orientación unos con respecto a otros que el ARN antisentido puede transcribirse en una célula de planta bajo control del promotor y del terminador. 4. La célula de planta, el tejido de planta, la semilla de planta, o la planta de cualquiera de las reivindicaciones 1, 2 o 3, en donde el virus es del grupo de virus del mosaico de la alfalfa. 5. La célula de planta, el tejido de planta, la semilla de planta, o la planta de la reivindicación 4 en donde el virus de planta es AMV. 6. La célula de planta, el tejido de planta, la semilla de planta, o la planta de la reivindicación 4 en donde el virus de planta es AMV cepa 425. 7. La célula de planta, el tejido de planta, la semilla de planta, o la planta de cualquiera de las reivindicaciones 1, 2 ó 3, en donde el virus de planta se selecciona entre el grupo que consta de un ilarvirus, un bromovirus, un cucumovirus y un hordeivirus. 8. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de 16 5 10 15 20 25 30 35 40 45 50 55 60 65 30 las reivindicaciones precedentes, en donde el ARN antisentido es complementario a una secuencia viral 5’ no traducida. 9. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de las reivindicaciones precedentes, en donde el ARN antisentido es complementario a una posición de iniciación de traducción viral. 10. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de las reivindicaciones precedentes, en donde el ARN antisentido es complementario a una secuencia viral 3’ conservada. 11. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de las reivindicaciones 1 a 7, en donde el ADNc es esencialmente ADNc de longitud completa. 12. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de las reivindicaciones precedentes, en donde el terminador del transcrito es una posición de poliadenilación. 13. La célula de planta, el tejido de planta, la semilla de planta, o la planta según la reivindicación 12, en donde la posición de poliadenilación es una posición de poliadenilación de CaMV. 14. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de las reivindicaciones precedentes, en donde dicha molécula de ADN comprende adicionalmente un marcador seleccionable o identificable en planta. 15. La célula de planta, el tejido de planta, la semilla de planta, o la planta según la reivindicación 14, en donde el marcador seleccionable codifica la neomicina fosfotransferasa. 16. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de las reivindicaciones precedentes, en donde dicha molécula de ADN comprende adicionalmente un gen ocs. 17. La célula de planta, el tejido de planta, la semilla de planta, o la planta según cualquiera de las reivindicaciones precedentes, en donde la molécula de ADN comprende adicionalmente una repetición de borde ADN-T. 18. La célula de planta, el tejido de planta, la semilla de planta, o la planta según la reivindicación 17, en donde el ADN-T es el ADN-T de pH400, en donde el pH400 puede obtenerse extrayendo la posición BglII entre el gen kan y el promotor ORF1 por digestión parcial de pH4-1 y tratamiento de pH4-1 lineal de longitud completa con el fragmento de Klenow de la ADN polimerasa I de E. coli y subsiguiente religado de los extremos romos. 19. La célula de planta, el tejido de planta, la semilla de planta, o la planta según la reivindicación 18, en donde el ADN-T es el ADN-T de pH400A4I (NRRL B-18062). 20. La utilización de una molécula de ADN según se cita en cualquiera de las reivindicaciones 1 a 19 para conferir resistencia viral a una célula de planta, tejido de planta, semilla de planta o planta. 21. La utilización de una molécula de ADN según la reivindicación 20 en donde la resistencia viral es para el virus AMV. 22. La utilización de una molécula de ADN 31 ES 2 076 143 T3 según la reivindicación 20 en donde la resistencia viral es para el virus seleccionado entre el grupo que consta de un ilarvirus, un bromovirus, un cucumovirus y un hordeivirus. 23. Un procedimiento para producir una 32 planta resistente a un virus de planta que comprende transformar una célula de planta con una molécula de ADN según se ha citado en cualquiera de las reivindicaciones 1 a 19. 5 10 15 20 25 30 35 40 45 50 55 60 NOTA INFORMATIVA: Conforme a la reserva del art. 167.2 del Convenio de Patentes Europeas (CPE) y a la Disposición Transitoria del RD 2424/1986, de 10 de octubre, relativo a la aplicación del Convenio de Patente Europea, las patentes europeas que designen a España y solicitadas antes del 7-10-1992, no producirán ningún efecto en España en la medida en que confieran protección a productos quı́micos y farmacéuticos como tales. 65 Esta información no prejuzga que la patente esté o no incluı́da en la mencionada reserva. 17