Diapositiva 1 - genoma . unsam . edu . ar
Anuncio

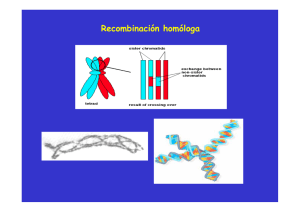
Recombinación y Ligamiento Génico Recombinación y Ligamiento Génico • Teoría cromosómica de la herencia – – – – • Evidencias citológicas de Sutton-Boveri Evidencias adicionales (Carothers, Blakeslee, etc.) Caracteres asociados al sexo Evidencias de Morgan en Drosophila Ligamiento génico y mapas genómicos – Mapas de ligamiento 9 Terminología 9 Modelos alternativos – Recombinación génica – Entrecruzamiento o crossing over 9 Quiasmas 9 Interferencia – Análisis y cálculo de distancias genéticas – Mapas de 2 y 3 puntos • Ligamiento génico en humanos – Lod scores – Uso de pedigrí Teoría Cromosómica de la Herencia Ligamiento, recombinación y mapeo • 2da ley de Mendel: Durante la formación de los gametos la segregación de una pareja génica se produce de manera independiente de las otras parejas génica. S/s SY 25% Y/y Sy sY sy 25% 25% 25% Teoria cromosómica de la herencia • Observación de gametas • Evidencias citológicas – Sutton-Boveri – Carothers y otros • Correlaciones genotipo-fenotipo – Blakeslee y otros • Genes ligados al sexo – No disyunción de cromosomas sexuales – Experimentos de Morgan-Bridges en Drosophila Interfase Profase Teoría cromosómica de la herencia (Sutton-Boveri): el paralelismo entre el comportamiento de los genes (Mendel) y los cromosomas llevó a pensar que los genes están situados en cromosomas. Metafase I Anafase I Explica la distribución igualitaria y la segregación independiente Telofase I Anafase II Telofase II Meiosis de una célula diploide con genotipo A/a:B/b Correlación Cromosoma-Fenotipo (Blakeslee) • Plantas datura stramonium con distintas trisomías • Objeciones: Integridad de los cromosomas en interfase? Solamente se aparean los cromosomas homólogos? Correlación genotipo-fenotipo Estricta correlación sexo – cromosoma dimórfico Determinación sexual • 2 tipos de determinación sexual en vertebrados: – Genética (GSD) 9 SRY (familia SOX), DMRT1 (aves) 9 Gen sin intrones, baja conservac. 9 DNA b protein – Ambiental (TSD) 9 T° como factor clave 9 Bias poblacional 9 T° altera los niveles de estrógenos y/o receptores de estrógenos Ambas estrategias son cuantitativas y no cualitativas (umbrales) Determinación sexual •Genética: •Cromosomas sexuales (heteromórficos) Sistemas XY (macho heteromorfo), X0, ZW (hembra heteromorfa) y cromosomas múltiples •Ploidía (himenópteros) •Cromosomas múltiples (nematodos) •Genes no asociados a cromosomas heteromórficos (hongos,...) •Ambiental •Temperatura (salamandras, reptiles, peces) •Substrato de fijación (gusanos y gasterópodos marinos) •Tamaño corporal (anélidos marinos, algunos peces) Determinación sexual • Determinación sexual genética: – Cromosomas sexuales tienen origen en autosomas por adquisición de male-determining role(s). – Adquisición de male-determining role(s) conlleva una diversificación de uno de ellos por falta de recombinación meiótica. – Acumulación de mutaciones, deleciones y rearreglos genómicos en el cromosoma heterogamético – Dimorfismo cromosómico (excepciones) – ¿El punto final de este proceso sería la desaparición de un miembro del par sexual? Cromosomas Cromosomas sexuales sexuales en en humanos humanos Porción no homóloga, genes ligados al X X X Región pseudoautosómica Y Genes holándricos Experimentos con cromosomas sexuales Estricta correlación sexo – cromosoma dimórfico Experimentos con Drosophila Experimentos de Morgan-Bridges en Drosophila Cruzamiento: Parental XwXw F1 x X+wY XwX+w ; XwY (1:1) - Se observan proporciones fenotípicas distintas entre los dos sexos de la progenie. - Los cruzamientos recíprocos dan proporciones diferentes. - Sin embargo 1/2000 de F1 eran hembras de ojos blancos o machos de ojos rojos! Experimentos con Drosophila Morgan sugiere que el “sexo” es un carácter y, como tal, tiene determinacion genetica (cromosomas heteromorfos). Propone que gen white está localizado en el cromosoma X y ausente en el Y. Morgan sugiere que los resultados observados en gallinas y polillas son análogos a los suyos, sólo que son las hembras las que tienen cromosomas sexuales heterogaméticos. Bridges explica la progenie excepcional postulando que eventos meióticos anormales originan hembras viables XXY y/o machos viables XO. Para probar este modelo, examina microscópicamente los cromosomas de esta progenie y obtiene el patrón genotípico predicho. Ligamiento Génico Hongos ascomicetos gametos Genotipos F1 A B -- -a b AB Ab A A a a A A B A A b B B b aB a a B 50% recombinanB tes b B b ab a a b b Proporción 1:1:1:1 Ligamiento: Estudios de Morgan con Drosophila melanogaster Mutantes de Drosophila melanogaster +: salvaje Cy: Curly sd: scalloped +: salvaje w: white ap: aptera vg: vestigial dp: dumpy sepia D: Dichaete c: curved Bar Ligamiento: Uso de cruz. prueba (Morgan) 1:1 1:1 1:1 1:1 El cruz. prueba permite inferir las proporciones de los gametos del heterocigota Ligamiento: hipótesis de Morgan Cuando dos genes se encuentran en el mismo par de cromosomas (ligados) no segregan en forma independiente. ¿Cómo se explican los recombinantes? El grado de ligamiento no es completo, porque durante la meiosis hay intercambio entre cromosomas homólogos. Crossing-overs • Uso de algas y hongos, que permiten visualizar las 4 tetradas resultantes de 1 meiosis, y múltiples marcadores: – Los cruzamientos ocurren en el estadio de 4 cromátidas – Los cruzamientos pueden ser múltiples e involucrar distintas cromátidas, incluyendo las cromátidas hermanas quiasmas Crossing-overs • Evidencia física del crossing-over quiasmas Conclusiones • Entre genes no ligados, la recombinación se produce por segregación independiente y es su frecuencia es ~ 50% (gametas recombinantes/gametas totales: Ab+aB / AB+ab+Ab+aB) • Entre genes ligados, la recombinación se produce por entrecruzamiento y su frecuencia es menor al <50% (o, a lo sumo, igual). • Si el ligamiento es total no hay recombinación y todos los gametos producidos tienen el genotipo parental. Ligamiento: simbología y terminología • Nomenclatura: pr vg / pr+ vg+ (conformaciones parentales en cis o en acoplamiento): dos alelos dominantes o tipo salvaje se encuentran en el mismo cromosoma. pr vg+ / pr+ vg (conformaciones recombinantes en trans o en repulsión): dos alelos dominantes o tipo salvaje se encuentran en cromosomas homólogos diferentes. • Los alelos que se encuentran sobre el mismo cromosoma homólogo no llevan puntuación entre ellos. • Los alelos siempre se escriben en el mismo orden. • Genes que se sabe se encuentran en cromosomas diferentes (o genes no-ligados) se separan con punto y coma. Ej: A/a; C/c • Genes que no se sabe si están ligados se representan separados por un punto. Ej: A/a . D/d Recombinación •Recombinación: producción de nuevas combinaciones alélicas. •Recombinación meiótica: cualquier proceso meiótico que genera un producto haploide con combinaciones de alelos nuevas, distintas a las presentes en los genotipos haploides que se unieron para formar el meiocito dihíbrido. – Los genotipos resultantes (output) son distintos de los originales (input) – Sólo se puede determinar directamente en organismos haploides •Se producen recombinantes por dos procesos celulares diferentes: - segregación independiente - entrecruzamiento Ambos procesos rinden frecuencias de recombinación diferentes, indicando si los genes están ligados o no. Detección de Recombinación Meiótica Detección de recombinación en haploides Detección de recombinación en diploides Mapas de ligamiento: Postulado de Sturtevant-Morgan • • • • • • La frecuencia de entrecruzamientos es directamente proporcional a la distancia entre 2 genes ligados. La frecuencia de recombinantes es inversamente proporcional a la distancia entre los genes y sirve para mapearlos entre sí. Se puede definir 1 unidad de mapa (m.u) como la distancia entre dos genes para los que 1/100 de sus gametas es recombinante. Recombinantes / totales x 100: Unidades de recombinación o de mapa aprox. igual a un CentiMorgan (función de mapeo) 1 m.u = 1 cM frec. de recomb de 0,01 o 1% Mapas de ligamiento: Postulado de Sturtevant-Morgan • Las distancias son aditivas • Se define el concepto de locus Prueba de los tres puntos: permite determinar si tres genes están ligados y, si lo están, en qué orden se ubican y qué distancia guardan entre sí. Cruzamiento de prueba 18,5%=18,5 m.u 13,2%=13,2 m.u 6,4%=6,4 m.u Todos los loci están ligados ya que las frecuencias de recombinación son menores al 50% en todos los casos. Los loci v y cv muestran el mayor valor de FR, lo cual indica que deben estar más distantes. El mapa podría ser: El cruzamiento de prueba entonces era: v+ ct cv/v ct+ cv+ x v ct cv/v ct cv 18. 5 ó 19.6 m.u? 268 + 3 + 3 + 5 + 5 = 284 19.6% Recombinantes dobles Los cruzamientos de prueba de tres puntos permiten evaluar el ligamiento entre 3 (ó más) genes en un solo cruzamiento. Interferencia • La frecuencia de dobles recombinantes es menor que la de recombinantes simples porque se necesitan dos eventos para producirlas. • Sin embargo, la frecuencia de estos recombinantes dobles es aún menor de lo esperado… En nuestro ejemplo: Frec. Recomb. v-cv (recomb. dobles) = frec. v-ct x frec. ct-cv Frec.v-cv = 0.132 x 0.064 = 0.0084 (0.84%), o sea 12 recomb. Dobles esperados, sin embargo se observaron sólo 8. Los entrecruzamientos adyacentes son eventos independientes? • Interferencia (I) = 1 - c.o.c = I = 1 - 8/12 = 4/12 = 1/3 ó 33% Por regla general, a medida que disminuye la distancia entre los loci, < c.o.c y > I. Cabe esperar que tres loci con distancias cortas muestren pocos o ningún entrecruzamiento doble. c.o.c = coeficiente de coincidencia Mapas de ligamiento: test de χ2 Si hay un distanciamiento de la segregación independiente hay ligamiento y si la frecuencia de recombinación es menor al 50 % los genes están ligados.., ¿pero cuánto menor? Por otro lado, la recombinación no es lo único que afecta las proporciones de la progenie producida. Algunas clases recombinantes pueden originarse por azar, por fallas experimentales o no observarse porque son letales. Para dar validez estadística se aplica el test de χ2, tomado como hipótesis nula el no ligamiento de los genes o mejor dicho que estos segregan independientemente. El rechazo de esta hipótesis se considera como prueba de P que existe ligamiento. Ejemplo: Proporciones gaméticas del dihíbrido Se cruzan 2 homocigotas (A/A . B/B) x (a/a . b/b) La F1 se cruza con un tester homocigota doble recesivo 225/500 = 45 % recombinantes El porcentaje de recombinantes es <50%, pero… Hay ligamiento? Prueba de χ2 Proporciones alélicas: Según la 1era y 2da ley de Mendel, los valores esperados de gametas serían: Sin embargo…no todos los genotipos son igualmente viables, de modo que la proporción A:a o B:b no siempre es 0.5:0.5 Se calculan las proporciones alélicas reales en caso de segregación independiente. A = 255/500 a = 245/500 B = 254/500 b = 246/500 gl: (nº de filas -1) x (nº de columnas -1) gl = (2 -1) x (2 - 1) = 1 Ho: los loci (o genes) no están ligados Para un valor de χ2 de 4.97 con 1 gl, según tablas p < 0.025 (2.5%) Como esta probabilidad es menor del 5% la hipótesis nula debe ser rechazada y por lo tanto hay probabilidad de que haya ligamiento. Estudios de ligamiento en humanos El mapeo de grupos de ligamiento en humanos es mas difícil de calcular debido a: 1. 2. 3. No es posible hacer entrecruzamientos controlados en humanos dificultando el calculo de frecuencias de recombinación a partir de dihibridos producidos por azar. En general se produce un bajo numero de progenie dificultando la obtención de datos que puedan ser tratados estadísticamente. El genoma humano es demasiado grande, lo que significa en que promedio las distancias entre genes son grandes. Para obtener valores de frecuencias de recombinación confiables se pueden combinar datos de muchos cruzamientos idénticos. La forma de hacer esto es calculando los Lod Scores (Lod= log of odds (logaritmo de la relación) entre la probabilidad de que 2 genes estén ligados / que segreguen independientemente) Mapas genéticos vs mapas físicos Mapeo genético: • Análisis de cruzamientos planificados, pruebas de progenie y análisis de pedigrí. Es decir, mediante el análisis comparativo de fenotipos recombinantes. Mapeo físico: • Análisis de clones genómicos mediante mapeo de contigs (de los cuales se conocen, por ejemplo, sus mapas de restricción), combinado con secuenciación nucleotídica, por hibridación in situ, u otros métodos que NO involucran cruzamientos y análisis de recombinantes. • Las distancias se calculan en nucleótidos o mediante medición de cromosomas. • El orden físico de los genes en el cromosoma coincide con el orden que surge de su mapeo genético pero no es proporcional para todo el cromosoma (distinto cerca del centrómero que cerca del telómero) porque las frecuencias de formación de quiasmas son distintas ¿Para qué se usa el mapeo genético en la era de los genomas completamente secuenciados? Organismos no secuenciados Determinación de función y expresión génica Asignación de genes a fenotipos (enfermedades) Estudios evolutivos Análisis de QTL (quantitative trait loci) Mapa genético (fenotípico) en tomate Desde el mapa genético al mapa físico