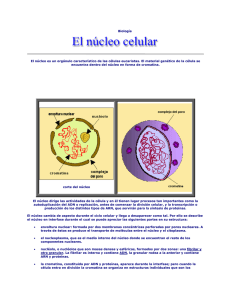
CROMOSOMAS EN EUCARIOTAS Estructura externa Forma Tamaño Número Diferenciación longitudinal Brazos Telómeros Cromómeros Centrómero Estructura interna Diferenciación lateral Estructura química cromátida DNA RNA Proteínas histónicas Proteínas no histónicas enzimas.... Estructura externa: Forma brazo TELOCENTRICO SUBMETACENTRICO Organizador nucleolar METACENTRICO CARIOTIPO CENTROMERO - Contiene el cinetocoro necesario en la división celular - Es heterocromatina constitutiva - Contiene puntos de anclaje de proteínas unidas a las fibras del uso - puede ser localizado o difuso - ¿Que le ocurre en meiosis I, que no se divide? ¿se bloquea? Modelo de disposición de moléculas de DNA en el centrómero y en las cromátidas hermanas durante la 1ª anafase de la meiosis ORGANIZADOR NUCLEOLAR - muchos genes en tanden que codifican para el ARN ribosómico - Heteropignosis + en profase - Responsable de la organización del nucleolo en cada ciclo - Es heterocromatina constitutiva TELOMEROS - son los extremos de los crs. - sirven para fusionar los crs durante la profase - son secuencias repetidas de DNA, que no dan RNA, pero con la función de replicar el cromosoma. En el hombre la secuencia repetida es TTAGGG. PROBLEMA EN LA REPLICACION DE LOS EXTREMOS CROMOSÓMICOS ¿COMO CEBAR EL ULTIMO TRAMO DE LA CADENA RETRASADA? CROMONEMA Fibra elemental de cada cromatina (ADN + proteína) CROMOMEROS Enrollamiento intenso del cromonema. Unidos unos a otros a modo de cuentas de rosario REGIONES HETEROCROMATINA Zona teñida con gran intensidad, muy compactada con genes inactivos REGIONES EUCROMATINA Zonas menos teñidas, menos compactada con genes activos BANDAS CROMSOMICAS Tinciones específicas que permiten ver modelos de bandas a lo largo del crs. La posición y tamaños de esta banda son específicas de cada crs. Y se usan como marcadores. Bandas Q - Quinacrina Bandas G - Giensaa: Oscuras: ricas A+T replicación tardía genes específicos Clara: ricas G+C replicación temprana genes básicos Bandas R - Giensa reversa CROMATINA Estructura y función Cromatina: estructura y función • Primera clase • Segunda clase - "Cromosomas" virales, - Procesos celulares en bacterianos y eucariotas el contexto de la cromatina: - Cromatina: Replicación Niveles de compactado: Regulación de la Nucleosoma, expresión génica Fibras, Cromosomas en interfase y mitosis EUCARIOTAS, PROCARIOTAS, VIRUS – ADN CONDENSADO POR ASOCIACIÓN CON PROTEÍNAS ADN de ØX174 Cromosomas eucarióticos en mitosis Bacteria lisada – ADN “compacto” EN VIRUS: ADN o ARN condensado dentro de las partículas virales por interacciones inespecíficas con proteínas de la cápside VMT: ARN en hélice dentro de la cápside CROMOSOMA PROCARIÓTICO •Cromosomas circulares •Cromosomas lineales •Ambos (ej. Agrobacterium tumefaciens) LISIS E.coli Lisis: fibras en forma de bucles unidas a la envoltura rota de la célula CROMOSOMA PROCARIÓTICO • Dominios en bucles de 40 kb aprox. de ADN superenrrollado; asociado a proteínas básicas • No se sabe cómo se mantienen unidos en su base • Cada dominio es independiente de los otros PROTEÍNAS BÁSICAS DETECTADAS EN BACTERIAS Proteína Composición Contenido por células Semejanza con eucariontes HU Dímero subunidades a y b de 9.000 d. 40.000 dímeros Histona H2A H Dímero subunidades idénticas de 28.000 d. 30.000 dímero Histona H2B IHF Subunidad a de 10.500 d. Subunidad b de 9.500 d. Desconocido Desconocida H1 Subunidad de 15.000 d. 10.000 copias Desconocida HLP1 Monómero de 15.000 d. 20.000 copias Desconocida P Sububnidad de 3.000 d. Desconocido Protaminas CROMOSOMA EUCARIÓTICO - CROMATINA DIFERENTES NIVELES DE COMPACTADO 6 veces compactado 40 veces compactado > 1000 veces compactado > 10000 veces compactado CROMATINA - NUCLEOSOMAS Suspension de nucleos a baja fuerza ionica: lisis y liberación de fibras de cromatina de 30 nm FIBRA DE 30 nm fuerza iónica FIBRA DE 10 nm « collar de perlas » Nucleasa microcóccica 805 605 405 205 Digestión suave con nucleasa microcóccica: se obtienen múltiplos de una determinada unidad de longitud: Repetición nucleosomal Tratamiento más drástico con nucleasa microcóccica: 1) Se liberan mononucleosomas 2) Se liberan nucleosomas recortados 3) Se liberan cores de histonas PARTÍCULA NUCLEOSÓMICA CENTRAL - 146 pb de ADN enrollados alrededor de la PARTÍCULA NUCLEOSÓMICA CENTRAL o CORE de histonas (2 vueltas) -Tetrámero de H3 y H4 -2 dímeros de H2A y H2B OCTÁMERO 6 nm NUCLEOSOMA -146 pb ADN enrollados en el core -ADN ligador de largo variable - Histona H1 Especie Repetición nucleosomal Longitud del ligador S. cerevisiae 160-165 13-18 Erizo de mar 260 (espermatozoide) 110 D. melanogaster 180 33 Humano 185-200 38-53 EN PROMEDIO EN LA CROMATINA HAY OTRAS PROTEINAS: HMGs, protaminas, y todas las enzimas necesarias para la duplicación, transcripción, etc. del ADN. HISTONAS DEL CENTRALES o del CORE - Proteínas pequeñas (11- 15 Kda) - Muy conservadas - RELEVANCIA - Alta carga + (ricas en Lys y Arg) - Dominio “pliegue histónico” - 3 hélices- separadas por asas cortas no estructuradas - Colas N-terminales ¡¡IMPORTANTES!! Contenido en moles % de Histona Nº residuos Pm Modificación Lys Arg H1 213 21.000 27,7 1,4 Fosforilación H2A 129 13.900 10,9 9,3 Fosforilación, acetilación H2B 125 13.774 15,2 6,1 Fosforilación, acetilación H3 135 15.273 9,6 13,3 Acetilación, metilación fosforilación 13,7 Acetilación, metilación fosforilación H4 102 11.236 10,8 ¿Cómo se arma un nucleosoma?: Las histonas se asocian de manera ORDENADA para formar un nucleosoma 1) Se forman heterodímeros H3-H4 2) Se forma tetrámero H3 - H4 3) Unión al ADN 4) Dos dímeros H2A-H2B son reclutados ESTRUCTURA ATÓMICA DEL NUCLEOSOMA A ALTA RESOLUCIÓN EJE DIÁDICO -Tetrámero se une a 60 pb centrales de los 146 -Dímeros H2A-H2B a 30 pb a cada lado de esos 60 pb -Hélice N-ter de cada H3 contactan 13 pb en cada extremo del ADN INTERACCIONES ADN-histonas - 14 sitios de contacto diferentes, uno por cada vez que el surco menor del ADN está frente al octámero de histonas - 142 puentes de hidrógeno (entre a.a. y O del enlace fosfodiéster cerca del surco menor) - enlaces por cargas + de histonas y – del ADN (INTERACCIONES INESPECÍFICAS) Colas N-terminales de histonas del core dirigen el enroscado del ADN en sentido levógiro: ¿Cómo se disponen los nucleosomas en la fibra de 10 nm? 2 factores favorecen la ubicación de un nucleosoma: 1) Facilidad para que se curve la doble hélice: secuencias ricas en AT en surco menor 2) Presencia de proteínas no histonas unidas al ADN con gran afinidad - Longitud variable en el genoma - Posicionamiento dinámico Fibra de 30 nm: Una vez que se forman los nucleosomas, la fibra de 10 nm se condensa en el siguiente nivel de compactado 2 modelos para la fibra de 30 nm: SOLENOIDE ZIGZAG ADN ligador INTERVIENEN: - H1 - Colas N-terminales de las histonas del core ADN ligador Histona H1 o ligadora - Proteína pequeña; un poco más grande que las del core - Secuencia menos conservada - Estructura típica ¿Dónde se ubica en el nucleosoma? Modelos: Protección de 20 pb adicionales de ADN de digestión con nucleasa microcóccica Al aumentar la concentración salina en presencia de histona H1 se forma una fibra de 30 nm: Las colas N-terminales de las histonas intervienen en la formación de la fibra de 30 nm Recordar : Las colas de las histonas son blanco de modificaciones post-traduccionales Compactado en la fibra de 30 nm, un cromosoma humano aún mediría 0.1 cm = 10 veces más grande que el núcleo celular!! Niveles de mayor compactado (?) que varían en el ciclo celular en respuesta a señales del ciclo celular Cromatina saliendo de un núcleo lisado en interfase Cromosoma mitótico CROMATINA EN INTERFASE * La cromatina es una estructura fluida y dinámica en la que se deben "exponer" las secuencias de ADN para que se den los diferentes procesos celulares. * Estructura de cromosomas en interfase: cromosomas plumosos, cromosomas politénicos * 2 «formas» de cromatina en interfase: EUCROMATINA y HETEROCROMATINA CROMOSOMAS “ESPECIALES” EN INTERFASE CROMOSOMAS PLUMOSOS («lampbrush») Cromosomas apareados en premeiosis en oocitos inmaduros de anfibios – CROMOSOMAS GIGANTES BUCLES: genes que se expresan EJE: cromatina condensada (ANDAMIAJE CROMOSÓMICO) CROMOSOMAS POLITENICOS Cromosomas de larvas de insectos; múltiples ciclos de síntesis de ADN sin división celular Los cromosomas homólogos se mantienen unidos, paralelos BANDA INTERBANDA -5000 bandas e interbandas -3 a 30 kb = 0.005 a 0.0005 µm - Interbandas: 5 % genoma - Bandas: > condensación cromatina y/o > contenido de proteínas - Patrón reconocible -Genes en interbandas se expresan más D. melanogaster CROMOSOMAS POLITENICOS ECDISONA Tiempo CAMBIOS EN LA EXPRESION GENICA APARICIÓN DE «PUFFS»: la cromatina se descomprime y hay transcripción MAS ADELANTE VEREMOS LOS MECANISMOS IMPLICADOS EN ESTA «APERTURA» DE LA CROMATINA ANILLOS DE BALBIANI (Chironomus tentans): LO QUE OCURRE EN CROMOSOMAS PLUMOSOS Y POLITENICOS EN CUANTO A PODRIA SER LO QUE OCURRE EN TODOS LOS CROMOSOMAS EN INTERFASE EUCROMATINA Y HETEROCROMATINA En la cromatina se distinguen zonas más y menos densas EUCROMATINA Estructura menos condensada, compuesta mayormente de fibra de 30 nm HETEROCROMATINA - Forma altamente condensada - Incluye proteínas adicionales - Genes no expresados (silenciados) en la mayoría de los casos; repetidos (tandems) de secuencias cortas - Mantenimiento de telómeros y centrómeros de cromosomas TELÓMERO -Secuencias cortas repetidas Ej. 5´TTAGGG 3’ (humano) - Gran parte monocatenario (3’) formando una estructura especial, resistente a nucleasas Estructura especial de la cromatina: - desacetilada - asociada a proteínas especiales CENTRÓMERO - Resistencia en la mitosis; unión a huso durante la mitosis a través del cinetocoro - Diferente largo en los diferentes organismos Ej. levadura: 125 pb humano: varios miles de Kb - Secuencias repetidas no características- satélites -Histonas desacetiladas, metiladas Histonas especiales ej CENP-A, variante de H3 (N-terminal con extensión; unión a cinetocoro?) -Docenas de proteínas asociadas CROMOSOMAS EN MITOSIS - Nivel máximo de compactado; los cromosomas se observan como cuerpos bien definidos - Asociados a diferentes complejos de proteínas. si se extraen las histonas, se observa en microscopio electrónico bucles de ADN unidos a un andamiaje nuclear (40-90 kb = 10-30 nm) Proteínas de andamiaje nuclear: - topoisomerasa II en base de los lazos : control en replicacion y transcripción? - SMCs “Structural maintenance of chromosomes” SMCs - COHESINAS - CONDENSINAS (+ ATP : formación de bucles) Formación de complejos en forma de anillos ORGANIZACIÓN DE LOS CROMOSOMAS: BANDEADO DE PROTEÍNAS G Bandas G: bajo contenido de GC Bandas R: alto contenido de GC – genes + transcriptos Organización mantenida en la evolución:: IMPORTANTE La cromatina es una estructura dinámica:porciones de ésta se descompactan para que tengan lugar los distintos procesos celulares - Replicación - Regulación de la expresión génica REPLICACIÓN En el sitio de replicación la fibra de 30 nm se desorganiza. Inmediatamente después de la replicación el ADN se asocia a nucleosomas. Nuevos nucleosomas : 2 posibilidades 1) los nucleosomas viejos se desarman y sus histonas + nuevas histonas forman nuevos nucleosomas 2) Los cores “viejos” se mantienen Experimento: Sobrecruzamiento de histonas Células crecen en presencia de a. a. PESADOS • Replicación en presencia de a.a. livianos:: histonas nuevas son livianas • Se forman octámeros • Purificación de nucleosomas y fraccionamiento en gradiente de densidad nucleosoma parental A) Se conservan los octámeros A) dos densidades histonas sintetizadas de novo (livianas) B) Se forman octámeros nuevos (mezcla de histonas) B) gradiente de densidades B) parece ser la correcta In vivo el ensamblaje de los nucleosomas no es un proceso espontáneo. Se requieren chaperonas histónicas (ej. CAF-I, RCAF) CROMATINA Y TRANSCRIPCIÓN POSICIONAMIENTO DE NUCLEOSOMAS: A veces es un estorbo, a veces una ventaja: depende de dónde queden los sitios de unión de factores transcripcionales Si quedan escondidos, la cromatina deberá ser modificada para que los factores de transcripción puedan actuar. POSICIONAMIENTO DE LOS NUCLEOSOMAS POSICIONAMIENTO EXTRÍNSECO: El primer nucleosoma se posiciona en un determinado lugar, por secuencia o por proteína unida al ADN. El resto determinado por el primero 200 pb ¿Qué pasa si los sitios de unión de factores de transcripción en un promotor NO están accesibles? - Modificaciones de la estructura de la cromatina asociados a transcripción se han observado in vitro e in vivo - Esas modificaciones, ¿son necesarias para la transcripción o son consecuencia de la misma? Se ha demostrado que en algunos promotores el “remodelado” de la cromatina puede tener lugar aún en ausencia de transcripción PERO.... ¿Cómo se remodela la cromatina? 1) COMPLEJOS REMODELADORES DE LA CROMATINA ATP-dependientes 2) MODIFICACIONES POST-TRADUCCIONALES DE LAS HISTONAS MODIFICACIONES POST-TRADUCCIONALES DE LAS HISTONAS DEL CORE -Lisinas y Serinas de histonas del core sufren acetilación, metilación, fosforilación, ubiquitinación.... - Mutaciones causan alteraciones de la expresión: Ej. H4: *dominio R (aa 17 a 29) : implicado en represión *dominio A (aa 1 a 16) : implicado en activación -Modo de acción: 1) modificación de la interacción con ADN (carga) 2) Lugares de unión para proteínas DESACETILACIÓN DE HISTONAS - Grandes complejos proteicos - Reclutados a determinados represores específicos promotores por 1) Ume6 se una a promotor; interacción con Sin3 2) Sin3 es parte de complejo con RPD3 (HDAC) COMPLEJOS DE ACCIÓN REPRESIVA SILENCIAMIENTO Complejos de proteínas que establecen estructuras muy organizadas de la cromatina (de tipo de heterocromatina) Ej. telómeros de S. cerevisiae 1) Rap1 se telómeros une 2) Rap1 recluta Sirs 3)Interacción con histonas hipoacetiladas 4) Sirs se agregan a Las figuras de esta presentación han sido extraídas de: - Lewin, B. 2004. Genes VIII. Pearson Prentice-Hall. -Watson, J.D. et al. 2004. Molecular Biology of the Gene. 5ª ed. Pearson BenjaminCummings. -Lodish, H. et al. 1999. Molecular Cell Biology. 4a ed. W.H. Freeman & Co. - Alberts, B. et. al. 2002. Molecular Biology of the Cell. 4a ed. Garland Publishers -Travers, A. (1999) “The location of linker histone in the nucleosome”; TIBS 24: 4. - Thoma, F.; Koller. T.H. and Klug A. (1979) “Involvement of the histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin”. J. Cell. Biol. 83; 403 ORGANIZACIÓN DE LOS ACIDOS NUCLEICOS EN PROCARIOTAS - Ac. Nucléicos desnudos sin membrana - no histonas - no cinetocoro, ni aparato mitótico . Condensación permanente de crs. - sin secuencias repetidas - el DNA esta en el genóforo ( crs bacteriano ) ORGANIZACIÓN DE LOS ACIDOS NUCLEICOS EN EUCARIOTAS - Ac. Nucléicos en núcleo con membrana - cubierto de proteinas histónicas y no histónicas - hay cinetocoro y aparato mitótico . Distintos grados de condensación del crs. ( eucromatina y heterocromatina) - con secuencias repetidas - el DNA esta organizado en distintos cromosomas ORGANIZACIÓN DE LOS ACIDOS NUCLEICOS EN MESOCARIONTES ( CRS. DE TRANSICION) DE PROCARIOTAS - Ac. Nucléicos desnudos sin proteínas - ausencia de cinetocoro y ap. Mitótico - condensación permanente de crs. DE EUCARIOTAS - presencia de membrana - con secuencias repetidas - el DNA esta organizado en distintos cromosomas Tema 12 El cromosoma eucariótico 73 El cromosoma eucariótico Relación existente entre el DNA, las histonas y la cromatina Niveles de empaquetamiento del DNA en los cromosomas Diferencias entre eucromatina y heterocromatina Bandeos cromosómicos y su relación con la compactación del DNA Función y estructura de centrómeros y telómeros El paisaje genómico: la complejidad de las secuencias del DNA eucariótico 74 La célula eucariota En un principio, el material genético se identifico como una masa densa y granular denominada cromatina: reacción con ciertas tinciones – Material oscuro, coloreado. 75 La célula eucariota Durante la división celular la cromatina se compacta en unidades discretas, los cromosomas. El ciclo celular consta, básicamente, de dos fases: Interfase Actividad celular sintética y núcleo discreto Mitosis Huso acromático Cada célula hija posee dos copias de cada cromosoma (homólogos) 76 Empaquetamiento del DNA Una de las características comunes de la organización del material genético es su alto grado de compactación: masa compacta que ocupa un espacio bien delimitado. El genoma humano tiene unas 3.000 megabases (1-2m) y un núcleo ocupa unas 6µm. 77 Empaquetamiento del DNA Doble hélice Nucleosoma (Octámero de Histonas) Solenoide (Histona H1) Composición del Cromosoma (Cromatina) Proteínas : básicas -> histonas ácidas -> no histonas (~30%) DNA RNA (1%) Tasa de empaquetamiento: 1:50.000-100.000 78 Empaquetamiento del DNA 79 Empaquetamiento del DNA 80 Empaquetamiento del DNA Existe una matriz proteica que forma el esqueleto del cromosoma. 81 Empaquetamiento del DNA Las fibras de DNA se unen al esqueleto en sitios específicos denominados MARs (o SARs) 82 El cariotipo humano y de ratón Bandas cromosómicas (Bandas G) Los genes tienden a localizarse en las regiones entre bandas (>%G-C) 83 Centrómero y telómero Centrómero Telómero Elemento de DNA responsable de la segregación en mitosis y meiosis (no distingue entre cromosomas). Elemento de DNA que sella los extremos de los cromosomas y les confiere estabilidad. Está compuesto de proteínas de unión a los microtúbulos y puede poseer gran cantidad de DNA repetitivo Integridad estructural Asegura la replicación de los extremos del cromosoma Papel en el apareamiento cromosómico y la arquitectura tridimensional del núcleo Envejecimiento celular y cáncer 84 El Telómero El problema de la replicación de los extremos cromosómicos 85 El Telómero 86 El Telómero Estructura del tapón telomérico 87 El Telómero Síndrome de Werner Mutación en el gen WRN, que codifica una helicasa que participa en la estructura del tapón telomérico 88 13 / 11 / 2008 Los números del genoma humano Total de genes estimado Genes identificados 19073 Genes secuencia conocida Descripción fenotípica Otros Genes mitocondriales 12527 374 6172 Genes con localización física 1097 nes localizados por cromosoma 37 11419 717 617 206 20.000-25.000 429 532 683 508 406 427 413 717 601 641 356 328 443 664 160 727 277 139 286 45 89 Características de los genes humanos que codifican proteínas. Característica Mediana Promedio Tamaño muestra Tamaño exones 122 bp 145 bp 43.317 Número exones 7 8.8 3.501 Tamaño intrones 1.023 bp 3.365 bp 27.238 3’ UTR 400 bp 770 bp 689 (crom. 22) 5’ UTR 240 300 463 (crom. 22) Secuencia codificadora 1.100 bp 1.340 bp 1.804 (CDS) 367 aa 447 aa Extensión genómica 14 kb 27 kb 1.804 90 Los genes humanos varían enormemente en tamaño y cantidad de exones 91 La densidad de genes no es homogénea Región del HLA de tipo III en el cromosoma 6p21.3 Gen de la distrofina en el cromosoma Xp21 92 La densidad génica se correlaciona con el bandeo cromosómico Xp21 6p21.3 BANDAS G Claras ▪Comparativamente ricas en GC ▪Sensibles a la DNAsa ▪Condensación tardía en el ciclo celular ▪Replicación temprana ▪Alta densidad génica ▪Pobres en secuencias repetidas oscuras ▪Comparativamente ricas en AT ▪Insensibles a la DNAsa ▪Condensación temprana en el ciclo celular ▪Replicación tardía ▪Baja densidad génica ▪Ricas en secuencias repetidas 93 Aunque es raro, existen genes solapados y anidados A) Región del HLA de tipo III en el cromosoma 6p21.3 B) Intrón 26 del gen de la neurofibromatosis tipo 1 94 El tamaño de los genomas No existe una buena correlación entre el tamaño del genoma y complejidad genética Existe una tamaño mínimo del genoma necesario para incrementar la complejidad Se detecta una gran variación en el tamaño del genoma dentro de un mismo phylum 95 Secuencias de DNA repetitivas Generalmente, los genes son codificados por secuencias únicas de DNA Los genomas más grandes dentro de un mismo phylum no contienen necesariamente más genes Una gran parte del ADN repetitivo puede proceder de los elementos transponibles 96 Contenido en repeticiones del genoma humano Derivados de elementos transponibles Pseudogenes procesados Repeticiones de secuencias sencillas Duplicaciones segmentales 10-300 kb Bloques de secuencias repetidas en tándem (centrómeros, telómeros,...) Derivados de trasposones 97 Contenido en repeticiones del genoma humano Elementos H. sapiens D. melanogaster C. elegans A. thaliana LINE/SINE 33.40% 0.70% 0.40% 0.50% LTR 8.10% 1.50% 0.00% 4.80% DNA 2.80% 0.70% 5.30% 5.10% Total 44.40% 3.10% 6.50% 10.50% 98 ORGANIZACION GENERAL DEL GENOMA HUMANO GENOMA NUCLEAR GENOMA MITOCONDRIAL genes y secuencias relacionadas 25% ADN extragénico 75% Codificante 2.5% No codificante 22.5% Moderada o altamente Repetido 30% 35000 genes proteínas 500 RNA funcionales Repetidos dispersos 12% Repetidos en tándem o agrupados 18% Satélite 4 genes rRNA Cortos (SINEs) Largos (LINEs) >40 tRNA Minisatélites Microsatélite >500 otros RNA funcionales Intrones, secuencias no traducidas Secs. únicas o bajo número de copias 45% Pseudogenes Fragmentos génicos 99