Regulación de la expresión génica Regulación de la expresión génica
Anuncio

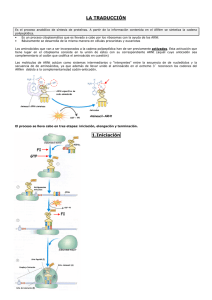
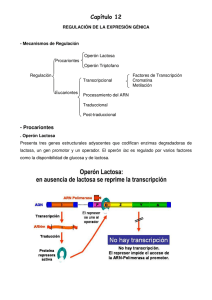
Regulación de la expresión génica Regulación de la expresión génica z z z z z z z z Mecanismos y sistemas que controlan la expresión de los genes Utilidad de la expresión génica Niveles de control de la expresión génica Diferencia entre genes y elementos regulatorios Regulación génica en procariotas Regulación génica en eucariotas En las bacterias, la regulación de la expresión génica mantiene la flexibilidad interna, activando e inhibiendo genes en respuesta a cambios ambientales En organismos eucariotas multicelulares, la regulación de la expresión génica lleva a cabo la diferenciación celular 1 Niveles de control génico ARNm procesado 2 Genes y elementos reguladores z z z Genes estructurales: codifican proteínas metabólicas o estructurales Genes reguladores: codifican ARN o proteínas que interactúan con otras secuencias y afectan la transcripción o la traducción Elementos reguladores: secuencias de ADN que no se transcriben pero afectan la expresión de los genes 3 Proteínas de unión a ADN 4 Regulación de la expresión génica en procariotas Tema 8 Operón z Los genes funcionalmente relacionados de las bacterias con frecuencia se agrupan como una única unidad de transcripción llamada operón. z Un operón típico: – – – un promotor genes estructurales un sitio operador 5 Control de la transcripción z Control negativo: una proteína reguladora actúa como represora, uniéndose al ADN e inhibiendo la transcripción. z Control positivo: una proteína reguladora actúa como activadora, estimulando la transcripción. 6 Operones z Inducibles – – z Positivo Negativo Reprimibles – – Positivo Negativo Con control negativo 7 8 El operón lac de E. coli 9 10 z El operón lac es inducible negativo: un gen regulador produce un represor que se une al operador y evita la transcripción de los genes estructurales. La presencia de alolactosa inactiva al represor y permite la transcripción. Control positivo y represión por catabolitos 11 12 El operón trp de E. coli z El operón trp es un operón reprimible negativo que controla la biosíntesis del triptófano. En un operón reprimible la transcripción está normalmente encendida y debe reprimirse. 13 Atenuación: Terminación prematura de la transcripción 14 z En la atenuación, la transcripción se inicia pero termina en forma prematura. 15 ARN antisentido en la regulación génica z z El ARN antisentido es complementario a otra secuencia de ARN o ADN. En las bacterias puede inhibir la traducción al unirse a secuencias en la UTR 5’ del ARNm y evitar la unión al ribosoma ARN antisentido en la regulación génica 16 Regulación de la expresión génica en eucariotas Tema 10 La estructura de la cromatina y la regulación génica 17 18 Control transcripcional z Activadores de la transcripción, coactivadores y represores 19 Intensificadores y aislantes 20 Control génico a través del procesamiento del ARN 21 Control génico a través de la estabilidad del ARN 22 Control génico por microARNs. Silenciamiento del ARN 23 24 Control traduccional y postraduccional z La iniciación de la traducción puede verse afectada por proteínas que se unen a secuencias específicas en el extremo 5’ del ARNm. z La disponibilidad de ribosomas, ARNt, factores de iniciacion y elongación y otros componentes del aparato traduccional puede afectar la velocidad de traducción. Tema 11: Síntesis de proteínas 25 Código genético z Triplete – z z z 4 Bases 61 codones para aa Degeneración del código Codones sinónimos ARNt z z z z 1 tipo de ARNt → 1 tipo de aa 30 a 50 tipos por célula + de 1 ARNt para cada aa: isoaceptores (difieren en el anticodón) 50 ARNt y 61 codones: ¿Qué pasa con los que no están?: Tambaleo 26 27 Marco de lectura: z Codón de iniciación – AUG: – N-formil metionina; metionina – – – z GUG UUG Codón de terminación – – – UAA UAG UGA 28 29 Traducción Síntesis de proteínas z 1. 2. 3. Unión del aa a su ARNt Iniciación Elongación Terminación 30 Unión del aa a su ARNt 20 aminoacil ARNt sintasas: 1 por aa 31 32 1. Iniciación 1. 2. 3. 4. 5. ARNm Las subunidades mayor y menor del ribosoma Factores de iniciación initiator ARNt iniciador con N-formil metionina (fMet-ARNtfMet) GTP Etapas 1. 2. 3. Unión de ARNm a la subunidad pequeña del ribosoma. ARNt iniciador se une al ARNm por apareamiento de bases: codón-anticodón. La subunidad grande se une y se forma el complejo de iniciación. 33 34 2. Elongación 1. 2. 3. 4. El complejo 70S ARNts cargados con su aa Factores de elongación: EF-Ts, EF-Tu, EFG GTP 35 36 3. Terminación z z z z z El ribosoma transloca a un codón de terminación No existen ARNts para los codones stop Ningún ARNt ingresa al sitio A Se unen los factores de liberación: RF1, RF2 y RF3, que reconocen los codones stop: UAA y UAG, y RF2 reconoce UGA y UAA. El RF3 forma un complejo con GTP y se une al ribosoma 37 38 Interacciones ARN-ARN z z z Shine-Dalgarno – ARNr 16S ARNr 16S – ARNr 23S Codón (ARNm) – Anticodón (ARNt) Polirribosomas z En células procariotas y eucariotas se unen a una única molécula de ARNm varios ribosomas, generando un polirribosoma 39