Inmunolocalización de Enzimas Degradadoras de Polisacáridos de
Anuncio

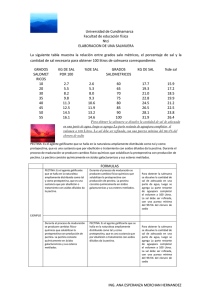
Inmunolocalización de Enzimas Degradadoras de Polisacáridos de Pared Celular de Plantas Producidas por Cepas de Aspergillus sp. Irene Rivera del Río Tutor: Dr. Guillermo Aguilar Osorio Departamento de Alimentos y Biotecnología Introducción: La pared celular de las plantas funciona como una primera barrera de protección a los microorganismos patógenos. Al iniciarse el proceso de colonización, ya sea saprofítico o patogénico, la fuente de carbono compleja más accesible es la pectina, presente en la lamela media, la cual debe ser degradada para permitir la penetración y colonización. Un microorganismo común en suelo y plantas es Aspergillus sp., hongo microscópico, saprofítico, filamentoso. La pectina es un heteropolisacárido constituyente de la pared celular de plantas, la cual presenta una estructura molecular compleja debido a sus múltiples tipos de azúcares monómericos y a sus múltiples enlaces. La molécula contiene homogalacturanos, ramnogalacturanos, polímeros neutros como arabinanos, galactanos y arabinogalactanos. Su esqueleto consiste de una región “lisa” de ácido α-(1?4)-D-galacturónico, interrumpidos por residuos de (1?2)-L-ramnopiranosilo ramificado altamente sustituidas por residuos de azucares neutros. Dichas interrupciones se denominan región “peluda”. La hidrólisis de pectina involucra la producción de enzimas pectinolíticas por Aspergillus sp.. La maquinaria enzimática debe ser tan versátil como la complejidad estructural de la pectina, entre las enzimas producidas las más notables son la poligalacturunosa, pectin y pectato liasa y pectin esterasas, dirigidas la región lisa. Las poligalacturonasas (E.C. 3.2.1.15) son enzimas que hidrolizan el esqueleto no metilado, atacan los enlaces α-(1?4) que unen a las unidades de ácido D-galacturónico. Esta enzima puede hidrolizar cualquier enlace de la cadena de polisacáridos y producir varios oligómeros de ácido galacturónico, entonces la enzima se llamará endopoligalacturonasa, ó hidrolizar el extremo no reductor de la cadena. El objetivo del presente trabajo es optimizar la producción de la enzima endo-poligalacturonasa, purificar la proteína y producir anticuerpos contra dicha enzima para inmunolocalizarla en esporas y/o micelio de Aspergillus sp. Desarrollo: Producción enzimática (Microorganismo y condiciones de cultivo) Se utilizó la cepa silvestre de Aspergillus sp. FP-500 aislada materia orgánica en estado de descomposición. Los medios líquidos utilizados para la fermentación contenían medio basal (Aguilar y Huitron, 1993) y pectina al 1% V/V o glucosa al 1%V/V como fuente de carbono. El inóculo utilizado fue de 1x108 esporas/mL. La fermentación se lleva a cabo por 72 horas a una temperatura de 37°C en agitación. Purificación de la enzima. Al término de la fermentación se separa por filtración el micelio y se recupera el filtrado enzimático. La purificación de la proteína se realizará por cromatografía de intercambio iónico. Determinaciones de Actividad Enzimática. La actividad enzimática endopectinolítica fue determinada por el efecto de reducción de viscosidad de pectina al 1% diluida en buffer acetatos-NaCl pH 4.2 a 30°C. La caracterización de la enzimas se llevó a cabo por electroforesis PAGE-SDS en acrilamida al 10% p/v y bis-acrilamida al 2.7% p/v (Bio-Rad Laboratories). Al terminar los geles fueron teñidos con azul de Coomassie R-250 (Bio-Rad Laboratories). Así mismo se llevó a cabo un zimograma con un gel SDS-PAGE renaturalizando las proteínas con Tris 100mM pH 6.8 a 37 ºC y agitación de 20 rpm durante 1 a 1.5 horas. Posteriormente, se sumerge en una solución de pectina al 1% p/v pH 5 durante una hora más a 37ºC. Finalmente, el gel se tiñe utilizando rojo rutenio, lo cual permite observar bandas de proteína donde se degrade la pectina, para así localizar la (s) banda (s) de enzimas pectinolíticas. Obtención de anticuerpos Los anticuerpos fueron obtenidos en conejos Nueva Zelanda blancos. Como antígenos se utilizaron esporas no germinadas, micelio, filtrado enzimático y enzima purificada o semipurificada. La prueba de anticuerpo vs. antígeno se llevó a cabo por el método de inmunodifusión radial u Ouchterlony donde si existe reacción antígeno – anticuerpo se forma una banda de precipitación o bien por medio Western Blot. Resultados Fermentaciones en glucosa y pectina como fuentes de carbono para obtener la enzima endo pectinolíticas: 3 2.5 U/mL 2 Pectina 1% 1.5 Glucosa 1% 1 0.5 0 24h 48h 72h Tiempo (h) Figura 1. Actividad enzimática producida por Aspergillus sp. FP-500, en pectina y gluocas como fuentes de carbono. La actividad enzimática mas alta se produjo en el medio que contenía pectina como fuente de carbono y con este filtrado se procedio a la purificación de la proteína por intercambio iónico, se utilizó un filtrado enzimático con una actividad de endo poligalacturonasa inicial de 3.9074 U/mL. Actividad endo pectinolítica (U) 250 200 150 100 50 0 Filtrado crudo Resina S (SN) Resina Q (SN) Resina Q (E) Tratamiento Endo-PG Figura 2. Seguimiento de los diferentes pasos de purificación de la Endo-PG y electroforesis de las diferentes fracciones obtenidas. Inmunoquímica. Se prepararon diferentes anticuerpos a partir de diversos filtrados enzimaticos completos así como de esporas y micelio. Con estos se llevó a cabo la detección inicial de la reacción antigenoanticuerpo por Western Blot en filtrados producidos por Aspergillus sp FP-500. Los resultados se pueden observar en la Figura 3, donde se observan las zonas de reacción. AA BA Figura 3. Zonas de reacción detectadas por Western Blot antígeno-anticuerpo A: Reacción de anticuerpo anti-espora 0.02 mg/mL con antígeno a 8.33 µg/mL y 0.833 µg/mL B: Reacción de anticuepo anti-filtrado enzimático (glucosa como fuente de carbono) 0.02 mg/mL con antígeno a 8.33 µg/mL.C: Reacción de anticuepo anti-filtrado enzimático (pectina como fuente de carbono) 0.02 mg/mL con antígeno a 8.33 µg/mL y 0.833 µg/mL. D: Reacción de anticuerpo anti-micelio 0.02 mg/mL con antígeno a 8.33 µg/mL. CA DA Conclusiones 1. Se han probado que los anticuerpos que se obtuvieron reconocen estructuras de Aspergillus sp. así como los filtrados enzimáticos. 2. Se han encontrado las condiciones de obtención de las más altas actividades enzimáticas de la proteína de interés. 3. Se tienen fracciones enriquecidas de la enzima Endo-PG que nos permitirán producir anticuerpos específicos contra dicha proteína para su posterior inmunolocalización. Bibliografía: Clausen C.A, Green, F. (1996). Characterization of polygalacturonase from the brown-rot fungus Postia placenta. Appl. Microbiol. Biotechnol 45, 750-754. Aguilar, G., Huitron, C., (1993) Conidial and mycelial-bound exo-pectinase of Aspergillus sp. Microbiology Letters 108, 127-132. Parenicová, L., Kester H., Benen, J., Visser, J., (2000) Characterization of a novel endopolygalacturonase from Aspergillus niger with unique kinetic properties, FEBS Letters 467, 333-336. Esquerré-Tugayé, M.T, Boudart, G., Dumas, B., (2000) Cell wall degrading enzymes, inhibitory proteins, and oligosaccharides participate in the molecular dialogue between plants and pathogens. Plant Physiol. Biochem. 38, 157-163. Prade, R., Zhan D., Youbi, P., Mort, A., (1999) Pectins, pectinases and plan-microbe interactions. Biotech. and Genetic Engineering Reviews 16, 361-391. Shwan, R., Cooper, R., Wheals, A., (1997) Endopolygalacturonase secretion by Kleuyveromyces marxianus and other cocoa pulp-degrading yeasts. Enzyme Microb. Technol. 21, 234-244. Towbin H, Staehelin T, Gordon, J (1979) Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proc. Natl. Acad. Sci. 76, 4350-4354. Evans C, Gallagher I, Atkey P, Wood D, (1991) Localisation of degradative enzymes in white-rot decay of lignocellulose, Biodegradation 2, 93-106. Moukha S, Wösten h, Asther M, Wessels J, (1993) In situ localization of the secretion of lignin peroxidases in colonies of Phanerochaete chrysoporium using a sandwiched mode of culture, Jour. Gen. Microbiol. 139, 969-978. De Vries, R, “Accessory Enzymes from Aspergillus Involved in Xylan and Petin Degradation”. Leisola, M, Kozulic B, Meussdoerffer F, (1987) Holmology among multiple extracellular peroxidases form Ph. chysosporium. Jour. Biol. Chem. 262, 419-42