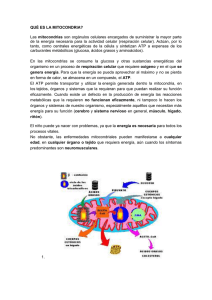
Descargar el archivo PDF
Anuncio

CAPÍTULO VIII Dinámica mitocondrial y sus implicaciones en la desregulación metabólica y en la neurodegeneración Antonio Zorzano, Jessica Segalés, María Isabel Hernández-Álvarez y Eleonora Sorianello Institute for Research in Biomedicine (IRB Barcelona) C/Baldiri Reixac 10, 08028 Barcelona, Spain; Departament de Bioquímica i Biologia Molecular, Facultat de Biologia, Universitat de Barcelona, Av. Diagonal 645, 08028 Barcelona, Spain; CIBER de Diabetes y Enfermedades Metabólicas Asociadas (CIBERDEM, Instituto de Salud Carlos III, España. edicion@ranf.com RESUMEN La dinámica mitocondrial es un concepto que incluye el movimiento de mitocondrias a lo largo del citoesqueleto, la regulación de la arquitectura mitocondrial, y su conectividad entre ellas, mediada por sucesos de fusión y fisión. Recientemente, nos hemos dado cuenta de la importancia que tiene la dinámica mitocondrial en en la fisiología celular, tras la identificación de los genes responsables de la fusión y de la fisión mitocondriales. Además, en la última década se han identificado mutaciones en genes que codifican para proteínas implicadas en la fusión mitocondrial tales como MFN2 y OPA1, y que provocan enfermedades neurodegenerativas (Charcot-Marie Tooth de tipo 2A y atrofia óptica autosómica dominante). Además, la alteración en la actividad de proteínas de fusión o fisión mitocondrial modula el metabolismo muscular. En este capítulo revisaremos los principales hallazgos obtenidos en el campo de la dinámica mitocondrial en células de mamífero y sus implicaciones en el metabolismo celular y en la neurodegeneración. CAPÍTULO VIII 155 INTRODUCCIÓN Las mitocondrias fueron descritas por primera vez a mediados de 1800 como compartimientos granulares citoplasmáticos provistos de su propia membrana. A finales del siglo XIX estos orgánulos fueron llamados mitocondrias, que procede de la fusión de las palabras griegas “mitos” y “chondros”, es decir, hilos y granos, respectivamente. A pesar de esto, las mitocondrias fueron consideradas durante décadas como orgánulos independientes y aislados entre sí. A principios del siglo XX, y gracias a los avances en microscopía de campo claro y en el cultivo celular, se reveló que las mitocondrias muestran morfologías distintas, probablemente debido a su naturaleza dinámica (1). No fue sino hasta la década de 1960 cuando se describió la primera evidencia de fusión mitocondrial sobre la base de la demostración de la recombinación y complementación del ADN mitocondrial en la levadura (2). Más tarde, hubo nuevas descripciones de procesos de fusión mitocondrial y de formación de la red mitocondrial también en células de mamíferos (3-5). En la década de 1990, el desarrollo de sondas o de proteínas fluorescentes dirigidas a la mitocondria han permitido la visualización de la dinámica mitocondrial en varios modelos celulares, desde la levadura hasta los hepatocitos (6-8). Este dinamismo no sólo se refiere al movimiento de las mitocondrias a lo largo del citoesqueleto, sino también a cambios en la forma mitocondrial, que son controlados por la fusión y la fisión eventos. En 1997 se descubrió el primer gen que participa en la fusión de las mitocondrias en Drosophila melanogaster (9). En la última década, se han identificado distintos genes que participan en la fusión y fisión mitocondriales, inicialmente descritos en la levadura y a continuación en células de mamíferos (10). A pesar de los grandes avances conseguidos en el estudio de la dinámica mitocondrial en los últimos años, los mecanismos que regulan los procesos de fusión y fisión aún no se comprenden en su totalidad. La importancia fisiológica de la dinámica mitocondrial en los tejidos de los mamíferos no se entiende bien, y los factores que determinan las diferencias en la morfología mitocondrial y la dinámica entre los distintos tipos de células aún deben ser definidos. En esta revisión se resumen los conocimientos actuales de la fusión y la fisión mitocondrial en mamíferos, y su participación en el metabolismo mitocondrial y en la neurodegeneración. 156 CAPÍTULO VIII LA MAQUINARIA DE FUSIÓN MITOCONDRIAL Las mitocondrias forman redes tubulares o ramificadas, que se someten a un equilibrio dinámico y regulado gracias a la actividad de los procesos de fusión y de fisión mitocondriales (8, 11). La fusión mitocondrial es necesaria para facilitar la intercomplementación del ADN mitocondrial y también como parte de una respuesta al estrés celular (12, 13). Para asegurar el mantenimiento de compartimentación mitocondrial, tanto la membrana mitocondrial interna como la externa necesitan fusionarse (14). Las principales proteínas implicadas en la fusión mitocondrial son las GTPasas de la membrana mitocondrial externa: Mitofusinas (Mfn1 y Mfn2) (15, 16) y la GTPasa de atrofia óptica (OPA1) de la membrana mitocondrial interna (17, 18). Las Mitofusina 1 y 2 son proteínas ancladas en la membrana mitocondrial externa y se ha demostrado que regulan la arquitectura de la red mitocondrial mediante la inmovilización y la fusión de las mitocondrias vecinas (15, 19-21). Saccharomyces cerevisiae muestra un solo gen ortólogo de Mfn y que recibe el nombre de FZO (de “fuzzy onion” o cebolla borrosa). Las funciones de Mfn1 y Mfn2 se solapan al menos en parte, ya que Mfn1 puede rescatar parcialmente mutantes de Mfn2 mediante la formación de heterodímeros con Mfn2 (22). Aunque los genes Mfn1 y Mfn2 presentan una expresión relativamente ubicua, los dos genes muestran diferentes niveles de expresión de ARNm en diferentes tejidos. Así, se detectan tránscritos de Mfn1, en niveles similares, en una variedad de tejidos humanos y son muy abundantes en el corazón; en cambio, los niveles de ARNm para Mfn2 son muy abundantes en el corazón y el tejido muscular, y se encuentran en niveles más bajos en muchos otros tejidos (23). Mfn1 La Mfn1, una proteína con actividad GTPasa, se encuentra en la membrana mitocondrial externa y muestra dos dominios transmembrana localizados hacia el extremo C-terminal de la proteína, cerca de un dominio héptada repetitiva (HR2) (23) (Figura 1). Este dominio héptada repetitiva media el primer paso de la fusión mitocondrial, el cual consiste en la interacción e inmovilización de dos mitocondrias adyacentes a través de la formación de un dímero antiparalelo entre dos moléculas de Mfn1 (24). Estos complejos diméricos pueden ser homotípicos (Mfn1-Mfn1 o Mfn2 Mfn2) o heterotípicos (Mfn1-Mfn2) (15, 24) (Figura 1). La tasa de de fusión mitocondrial depende de los dímeros homo- o heterotípicos; Mfn1 muestra una mayor actividad GTPasa en comparación con Mfn2 (16). CAPÍTULO VIII 157 En este sentido, las mitocondrias que expresan Mfn1 muestran una eficiencia mayor de interacción e inmovilización de mitocondrias, que aquellas que expresan Mfn2 (16). La región N-terminal de la proteína Mfn1 contiene el dominio de unión a GTP y otro dominio héptada repetitivo (HR1) (23, 24). Una alta sobreexpresión de Mfn1 en humanos causa una agregación perinuclear de las mitocondrias (23), que es independiente de la actividad de fusión de Mfn1. Por lo tanto, la sobreexpresión de una forma mutante de Mfn1 exenta de actividad GTPasa también causa agregación mitocondrial (23). La identificación de isoformas de “splicing” alternativo para Mfn1, que se detectan en cáncer de pulmón (25), o el hecho de que la variación en los niveles de proteína entre tejidos no siempre son equivalentes a los niveles de ARNm, sugiere que Mfn1 muestra una regulación post-transcripcional o postraduccional (23). Sin embargo, los mecanismos moleculares relevantes en esta regulación se desconocen. La proteína MIB (o “mitofusin binding protein”) es un miembro de la familia de enzimas deshidrogenasas/reductasas de cadena media. La mayoría de los MIB se localiza en el citoplasma, pero una pequeña cantidad está asociada a las mitocondrias (26). La sobreexpresión de MIB en células HeLa induce la fragmentación mitocondrial, lo que se previene por la co-expresión de Mfn1, y ello sugiere una interacción funcional de MIB con proteínas Mfn. Por otro lado, la pérdida de función de MIB da lugar a la detención del crecimiento de las células HeLa y a la inducción de una extensa red mitocondrial (26). Se ha demostrado la existencia de una interacción directa entre Mfn1-2 y MIB mediante estudios de co-inmunoprecipitación. Los mecanismos por los que MIB regula la actividad de Mfn1 o Mfn2 aún no se conocen (26). Mfn2 Mfn2 es una proteína integral de membrana mitocondrial externa con los extremos N-terminal y C-terminal expuestos en el citosol (20). Mfn2 contiene un dominio héptada repetitiva (HR2) localizado en la región C-terminal de la proteína (20, 21), y la actividad GTPasa localizada a N-terminal es crucial para su actividad de fusión mitocondrial (15, 27) (Figura 1). Sin embargo, y de manera similar a lo reportado para Mfn1, la actividad GTPasa de Mfn2 no es necesaria para la formación de agregados perinucleares mitocondriales causados por la sobreexpresión de Mfn2 (20, 21, 23, 28). Este mecanismo de agregación puede ser causado por la inmovilización mediada por el dominio HR2 de Mfn2 (23, 24). 158 CAPÍTULO VIII Mfn2 es esencial para el desarrollo embrionario, y se ha descrito que los ratones Mfn2 “knockout” (KO) mueren hacia la mitad de la vida fetal debido a defectos de la placenta (15). Cuando el defecto de la placenta se previene, los ratones KO de Mfn2 desarrollan una degeneración cerebelosa intensa (29). Figura 1.- Maquinaria de fusión mitocondrial A) Esquema de las proteínas Mfn1, Mfn2, y OPA1. Las 3 proteínas contienen dominios GTPasa y regiones héptada repetitivas (HR). Los dominios de transmembrana (TM) de las mitofusinas son inusualmente largas y contienen residuos cargados que se piensa permiten un giro en forma de U en la membrane mitochondrial externa, de manera que casi toda la proteína se orienta al citosol. OPA1 comprende una secuencia N-terminal de importe mitocondrial, regions hidrofóbicas héptada repetitivas (HR), un dominio GTPasa, y un dominio central. OPA1 media la fusión de las membranas mitocondriales internas. B) Inmovilización mitocondrial por Mitofusinas. Las interacciones homotípicas entre moléculas de Mfn1 o heterotípicas entre moléculas de Mfn1 y Mfn2, que tienen lugar entre mitocondrias adyacentes, son mediadas por la región HR2, y conducen a la unión de las membranas externas. La proteína OPA1, localizada en el espacio intermembrana y asociada a la membrane interna, es el mejor candidato como efector de la fusión de la membrana mitocondrial interna. CAPÍTULO VIII 159 De acuerdo con los datos de Brito y Scorrano, Mfn2 también juega un papel importante en la interacción entre el retículo endoplásmico (RE) y las mitocondrias (30). En fibroblastos embrionarios de ratón (MEFs) carentes de Mfn2, las regiones de contacto entre la mitocondria y el retículo endoplasmático (ER), que se considera forman subdominios especializados de las ER llamados membranas asociadas a mitocondrias (MAM), se reducen de manera significativa. Además, las células “knockout” de Mfn2 muestran una red fragmentada del ER (30). Algunas evidencias, indican que tanto Mfn1 como Mfn2 se regulan por el sistema de proteasoma, de manera que inhibidores del proteasoma conducen a un aumento de su expresión (31). La proteína ortóloga en levadura Fzo1 experimenta ubiquitinación (32, 33) (31, 34). Mdm30 es una proteína con un motivo “F-box” que en levadura que reconoce a Fzo como su substrato (32). Mdm30 regula la fusión mitocondrial de la levadura a través de ubiquitinación de Fzo1, con su consiguiente degradación por el proteasoma (32). No se han descrito homólogo de mamíferos de Mdm30 en la actualidad. Sin embargo, estudios recientes han identificado tanto a Mfn de Drosophila como a Mfn1 y Mfn2 de mamiferos como substratos de la ubiquitina ligasa E3, Parkin (35-37). Parkin promueve mitofagia a través de la ubiquitinación de proteínas mitocondriales (38, 39). La disfunción de Parkin es una causa importante de la enfermedad de Parkinson, y la consiguiente acumulación de las mitocondrias dañadas por un defecto en la mitofagia puede conducir a un augmento de la muerte celular (38, 39). La ubiquitinación de Mfn1 y Mfn2 por Parkin pueden conducir a las mitocondrias hacia su degradación por mitofagia, mediante la degradación selectiva de Mfn1/2, seguido de la inhibición de la fusión mitocondrial y la segregación de las mitocondrias dañadas (35, 36). OPA1 La proteína Mgm1 de la familia de las dinaminas fue identificada inicialmente en levadura como un actor implicado en el mantenimiento de la red mitocondrial, una función que es coherente con su localización en el espacio entre la membrana y su asociación con la membrana mitocondrial interna (40) (Figura 1). La proteína ortóloga de Mgm1 en mamíferos es OPA1, y es también esencial para la fusión mitocondrial (17, 41). OPA1 se localiza en la membrana mitocondrial interna y en el espacio intermembrana. OPA1 ha sido también detectada en las crestas mediante estudios de microscopía electrónica (42-44), y una pequeña fracción de OPA1 constituída por las denominadas isoformas cortas, se asocia a la membrana mitocondrial externa (44). 160 CAPÍTULO VIII Se conoce la existencia de distintas isoformas de OPA1. Estas isoformas están además reguladas por la actividad de proteasas mitocondriales que reconocen a OPA1 en sitios de corte específicos en respuesta a la pérdida del potencial de membrana mitocondrial, la pérdida de ADN mitocondrial o a la inducción de apoptosis (18, 45, 46). Se han identificado una serie de proteasas que pueden procesar OPA1 en diferentes puntos de la molécula, y que regulan el equilibrio entre las isoformas largas y cortas de OPA1. Esto puede estar vinculado a dos fenómenos no excluyentes: a) una alteración de OPA1 modifica el potencial de membrana mitocondrial, el cual a su vez es requerido para el proceso de fusión (47, 48), y b) OPA1 se ha mostrado que interactúa con Mfn1 (17) y con Mfn2 (49). Recientemente, se ha descrito que la actividad de pro-fusión de OPA1 es crucial en el proceso de control de calidad de las mitocondrias, a través del proceso llamado de “kissand-run” y que implica la fusión transitoria entre mitocondrias (50). Las mitocondrias que muestran una actividad baja de OPA1 y, en consecuencia, una capacidad de fusión reducida y bajo potencial de membrana mitocondrial, tendrá asimismo una reducida capacidad de “kiss-and-run” y por lo tanto, se someterá a degradación por autofagia (51). Además, un exceso de actividad de fusión OPA1, aunque de forma transitoria puede proteger para algunos tipos de estrés celulares (13), provocará un proceso de senescencia, en presencia de una excesiva producción de especies reactivas de oxígeno y de daño del genoma mitocondrial (52). Por último, datos recientes apuntan a una nueva e importante vía reguladora de fusión-fisión mitocondrial que involucra a la proteína serina-treonina quinasa Pink y a la ubiquitina ligasa Parkin. Ambas proteínas están involucradas en formas recesivas de la enfermedad de Parkinson. Pink y Parkin regulan negativamente OPA1 con lo que promueven la fragmentación de las mitocondrias (53). Estos datos sugieren que el control estricto de la OPA1 pro-fusión de la actividad es crucial para el mantenimiento de la red mitocondrial y un estado saludable de las células. LA MAQUINARIA DE FISIÓN MITOCONDRIAL El proceso de fisión mitocondrial es necesario para incorporar las mitocondrias viejas o dañadas de la célula a través de un proceso de autofagia llamado mitofagia (54). La velocidad de fisión también aumenta notablemente cuando las células se comprometen en el proceso de apoptosis, sin que tenga lugar un aumento compensatorio en la tasa de fusión (55). Alteraciones en la maquinaria de fisión causan una mayor generación de especies CAPÍTULO VIII 161 reactivas de oxígeno, una mayor susceptibilidad de las células a inducir apoptosis, una capacidad diferente para generar ATP, y una población heterogénea de mitocondrias con una distribución no uniforme del ADNmt (56). La importancia fisiológica de la fisión mitocondrial se manifestó tras la identificación inicial de la proteína Drp1 (miembro de la familia de las dinaminas) y Fis1 (57-58) (). Recientemente, se ha identificado un nuevo factor de fisión mitocondrial (Mff), el cual media la fisión mitocondrial y la peroxisomal de una manera similar a Fis1 y Drp1. Mff también se encuentra en la membrana mitocondrial externa, pero no en los mismos complejos que Fis1, lo que sugiere un papel independiente (59) (Figura 2). Drp1 Drp1 y su ortólogo en levadura Dnm1, comparten características estructurales comunes a la familia de las dinaminas, lo que incluye un dominio con actividad GTPasa, un dominio efector de GTPasa (GED) y un dominio central (60, 61). Drp1 se encuentra principalmente en el citosol y con el fin de facilitar la escisión de la doble membrana mitocondrial, se ensambla en complejos multiméricos en forma de anillo en los sitios de fisión mitocondrial; a continuación, éstos anillos se constriñen por la hidrólisis del GTP lo que promueve la división mitocondrial (61-66) (Figura 2). Debido a que Drp1 carece de un dominio de homología a pleckstrina que está implicado en la unión da membranas, el reclutamiento y el ensamblaje de Drp1 en la membrana mitocondrial externa requiere de la participación de proteínas receptoras de membrana (61, 64, 67-69). El ratón “knockout” para Drp1 muestra letalidad embrionaria. Los embriones muestran daño en el desarrollo del hígado y del corazón, un aumento en la apoptosis neuronal en la corteza cerebral profunda, y una alterada formación de sinapsis (57, 58). Además, las mutaciones en la proteína Drp1 retrasan la liberación de citocromo c asociada a la apoptosis, lo que sugiere que la liberación del citocromo c de apoptosis está estrechamente relacionada con la fisión mitocondrial (57, 58). Los mecanismos implicados en la activación y translocación de Drp1 a las mitocondrias pueden tener que ver con fenómenos de S-nitrosilación, sumoilación, ubiquitinación, o fosforilación de residuos de serina en el dominio GED. Drp1 puede ser activado por S-nitrosilación, una modificación post-traduccional que aumenta su actividad de fisión mediante la inducción de su actividad GTPasa y dimerización (70). Se ha observado S-nitrosilación de Drp1 y fragmentación de la red mitocondrial en respuesta a la 162 CAPÍTULO VIII expresión celular de la proteína ß-amiloide (Aß), un mediador clave de la enfermedad de Alzheimer. Se ha propuesto que la formación de oligómeros de Aß conduce a estrés nitrooxidativo lo que conduce a producción de SNO-Drp1 (70). Figura 2.- Maquinaria de fisión mitocondrial A) Esquema de las proteínas Drp1, Fis1 y Mff. Drp1 tiene dominios típicos de la familia de las dinaminas-GTPasas, lo que incluye un dominio GTPasa, un dominio central, y un dominio efector GTPasa (GED). Drp1 se localiza en el citosol. Fis1 es una proteína de bajo peso molecular presente en la membrana mitocondrial externa cuyo extremo Nterminal se orienta al citosol, y con un dominio transmembrana localizado en el extremo C-terminal (TM). El dominio citosólico forma un haz compuesto por 6 hélices que incluye dos repeticiones de tipo “tetratricopeptide” (TPR). Mff está anclada en la membrana mitochondrial externa por un segmento transmembrana localizado en el extremo C-terminal; también contiene 2 repeticiones cortas en la mitad N-terminal y un dominio “coiled-coil” justo a N-terminal del segmento TM orientado al citosol. Drp1 se recluta a la mitocondria y media la fisión mitocondrial mediante 2 mecanismos que se ilustran en los paneles B y C. B) Fis1 reside en la membrana mitocondrial externa y se localiza de manera uniforme; la mayor parte de la proteína se orienta al citosol. C) Mff se localiza principalmente en zonas precisas de la mitocondria donde actúa a modo de lugar de unión de Drp1. En ambos casos, Drp1 constriñe el túbulo mitocondrial con el objeto de inducir la fisión de las membranas. CAPÍTULO VIII 163 Existen evidencias experimentales, que demuestran que la sumoilación es un proceso mediante el cual se puede activar Drp1 (71). SUMO-1 y su enzima de conjugación Ubc9 inducen la fisión mitocondrial mediante la estabilización de Drp1 (72). Contrariamente, la proteasa específica de SUMO/sentrin, SENP5, reduce los niveles de Drp1, lo cual previene la fisión mitocondrial (73). Además, se ha sugerido que MARCH V, que es una ubiquitina ligasa E3 mitocondrial, podría estar implicada en la translocación de Drp1 a la mitocondria, sin que ello afecte su estabilidad (74). La regulación de la actividad de Drp1, en respuesta a cambios en el nivel de fosforilación ha sido tema debate, en especial, el residuo específico de Drp1 implicado (57). Por un lado, se ha demostrado que durante la mitosis, el dominio GED de Drp1 de rata se fosforila en el residuo de Ser585 (correspondientes a Ser616 en la variante de “splicing” 1 de Drp1) por la proteína quinasa CDK1 dependiente de ciclina B, lo que da lugar a una fisión mitocondrial transitoria, y por tanto a una distribución correcta de las mitocondrias en las células hijas (75). También se ha descrito que el óxido nítrico induce la fisión mitocondrial por fosforilación de Drp1 en el residuo de Ser616 (76). Otros autores han demostrado que el tratamiento con óxido nítrico induce la S-nitrosilación de Drp1 en el residuo de Cys644, lo que provoca un aumento de la actividad GTPasa y la dimerización de Drp1 (70, 77). Por otro lado, la fosforilación de la proteína quinasa A (PKA) en el residuo de serina Ser637 (en la variante de “splicing” 1 de Drp1 humana) (78) o Ser656 (en la variante de “splicing” 1 de Drp1 de rata) (79), que se encuentra en el extremo NH2-terminal de la GED, conduce a la inhibición de la fisión mitocondrial y a la muerte celular por apoptosis. La desfosforilación de la Ser637 o de la Ser656 por la proteína fosfatasa, calcineurina, resulta en la translocación de Drp1 a la mitocondria y la activación de la fisión mitocondrial (79, 80). También ha demostrado que la fosforilación del residuo de Ser600 de la isoforma 3 de Drp1 (equivalente a la Ser637 en la variante de “splicing” 1 de Drp1 humana) por la proteina quinasa dependiente de Ca2+/calmodulina promueve la fisión mitocondrial, posiblemente debido a una mayor afinidad entre Drp1 y Fis1 (81). Fis 1 El reclutamiento de la proteína de levadura Dnm1 desde el citosol a la membrana mitocondrial externa, se produce a través de asociación con la proteína Fis1, lo que resulta en la formación de un complejo de fisión y en la escisión de las membranas mitocondriales (65, 68, 82, 83). Esta interacción se produce con la participación de la proteína adaptadora Mdv1, que tiene un papel en el co-ensamblaje de Dnm1 en estructuras helicoidales (64, 67). 164 CAPÍTULO VIII Fis1 contiene cuatro repeticiones de tipo “tetratrico-peptide” (TPR), que son cruciales para la interacción con Mdv1, que a su vez, facilitan la unión y el ensamblaje de Dnm1 (84). Mientras que Mdv1 interactúa con Dnm1 y Fis1 para facilitar la fisión de membranas mitocondriales, pero su función parece limitarse a levadura (82, 85). En mamíferos, Fis1 es una pequeña proteína de 17,2 kDa que se expresa ubicuamente y se detecta en toda la red mitocondrial (Figura 2). Fis1 está anclada en la membrana mitocondrial externa a través de su extremo C-terminal, que contiene una hélice alfa, un dominio transmembrana, y una cola C-terminal expuesta al espacio intermembrana. La parte N-terminal de la proteína contiene cuatro regiones distintas con cinco hélices alfa (86, 87); la primera hélice alfa de Fis1 parece ser crítica para la oligomerización y su actividad de fisión (88). Las siguientes cuatro hélices alfa constituyen dos repeticiones de tipo “tetratrico-peptide” (TPR1 y TPR2), y aunque no son necesarias para la oligomerización de Fis1, están implicados en las interacciones proteína-proteína necesaria para la fisión (88). La sobreexpresión de Fis1 causa fragmentación mitocondrial, mientras que la pérdida de función de Fis1 resulta en la formación de una red mitocondrial altamente fusionada indicando que la fisión se bloquea en condiciones en las que los eventos de fusión se mantienen (68, 82, 83, 89). Se ha observado que Drp1 se recluta en células con expresión silenciada para Fis1 (71, 90), lo que sugiere la participación de otras proteínas en la fisión mitocondrial de mamíferos. Mff El Factor de fisión mitocondrial (Mff) (59, 69), está anclado en la membrana mitocondrial externa y se ha propuesto que tiene un papel en la fisión mitocondrial de manera independiente a Fis1(69) (Figura 2). Mff no se expresa en la levadura, y representa un nuevo componente de la maquinaria de la fisión mitocondrial de mamíferos. Mff se localiza en puntos específicos de las mitocondrias, y la sobreexpresión de Mff causa la translocación de Drp1 citosólico a las mitocondrias, y promueve la fisión mitocondrial. Por el contrario, la pérdida de función de Mff resulta en una reducción de Drp1 en la superficie de las mitocondrias y en un alargamiento de la red mitocondrial (69). Otras proteínas de fisión La proteína GDAP1 (“Ganglioside-induced differenciation-associated protein 1”), se encuentra en la membrana mitocondrial externa. Aunque la función de la proteína GDAP1 no se entiende bien, parece jugar un papel en el control de la forma de las mitocondrias CAPÍTULO VIII 165 mediante la promoción de la fisión mitocondrial (91). Su sobreexpresión, causa la fragmentación de las mitocondrias, y su pérdida de función da lugar a un incremento en la fusión de la red mitocondrial (92). Se han descrito, más de 20 mutaciones en el gen GDAP1 como causantes de la neuropatía Charcot-Marie-Tooth de tipo 4A o de tipo 2K (91, 93), dando lugar a desmielinización axonal, y a formas intermedias de neuropatía periférica sensorial y motora (94). MTP18 se encuentra en la membrana mitocondrial interna y su sobreexpresión da lugar a fragmentación mitocondrial. La pérdida de función de MTP18 por interferencia de ARN (ARNi) promueve una alta fusión mitocondrial y la liberación de citocromo c (95). La inducción de fragmentación mitocondrial por MTP18 depende de la expresión de Drp1, lo que sugiere un papel de esta proteína en las acciones mitocondriales de Drp1 (95). MiD49 y MiD51 son proteínas homólogas que presentan un 65% de identidad y su extremo N-terminal está anclado en la membrana mitocondrial externa (96, 97). MiD49/51 forma focos y anillos alrededor de las mitocondrias, de manera similar a Drp1. MiD49/51 directamente recluta Drp1 a la superficie mitocondrial, mientras que su pérdida de función reduce la asociación mitocondrial de Drp1, lo que conduce a aumentada fusión. La sobreexpresión de MiD49/51 parece secuestrar a Drp1 fuera del entorno mitocondrial de manera que causa fusiones tubulares que interaccionan con actina (96). Experimentos de co-inmunoprecipitación han demostrado que MiD49 interactúa con Drp1. Sin embargo, los mecanismos precisos de acción de esta proteína aún no han sido esclarecidos (96). DINÁMICA MITOCONDRIAL Y METABOLISMO CELULAR Diferentes estudios, demuestran que la dinámica mitocondrial juega un importante papel en la función de las mitocondrias, regulando distintos procesos fisiológicos como la fosforilación oxidativa, el ciclo celular o la apoptosis. El grupo de Skulachev fue el primero en proponer un papel funcional a las redes mitocondriales observadas en células musculares. En estudios anteriores a la identificación de las proteínas de fusión y fisión mitocondrial, Skulachev y colaboradores demostraron que los filamentos mitocondriales presentaban continuidad de la matriz y que esto era importante para el mantenimiento del potencial de membrana mitocondrial (98, 99). Sobre la base de estas observaciones, Skulachev propuso la hipótesis de que los filamentos mitocondriales podrían actuar como cables transmisores de energía. Así, en células como las fibras musculares, estos filamentos mitocondriales 166 CAPÍTULO VIII permitirían la transmisión del potencial de membrana mitocondrial desde las mitocondrias localizadas en la periferia de la célula a las mitocondrias internas, con menor disponibilidad de oxígeno (99). En los últimos años, se ha demostrado que alteraciones en la actividad de las proteínas de dinámica mitocondrial afectan al metabolismo energético. En este sentido, se han obtenido datos relevantes mediante la manipulación de la expresión de Mfn2 en distintos modelos celulares. Así, fibroblastos 10T1/2 transfectados con una secuencia antisentido de Mfn2 muestran una disminución en la oxidación de glucosa y una reducción similar del consumo de oxígeno mitocondrial (19). La represión de Mfn2 en células musculares L6E9, mediante la transducción con adenovirus antisentido, produce un perfil metabólico parecido, causando una disminución en la tasa de oxidación de glucosa (19, 100). En estas condiciones, la captación de glucosa y la producción de lactato se encuentran claramente incrementadas en las células musculares con menor expresión de Mfn2, mientras que la incorporación de glucosa al glucógeno se ve reducida significativamente (100). La represión de Mfn2 en miotubos L6E9 también disminuye la tasa de oxidación de piruvato y de palmitato, y está asociada a una reducción del potencial de membrana mitocondrial en presencia de varios sustratos (100). Estas alteraciones en la oxidación de sustratos inducidas por la pérdida de función de Mfn2 no son consecuencia de una disminución de la masa mitocondrial. En conjunto, estos datos indican que la represión de Mfn2 en células de mamífero produce alteraciones en el metabolismo mitocondrial, caracterizadas por una menor oxidación de diferentes sustratos y una disminución del consumo de oxígeno celular y del potencial de membrana mitocondrial (Figura 3). Estos resultados van en el mismo sentido que los observados en fibroblastos embrionarios de ratón (MEFs) con mutaciones de pérdida de función en ambas mitofusinas, Mfn1 y Mfn2. Estas células presentan pérdida de potencial de membrana mitocondrial, consumo de oxígeno endógeno reducido e incapacidad para incrementar la respiración en respuesta al ionóforo 2,4-dinitrofenol (12). También, se han detectado alteraciones metabólicas en fibroblastos de pacientes con determinadas mutaciones en el gen que codifica la proteína Mfn2. Así, fibroblastos que presentan las mutaciones R364Q o A166T muestran un aumento del consumo de oxígeno basal y de la respiración insensible a oligomicina, así como una disminución del potencial de membrana mitocondrial (101). Los mutantes mencionados presentan una tasa de síntesis CAPÍTULO VIII 167 de ATP similar a los controles pero una menor eficiencia de acoplamiento entre la cadena respiratoria y la síntesis de ATP (ratio ATP/O). Estos cambios ocurren en condiciones en las que la masa mitocondrial no se encuentra alterada y la actividad de los complejos de la cadena respiratoria es normal (101). Por otro lado, se han descrito otras mutaciones de Mfn2 como M21V, T105M, I213T o V273G que no presentan efectos a nivel del metabolismo mitocondrial (Amiott et al, 2008; Loiseau et al, 2007). En la actualidad se desconocen los mecanismos por los que determinadas mutaciones en Mfn2 causan alteraciones metabólicas en fibroblastos humanos. Figura 3.- Modelo de la función de la proteína de fusión mitocondrial Mfn2 sobre el metabolismo celular El esquema resume la información conocida actualmente sobre los efectos metabólicos de la pérdida de función de Mfn2 y de la sobreexpresión de una forma de Mfn2 truncada por el extremo C-terminal (Mfn2Δ602–757) en células en cultivo. 168 CAPÍTULO VIII Alteraciones en la expresión de OPA1, también afectan al metabolismo mitocondrial. En este sentido, la represión de OPA1 en células MEF mediante ARNi provoca una reducción en el potencial de membrana mitocondrial, una marcada disminución de la respiración basal y una incapacidad para incrementar el consumo de oxígeno en presencia del desacoplante 2,4-dinitrofenol (12). Además, se ha descrito un aumento significativo en la producción de especias reactivas de oxígeno (ROS) en moscas deficientes en OPA1 (102,103). Los primeros datos referentes a las consecuencias de mutaciones de OPA1 sobre el metabolismo energético, demuestran que el músculo de pacientes con atrofia óptica autosómica dominante (ADOA) presenta una disminución en la producción de ATP (104). Además, fibroblastos de pacientes de ADOA con determinadas mutaciones en OPA1 muestran una disminución de la actividad enzimática del complejo IV y un aumento de la actividad del complejo V (105). También se ha descrito que fibroblastos procedentes de pacientes con ciertas mutaciones en OPA1 presentan alteraciones en la síntesis de ATP usando sustratos para el complejo I (106). En este estudio se demuestra, mediante coinmunoprecipitación, la interacción entre OPA1, AIF y los complejos I, II y III, sugiriendo un posible mecanismo por el que estas mutaciones en OPA1 causarían defectos a nivel de la cadena respiratoria. En cambio, otro trabajo señala la existencia de otras mutaciones en OPA1 que no alteran la actividad mitocondrial ni la bioenergética celular (107). En un estudio muy reciente, se ha demostrado que el ratón knockout de OPA1 específico de células β del páncreas muestra hiperglucemia tanto en ayuno como en alimentación, y una menor tolerancia a la glucosa que los ratones control (108). La ablación de OPA1 también altera la secreción de insulina en islotes pancreáticos aislados. Además, la producción de ATP estimulada por glucosa y la actividad del complejo IV se encuentran significativamente reducidas en las células β deficientes en OPA1 (108). Estudios recientes, demuestran que alteraciones en las proteínas de fisión mitocondrial producen efectos sobre el metabolismo mitocondrial similares a los originados por modificaciones en los componentes de fusión. La represión de Drp1 en células HeLa mediante ARNi induce la aparición de mitocondrias fusionadas e interconectadas. Estas alteraciones morfológicas vienen acompañadas de una disminución del consumo de oxígeno, de la síntesis de ATP y del potencial de membrana mitocondrial, así como de un incremento en los niveles de ROS y de daño oxidativo (56, 109). CAPÍTULO VIII 169 La represión de Drp1 también provoca la pérdida de ADN mitocondrial y la activación de la autofagia (56, 109). Actualmente, se ha demostrado que determinadas mutaciones en Drp1 disminuyen la expresión de subunidades de los complejos OXPHOS y del contenido de ATP en el corazón, causando una cardiomiopatía en ratones (110). La sobreexpresión de una forma dominante negativa de Drp1 y la represión de Fis1 también producen una reducción de la capacidad respiratoria de las células de insulinoma de rata INS1 (51). Así, se puede concluir que la perturbación de la dinámica mitocondrial, bien modificando las proteínas de fusión o bien las de fisión, altera la producción de energía por parte de las mitocondrias en células de mamífero. Los mecanismos mediante los cuales la alteración de la dinámica mitocondrial afecta al metabolismo energético, no se conocen completamente. En varios estudios, se ha analizado la expresión o la actividad de los complejos de la cadena respiratoria mitocondrial. La represión de Mfn2 en miotubos L6E9 causa una marcada disminución en la expresión de algunas subunidades de los complejos I, II, III y V (100). Paralelamente, la actividad enzimática de los complejos I+III y III disminuye en las células con menor expresión de Mfn2 (100). También se han detectado deficiencias en las actividades de los complejos I, II y III en células MEF doble knockout para ambas mitofusinas (12). Estos resultados indican que la pérdida de función de Mfn2 causa alteraciones específicas en la expresión de algunas subunidades de los complejos de la cadena respiratoria, lo que conlleva a una disminución en la actividad de dichos complejos. Células MEF deficientes en OPA1 también muestran una menor actividad de los complejos I, II y III (12). Otra cuestión relevante referente a los mecanismos implicados en la regulación del metabolismo mitocondrial por parte de las proteínas de dinámica mitocondrial, es si la ganancia de función ejerce también efectos biológicos. Este aspecto ha sido analizado principalmente en el caso de Mfn2. La sobreexpresión de Mfn2 en células HeLa provoca la agregación de las mitocondrias alrededor del núcleo, así como un aumento del potencial de membrana mitocondrial y de la oxidación de glucosa (100). Del mismo modo, la sobreexpresión de Mfn2 en mioblastos L6E9 incrementa la expresión de diferentes subunidades de los complejos I, IV y V (100). Con el propósito de determinar si los efectos inducidos por la sobreexpresión de Mfn2 sobre la actividad mitocondrial dependen o no de su actividad de fusión, se han llevado a cabo estudios con una forma de Mfn2 truncada por el extremo C-terminal (hMfn2Δ602–757), que no mantiene la capacidad de promover fusión mitocondrial. La sobreexpresión de hMfn2Δ 602–757 en células HeLa no altera la morfología de la red mitocondrial pero estimula la 170 CAPÍTULO VIII oxidación de glucosa e incrementa el potencial de membrana mitocondrial (100). En resumen, los datos existentes indican que la ganancia de función de Mfn2 activa el metabolismo mitocondrial y aumenta la expresión de subunidades de los complejos del sistema OXPHOS. Los efectos estimuladores de Mfn2 sobre el metabolismo mitocondrial no son totalmente dependientes de su actividad de fusión mitocondrial, aunque los mecanismos específicos implicados aún se desconocen. Sobre la base de la información existente, proponemos que las proteínas de dinámica mitocondrial afectan al metabolismo por dos vías distintas: (a) por un lado, la perturbación tanto de la fusión como de la fisión mitocondrial altera la producción de energía en células de mamífero, sugiriendo que la dinámica mitocondrial per se controla el metabolismo energético y (b) por otro lado, Mfn2 parece tener un papel regulador específico del metabolismo mitocondrial a través de cambios en la expresión de las subunidades de los complejos OXPHOS (111). Se requieren más estudios para determinar cómo operan estas dos vías in vivo y para analizar en detalle los mecanismos moleculares implicados. La función mitocondrial se encuentra alterada en situaciones de resistencia a la insulina tales como la obesidad y la diabetes de tipo 2 (112, 113). Existen evidencias de que en estas condiciones la expresión de los genes de dinámica mitocondrial también está desregulada (114,115). En este sentido, la expresión de Mfn2 se halla disminuida en músculo esquelético de ratas obesas Zucker y en personas con obesidad (19, 116). Paralelamente, se ha observado una reducción del 25% de la red mitocondrial en músculo de ratas Zucker (19). Por otro lado, la derivación bilio-pancreática causa un incremento de la expresión del mARN de Mfn2 en músculo esquelético de individuos con obesidad mórbida (117). En estas condiciones de pérdida masiva de peso, se produce una importante mejoría de la sensibilidad a la insulina (117). Los pacientes con diabetes de tipo 2 también muestran una reducción en la expresión de Mfn2 en músculo esquelético en relación a los sujetos control (116). No es probable que esta disminución en la expresión de Mfn2 sea consecuencia de una reducida acción de la insulina, ya que en individuos sanos, obesos o diabéticos de tipo 2 la expresión de Mfn2 no se altera en respuesta a 3 horas de hiperinsulinemia al ser sometidos a un clamp. Además, la incubación de células musculares con concentraciones supramáximas de insulina durante 48 horas tampoco afecta a la expresión de Mfn2 (116). Los datos existentes indican que Mfn2 controla el metabolismo mitocondrial en células en cultivo (Bach et al., 2003; Chen et al., 2005; Pich et al., 2005). La reducción en la CAPÍTULO VIII 171 expresión de Mfn2 observada en las situaciones de obesidad y diabetes de tipo 2 nos permite proponer que esta proteína juega un papel relevante en la disfunción metabólica que caracteriza estas condiciones. Además, se ha demostrado que los coactivadores transcripcionales PGC-1α y PGC1β inducen la expresión de Mfn2 mediante la interacción con el factor de transcripción ERRα (118, 119). Este hecho, puede ser particularmente relevante ya que se ha descrito que la expresión de ambos coactivadores, así como la de Mfn2, se halla reprimida en la obesidad y la diabetes de tipo 2 (113, 120). Dado el relevante papel de PGC-1α y PGC-1β en la biogénesis mitocondrial y el metabolismo celular, alteraciones en las vías PGC-1α /Mfn2 y PGC-1β/Mfn2 podrían conferir susceptibilidad al desarrollo de resistencia a la insulina y a la diabetes de tipo 2. MUTACIONES EN GENES INVOLUCRADOS EN LA DINÁMICA MITOCONDRIAL CAUSAN ENFERMEDADES NEURODEGENERATIVAS Las mitocondrias son organelos sumamente importantes en todos los tipos celulares, pero lo son particularmente relevantes en el sistema nervioso. Ellas, son esenciales para la producción de energía, la regulación del calcio, el mantenimiento del potencial de la membrana plasmática, el plegamiento de proteínas mediante chaperonas, el transporte axonal y dendrítico, y la liberación y recaptación de neurotransmisores en las sinapsis. Durante muchos años se ha propuesto que la respiración mitocondrial es la responsable de la disfunción neuronal. En la última década, numerosos estudios han aportado evidencias adicionales implicando la dinámica mitocondrial en la patofisiología de las enfermedades neuronales. En relación a ello, se ha demostrado que mutaciones en genes involucrados en la dinámica mitocondrial causan desórdenes neuronales. Mutaciones en OPA1: atrofia óptica autosómica dominante La atrofia óptica autosómica dominante (AOAD), es la más frecuente de las neuropatías ópticas hereditarias, con una incidencia de 1:12.000 a 1:50.000 habitantes. AOAD es una enfermedad genéticamente heterogénea y cinco loci se han asociado a esta patología (OPA1 a OPA5) (121). Esta enfermedad se caracteriza por una deficiencia de la 172 CAPÍTULO VIII visión en la infancia temprana, que se caracteriza por una pérdida moderada a severa de la agudeza visual, palidez temporal del disco óptico, defectos en la visión de colores y escotomas (zonas de pérdida de campo visual) cecocentrales (122-124). El examen histopatológico de ojos de donantes sugiere que la patología fundamental de AOAD es una degeneración primaria de las células ganglionares retinales seguida por una creciente atrofia del nervio óptico (125, 126). Se han identificado 117 mutaciones del gen de OPA, las cuales son básicamente familia-específicos (127) (http://lbbma.univ-angers.fr/eOPA1). Las mutaciones de OPA1 involucran principalmente sustituciones y también deleciones e inserciones (121, 128). Casi el 50% de las mutaciones causa una terminación prematura de la proteína OPA1. La mayoría de las mutaciones han sido observadas en el dominio GTPasa y probablemente supriman la actividad de fusión mitocondrial. Se han detectado mutaciones adicionales en la región NH2-terminal de OPA1 abarcando la secuencia de importación mitocondrial, las secuencias hidrofóbicas y la región “coiled-coil”; en el dominio central; y en el dominio GTPasa o dominio GED. La patogénesis de AOAD se produce en algunos casos como el resultado de una haploinsuficiencia (pérdida de función). Datos clínicos apoyan este concepto: 1) la deleción del gen completo de OPA1 o en el residuo aminoacídico 2 (Trp2X) (129, 130); 2) casi el 50% de las mutantes de OPA1 causan la terminación prematura de OPA1, un número importante de éstos en el dominio GTPasa, y es altamente probable que causen pérdida de función; y 3) fibroblastos de la piel con mutaciones en OPA1 evidencian fosforilación oxidativa y fusión mitocondrial deficientes (106). En este aspecto, la ablación de OPA1 inducida por ARNi causa fragmentación mitocondrial, disminución del potencial de membrana mitocondrial y de la respiración celular, desorganización de las crestas mitocondriales y apoptosis (17, 48, 131, 132). En ratones la pérdida de función de OPA1 inducida por deleción (sin corrimiento del marco de lectura) de 27 aa en el dominio GTPasa (en heterocigosis) exhibe degeneración de las células gangionares retinales y desorganización de las crestas mitocondriales en los axones del nervio óptico (133). Ratones mutantes inducidos mediante ENU (N-etil-N-nitrosurea) que contienen una mutación sin sentido en el gen de OPA1 (en heterocigosis), la cual CAPÍTULO VIII 173 produce una proteína truncada, también muestra un 50% de reducción de la proteína OPA1, la aparición lenta de la degeneración del nervio óptico y la disminución de la función visual (134). Además de la haploinsuficiencia, la AOAD puede producirse como consecuencia de un mecanismo de dominancia negativa. En este aspecto, se han reportado varias mutaciones sin sentido en el gen de OPA1 que eliminan los elementos consenso para la unión a GTP (130, 135-137) (http://lbbma.univ-angers.fr/eOPA1), y mutantes de la GTPasa de OPA1 o de su ortólogo de levadura Mgm1 evidencian un efecto negativo en presencia de la proteína salvaje (90, 138, 139). Recientemente, se ha publicado que mutaciones de OPA1 causan oftalmoplegia externa progresiva además de atrofia óptica dominante. Este síndrome extra de OPA1 (“OPA1 plus syndrome”) es un fenotipo clínico caracterizado por una reducción en el número de copias de ADN mitocondrial. En relación a ello, pacientes con determinadas mutaciones en OPA1 muestran múltiples deleciones en el ADN mitocondrial en el músculo esquelético (140, 141). En los últimos años, se ha obtenido información creciente con respecto a las patologías relacionadas a OPA1, sin embargo quedan aún pendientes de resolver los interrogantes de por qué las mutaciones de OPA1 afectan específicamente a la célula ganglionar retinal y no a otros tipos celulares, y si mutaciones de OPA1 causan enfermedad debido a la disrupción de la morfología mitocondrial o si esta alteración es secundaria a la mayor susceptibilidad a la apoptosis. Por ende, son necesarios estudios adicionales sobre la función de OPA1 para poder responder a dichas preguntas. Mutaciones en MFN2: Charcot-Marie-Tooth Tipo 2A La enfermedad de Charcot-Marie-Tooth (CMT) (también conocida como neuropatía motora y sensorial hereditaria HMSN) se caracteriza clínicamente por debilidad y atrofia muscular distal, predominantemente en las extremidades inferiores, y pérdida sensorial. Aproximadamente 1 de cada 2.500 individuos es diagnosticado con CMT, lo que conlleva a que sea la enfermedad hereditaria más común y la neuropatía hereditaria más habitual (142). CMT2 se caracteriza por la degeneración y la regeneración axonal crónica, produciendo una pérdida continua de fibras nerviosas asociada a una velocidad de conducción del nervio motor normal o levemente reducida (≥38 m/s) (143, 144). 174 CAPÍTULO VIII Mutaciones en MFN2 son la causa del 20% de los casos de CMT2 (145), haciéndola la forma axonal de CMT más prevalente. Los casos de CMT2 presentan alta variabilidad en cuanto a los síntomas clínicos. Se han hallado mutaciones de MFN2 en familias con CMT2 que se asocian a características adicionales tales como espasticidad (también referida como neuropatía motora y sensorial hereditaria de tipo V, HMSN V) (146) o atrofia (HMSN VI) (147). Se han identificado más de 40 formas mutantes de MFN2 en pacientes con CMT2A (145-152), y la mayoría de dichas mutaciones son de sentido erróneo. Más del 50% han sido detectadas en el dominio GTPasa de MFN2 (entre los residuos aminoacídicos 99 y 310). Además, se detectaron mutaciones de MFN2 en la región NH2-terminal, cerca o en el dominio de unión a Ras; en la vecindad o en la región “coiled-coil” 1 (406-433); y en el extremo COOH terminal, en la región “coiled-coil” 2, del lado citoplasmático. Curiosamente, teniendo en cuenta las aproximadamente 23 mutaciones halladas en el dominio GTPasa, ninguna de ellas afecta la unión a GTP (153). En el gen de MFN2, hay zonas de alta frecuencia de mutación (“mutational spots”). Con respecto a ello, estudios clínicos indican que algunas de las mutaciones de MFN2 han sido halladas en más de una familia. Dos estudios clínicos importantes demostraron la existencia de dos subgrupos de pacientes de CMT2 que pueden distinguirse sobre la base de las diferencias en la edad de aparición y la severidad de la enfermedad. En relación a ello, se han detectado algunas de las mutaciones en pacientes con aparición temprana de la enfermedad (<10 años de edad) y un fenotipo clínico fuerte, mientras que otras mutaciones de MFN2 se han detectado en pacientes con aparición tardía y enfermedad más leve (148, 151). Es llamativo que la mutación R280H ha sido identificada en pacientes con aparición tardía de la enfermedad (en 6 individuos de la misma familia), mientras que la misma mutación ha sido detectada en otra familia de distinto origen asociada a una aparición temprana de la enfermedad. Estos datos sugieren que el ambiente o genes moduladores estarían regulando la intensidad del fenotipo. Las mutaciones de MFN2 responsables de CMT2A se heredan en forma autosómica dominante; como consecuencia pueden producir haploinsuficiencia o una ganancia de función dominante. En esta línea, la sobreexpresión de algunas formas mutantes de MFN2 induce la agregación de mitocondrias en cultivos de neuronas ganglionares de la raíz dorsal de rata o en fibroblastos embrionarios de ratón (MEFs) resultando en una ganancia de CAPÍTULO VIII 175 función, mientras que otras formas mutantes o la MFN2 salvaje no producen agregación mitocondrial (22, 153). También, en relación a esto, se han generado ratones transgénicos que expresan una forma mutante de MFN2 con una presumible ganancia de función (T105M) específicamente en neuronas motoras. Estos ratones evidencian un fenotipo consistente con los síntomas clínicos detectados en CMT2A, sugiriendo que mutaciones en MFN2 afectan la patogénesis de esta enfermedad (154). Además de los efectos dominantes mostrados por algunos mutantes de MFN2, estudios realizados en MEFs doble knockout de MFN2 (KO simultáneo de MFN1 y MFN2) evidencian que algunos mutantes de MFN2 también confieren ausencia de actividad de fusión mitocondrial (22). En un estudio diferente se ha demostrado que otros mutantes de MFN2 inducen movilidad mitocondrial reducida en cultivos neuronales (153). Es interesante comentar que la actividad de fusión mitocondrial defectuosa de algunos mutantes de Mfn2 es rescatada por la expresión de Mfn1, que está de acuerdo con la observación de que Mfn1 se asocia físicamente con Mfn2 salvaje y con formas mutadas causantes de CMT2A (101, 155). CMT2A es una axonopatía y esto está de acuerdo con el fenotipo obtenido en ratones knockout condicionales para MFN2. De hecho, la ablación específica de Mfn2 en el cerebelo de ratones causa neurodegeneración (29). Bajo estas condiciones, las células de Purkinje evidencian una distribución mitocondrial aberrante y un crecimiento dendrítico y formación de espinas reducidos (29). Estos resultados indican que Mfn2 es esencial para mantener la función neuronal. Hasta el momento, la interacción entre mutantes de Mfn1 y de Mfn2, y la capacidad de Mfn1 de rescatar la función es la única explicación mecanística disponible que explique las alteraciones tejido-específicas en los pacientes con CMT2A. Otra conclusión extraída a partir de la información disponible sobre mutaciones de MFN2 que causan CMT2A es la considerable heterogeneidad con respecto a las alteraciones en sus propiedades biológicas. Con respecto a ello, algunos mutantes de MFN2 muestran ganancia de función, otros, pérdida de función, mientras que otros parecieran provocar ambos, ganancia y pérdida de función (22, 153). 176 CAPÍTULO VIII Mutaciones en GDAP1: Charcot-Marie-Tooth Tipo 4A Las mutaciones en GDAP1 están asociadas con el tipo 4A de CMT (CMT4A), la forma recesiva de CMT detectada con mayor frecuencia (93, 94). CMT4 es clínicamente similar pero más severa que CMT1 y tiene una edad promedio de aparición temprana. CMT4 se define clásicamente como una forma demielinizante de la enfermedad y está asociada a de- y remielinización segmental, en contraste a CMT2, que muestra degeneración axonal sin demielinización (156, 157). Sin embargo, el análisis de la tasa de conducción nerviosa de individuos con mutaciones en GDAP1 indica que mientras algunas mutaciones muestran bajas tasas de conducción (94, 158, 159), otras se caracterizan por presentar tasas normales (158, 160-163). Estos datos demuestran que algunas mutaciones en GDAP1 causan pérdida axonal más que demielinización. GDAP1 se expresa en las células de Schwann en proceso de mielinización y en las neuronas del sistema nervioso periférico en roedores (92, 164), y existen evidencias que indican que la expresión génica de GDAP1 es mucho mayor en las últimas (164), y que las células de Schwann en cultivo no expresan GDAP1 (164). Estas observaciones sugieren que las neuronas del sistema nervioso periférico son el principal tipo celular afectado en esta enfermedad. Sin embargo, se requieren estudios adicionales que demuestren este punto. Tras su expresión en células COS-7, los mutantes de GDAP1 presentan actividad de fusión mitocondrial deficiente, a pesar de localizarse en la mitocondria (92). Algunos mutantes son completamente inactivos con respecto a la promoción de la fragmentación mitocondrial, y otros mutantes muestran algún efecto residual sobre la fragmentación mitocondrial aunque muy reducido en comparación con la actividad de GDAP1 salvaje (92). Es interesante mencionar que la mutación T157P, descripta originalmente como una mutación dominante de novo en un caso esporádico (165), induce agregación mitocondrial, sugiriendo una potencial ganancia de función (166). Muy recientemente, se han identificado 4 mutaciones patogénicas heterocigotas, 3 de las cuales novedosas, en familias con CMT (167). Tres de las mutaciones presentan penetrancia reducida. La aparición de la enfermedad en los individuos afectados fue variable, extendiéndose desde la infancia temprana hasta la adultez. La progresión de la enfermedad fue lenta en la mayoría de los pacientes y la severidad global más moderada que la observada típicamente en las mutaciones de GDAP1 autosómicas recesivas. En la mayoría de los pacientes los cambios electrofisiológicos son heterogéneos pero compatibles CAPÍTULO VIII 177 con la neuropatía axonal. Por ello, este reporte confirma la variabilidad fenotípica y la penetrancia reducida de las mutaciones de GDAP1 autosómicas dominantes en CMT. Mutaciones en DRP1: Desarrollo cerebral anormal Se ha publicado un reportaje de una niña recién nacida con microencefalia, desarrollo cerebral anormal, atrofia e hipoplasia óptica, acidemia láctica persistente, y concentración plasmática levemente elevada de ácidos grasos de cadena muy larga (168). A los 6 días de vida, estudios de resonancia magnética del cerebro revelaron un patrón giral anormal en ambos lóbulos frontales que se extendía hacia las áreas perisilvias y que estaba asociado a desmielinización. Además, la infante evidenciaba hipotonía troncal con movimientos espontáneos pequeños, carecía de reflejos en los tendones y no respondía a la estimulación lumínica (168). El desorden fue letal, y la paciente falleció a los 37 días de vida. La paciente también presentaba alteraciones metabólicas que consistieron en concentraciones elevadas de lactato en sangre, de alanina plasmática y de ácido cerótico (ácido graso de cadena muy larga). La paciente también evidenciaba concentraciones elevadas de lactato, alanina, piruvato y proteína en el fluido cerebroespinal. Estos datos sugieren alteraciones en la oxidación mitocondrial (oxidación de piruvato) como así también en la oxidación peroxisomal (oxidación de ácidos grasos de cadena muy larga). Fibroblastos obtenidos de la paciente mostraron una deficiente fisión mitocondrial y fisión peroxisomal. Los análisis genéticos revelaron una mutación heterocigota sin sentido en DRP1 (A395D) y que se localiza en un residuo aminoacídico conservado en el dominio medio de la proteína. Esta mutación se comporta de forma dominante y causa la inhibición tanto de la fisión peroxisomal como de la mitocondrial cuando se expresa en fibroblastos (168). Esta inhibición podría ser explicada por un posible defecto en la oligomerización inicial de DRP1 requerida para la formación de las estructuras altamente ensambladas sobre la membrana mitocondrial. Este papel hipotético del dominio central ha sido recientemente demostrado para dinaminas convencionales (169). Evidencias experimentales indican que ratones que carecen de Drp1 presentan anormalidades en el desarrollo, particularmente en el cerebro anterior, y mueren en promedio un poco después de E12.5. Ratones DRP1-/- específico de células neurales (NS) mueren poco después de su nacimiento como resultado de una hipoplasia cerebral con apoptosis. Cultivos primarios de cerebro anterior de ratones NS-DRP1-/- evidenciaron una 178 CAPÍTULO VIII disminución en el número de neuritas y también una formación sináptica defectuosa, probablemente debido a la presencia de mitocondrias agregadas que no pudieron distribuirse correctamente entre las proyecciones neuronales (57). Los autores remarcaron la importancia de la fisión mitocondrial DRP1-dependiente en células altamente polarizadas como es el caso de las neuronas. La depleción Drp1 específica de cerebro causó defectos en el desarrollo del cerebelo: las células de Purkinje poseían unas pocas mitocondrias gigantes en vez de muchas mitocondrias cortas y tubulares observadas en células controles (58). Además, el proceso de apoptosis regulada durante el desarrollo no pudo llevarse a cabo durante la formación del tubo neural in vivo en los embriones DRP1 homocigotas. Resumiendo, la mutación A395D de DRP1 causa una enfermedad letal que puede reflejar defectos en la función mitocondrial o peroxisomal. La severidad de las características clínicas sugiere que las mitocondrias y los peroxisomas están involucrados en la patogénesis de la enfermedad. Estudios adicionales en animales mutantes confirman el papel esencial de Drp1 en la dinámica mitocondrial como así también en el proceso de apoptosis en el desarrollo neuronal. Sin embargo, estos hallazgos requieren de evidencia experimental suplementaria. Mutaciones en LETM1: El Síndrome de Wolf-Hirschhorn Hasta este momento, hemos revisado la información disponible sobre el efecto de mutaciones humanas en genes involucrados en la fusión y fisión mitocondrial. Mutaciones en genes involucrados en la morfología mitocondrial tal como LETM1 también provoca enfermedad neurodegenerativa. El síndrome de Wolf-Hirschhorn (WHS) es un complejo desorden de malformación causado por la deleción de un fragmento del cromosoma 4 y tiene una incidencia de 1/50.000 nacimientos vivos (170). El WHS se caracteriza por retardo de crecimiento y mental, hipotonía congénita, apariencia facial distinta, defectos cardíacos congénitos, defectos en la línea media, y convulsiones. Un número desconocido de genes contribuyen con el fenotipo. El extremo 5’ del gen de LETM1 está deleccionado en todos los pacientes con WHS con el fenotipo completo. Un paciente portador de una pequeña deleción intersticial cercana a LETM1 mostró una afección muy débil y carecía de convulsiones, lo cual sugiere que LETM1 está involucrado CAPÍTULO VIII 179 en algunas de las características neuromusculares del WHS (171). LETM1 es una proteína de 83.5-kDa que se expresa en forma ubicua. LETM1 es una proteína de la membrana interna mitocondrial (172) y posee identidad de secuencia aminoacídica con proteínas mitocondriales de numerosas especies. La delección del gen en levaduras causa disfunción mitocondrial, contenido incrementado de cationes (especialmente K+) y mitocondrias hinchadas (173). Por ello, se ha propuesto que está involucrado en la morfología de organelas y que participa en el intercambio de K+/H+ a través de la membrana mitocondrial interna (173). Datos recientes, confirmaron que LETM1 es un transportador antipórtico mitocondrial (que intercambia Ca2+ y H+) (174). Hallazgos adicionales han demostrado que LETM1 modula la morfología mitocondrial en células de mamíferos. En relación a ello, se ha observado que su represión causa muerte celular no mitocondrial, fragmentación de la red mitocondrial independiente de DRP1, hinchamiento mitocondrial y desorganización de las crestas (172, 175). Los defectos en la morfología mitocondrial causados por la pérdida de función de LETM1 pueden restablecerse mediante la normalización de los flujos de K+ a través de la membrana interna (172, 175). Por otra parte, la represión neurona-específica de DmLETM1 de Drosophila resulta en trastornos del comportamiento locomotor de la mosca y una liberación sináptica reducida de neurotransmisores (176). Por lo tanto, estos datos resaltan la importancia de los flujos de iones y LETM1 en el mantenimiento de la morfología y la función mitocondrial normales. En conclusión, evidencias experimentales y clínicas confirman que mutaciones en genes relacionados a la dinámica mitocondrial son responsables de varias neuropatías. Además, vale la pena mencionar que las proteínas de la dinámica mitocondrial están asociadas a procesos celulares relevantes tal como la unidad mitocondria-retículo endoplásmico, el control de calidad mitocondrial y el tráfico mitocondrial, los cuales son esenciales para el correcto funcionamiento/sobrevida de la neurona. En este reporte, alteraciones en la expresión/actividad de las proteínas de la dinámica mitocondrial podrían estar jugando un papel patofisiológico relevante en las enfermedades neurodegenerativas más importantes. 180 CAPÍTULO VIII AGRADECIMIENTOS Este estudio fue financiado por proyectos de investigación del Ministerio de Educación y Cultura” (SAF2008-03803), de la Generalitat de Catalunya (Proyecto 2005SGR00947), CIBERDEM (Instituto de Salud Carlos III), de la Comisión Europea FP7 (MITIN, HEALTH-F4-2008-223450), y de INTERREG IV-B-SUDOE-FEDER (DIOMED, SOE1/P1/E178). J.S. fue becaria predoctoral de la Fundación Ramon Areces. M.I.H.-A. fue becaria predoctoral de CONACYT, Mexico. A.Z. disfrutó de un Premio de Intensificación Científica de la Universitat de Barcelona. BIBLIOGRAFÍA 1. Lewis, M.R., and Lewis, W.H. 1914. Mitochondria in Tissue Culture. Science 39:330-333. 2. Thomas, D.Y., and Wilkie, D. 1968. Recombination of mitochondrial drug-resistance factors in Saccharomyces cerevisiae. Biochem Biophys Res Commun 30:368-372. 3. Bakeeva, L.E., Chentsov, Y.S., and Skulachev, V.P. 1981. Ontogenesis of mitochondrial reticulum in rat diaphragm muscle. Eur J Cell Biol 25:175-181. 4. Bakeeva, L.E., Chentsov Yu, S., and Skulachev, V.P. 1978. Mitochondrial framework (reticulum mitochondriale) in rat diaphragm muscle. Biochim Biophys Acta 501:349-369. 5. Kimberg, D.V., and Loeb, J.N. 1972. Effects of cortisone administration on rat liver mitochondria. Support for the concept of mitochondrial fusion. J Cell Biol 55:635-643. 6. Bereiter-Hahn, J., and Voth, M. 1994. Dynamics of mitochondria in living cells: shape changes, dislocations, fusion, and fission of mitochondria. Microsc Res Tech 27:198-219. 7. Cortese, J.D., Voglino, L.A., and Hackenbrock, C.R. 1998. Novel fluorescence membrane fusion assays reveal GTP-dependent fusogenic properties of outer mitochondrial membrane-derived proteins. Biochim Biophys Acta 1371:185-198. 8. Nunnari, J., Marshall, W., Straight, A., Murray, A., Sedat, J., and Walter, P. 1997. Mitochondrial transmission during mating in Saccharomyces cerevisiae is determined by mitochondrial fusion and fission and the intramitochondrial segregation of mitochondrial DNA. Mol. Biol. Cell 8:1233-1242. 9. Hales, K.G., and Fuller, M.T. 1997. Developmentally regulated mitochondrial fusion mediated by a conserved, novel, predicted GTPase. Cell 90:121-129. 10. Yaffe, M.P. 1999. The machinery of mitochondrial inheritance and behavior. Science 283:1493-1497. 11. Yaffe, M.P. 2003. The cutting edge of mitochondrial fusion. Nat Cell Biol 5:497-499. CAPÍTULO VIII 181 12. Chen, H., Chomyn, A., and Chan, D.C. 2005. Disruption of fusion results in mitochondrial heterogeneity and dysfunction. Journal of Biological Chemistry 280:26185-26192. 13. Tondera, D., Grandemange, S., Jourdain, A., Karbowski, M., Mattenberger, Y., Herzig, S., Da Cruz, S., Clerc, P., Raschke, I., Merkwirth, C., et al. 2009. SlP-2 is required for stress-induced mitochondrial hyperfusion. EMBO Journal 28:1589-1600. 14. Meeusen, S., McCaffery, J.M., and Nunnari, J. 2004. Mitochondrial Fusion Intermediates Revealed in Vitro. Science 305:1747-1752. 15. Chen, H., Detmer, S.A., Ewald, A.J., Griffin, E.E., Fraser, S.E., and Chan, D.C. 2003. Mitofusins Mfn1 and Mfn2 coordinately regulate mitochondrial fusion and are essential for embryonic development. The Journal of Cell Biology 160:189-200. 16. Ishihara, N., Eura, Y., and Mihara, K. 2004. Mitofusin 1 and 2 play distinct roles in mitochondrial fusion reactions via GTPase activity. Journal of Cell Science 117:6535-6546. 17. Cipolat, S., De Brito, O.M., Dal Zilio, B., and Scorrano, L. 2004. OPA1 requires mitofusin 1 to promote mitochondrial fusion. Proceedings of the National Academy of Sciences of the United States of America 101:15927-15932. 18. Ishihara, N., Fujita, Y., Oka, T., and Mihara, K. 2006. Regulation of mitochondrial morphology through proteolytic cleavage of OPA1. EMBO Journal 25:2966-2977. 19. Bach, D., Pich, S., Soriano, F.X., Vega, N., Baumgartner, B., Oriola, J., Daugaard, J.R., Lloberas, J., Camps, M., Zierath, J.R., et al. 2003. Mitofusin-2 determines mitochondrial network architecture and mitochondrial metabolism. A novel regulatory mechanism altered in obesity. J Biol Chem 278:1719017197. 20. Rojo, M., Legros, F., Chateau, D., and Lombès, A. 2002. Membrane topology and mitochondrial targeting of mitofusins, ubiquitous mammalian homologs of the transmembrane GTPase Fzo. Journal of Cell Science 115:1663-1674. 21. Santel, A., and Fuller, M.T. 2001. Control of mitochondrial morphology by a human mitofusin. Journal of Cell Science 114:867-874. 22. Detmer, S.A., and Chan, D.C. 2007. Complementation between mouse Mfn1 and Mfn2 protects mitochondrial fusion defects caused by CMT2A disease mutations. The Journal of Cell Biology 176:405414. 23. Santel, A., Frank, S., Gaume, B., Herrler, M., Youle, R.J., and Fuller, M.T. 2003. Mitofusin-1 protein is a generally expressed mediator of mitochondrial fusion in mammalian cells. Journal of Cell Science 116:2763-2774. 24. Koshiba, T., Detmer, S.A., Kaiser, J.T., Chen, H., McCaffery, J.M., and Chan, D.C. 2004. Structural Basis of Mitochondrial Tethering by Mitofusin Complexes. Science 305:858-862. 182 CAPÍTULO VIII 25. Chung, J.-G., Yeh, K.-T., Wu, S.-L., Hsu, N.-Y., Chen, G.-W., Yeh, Y.-W., and Ho, H.-C. 2001. Novel Transmembrane GTPase of Non-Small Cell Lung Cancer Identified by mRNA Differential Display. Cancer Research 61:8873-8879. 26. Eura, Y., Ishihara, N., Oka, T., and Mihara, K. 2006. Identification of a novel protein that regulates mitochondrial fusion by modulating mitofusin (Mfn) protein function. Journal of Cell Science 119:49134925. 27. Eura, Y., Ishihara, N., Yokota, S., and Mihara, K. 2003. Two Mitofusin Proteins, Mammalian Homologues of FZO, with Distinct Functions Are Both Required for Mitochondrial Fusion. Journal of Biochemistry 134:333-344. 28. Huang, P., Yu, T., and Yoon, Y. 2007. Mitochondrial clustering induced by overexpression of the mitochondrial fusion protein Mfn2 causes mitochondrial dysfunction and cell death. European Journal of Cell Biology 86:289-302. 29. Chen, H., McCaffery, J.M., and Chan, D.C. 2007. Mitochondrial Fusion Protects against Neurodegeneration in the Cerebellum. Cell 130:548-562. 30. de Brito, O.M., and Scorrano, L. 2008. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature 456:605-610. 31. Karbowski, M., Norris, K.L., Cleland, M.M., Jeong, S.-Y., and Youle, R.J. 2006. Role of Bax and Bak in mitochondrial morphogenesis. Nature 443:658-662. 32. Cohen, M.M.J., Leboucher, G.P., Livnat-Levanon, N., Glickman, M.H., and Weissman, A.M. 2008. Ubiquitin-proteasome-dependent degradation of a mitofusin, a critical regulator of mitochondrial fusion. Molecular Biology of the Cell 19:2457-2464. 33. Escobar-Henriques, M., Westermann, B., and Langer, T. 2006. Regulation of mitochondrial fusion by the F-box protein Mdm30 involves proteasome-independent turnover of Fzo1. The Journal of Cell Biology 173:645-650. 34. Neutzner, A., and Youle, R.J. 2005. Instability of the Mitofusin Fzo1 Regulates Mitochondrial Morphology during the Mating Response of the Yeast Saccharomyces cerevisiae. Journal of Biological Chemistry 280:18598-18603. 35. Chan, N.C., Salazar, A.M., Pham, A.H., Sweredoski, M.J., Kolawa, N.J., Graham, R.L., Hess, S., and Chan, D.C. 2011. Broad activation of the ubiquitin-proteasome system by Parkin is critical for mitophagy. Human Molecular Genetics 20:1726-1737. 36. Gegg, M.E., Cooper, J.M., Chau, K.Y., Rojo, M., Schapira, A.H.V., and Taanman, J.W. 2010. Mitofusin 1 and mitofusin 2 are ubiquitinated in a PINK1/parkin-dependent manner upon induction of mitophagy. Human Molecular Genetics 19:4861-4870. CAPÍTULO VIII 183 37. Ziviani, E., Tao, R.N., and Whitworth, A.J. 2010. Drosophila Parkin requires PINK1 for mitochondrial translocation and ubiquitinates Mitofusin. Proceedings of the National Academy of Sciences 107:5018-5023. 38. Narendra, D., Tanaka, A., Suen, D.F., and Youle, R.J. 2008. Parkin is recruited selectively to impaired mitochondria and promotes their autophagy. Journal of Cell Biology 183:795-803. 39. Shimura, H., Hattori, N., Kubo, S.I., Mizuno, Y., Asakawa, S., Minoshima, S., Shimizu, N., Iwai, K., Chiba, T., Tanaka, K., et al. 2000. Familial Parkinson disease gene product, parkin, is a ubiquitinprotein ligase. Nature Genetics 25:302-305. 40. Meeusen, S., Devay, R., Block, J., Cassidy-Stone, A., Wayson, S., McCaffery, J.M., and Nunnari, J. 2006. Mitochondrial inner-membrane fusion and crista maintenance requires the dynamin-related GTPase Mgm1. Cell 127:383-395. 41. Chen, H., and Chan, D.C. 2005. Emerging functions of mammalian mitochondrial fusion and fission. Hum Mol Genet 14 Spec No. 2:R283-289. 42. Misaka, T., Miyashita, T., and Kubo, Y. 2002. Primary structure of a dynamin-related mouse mitochondrial GTPase and its distribution in brain, subcellular localization, and effect on mitochondrial morphology. Journal of Biological Chemistry 277:15834-15842. 43. Olichon, A., Emorine, L.J., Descoins, E., Pelloquin, L., Brichese, L., Gas, N., Guillou, E., Delettre, C., Valette, A., Hamel, C.P., et al. 2002. The human dynamin-related protein OPA1 is anchored to the mitochondrial inner membrane facing the inter-membrane space. FEBS Letters 523:171-176. 44. Satoh, M., Hamamoto, T., Seo, N., Kagawa, Y., and Endo, H. 2003. Differential sublocalization of the dynamin-related protein OPA1 isoforms in mitochondria. Biochemical and Biophysical Research Communications 300:482-493. 45. Ehses, S., Raschke, I., Mancuso, G., Bernacchia, A., Geimer, S., Tondera, D., Martinou, J.C., Westermann, B., Rugarli, E.I., and Langer, T. 2009. Regulation of OPA1 processing and mitochondrial fusion by m-AAA protease isoenzymes and OMA1. Journal of Cell Biology 187:1023-1036. 46. Griparic, L., Kanazawa, T., and Van Der Bliek, A.M. 2007. Regulation of the mitochondrial dynaminlike protein Opa1 by proteolytic cleavage. Journal of Cell Biology 178:757-764. 47. Legros, F., Lombès, A., Frachon, P., and Rojo, M. 2002. Mitochondrial fusion in human cells is efficient, requires the inner membrane potential, and is mediated by mitofusins. Molecular Biology of the Cell 13:4343-4354. 48. Olichon, A., Baricault, L., Gas, N., Guillou, E., Valette, A., Belenguer, P., and Lenaers, G. 2003. Loss of OPA1 Perturbates the Mitochondrial Inner Membrane Structure and Integrity, Leading to Cytochrome c Release and Apoptosis. Journal of Biological Chemistry 278:7743-7746. 184 CAPÍTULO VIII 49. Guillery, O., Malka, F., Landes, T., Guillou, E., Blackstones, C., Lombès, A., Belenguer, P., Arnoult, D., and Rojo, M. 2008. Metalloprotease-mediated OPA1 processing is modulated by the mitochondrial membrane potential. Biology of the Cell 100:315-325. 50. Liu, X., Weaver, D., Shirihai, O., and Hajnoczky, G. 2009. Mitochondrial /`kiss-and-run/': interplay between mitochondrial motility and fusion-fission dynamics. EMBO J 28:3074-3089. 51. Twig, G., Elorza, A., Molina, A.J., Mohamed, H., Wikstrom, J.D., Walzer, G., Stiles, L., Haigh, S.E., Katz, S., Las, G., et al. 2008. Fission and selective fusion govern mitochondrial segregation and elimination by autophagy. EMBO J 27:433-446. 52. Lee, S., Jeong, S.Y., Lim, W.C., Kim, S., Park, Y.Y., Sun, X., Youle, R.J., and Cho, H. 2007. Mitochondrial fission and fusion mediators, hFis1 and OPA1, modulate cellular senescence. Journal of Biological Chemistry 282:22977-22983. 53. Deng, H., Dodson, M.W., Huang, H., and Guo, M. 2008. The Parkinson's disease genes pink1 and parkin promote mitochondrial fission and/or inhibit fusion in Drosophila. Proceedings of the National Academy of Sciences of the United States of America 105:14503-14508. 54. Kim, I., Rodriguez-Enriquez, S., and Lemasters, J.J. 2007. Selective degradation of mitochondria by mitophagy. Archives of Biochemistry and Biophysics 462:245-253. 55. Suen, D.-F., Norris, K.L., and Youle, R.J. 2008. Mitochondrial dynamics and apoptosis. Genes & Development 22:1577-1590. 56. Parone, P.A., Da Cruz, S., Tondera, D., Mattenberger, Y., James, D.I., Maechler, P., Barja, F., and Martinou, J.C. 2008. Preventing mitochondrial fission impairs mitochondrial function and leads to loss of mitochondrial DNA. PLoS One 3:e3257. 57. Ishihara, N., Nomura, M., Jofuku, A., Kato, H., Suzuki, S.O., Masuda, K., Otera, H., Nakanishi, Y., Nonaka, I., Goto, Y.I., et al. 2009. Mitochondrial fission factor Drp1 is essential for embryonic development and synapse formation in mice. Nature Cell Biology 11:958-966. 58. Wakabayashi, J., Zhang, Z., Wakabayashi, N., Tamura, Y., Fukaya, M., Kensler, T.W., Iijima, M., and Sesaki, H. 2009. The dynamin-related GTPase Drp1 is required for embryonic and brain development in mice. Journal of Cell Biology 186:805-816. 59. Gandre-Babbe, S., and van der Bliek, A.M. 2008. The Novel Tail-anchored Membrane Protein Mff Controls Mitochondrial and Peroxisomal Fission in Mammalian Cells. Mol. Biol. Cell 19:2402-2412. 60. Smirnova, E., Griparic, L., Shurland, D.L., and Van der Bliek, A.M. 2001. Dynamin-related protein Drp1 is required for mitochondrial division in mammalian cells. Molecular Biology of the Cell 12:22452256. 61. Van Der Bliek, A.M. 1999. Functional diversity in the dynamin family. Trends in Cell Biology 9:96-102. CAPÍTULO VIII 185 62. Bleazard, W., McCaffery, J.M., King, E.J., Bale, S., Mozdy, A., Tieu, Q., Nunnari, J., and Shaw, J.M. 1999. The dynamin-related GTPase Dnm1 regulates mitochondrial fission in yeast. Nature Cell Biology 1:298-304. 63. Ingerman, E., Perkins, E.M., Marino, M., Mears, J.A., McCaffery, J.M., Hinshaw, J.E., and Nunnari, J. 2005. Dnm1 forms spirals that are structurally tailored to fit mitochondria. Journal of Cell Biology 170:1021-1027. 64. Lackner, L.L., Horner, J.S., and Nunnari, J. 2009. Mechanistic analysis of a dynamin effector. Science 325:874-877. 65. Legesse-Miller, A., Massol, R.H., and Kirchhausen, T. 2003. Constriction and Dnm1p recruitment are distinct processes in mitochondrial fission. Molecular Biology of the Cell 14:1953-1963. 66. Mears, J.A., Lackner, L.L., Fang, S., Ingerman, E., Nunnari, J., and Hinshaw, J.E. 2011. Conformational changes in Dnm1 support a contractile mechanism for mitochondrial fission. Nat Struct Mol Biol 18:20-26. 67. Koirala, S., Bui, H.T., Schubert, H.L., Eckert, D.M., Hill, C.P., Kay, M.S., and Shaw, J.M. 2010. Molecular architecture of a dynamin adaptor: Implications for assembly of mitochondrial fission complexes. Journal of Cell Biology 191:1127-1139. 68. Mozdy, A.D., McCaffery, J.M., and Shaw, J.M. 2000. Dnm1p Gtpase-Mediated Mitochondrial Fission Is a Multi-Step Process Requiring the Novel Integral Membrane Component Fis1p. The Journal of Cell Biology 151:367-380. 69. Otera, H., Wang, C., Cleland, M.M., Setoguchi, K., Yokota, S., Youle, R.J., and Mihara, K. 2010. Mff is an essential factor for mitochondrial recruitment of Drp1 during mitochondrial fission in mammalian cells. The Journal of Cell Biology 191:1141-1158. 70. Cho, D.H., Nakamura, T., Fang, J., Cieplak, P., Godzik, A., Gu, Z., and Lipton, S.A. 2009. - Amyloid-related mitochondrial fission and neuronal injury. Science 324:102-105. 71. Wasiak, S., Zunino, R., and McBride, H.M. 2007. Bax/Bak promote sumoylation of DRP1 and its stable association with mitochondria during apoptotic cell death. Journal of Cell Biology 177:439-450. 72. Harder, Z., Zunino, R., and McBride, H. 2004. Sumo1 conjugates mitochondrial substrates and participates in mitochondrial fission. Current Biology 14:340-345. 73. Zunino, R., Braschi, E., Xu, L., and McBride, H.M. 2009. Translocation of SenP5 from the nucleoli to the mitochondria modulates DRP1-dependent fission during mitosis. Journal of Biological Chemistry 284:17783-17795. 74. Karbowski, M., Neutzner, A., and Youle, R.J. 2007. The mitochondrial E3 ubiquitin ligase MARCH5 is required for Drp1 dependent mitochondrial division. Journal of Cell Biology 178:71-84. 186 CAPÍTULO VIII 75. Taguchi, N., Ishihara, N., Jofuku, A., Oka, T., and Mihara, K. 2007. Mitotic phosphorylation of dynamin-related GTPase Drp1 participates in mitochondrial fission. Journal of Biological Chemistry 282:11521-11529. 76. Bossy, B., Petrilli, A., Klinglmayr, E., Chen, J., Lutz-Meindl, U., Knott, A.B., Masliah, E., Schwarzenbacher, R., and Bossy-Wetzel, E. 2010. S-Nitrosylation of DRP1 Does Not Affect Enzymatic Activity and is Not Specific to Alzheimer's Disease. Journal of Alzheimers Disease 20:S513-S526. 77. Barsoum, M.J., Yuan, H., Gerencser, A.A., Liot, G., Kushnareva, Y., Graber, S., Kovacs, I., Lee, W.D., Waggoner, J., Cui, J., et al. 2006. Nitric oxide-induced mitochondrial fission is regulated by dynamin-related GTPases in neurons. EMBO J 25:3900-3911. 78. Chang, C.R., and Blackstone, C. 2007. Cyclic AMP-dependent protein kinase phosphorylation of Drp1 regulates its GTPase activity and mitochondrial morphology. Journal of Biological Chemistry 282:2158321587. 79. Cribbs, J.T., and Strack, S. 2007. Reversible phosphorylation of Drp1 by cyclic AMP-dependent protein kinase and calcineurin regulates mitochondrial fission and cell death. EMBO Reports 8:939-944. 80. Cereghetti, G.M., Stangherlin, A., Martins De Brito, O., Chang, C.R., Blackstone, C., Bernardi, P., and Scorrano, L. 2008. Dephosphorylation by calcineurin regulates translocation of Drp1 to mitochondria. Proceedings of the National Academy of Sciences of the United States of America 105:15803-15808. 81. Han, X.J., Lu, Y.F., Li, S.A., Kaitsuka, T., Sato, Y., Tomizawa, K., Nairn, A.C., Takei, K., Matsui, H., and Matsushita, M. 2008. CaM kinase I -induced phosphorylation of Drp1 regulates mitochondrial morphology. Journal of Cell Biology 182:573-585. 82. Karren, M.A., Coonrod, E.M., Anderson, T.K., and Shaw, J.M. 2005. The role of Fis1p-Mdv1p interactions in mitochondrial fission complex assembly. Journal of Cell Biology 171:291-301. 83. Yoon, Y., Krueger, E.W., Oswald, B.J., and McNiven, M.A. 2003. The mitochondrial protein hFis1 regulates mitochondrial fission in mammalian cells through an interaction with the dynamin-like protein DLP1. Molecular and Cellular Biology 23:5409-5420. 84. Zhang, Y., and Chan, D.C. 2007. Structural basis for recruitment of mitochondrial fission complexes by Fis1. Proceedings of the National Academy of Sciences of the United States of America 104:18526-18530. 85. Tieu, Q., and Nunnari, J. 2000. Mdv1p Is a Wd Repeat Protein That Interacts with the DynaminRelated Gtpase, Dnm1p, to Trigger Mitochondrial Division. The Journal of Cell Biology 151:353-366. 86. Dohm, J.A., Lee, S.J., Hardwick, J.M., Hill, R.B., and Gittis, A.G. 2004. Cytosolic domain of the human mitochondrial fission protein fis1 adopts a TPR fold. Proteins: Structure, Function, and Bioinformatics 54:153-156. 87. Suzuki, M., Jeong, S.-Y., Karbowski, M., Youle, R.J., and Tjandra, N. 2003. The Solution Structure of Human Mitochondria Fission Protein Fis1 Reveals a Novel TPR-like Helix Bundle. Journal of Molecular Biology 334:445-458. CAPÍTULO VIII 187 88. Jofuku, A., Ishihara, N., and Mihara, K. 2005. Analysis of functional domains of rat mitochondrial Fis1, the mitochondrial fission-stimulating protein. Biochemical and Biophysical Research Communications 333:650-659. 89. Stojanovski, D., Koutsopoulos, O.S., Okamoto, K., and Ryan, M.T. 2004. Levels of human Fis1 at the mitochondrial outer membrane regulate mitochondrial morphology. Journal of Cell Science 117:12011210. 90. Lee, Y.J., Jeong, S.Y., Karbowski, M., Smith, C.L., and Youle, R.J. 2004. Roles of the mammalian mitochondrial fission and fusion mediators Fis1, Drp1, Opa1 in apoptosis. Molecular Biology of the Cell 15:5001-5011. 91. Cassereau, J., Chevrollier, A., Gueguen, N., Desquiret, V., Verny, C., Nicolas, G., Dubas, F., AmatiBonneau, P., Reynier, P., Bonneau, D., et al. 2011. Mitochondrial dysfunction and pathophysiology of Charcot-Marie-Tooth disease involving GDAP1 mutations. Experimental Neurology 227:31-41. 92. Niemann, A., Ruegg, M., La Padula, V., Schenone, A., and Suter, U. 2005. Ganglioside-induced differentiation associated protein 1 is a regulator of the mitochondrial network: New implications for Charcot-Marie-Tooth disease. Journal of Cell Biology 170:1067-1078. 93. Cuesta, A., Pedrola, L., Sevilla, T., Garcia-Planells, J., Chumillas, M.J., Mayordomo, F., LeGuern, E., Marin, I., Vilchez, J.J., and Palau, F. 2002. The gene encoding ganglioside-induced differentiationassociated protein 1 is mutated in axonal Charcot-Marie-Tooth type 4A disease. Nat Genet 30:22-25. 94. Baxter, R.V., Ben Othmane, K., Rochelle, J.M., Stajich, J.E., Hulette, C., Dew-Knight, S., Hentati, F., Ben Hamida, M., Bel, S., Stenger, J.E., et al. 2002. Ganglioside-induced differentiation-associated protein-1 is mutant in Charcot-Marie-Tooth disease type 4A/8q21. Nat Genet 30:21-22. 95. Tondera, D., Czauderna, F., Paulick, K., Schwarzer, R., Kaufmann, J., and Santel, A. 2005. The mitochondrial protein MTP18 contributes to mitochondrial fission in mammalian cells. Journal of Cell Science 118:3049-3059. 96. Palmer, C.S., Osellame, L.D., Laine, D., Koutsopoulos, O.S., Frazier, A.E., and Ryan, M.T. 2011. MiD49 and MiD51, new components of the mitochondrial fission machinery. EMBO Rep 12:565-573. 97. Simpson, J.C., Wellenreuther, R., Poustka, A., Pepperkok, R., and Wiemann, S. 2000. Systematic subcellular localization of novel proteins identified by large-scale cDNA sequencing. EMBO Reports 1:287-292. 98. Amchenkova, A.A., Bakeeva, L.E., Chentsov, Y.S., Skulachev, V.P., and Zorov, D.B. 1988. Coupling membranes as energy-transmitting cables. I. Filamentous mitochondria in fibroblasts and mitochondrial clusters in cardiomyocytes. J Cell Biol 107:481-495. 99. Skulachev, V.P. 2001. Mitochondrial filaments and clusters as intracellular power-transmitting cables. Trends Biochem Sci 26:23-29. 188 CAPÍTULO VIII 100. Pich, S., Bach, D., Briones, P., Liesa, M., Camps, M., Testar, X., Palacin, M., and Zorzano, A. 2005. The Charcot-Marie-Tooth type 2A gene product, Mfn2, up-regulates fuel oxidation through expression of OXPHOS system. Hum Mol Genet 14:1405-1415. 101. Loiseau, D., Chevrollier, A., Verny, C., Guillet, V., Gueguen, N., Pou de Crescenzo, M.A., Ferre, M., Malinge, M.C., Guichet, A., Nicolas, G., et al. 2007. Mitochondrial coupling defect in Charcot-MarieTooth type 2A disease. Ann Neurol 61:315-323. 102. Tang, S., Le, P.K., Tse, S., Wallace, D.C., and Huang, T. 2009. Heterozygous mutation of Opa1 in Drosophila shortens lifespan mediated through increased reactive oxygen species production. PLoS One 4:e4492. 103. Yarosh, W., Monserrate, J., Tong, J.J., Tse, S., Le, P.K., Nguyen, K., Brachmann, C.B., Wallace, D.C., and Huang, T. 2008. The molecular mechanisms of OPA1-mediated optic atrophy in Drosophila model and prospects for antioxidant treatment. PLoS Genet 4:e6. 104. Lodi, R., Tonon, C., Valentino, M.L., Iotti, S., Clementi, V., Malucelli, E., Barboni, P., Longanesi, L., Schimpf, S., Wissinger, B., et al. 2004. Deficit of in vivo mitochondrial ATP production in OPA1related dominant optic atrophy. Ann Neurol 56:719-723. 105. Chevrollier, A., Guillet, V., Loiseau, D., Gueguen, N., de Crescenzo, M.A., Verny, C., Ferre, M., Dollfus, H., Odent, S., Milea, D., et al. 2008. Hereditary optic neuropathies share a common mitochondrial coupling defect. Ann Neurol 63:794-798. 106. Zanna, C., Ghelli, A., Porcelli, A.M., Karbowski, M., Youle, R.J., Schimpf, S., Wissinger, B., Pinti, M., Cossarizza, A., Vidoni, S., et al. 2008. OPA1 mutations associated with dominant optic atrophy impair oxidative phosphorylation and mitochondrial fusion. Brain 131:352-367. 107. Spinazzi, M., Cazzola, S., Bortolozzi, M., Baracca, A., Loro, E., Casarin, A., Solaini, G., Sgarbi, G., Casalena, G., Cenacchi, G., et al. 2008. A novel deletion in the GTPase domain of OPA1 causes defects in mitochondrial morphology and distribution, but not in function. Hum Mol Genet 17:32913302. 108. Zhang, Z., Wakabayashi, N., Wakabayashi, J., Tamura, Y., Song, W.J., Sereda, S., Clerc, P., Polster, B.M., Aja, S.M., Pletnikov, M.V., et al. 2011. The dynamin-related GTPase Opa1 is required for glucose-stimulated ATP production in pancreatic beta cells. Mol Biol Cell 22:2235-2245. 109. Benard, G., Bellance, N., James, D., Parrone, P., Fernandez, H., Letellier, T., and Rossignol, R. 2007. Mitochondrial bioenergetics and structural network organization. J Cell Sci 120:838-848. 110. Ashrafian, H., Docherty, L., Leo, V., Towlson, C., Neilan, M., Steeples, V., Lygate, C.A., Hough, T., Townsend, S., Williams, D., et al. 2010. A mutation in the mitochondrial fission gene Dnm1l leads to cardiomyopathy. PLoS Genet 6:e1001000. 111. Liesa, M., Palacin, M., and Zorzano, A. 2009. Mitochondrial dynamics in mammalian health and disease. Physiol Rev 89:799-845. CAPÍTULO VIII 189 112. Kelley, D.E., He, J., Menshikova, E.V., and Ritov, V.B. 2002. Dysfunction of mitochondria in human skeletal muscle in type 2 diabetes. Diabetes 51:2944-2950. 113. Mootha, V.K., Lindgren, C.M., Eriksson, K.F., Subramanian, A., Sihag, S., Lehar, J., Puigserver, P., Carlsson, E., Ridderstrale, M., Laurila, E., et al. 2003. PGC-1alpha-responsive genes involved in oxidative phosphorylation are coordinately downregulated in human diabetes. Nat Genet 34:267-273. 114. Zorzano, A., Liesa, M., Sebastian, D., Segales, J., and Palacin, M. 2010. Mitochondrial fusion proteins: dual regulators of morphology and metabolism. Semin Cell Dev Biol 21:566-574. 115. Zorzano, A., Liesa, M., and Palacin, M. 2009. Role of mitochondrial dynamics proteins in the pathophysiology of obesity and type 2 diabetes. Int J Biochem Cell Biol 41:1846-1854. 116. Bach, D., Naon, D., Pich, S., Soriano, F.X., Vega, N., Rieusset, J., Laville, M., Guillet, C., Boirie, Y., Wallberg-Henriksson, H., et al. 2005. Expression of Mfn2, the Charcot-Marie-Tooth neuropathy type 2A gene, in human skeletal muscle: effects of type 2 diabetes, obesity, weight loss, and the regulatory role of tumor necrosis factor alpha and interleukin-6. Diabetes 54:2685-2693. 117. Mingrone, G., Manco, M., Calvani, M., Castagneto, M., Naon, D., and Zorzano, A. 2005. Could the low level of expression of the gene encoding skeletal muscle mitofusin-2 account for the metabolic inflexibility of obesity? Diabetologia 48:2108-2114. 118. Liesa, M., Borda-d'Agua, B., Medina-Gomez, G., Lelliott, C.J., Paz, J.C., Rojo, M., Palacin, M., Vidal-Puig, A., and Zorzano, A. 2008. Mitochondrial fusion is increased by the nuclear coactivator PGC-1beta. PLoS One 3:e3613. 119. Soriano, F.X., Liesa, M., Bach, D., Chan, D.C., Palacin, M., and Zorzano, A. 2006. Evidence for a mitochondrial regulatory pathway defined by peroxisome proliferator-activated receptor-gamma coactivator-1 alpha, estrogen-related receptor-alpha, and mitofusin 2. Diabetes 55:1783-1791. 120. Patti, M.E., Butte, A.J., Crunkhorn, S., Cusi, K., Berria, R., Kashyap, S., Miyazaki, Y., Kohane, I., Costello, M., Saccone, R., et al. 2003. Coordinated reduction of genes of oxidative metabolism in humans with insulin resistance and diabetes: Potential role of PGC1 and NRF1. Proc Natl Acad Sci U S A 100:8466-8471. 121. Olichon, A., Guillou, E., Delettre, C., Landes, T., Arnauné-Pelloquin, L., Emorine, L.J., Mils, V., Daloyau, M., Hamel, C., Amati-Bonneau, P., et al. 2006. Mitochondrial dynamics and disease, OPA1. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1763:500-509. 122. Hoyt, C. 1980. Autosomal dominant optic atrophy. A spectrum of disability. Ophthalmology. 87:245251. 123. Johnston, R.L., Seller, M.J., Behnam, J.T., Burdon, M.A., and Spalton, D.J. 1999. Dominant optic atrophy: Refining the clinical diagnostic criteria in light of genetic linkage studies. Ophthalmology 106:123-128. 190 CAPÍTULO VIII 124. Votruba, M., Fitzke, F.W., Holder, G.E., Carter, A., Bhattacharya, S.S., and Moore, A.T. 1998. Clinical Features in Affected Individuals From 21 Pedigrees With Dominant Optic Atrophy. Arch Ophthalmol 116:351-358. 125. Johnston, P., Gaster, R., Smith, V., and Tripathi, R. 1979. A clinicopathologic study of autosomal dominant optic atrophy. Am J Ophthalmol 88:868-875. 126. Kjer, P., Jensen, O., and Klinken, L. 1983. Histopathology of eye, optic nerve and brain in a case of dominant optic atrophy. Acta Ophthalmol (Copenh) 61:300-312. 127. Ferré, M., Amati-Bonneau, P., Tourmen, Y., Malthièry, Y., and Reynier, P. 2005. eOPA1: An online database for OPA1 mutations. Human Mutation 25:423-428. 128. Delettre, C., Griffoin, J.-M., Kaplan, J., Dollfus, H., Lorenz, B., Faivre, L., Lenaers, G., Belenguer, P., and Hamel, C. 2001. Mutation spectrum and splicing variants in the &lt;SMALL&gt;OPA1&lt;/SMALL&gt; gene. Human Genetics 109:584-591. 129. Marchbank, N.J., Craig, J.E., Leek, J.P., Toohey, M., Churchill, A.J., Markham, A.F., Mackey, D.A., Toomes, C., and Inglehearn, C.F. 2002. Deletion of the OPA1 gene in a dominant optic atrophy family: evidence that haploinsufficiency is the cause of disease. Journal of Medical Genetics 39:e47. 130. Pesch, U.E.A., Leo-Kottler, B., Mayer, S., Jurklies, B., Kellner, U., Apfelstedt-Sylla, E., Zrenner, E., Alexander, C., and Wissinger, B. 2001. OPA1 mutations in patients with autosomal dominant optic atrophy and evidence for semi-dominant inheritance. Human Molecular Genetics 10:1359-1368. 131. Frezza, C., Cipolat, S., Martins de Brito, O., Micaroni, M., Beznoussenko, G.V., Rudka, T., Bartoli, D., Polishuck, R.S., Danial, N.N., De Strooper, B., et al. 2006. OPA1 Controls Apoptotic Cristae Remodeling Independently from Mitochondrial Fusion. Cell 126:177-189. 132. Griparic, L., van der Wel, N.N., Orozco, I.J., Peters, P.J., and van der Bliek, A.M. 2004. Loss of the Intermembrane Space Protein Mgm1/OPA1 Induces Swelling and Localized Constrictions along the Lengths of Mitochondria. Journal of Biological Chemistry 279:18792-18798. 133. Alavi, M.V., Bette, S., Schimpf, S., Schuettauf, F., Schraermeyer, U., Wehrl, H.F., Ruttiger, L., Beck, S.C., Tonagel, F., Pichler, B.J., et al. 2007. A splice site mutation in the murine Opa1 gene features pathology of autosomal dominant optic atrophy. Brain 130:1029-1042. 134. Davies, V.J., Hollins, A.J., Piechota, M.J., Yip, W., Davies, J.R., White, K.E., Nicols, P.P., Boulton, M.E., and Votruba, M. 2007. Opa1 deficiency in a mouse model of autosomal dominant optic atrophy impairs mitochondrial morphology, optic nerve structure and visual function. Human Molecular Genetics 16:1307-1318. 135. Baris, O., Delettre, C., Amati-Bonneau, P., Surget, M.-O., Charlin, J.-F., Catier, A., Derieux, L., Guyomard, J.-L., Dollfus, H., Jonveaux, P., et al. 2003. Fourteen novel OPA1 mutations in autosomal dominant optic atrophy including two de novo mutations in sporadic optic atrophy. Human Mutation 21:656-656. CAPÍTULO VIII 191 136. Delettre, C., Lenaers, G., Griffoin, J.-M., Gigarel, N., Lorenzo, C., Belenguer, P., Pelloquin, L., Grosgeorge, J., Turc-Carel, C., Perret, E., et al. 2000. Nuclear gene OPA1, encoding a mitochondrial dynamin-related protein, is mutated in dominant optic atrophy. Nat Genet 26:207-210. 137. Kim, J.Y., Hwang, J.-M., Ko, H.S., Seong, M.-W., Park, B.-J., and Park, S.S. 2005. Mitochondrial DNA content is decreased in autosomal dominant optic atrophy. Neurology 64:966-972. 138. Shepard, K.A., and Yaffe, M.P. 1999. The Yeast Dynamin-like Protein, Mgm1p, Functions on the Mitochondrial Outer Membrane to Mediate Mitochondrial Inheritance. The Journal of Cell Biology 144:711-720. 139. Wong, E.D., Wagner, J.A., Scott, S.V., Okreglak, V., Holewinske, T.J., Cassidy-Stone, A., and Nunnari, J. 2003. The intramitochondrial dynamin-related GTPase, Mgm1p, is a component of a protein complex that mediates mitochondrial fusion. The Journal of Cell Biology 160:303-311. 140. Amati-Bonneau, P., Valentino, M.L., Reynier, P., Gallardo, M.E., Bornstein, B., Boissière, A., Campos, Y., Rivera, H., de la Aleja, J.G., Carroccia, R., et al. 2008. OPA1 mutations induce mitochondrial DNA instability and optic atrophy ‘plus’ phenotypes. Brain 131:338-351. 141. Hudson, G., Amati-Bonneau, P., Blakely, E.L., Stewart, J.D., He, L., Schaefer, A.M., Griffiths, P.G., Ahlqvist, K., Suomalainen, A., Reynier, P., et al. 2008. Mutation of OPA1 causes dominant optic atrophy with external ophthalmoplegia, ataxia, deafness and multiple mitochondrial DNA deletions: a novel disorder of mtDNA maintenance. Brain 131:329-337. 142. Skre, H. 1974. Genetic and clinical aspects of Charcot-Marie-Tooth's disease. Clin Genet 6:98-118. 143. Dyck, P.J., and Lambert, E.H. 1968. Lower Motor and Primary Sensory Neuron Diseases With Peroneal Muscular Atrophy: I. Neurologic, Genetic, and Electrophysiologic Findings in Hereditary Polyneuropathies. Arch Neurol 18:603-618. 144. Dyck, P.J., and Lambert, E.H. 1968. Lower Motor and Primary Sensory Neuron Diseases With Peroneal Muscular Atrophy: II. Neurologic, Genetic, and Electrophysiologic Findings in Various Neuronal Degenerations. Arch Neurol 18:619-625. 145. Lawson, V.H., Graham, B.V., and Flanigan, K.M. 2005. Clinical and electrophysiologic features of CMT2A with mutations in the mitofusin 2 gene. Neurology 65:197-204. 146. Zhu, D., Kennerson, M.L., Walizada, G., Züchner, S., Vance, J.M., and Nicholson, G.A. 2005. Charcot–Marie–Tooth with pyramidal signs is genetically heterogeneous: Families with and without MFN2 mutations. Neurology 65:496-497. 147. Züchner, S., De Jonghe, P., Jordanova, A., Claeys, K.G., Guergueltcheva, V., Cherninkova, S., Hamilton, S.R., Van Stavern, G., Krajewski, K.M., Stajich, J., et al. 2006. Axonal neuropathy with optic atrophy is caused by mutations in mitofusin 2. Annals of Neurology 59:276-281. 192 CAPÍTULO VIII 148. Chung, K.W., Kim, S.B., Park, K.D., Choi, K.G., Lee, J.H., Eun, H.W., Suh, J.S., Hwang, J.H., Kim, W.K., Seo, B.C., et al. 2006. Early onset severe and late-onset mild Charcot–Marie–Tooth disease with mitofusin 2 (MFN2) mutations. Brain 129:2103-2118. 149. Engelfried, K., Vorgerd, M., Hagedorn, M., Haas, G., Gilles, J., Epplen, J., and Meins, M. 2006. Charcot-Marie-Tooth neuropathy type 2A: novel mutations in the mitofusin 2 gene (MFN2). BMC Medical Genetics 7:53. 150. Kijima, K., Numakura, C., Izumino, H., Umetsu, K., Nezu, A., Shiiki, T., Ogawa, M., Ishizaki, Y., Kitamura, T., Shozawa, Y., et al. 2005. Mitochondrial GTPase mitofusin 2 mutation in Charcot– Marie–Tooth neuropathy type 2A. Human Genetics 116:23-27. 151. Verhoeven, K., Claeys, K.G., Züchner, S., Schröder, J.M., Weis, J., Ceuterick, C., Jordanova, A., Nelis, E., De Vriendt, E., Van Hul, M., et al. 2006. MFN2 mutation distribution and genotype/phenotype correlation in Charcot–Marie–Tooth type 2. Brain 129:2093-2102. 152. Zuchner, S., Mersiyanova, I.V., Muglia, M., Bissar-Tadmouri, N., Rochelle, J., Dadali, E.L., Zappia, M., Nelis, E., Patitucci, A., Senderek, J., et al. 2004. Mutations in the mitochondrial GTPase mitofusin 2 cause Charcot-Marie-Tooth neuropathy type 2A. Nat Genet 36:449-451. 153. Baloh, R.H., Schmidt, R.E., Pestronk, A., and Milbrandt, J. 2007. Altered Axonal Mitochondrial Transport in the Pathogenesis of Charcot-Marie-Tooth Disease from Mitofusin 2 Mutations. The Journal of Neuroscience 27:422-430. 154. Detmer, S.A., Velde, C.V., Cleveland, D.W., and Chan, D.C. 2008. Hindlimb gait defects due to motor axon loss and reduced distal muscles in a transgenic mouse model of Charcot–Marie–Tooth type 2A. Human Molecular Genetics 17:367-375. 155. Amiott, E.A., Lott, P., Soto, J., Kang, P.B., McCaffery, J.M., DiMauro, S., Abel, E.D., Flanigan, K.M., Lawson, V.H., and Shaw, J.M. 2008. Mitochondrial fusion and function in Charcot-MarieTooth type 2A patient fibroblasts with mitofusin 2 mutations. Exp Neurol 211:115-127. 156. Berger, P., Young, P., and Suter, U. 2002. Molecular cell biology of Charcot-Marie-Tooth disease. neurogenetics 4:1-15. 157. Suter, U., and Scherer, S.S. 2003. Disease mechanisms in inherited neuropathies. Nat Rev Neurosci 4:714-726. 158. Ammar, N., Nelis, E., Merlini, L., Barisic, N., Amouri, R., Ceuterick, C., Martin, J.-J., Timmerman, V., Hentati, F., and De Jonghe, P. 2003. Identification of novel GDAP1 mutations causing autosomal recessive Charcot-Marie-Tooth disease. Neuromuscular Disorders 13:720-728. 159.Senderek, J., Bergmann, C., Ramaekers, V.T., Nelis, E., Bernert, G., Makowski, A., Züchner, S., De Jonghe, P., Rudnik Schöneborn, S., Zerres, K., et al. 2003. Mutations in the ganglioside induced differentiation associated protein 1 (GDAP1) gene in intermediate type autosomal recessive Charcot– Marie–Tooth neuropathy. Brain 126:642-649. CAPÍTULO VIII 193 160.Boerkoel, C.F., Takashima, H., Nakagawa, M., Izumo, S., Armstrong, D., Butler, I., Mancias, P., Papasozomenos, S.C.H., Stern, L.Z., and Lupski, J.R. 2003. CMT4A: Identification of a Hispanic GDAP1 founder mutation. Annals of Neurology 53:400-405. 161.Nelis, E., Erdem, S., Van den Bergh, P.Y.K., Belpaire–Dethiou, M.-C., Ceuterick, C., Van Gerwen, V., Cuesta, A., Pedrola, L., Palau, F., Gabreëls–Festen, A.A.W.M., et al. 2002. Mutations in GDAP1. Neurology 59:1865-1872. 162.De Sandre-Giovannoli, A., Chaouch, M., Boccaccio, I., Bernard, R., Delague, V., Grid, D., Vallat, J.M., Lévy, N., and Mégarbané, A. 2003. Phenotypic and genetic exploration of severe demyelinating and secondary axonal neuropathies resulting from GDAP1 nonsense and splicing mutations. Journal of Medical Genetics 40:e87. 163. Sevilla, T., Cuesta, A., Chumillas, M.J., Mayordomo, F., Pedrola, L., Palau, F., and Vílchez, J.J. 2003. Clinical, electrophysiological and morphological findings of Charcot–Marie–Tooth neuropathy with vocal cord palsy and mutations in the GDAP1 gene. Brain 126:2023-2033. 164. Pedrola, L., Espert, A., Wu, X., Claramunt, R., Shy, M.E., and Palau, F. 2005. GDAP1, the protein causing Charcot–Marie–Tooth disease type 4A, is expressed in neurons and is associated with mitochondria. Human Molecular Genetics 14:1087-1094. 165. Claramunt, R., Pedrola, L., Sevilla, T., López de Munain, A., Berciano, J., Cuesta, A., SánchezNavarro, B., Millán, J.M., Saifi, G.M., Lupski, J.R., et al. 2005. Genetics of Charcot-Marie-Tooth disease type 4A: mutations, inheritance, phenotypic variability, and founder effect. Journal of Medical Genetics 42:358-365. 166. Pedrola, L., Espert, A., Valdés-Sánchez, T., Sánchez-Piris, M., Sirkowski, E.E., Scherer, S.S., Fariñas, I., and Palau, F. 2008. Cell expression of GDAP1 in the nervous system and pathogenesis of CharcotMarie-Tooth type 4A disease. Journal of Cellular and Molecular Medicine 12:679-689. 167. Zimon, M., Baets, J., Fabrizi, G., Jaakkola, E., Kabzinska, D., Pilch, J., Schindler, A., Cornblath, D., Fischbeck, K., Auer-Grumbach, M., et al. 2011. Dominant GDAP1 mutations cause predominantly mild CMT phenotypes. Neurology [Epub ahead of print]. 168. Waterham, H.R., Koster, J., van Roermund, C.W.T., Mooyer, P.A.W., Wanders, R.J.A., and Leonard, J.V. 2007. A Lethal Defect of Mitochondrial and Peroxisomal Fission. New England Journal of Medicine 356:1736-1741. 169. Ramachandran, R., Surka, M., Chappie, J.S., Fowler, D.M., Foss, T.R., Song, B.D., and Schmid, S.L. 2007. The dynamin middle domain is critical for tetramerization and higher-order self-assembly. EMBO J 26:559-566. 170. Bergemann, A.D., Cole, F., and Hirschhorn, K. 2005. The etiology of Wolf-Hirschhorn syndrome. Trends in Genetics 21:188-195. 194 CAPÍTULO VIII 171. Rauch, A., Schellmoser, S., Kraus, C., Dörr, H.G., Trautmann, U., Altherr, M.R., Pfeiffer, R.A., and Reis, A. 2001. First known microdeletion within the Wolf-Hirschhorn syndrome critical region refines genotype–phenotype correlation. American Journal of Medical Genetics 99:338-342. 172. Dimmer, K.S., Navoni, F., Casarin, A., Trevisson, E., Endele, S., Winterpacht, A., Salviati, L., and Scorrano, L. 2008. LETM1, deleted in Wolf–Hirschhorn syndrome is required for normal mitochondrial morphology and cellular viability. Human Molecular Genetics 17:201-214. 173. Nowikovsky, K., Froschauer, E.M., Zsurka, G., Samaj, J., Reipert, S., Kolisek, M., Wiesenberger, G., and Schweyen, R.J. 2004. The LETM1/YOL027 Gene Family Encodes a Factor of the Mitochondrial K+ Homeostasis with a Potential Role in the Wolf-Hirschhorn Syndrome. Journal of Biological Chemistry 279:30307-30315. 174. Jiang, D., Zhao, L., and Clapham, D.E. 2009. Genome-Wide RNAi Screen Identifies Letm1 as a Mitochondrial Ca2+/H+ Antiporter. Science 326:144-147. 175. Tamai, S., Iida, H., Yokota, S., Sayano, T., Kiguchiya, S., Ishihara, N., Hayashi, J.-I., Mihara, K., and Oka, T. 2008. Characterization of the mitochondrial protein LETM1, which maintains the mitochondrial tubular shapes and interacts with the AAA-ATPase BCS1L. Journal of Cell Science 121:2588-2600. 176. McQuibban, A.G., Joza, N., Megighian, A., Scorzeto, M., Zanini, D., Reipert, S., Richter, C., Schweyen, R.J., and Nowikovsky, K. 2010. A Drosophila mutant of LETM1, a candidate gene for seizures in Wolf-Hirschhorn syndrome. Human Molecular Genetics 19:987-1000. CAPÍTULO VIII 195