sintesis de proteinas
Anuncio

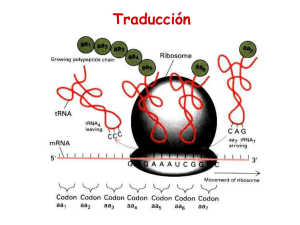
SINTESIS DE PROTEINAS: TRADUCCION La secuencia de nucleótidos da lugar a una secuencia de aminoácidos unidos por condensación. Formar el enlace peptídico es fácil, el COO- está activado y proporciona la energía necesaria. Lo complicado de la traducción es leer la información para poner los aminoácidos en orden. Ahora los aminoácidos no son complementarios de los nucleótidos, hay que modificarlos para que lo sean. Código genético. Cada aminoácido está representado por tres nucleótidos (64 combinaciones). A cada triplete se le llama codon. La correspondencia entre tripletes y aminoácidos es el código genético, que a veces es redundante porque más de un triplete codifica el mismo aminoácido. Nunca es ambiguo, ningún codon especifica más de un aminoácido. Como los codones está uno al lado del otro sin separación podrían leerse hasta tres mensajes distintos. Lo que determina la pauta de lectura es la señal (codon de iniciación → AUG) para el inicio de la traducción. Por lo tanto para encontrar un mensaje en un gen ha de empezar por ATG, que especifica el aminoácido metionina, por lo que las proteínas empiezan por metionina. Hay tres señales para acabar: UAA, UAG, UGA. La región codificante es el trozo de mRNA que codifica una proteína. Los aminoácidos que están representados por más de un triplete son los que más frecuentemente aparecen y son similares. La tercera base de un triplete puede que no sea importante que se reconozca bien, porque parece que no encaja perfectamente. Los tripletes que se diferencian en la tercera posición codifican aminoácidos muy parecidos, lo que disminuye el efecto de las mutaciones. Moléculas participantes en la traducción. En los eucariotas el DNA está en el núcleo pero la traducción se produce en el citosol. El DNA no participa. - mRNA: proporciona información sobre el orden de los aminoácidos con su secuencia de nucleótidos. Se hace en el núcleo y sale al citosol. Otros se producen en mitocondrias y cloroplastos. Hay dos tipos de mensajeros: - Célula procariota: secuencia de nucleótidos 5'→3'. Parte del RNA codifica proteína: región codificante o pauta de lectura abierta (ORF). Los nucleótidos que no aparecen en la proteína están a los dos lados y se llaman UTRC (Untranslated Region), son imprescindibles para la estabilidad y la función. Punto de inicio y final de la traducción bien definidos con triplete de iniciación y STOP. El triplete de iniciación codifica el primer aminoácido, el N-terminal. El triplete de parada está inmediatamente detrás del que especifica el ultimo aminoácido. Frecuentemente los mensajeros son policistrónicos, el gen codificante tiene un solo promotor aunque origine varias proteínas. La parte no codificante se une a ribosoma proporcionando estabilidad. Célula eucariota: Sólo hay una región codificante cuando ya está procesado. Hay dos regiones no codificantes: - 5' es la líder, no traducible. - 3' tiene cola poliA. Todos los mensajeros tienen dos modificaciones en los extremos, una guanosina metilada y una cola poliA en 3'. Ribosomas. Donde se localiza la síntesis de proteínas. Son partículas formadas por RNA y proteínas. Son funcionales en el citosol, excepto los de mitocondrias y cloroplastos. Los de eucariotas y procariotas son esencialmente iguales. La síntesis de una proteína o de otra depende del mRNA. Constan de dos subunidades, una (LS) aproximadamente el doble que la otra (SS). Recogen la mayor parte del RNA celular. Estructura y composición: durante el funcionamiento las subunidades pueden asociarse y disociarse. Cada subunidad tiene una molécula de RNA grande y proteínas, y a veces otros RNA. En u gradiente de densidad tienen un coeficiente de sedimentación: - Procariotas: 70s. La subunidad grande tiene rRNA grande y muchas proteínas. 23s. La subunidad pequeña tiene 5s, molécula de RNA 16s y unas 20 proteínas distintas - Eucariotas: 60s. mensajero subunidad grande 28s y pequeña 18s. Muchas proteínas. La funcionalidad recae en el RNA y no en las proteínas. El ribosoma se ve al microscopio electrónico, se sabe dónde se localizan las proteínas y por lo tanto dónde se producen las funciones. Funciones: - Reconocer mensajero y ponerse en sitio adecuado para la traducción (sitio A). - Sitio A: sitio definido para la entrada de aminoácidos que se van añadiendo. - Sitio P: donde se une la cadena de polímeros. Los sitios están en contacto con el mensajero para saber qué aminoácido poner. - Sitio E: es el de salida. - tRNA: Los aminoácidos han de ser complementarios de los tripletes, por lo que es necesaria esta molécula adaptadora, es un ácido nucleico porque debe ser capaz de reconocer codones y complementarlos. El tRNA lleva aminoácidos y se encarga de que participen en la síntesis de proteínas, formando un derivado del aminoácido y tRNA, el aminoacil-tRNA. Debe haber tantos tRNA como tripletes. Si el aminoácido es sustituido tras la formación del derivado se pondrá erróneamente. La derivación debe ser una reacción muy exacta catalizada por un enzima que produzca pocos errores. Los tRNA son muy pequeños, mucha estructura secundaria, compactos, forma tridimensional de L. Constan de dos partes esenciales: - Parte que reconoce el triplete, un anticodon complementario del codon. - Zona a la que se une el aminoácido, extremo 3'. En todos este extremo termina en CCA, es el aceptor del aminoácido que se une a la adenina final. El anticodon y el sitio aceptor son opuestos. La reacción de unión está llevada a cabo por enzimas aminoacil-tRNA-sintasa. Los sustratos son los aminoácidos y e tRNA y la energía la aporta el ATP. Reacción en dos etapas: - aminoácido + ATP → derivado activado del aminoácido (aa+AMP→derivado aminadenilado) + pirofosfato - El mismo enzima transfiere el aminoácido activado al extremo 3' del tRNA. Con la formación del derivado se consiguen dos cosas: - Formar un derivado complementario del codon. - Forma activada del grupo carboxilo que se une al aminoácido siguiente por condensación. Corrección de errores de la aminoacil-tRNA-sintasa: cada tRNA sólo se carga con el aminoácido adecuado, y a veces un compuesto mal formado es hidrolizado antes de salir del enzima, porque el enzima discrimina entre aminoácidos pero a veces se confunde. El gasto energético es muy alto. Hay un enzima para cada aminoácido. Parte pequeña del ribosoma reconoce al mensajero y se le une. Todos los tRNA tienen la misma forma porque entran en el mismo sitio. Se selecciona un tRNA y no otro según el codon que complemente. El ribosoma tiene un túnel para proteger los primeros 30 aminoácidos de la cadena. El enlace peptídico se forma en la subunidad grande, recayendo la actividad en el RNA grande (23s). Funcionamiento del ribosoma en la traducción: Al principio las dos subunidades están separadas, ensamblándose en el codon de iniciación. Luego se desplaza y va leyendo y colocando aminoácidos. Al final se disocia del mensajero, se suelta y vuelve a empezar. Si al mismo mensajero se asocian varios ribosomas (polirribosoma o polisoma) se producen varias proteínas. el ribosoma cerca de 5' sintetizará una cadena más corta que el de 3'. Todos los componentes se reciclan si no se estropean. El N-terminal se incorpora primero, el último es el C-terminal. En procariotas la traducción puede empezar antes de que finalice la transcripción. Una vez hecho el extremo 5' puede empezar, de ahí el poco procesamiento. En eucariotas no puede ser porque la transcripción y la traducción ocurren en lugares distintos Etapas de la traducción Iniciación Procariotas. Empieza con las subunidades separadas, comenzando la pequeña que une un mensajero por el primer aminoácido (metionina) unido a un tRNA especial. Hay factores proteicos que según la etapa en que participen se llaman IF, EF y RF (con E delante si son eucariotas).Participan tres factores: - IF1 e IF3: estabilizan la subunidad pequeña separada de la grande. - IF2: permite que el primer aminoacil-tRNA se una. Al unirse lo hace al sitio P, asociado a IF2 que ha de estar en forma activa (uniendo un nucleótido GTP) y es esencial. Es el único aminoacil-tRNA que se une cuando sólo está la subunidad pequeña. La metionina está formilada en el α-amino (formilmetionina). Cuando se une la subunidad grande se disocian todos los factores proteicos. IF1 e IF3 salen pero IF2 ha de hidrolizar el GTP para salir. El mensajero se posiciona sobre el codon de AUG adecuadamente para que luego se vean los codones. Para ello todos los mensajeros de procariotas tienen el codon de iniciación AUG y a su izquierda tienen una secuencia (Shine-Dalgarno) muy conservada rica en purina complementaria de la secuencia del RNA de la subunidad pequeña (16s). Se supone que interaccionan igual que la doble hélice colocando el mensajero. Eucariotas. Es más complicada por: - Muchos más factores de iniciación. - No hay secuencias de Shine-Dalgarno, la colocación es por otros procedimientos, el tRNA no tiene una zona complementaria. - Metionina no formilada. La metionina unida al tRNA iniciador es lo único que se une a sólo una de las dos subunidades en el sitio P mal definido. Reacciona con EIF2. El primer aminoácido (metionina) se une con EIF2, cuando el ribosoma tiene el primer aminoácido se une al mensajero. El mRNA en eucariotas tiene gorra 5', es el punto de enganche de la subunidad pequeña que ya tiene el primer aminoácido. Se desliza por el mensajero hasta encontrar el codon de iniciación y se para porque el tiene el aminoacil-tRNA que establece interacciones. Implica gasto de ATP porque hay estructuras que deshacer, hay factores proteicos que ayudan a deslizarse. Al entrar la subunidad grande se sueltan los factores proteicos. Elongación. Formación de los enlaces peptídicos desde el segundo aminoácido hasta el C-terminal. Ribosoma ya preparado. Entra el segundo aminoácido en el sitio A (la metionina está en el P) y se forma enlace peptídico. el ribosoma se desplaza, en el sitio P estarán la metionina y el segundo y en el sitio A el tercero. La entrada en el sitio A depende del codon. En procariotas y eucariotas es casi igual. Hay tres factores de elongación: EF-Tu EEF-1α EF-Ts EEF-1βγ EF-G EEF-2 Un aminoácido para entrar en el ribosoma ha de estar unido a un factor proteico (EF-Tu o EEF1α) llamado aminoacil-tRNA: El factor proteico nunca une ni tRNA iniciador ni tRNA sin aminoácido. El tRNA seleccionado según complementariedad dl codon. Sitio P: Los aminoácidos unidos por el carboxilo. Para formar peptídico el carboxilo 1° + amino 2°. Implica transferencia porque el CO que está activado se transfiere al α-amino participando una peptidiltransferasa. En el sitio A queda un dipéptido unido por el tRNA del segundo. En el P queda tRNA vacío que se sale pasando por el sitio E. No necesita factor proteico extra. El RNA grande de la subunidad grande es la peptidiltransferasa. Tiene actividad catalítica. Para unir el siguiente aminoácido en el sitio A el tercer codon ha de estar desocupado, por lo que el complejo se ha de mover 3 nucleótidos (traslocación). Requiere factor proteico G activado con GTP. El factor proteico G ha de salir para que entre otro aminoácido porque sólo ayuda a moverse. Para salir hidroliza GTP. Se disocia porque solapa el sitio de Tu. El factor Ts sirve para reciclar Tu reactivándolo. Los factores se pueden usar varias veces si hay energía. Terminación Implica la disociación del complejo ribosoma + tRNA + proteína nueva.. Cuando en el sitio A aparezca un codon de parada tras el C-terminal o sin sentido, la peptidiltransferasa en lugar de transferir el peptídico a otro aminoácido lo transfiere al agua, gracias a los factores proteicos RF ó ERF. Modificaciones post-traduccionales. 1.- Transformaciones para que la proteína sea nativa. Las diferencias entre la proteína sintetizada y la nativa son: - Puede perder aminoácidos por ruptura del enlace peptídico. Es común la pérdida del Nterminal (Met). - Modificaciones químicas de las cadenas laterales de los aminoácidos codificados. Pueden ser durante la síntesis (contraduccionales) o después (post-traduccionales). Siempre tras la incorporación del aminoácido a la cadena. A veces implica reaccionar con hidratos de carbono, metilación, hidroxilación ... 2.- Es esencial que la cadena polipeptídica se pliegue (estructuras secundaria y terciaria). El plegamiento ocurre cuando la proteína se sintetiza, no espera a que esté la cadena completa, porque los grupos hidrofóbicos molestarían. Puede ser que los primeros plegamientos no sean los óptimos, por lo que ayudan carabinas moleculares protegiendo las regiones hidrofóbicas, para lo que se unen reversiblemente. 3.- Para que sean funcionales deben localizarse dentro de la célula. En los eucariotas hay muchas membranas que deben ser traspasadas. En eucariotas hay síntesis de proteínas: - Dentro de mitocondrias y cloroplastos, que sintetizan para ellos mismos con ribosomas propios aunque necesiten proteínas del exterior. - A partir del DNA nuclear se sintetizan proteínas por los ribosomas en el citosol. Las proteínas sintetizadas en el citosol pueden tener varios destinos: - Para distinguir entre varios destinos tendrán que tener una señal de localización. - En el orgánulo habrá un receptor que reconozca esa señal. Una célula no se puede obtener a partir del DNA. hace falta más información, epigenética, que diga qué cosa ha de ir dónde. Las proteínas que se han de quedar en el citosol no tienen señal porque siempre hay un sitio por defecto. Transportar una proteína a través de una membrana se puede hacer de dos maneras:: - Si ha de pasar a través de la membrana no podrá haberse plegado. Si la proteína es globular no podrá pasar porque la membrana es hidrofóbica, por lo que tendrá que estar parcialmente desplegada y protegida por carabinas. - Se puede englobar las proteínas dentro de una vesícula que se fusione con la membrana y las libere dentro. Las proteínas deben estar dentro del retículo y pueden estar plegadas. - El núcleo tiene poros por los que pueden pasar las proteínas ya plegadas. Hay dos tipos de transporte: - Post-traduccional: las proteínas primero se sintetizan y luego se transportan. - Cotraduccional: siempre van al retículo endoplásmico. El retículo rugoso tiene ribosomas que sintetizan proteínas que se quedan dentro (tienen una señal que hace que se peguen al retículo). Luego se suelta el ribosoma. Las proteínas de las membranas entran siempre dentro del retículo. Como tienen muchos trozos hidrofóbicos al sintetizarse se incorporan a la membrana y ya no salen de ella. Viajan en vesículas. Regulación de la traducción. Todas las moléculas que intervienen pueden ser reguladores (tRNA, mRNA...). - Como hay aminoácidos representados por varios codones hay tRNA más abundantes que otros, lo que limita la expresión del codon, ya que un tRNA difícil de encontrar se usa menos. - Factores que potencien o no la traducción del mensajero. En procariotas si la secuencia de Shine - Dalgarno está tapada por una proteína reguladora no puede haber traducción del mensajero. - Alteración de los factores proteicos IF y EF por modificación covalente (fosforilación). El punto de control más importante es el I (iniciación) porque es la etapa más lenta). Los antibióticos matan bacterias bloqueando la síntesis de proteínas. A los eucariotas no les afecta porque el mecanismo es otro. El cloramfenicol bloquea la síntesis del enlace peptídico. http://www.loseskakeados.com