Biología Celular
Anuncio

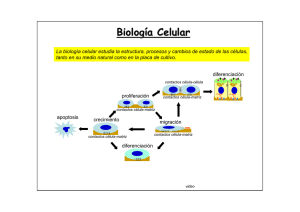
Biología Celular La biología celular estudia la estructura, procesos y cambios de estado de las células, tanto en su medio natural como en la placa de cultivo. diferenciación contactos célula-célula proliferación contactos célula-matriz contactos célula-matriz apoptosis crecimiento migración contactos célula-matriz contactos célula-matriz diferenciación video ... ... .. . ... .... Las células en cultivo pueden examinarse vivas mediante microscopía microscopio invertido lámpara halógena cámara calefaccionada lámpara de mercurio cámara CCD mesa a datos a la computadora ntivib ratoria Microscopía multi-dimensional permite el análisis simultaneo de la localización 3D, dinámica e interacciones de múltiples proteínas Giepmans et al., Science 2006 Adquisición y procesamiento de imágenes en 4 dimensiones serie temporal + varios planos de foco en el eje z. En el ejemplo de la derecha se muestra el esquema de adquisición de los datos y su posterior procesamiento. Organización de la cromatina durante mitosis visualizada mediante transfeccion de la histona 2B-CFP Gerlich, Nature 2003 El análisis de células vivas es posible mediante la transfección de proteínas fluorescentes Transfección de retinas enteras pistola de helio “gen gun” para introducir cDNA codificante de la GFP. CMV GFP Expresión de GFP en neuronas in situ Visualización panorámica (b) y mas detallada (c) neuronas ganglionares que expresan la GFP. Secuencia temporal ilustrando la remodelación dendrítica de una neurona ganglionar de retina in situ Marrs et al., MCN2006 El citoplasma celular esta poblado de macromoléculas y organelas Se estima que el citoplasma contiene una concentración de macromoléculas de ~ 300 mg/ml (Zimmerman & Trach, J. Molec. Biol 1991, 222:599) La densidad macromolecular del citosol modula la síntesis y dinámica de las proteínas. Moléculas < 500 daltons difunden libremente en el citosol, la difusión de grandes macromoléculas, como las proteínas es significativamente retardada (Seksek et al J Cell. Biol 1997, 138: 131). Densidad de microtúbulos de interfase en un fibroblasto inmunofluorescencia núcleo C. O. Arregui Densidad de filamentos de actina en la lamela de un keratinocito malla de actina Svitkina et al, JCB 1997 Densidad de túbulos pertenecientes a la red del retículo endoplásmico C. O. Arregui Reconstrucción de una porción del citoplasma de una célula de Dictiostelium discoideum actina en rojo ribosomas en gris membranas en azul Ellis & Minton, Nature 2004 1 1 8 Eucaryotic cell David Goodsell, Scripps Res. Inst. http://mgl.scripps.edu/people/goodsell/ proteínas en azul, DNA/RNA en rosado lípidos en amarillo carbohidratos en verde ribosomas en magenta 8 La estructura y función de las células depende fundamentalmente de las proteínas Alberts et al, MBC 2002 Revisión de algunos conceptos relacionados a las proteínas • El plegado de las proteínas es asistido por chaperonas y ocurre en el citosol o en compartimientos específicos (RE, mitocondrias). • La función de las proteínas depende de su estructura tridimensional. • La estructura tridimensional es determinada por la secuencia de aminoácidos. • Cada proteína adopta una estructura tridimensional única o conformación nativa. • La conformación nativa es la más estable termodinámicamente y se mantiene por múltiples interacciones no covalentes. • Los dominios son subestructuras que se pliegan de manera autónoma y conservan su función cuando se transplantan de una proteína a otra. • La pérdida de la conformación nativa se denomina desnaturalización. La función de las proteínas depende de su estructura tridimensional Fosfatasa de tirosina PTP1B Quinasa de tirosina Src Integrina Proteína con un único dominio globular. Proteína con varios dominios globulares. Proteína de transmembrana con un dominio intracelular, transmembrana y extracelular. En la célula el plegado de las proteínas es asistido por chaperonas en reacciones dependientes de ATP Hsp70-ADP Hsp70-ATP Las chaperonas difieren en estructura y función. Las denominadas chaperonas moleculares se unen a segmentos hidrofóbicos de los polipéptidos nacientes impidiendo su agregación y degradación (ej. Hsp70, BiP, DnaK). La unión de ATP y su hidrólisis modulan la afinidad por el substrato. Las denominadas chaperoninas son complejos multi-proteicos que encapsulan y activamente contribuyen al plegado de los polipéptidos sintetizados en una manera dependiente de ATP (ej. Hsp60, GroEL, CCT). (chaperonina de E. coli) La elongación de los polipéptidos en eucariotas ocurre a ~ 4 aminoácidos por segundo (Thulasiraman et al EMBOJ 1999). Las chaperoninas representan entre el 0.1-1% del total de proteínas solubles y ~ 10% del total de ribosomas. Lodish et al, MCB 2004 En la célula las proteínas defectuosas o que han cumplido su vida útil se degradan en los proteasomas por procesos dependientes de ATP A diferencia de los lisosomas, organelas que degradan material endocitado, los proteasomas degradan proteínas en el citosol. Los proteosomas son complejos de proteasas organizados en estructuras con forma de barril. Las subunidades reguladoras de los extremos unen a polipéptidos poli-ubiquitinilados, los despliegan y transfieren a la cámara central donde son degradados a péptidos cortos. Lys Reconstrucción de imágenes de microscopía electrónica 19S tapa cámara tapa ~ 2,5 MDa 20S Existen diversos mecanismos de reconocimiento de los substratos de ubiquitinación Lodish et al, MCB 2004 Smalle & Vierstra, ARPB 2004 Varias enfermedades humanas resultan de la agregación intra o extracelular de polipéptidos mal plegados Soto, Nature Rev Neuro 2003 La presencia de agregados proteicos puede visualizarse por histoquímica. Al microscopio electrónico los agregados forman fibras (centro) Soto, Nature Rev Neuro 2003 Las células exhiben una organización modular (0.5-1 μm) Gavin et al, COCB 2003 En las células las proteínas usualmente forman complejos (ensambles) funcionales Alberts et al, MBC 2002 Los complejos de señalización dependen de diversos módulos o dominios proteicos Pawson & Nash, TICB 2001 Los complejos de señalización son dinámicos señal1 GEF PP GEF PP SH3 Crk SH2 SH3 Crk SH2 pY pY pY pY pY pY Cas PP SH3 Src kinasa SH2 pY397 FERM pY861 pY925 pY576/577 FAT FAK kinasa LD LD PP SH3 autofosforilación de FAK unión de Src fosforilación y max activación de FAK fosforilación de Cas unión de Crk y GEFs (C3G, DOCK180) PP pax señal1 GEF PP GEF PP SH3 Crk SH2 SH3 Crk SH2 señal2 Sos SH3 SH3 pY pY pY pY pY pY Grb2 Cas PP SH3 SH2 Src kinasa pY397 FERM SH2 pY861 pY925 pY576/577 FAT FAK kinasa LD LD PP SH3 unión de Grb2 interacción con Sos PP pax Otros ejemplos de ensambles funcionales multiproteicos transcripción eliminación de intrones degradación de proteínas Ejemplos de ensambles funcionales multiproteicos contracción distribución de cromosomas adhesión celular Ejemplos de ensambles funcionales multiproteicos transporte núcleo-citoplásmático Los interactomas describen las interacciones entre proteínas a nivel global Interactoma de S. cerevisiae 1.870 proteínas 2.240 interacciones ROBUSTEZ ~73% genes de levadura no son esenciales; mutaciones al azar no afectan la topología de la red (Giaever et al, Nature 2002) (n=1.572 mutants) nodo Jeong et al, Nature 2001 DIP: Database of Interacting Proteins http://dip.doe-mbi.ucla.edu P53 es un ejemplo de proteína nodal (hiperconectada) Vogelstein et al, Nature 2000 Relaciones topológicas entre compartimientos subcelulares Alberts et al, MBC 2002 Las proteínas no citosólicas poseen motivos esenciales para su localización subcelular Lodish et al, MCB 2004 poro nuclear carioferinas Ran otros componentes… El transporte núcleo-citoplasmático ocurre a través de los poros nucleares Alberts et al, MBC 2002 Los poros nucleares son complejos multiproteicos Los complejos de los poros nucleares de células de vertebrados consisten de múltiples copias de ~ 30 proteínas diferentes denominadas nucleoporinas. vista desde el citosol cara intranuclear malla de laminas (intranuclear) microscopía electrónica de la envoltura nuclear de oocitos de Xenopus 50 nm FG nucleoporins proteínas < 50 kDa pueden pasar pasivamente por los poros nucleares. Proteínas > 50kDa requieren de un transporte activo. Lodish et al, MCB 2004 Los complejos del poro nuclear estan anclados a una malla filamentosa o lámina nuclear en contacto con la membrana nuclear interna La lámina nuclear esta formada por proteínas de filamentos intermedios denominadas laminas Alberts et al, MBC 2002 Diversos componentes estan involucrados en el transporte a través de los poros nucleares secuencias de localización nuclear (NLS) entrada al núcleo secuencias de exportación nuclear (NES) salida del núcleo GTPasas: Ran-GDP Ran-GTP FG-nucleoporinas transportadores o carioferinas (importinas, exportinas) La primera señal NLS se identificó en mutantes de proteínas (antígeno) T del virus SV40 (A) La proteína T salvaje se localiza en el núcleo y depende de una NLS antígeno T dentro de núcleos (B) proteínas T con una mutación en la NLS se localizan en el citoplasma antígeno T excluído de núcleos Alberts et al, MBC 2002 La fusión de la NLS a una proteína citosólica es suficiente para dirigirla al núcleo piruvato quinasa (distribución citosólica) piruvato quinasa fusionada a la NLS del SV40 núcleo núcleo Lodish et al, MCB 1999 Las NLS denominadas clásicas son de dos tipos: unipartita, ej. antígeno T de SV40; KKKRK bipartita, ej. nucleoplasmina; KRPAATKKAGQAKKKK Demostración de la identificación de una señal NLS en la proteína nuclear nucleoplasmina Alberts et al, MBC 2002 Micrografía electrónica mostrando la translocación de partículas de oro coloidal recubiertas con NLS Cada núcleo posee ~ 4000 poros nucleares Alberts et al, MBC 2002 La importación de proteínas al núcleo requiere de transportadores y de la GTPasa Ran Las importinas β interaccionan directamente con la NLS de la proteína cargo, o indirectamente a través de las importinas α. Las importinas β además interaccionan con las FG nucleoporinas. Existen ~ 20 importinas β en humanos. Ran es una GTPasa que se activa exclusivamente en el núcleo como consecuencia del intercambio de GDP por GTP promovido por un GEF. Ran-GDP (inactivo) Ran-GTP (activo) FG nucleoporins Ran-GTP se une e induce un cambio conformacional en la importina-β, provocando la disociación del cargo en el núcleo. Lodish et al, MCB 2004 Diversas modificaciones post-traducción regulan la importación al núcleo *ver clase de señalización, imagen 86 enmascaramiento de la NLS enmascaramiento de la NLS Ciertas proteínas que se translocan al núcleo no poseen una NLS y no interaccionan con importinas (ej. β-catenina, Erk2, SMADs) Gasiorowski & Dean, ADDR 2003 Regulación del transporte núcleo-citoplasma de NF-AT activación de células T, incremento de calcio, activación de calcineurina P 1 2 P Calcineurina se asocia y defosforila NF-AT. NF-AT defosforilado expone una NLS NF-AT NLS tirosin-kinasas NÚCLEO + Crm-1 (exportina) 4 E E NES retorno de calcio a nivel basal, disociación de calcineurina, exposición de una NES en NF-AT. (NF-AT: Nuclear Factor of Activated T cells) E E CITOSOL + αβ importinas 3 NF-AT se une al DNA y activa la transcripción activación de genes (ej. IL-2 & IL-4) (video disponible) El transporte de proteínas del núcleo al citosol requiere de una NES, exportinas y Ran-GTP Las exportinas median el transporte de proteínas del núcleo al citosol. La unión de exportinas a Ran-GTP aumenta su afinidad por la NES del cargo. Ran hidroliza el GTP en el citoplasma, en una reacción facilitada por la proteína reguladora GAP. La hidrólisis del GTP de Ran en el citosol produce el desensamble del complejo cargo/exportina/Ran. La mayoría de las proteínas nucleares, tRNAs y rRNAs utilizan este mecanismo de exportación. La exportina-1 o Crm1 reconoce una NES rica en leucinas (L - X(1-3) - L - X(2-4) - L - X – L) Lodish et al, MCB 2004 Los mRNAs se transportan al citosol independientemente de Ran y exportinas * las repeticiones de FG en las nucleoporinas fueron omitidas en la porción inferior de la figura para una mejor claridad. En eucariotas el mRNA es exportado al citoplasma por el complejo TAP-p15. Este complejo interacciona directamente o a través de proteínas adaptadoras con el mRNA. Otras regiones del complejo interaccionan con las FG nucleoporinas. Lodish et al, MCB 2004 las mitocondrias representan ~20% de la masa total de la célula eucariota las mitocondrias estan delimitadas por una doble membrana (externa e interna) mas del 98% de las proteínas mitocondriales son codificadas por genes nucleares las proteínas mitocondriales codificadas por genes nucleares poseen secuencias de localización a mitocondrias las secuencias interaccionan con receptores mitocondriales(TOMs y TIMs) la secuencia de tránsito a la matriz es clivada en la proteína madura las proteínas se importan a la matriz en forma desplegada Ejemplos de proteínas mitocondriales sintetizadas en el citosol Mitochondrial Location Protein* Matrix Alcohol dehydrogenase (yeast) Carbamoyl phosphate synthase (mammals) Citrate synthase and other citric acid enzymes DNA polymerase F1 ATPase subunits α (except in plants), β, γ, and δ (in certain fungi) Mn2+ -superoxide dismutase Ornithine aminotransferase (mammals) Ornithine transcarbamoylase (mammals) Ribosomal proteins RNA polymerase Inner membrane ADP/ATP antiporter CoQH2 cytochrome c reductase complex: subunits 1, 2, 5 (Fe-S protein), 6, 7, and 8 Cytochrome c oxidase subunits 4, 5, 6, and 7 Phosphate/OH antiporter Proteolipid of F0 ATPase Thermogenin (brown fat) Intermembrane space Cytochrome c Cytochrome c peroxidase Cytochrome b2 and c1 (subunits of CoQH2 cytochrome c reductase complex) Outer membrane Mitochondrial porin (P70) * Most proteins (except the ADP-ATP antiporter, cytochrome c, and porin) are fabricated as longer precursors. Las proteínas de la matriz mitocondrial poseen secuencias de importación en el terminal amino Las secuencias de importación a la matrix (10-80 aminoácidos) adoptan una estructura de alfa hélice anfipática. Residuos cargados e hidrofóbicos se orientan en lados opuestos de la hélice formando una señal conformacional. Las propiedades anfipáticas de la secuencia señal están conservadas evolutivamente y pueden predecirse mediante programas como PSORT II y MITOPROT II residuos cargados positivamente (rojo) e hidrofóbicos (amarillo) Alberts et al, MBC 2002 La translocación de las proteínas mitocondriales ocurre post-traducción y requiere ATP Diseño experimental para investigar la importación de proteínas a mitocondrias importación de la proteína y clivado de la secuencia N-terminal síntesis in vitro de la proteína en estudio proteína degradada la proteína internalizada es resistente a la tripsina secuencia de importación + tripsina 1 + tripsina 2 3 + mitocondrias activas y ATP proteína mitocondrial 3 1 2 la secuencia de importación es clivada en la matriz gel de poliacrilamida Lodish et al, MCB 1999 Las proteínas mitocondriales se importan en forma desplegada por canales proteicos de la membrana externa e interna Experimento que demuestra la importación de proteínas en forma desplegada microscopía electrónica mostrado las regiones de contacto entre membrana externa e interna La proteína citosólica dihidrofolato reductasa (DHFR) fusionada a una secuencia de importación a mitocondrias (en rojo) a través de un péptido espaciador (en negro) se transloca normalmente a la matriz. La inducción del plegamiento por el agregado de methotrexato permite visualizar intermediarios de la translocación. Lodish et al, MCB 2004 La translocación de proteínas a la matriz mitocondrial involucra tres etapas que requieren energía 1. Hidrólisis de ATP. Requerida por chaperonas para mantener los polipéptidos desplegados en el citosol. 2. Gradiente electroquímico. Requerido para translocar el polipéptido a través de la membrana interna. 3. Hidrólisis de ATP. Requerida por chaperonas de la matriz para traccionar activamente el polipéptido hacia la matriz y plegarlo. Tim22/54 Complejos proteicos de la membrana externa (TOMs) e interna (TIMs), median la importación de polipéptidos que poseen la secuencia de tránsito en el terminal amino. TOM 40 forma el canal (~2 nm) en la membrana externa; TIM23/17 y TIM22/54 forman los canales en la membrana interna. en la matriz, la secuencia de importación N-terminal es removida por una proteasa. Lodish et al, MCB 1999 La localización submitocondrial requiere de secuencias adicionales Señales en proteínas mitocondriales N Lodish et al, MCB 2004 Direccionamiento de polipéptidos a la membrana mitocondrial interna Tres mecanismos principales gobiernan la inserción de proteínas en la membrana interna e involucran diferentes complejos, TIM 23/17, OXA-1 y TIM22/54, localizados en la membrana interna. Vía 1: ejemplo citocromo oxidasa Vía 2: ejemplo ATP sintasa, sub 9 Vía 3: ejemplo intercambiador de ADP/ATP Lodish et al, MCB 2004 Direccionamiento de polipéptidos al espacio de intermembrana Vía 1: ejemplo citocromo b2 Vía 2: ejemplo citocromo c heme liasa Lodish et al, MCB 2004 Génesis de cloroplastos mas del 90% de las proteínas de cloroplastos son codificadas por genes nucleares los cloroplastos poseen una membrana doble; la membrana interna delimita el estroma en el estroma existe un tercer conjunto de membranas (tilacoides) que delimitan el lúmen del tilacoide Mecanismo de importación de proteínas al estroma de cloroplastos - receptores de la membrana externa (Toc34) se unen a la secuencia señal de las proteínas de cloroplastos. - los precursores se translocan al estroma en forma desplegada, a través de complejos multiproteicos de la membrana externa (Toc75) e interna (Tic110). Tic110 - la translocación requiere de hidrólisis de ATP. - no requiere de gradiente electroquímico. - la secuencia señal es removida en la proteína madura. Lodish et al, MCB 1999 Existen varios mecanismos para importar proteínas al lumen de tilacoides - Los precursores poseen dos señales, una amino terminal para la internalización al estroma y otra para su importación al tilacoide. - Unos precursores se internalizan a través de complejos similares a los OXA de mitocondrias. - Otros dependen de un complejo proteico (SRP) que se une a la secuencia de importación al tilacoide (2). - Proteínas que unen metales como cofactores son propulsadas al interior del tilacoide por un gradiente de pH. - La cuarta vía descripta requiere de una proteína similar a SecA de bacterias. Lodish et al, MCB 2004 Los peroxisomas son organelas pequeñas presentes en la mayoría de las células eucariotas. Participan en el metabolismo de ácidos grasos. Son organelas limitadas por una sola membrana. Todas las proteínas de peroxisomas son importadas desde el citosol post-traducción. Secuencias cortas del terminal carboxilo y amino dirigen la importación de proteínas a peroxisomas - Varias enzimas de peroxisomas contienen la secuencia de importación a peroxisoma Ser-Lys-Leu (SKL) en el terminal carboxilo (PST1). - Receptores citosólicos (Pex5) se unen a la secuencia señal y transfieren los polipéptidos a un receptor en la membrana del peroxisoma (Pex14). - El complejo es transferido a un complejo multiproteico que forma un canal y es translocado. - La importación requiere de hidrólisis de ATP, aunque el mecanismo es desconocido. - La impoprtación no requiere de gradiente electroquímico. - Las proteínas se importan plegadas. - La secuencia SKL no se remueve en la proteína madura. Lodish et al, MCB 1999 Translocación post-traducción de proteínas del citosol al espacio Periplásmico en bacterias gram-negativas secΒ secΒ El translocon secY es homólogo al translocon sec61 de eucariotas. La chaperona secB une y mantiene el polipéptido en conformación competente para su translocación. SecB interacciona y transfiere el polipéptido a la ATPasa citosólica secA, la cual forma un complejo con el translocon. La translocación del polipéptido requiere de cambios conformacionales de secA propulsados por hidrólisis de ATP. lisosomas ribosoma retículo endoplásmico aparato de Golgi transportadores túbulo-vesículares membrana plasmática las proteínas se insertan o atraviesan la membrana del RE a medida que emergen del ribosoma (co-traducción). las proteínas requieren de una secuencia señal hidrofóbica en el terminal amino. las proteínas se transportan en estructuras tubulo-vesiculares que emergen del compartimiento donor y se fusionan con el compartimiento aceptor. Translocación post-traducción de proteínas del citosol al lumen del RE en eucariotas Algunas proteínas eucariotas se translocan al lumen del RE post-traducción y no requieren de un complejo soluble de reconocimiento de la secuencia señal o SRP. Este proceso requiere del translocon sec61, del complejo sec63, y de la chaperona del RE BiP. chaperonas citoplasmáticas Sec63 forma un complejo con el translocon. (1) La secuencia señal se inserta en el translocon y movimientos oscilatorios del polipéptido inician su translocación. (2) Asociación de BiP-ATP al dominio luminal de sec63, unión de baja afinidad con polipéptidos entrantes e hidrólisis del ATP. BiP-ADP incrementa la afinidad por el polipéptido y evita su retrotranslocación. (3, 4) La unión de sucesivas moléculas de BiP-ADP al polipéptido entrante facilita la translocación. (5, 6) El intercambio de ADP por ATP induce la disociación de BiP del polipéptido. Lodish et al, MCB 2004 Translocación de proteínas a través del complejo sec61 o translocon en la membrana del RE bisagra abertura lateral citosol El complejo sec61 consiste de tres subunidades: αβγ. La subunidad α posee 10 alfa-hélices organizadas en dos dominios (en rojo y azul) que forman el canal hidrofílico (~2nm Ø) por donde se translocan los polipéptidos nacientes. En reposo, el canal del translocon es obstruído por una hélice alfa en la cara luminal. Esta alfa hélice se retrae cuando el ribosoma se asocia al translocon. lúmen del RE para ver estructura entrar en: http://www.rcsb.org/pdb/explore/images.do?structureId=1RH5 Rappoport et al., TICS 2004 Una secuencia amino terminal hidrofóbica es esencial para la translocación co-traducción de numerosas proteínas al RE ejemplos de péptidos señal en tres proteínas de secreción Protein Preproalbumin Pre-IgG light chain Prelysozyme * Amino Acid Sequence* Met-Lys-Trp-Val-Thr-Phe-Leu-Leu-Leu-Leu-Phe-Ile-Ser-Gly-SerAla-Phe-Ser ? Arg . . . Met-Asp-Met-Arg-Ala-Pro-Ala-Gln-Ile-Phe-Gly-Phe-Leu-Leu-LeuLeu-Phe-Pro-Gly- Thr-Arg-Cys ? Asp . . . Met-Arg-Ser-Leu-Leu-Ile-Leu-Val-Leu-Cys-Phe-Leu-Pro-Leu-AlaAla-Leu-Gly ? Lys . . . * los residuos hidrofóbicos estan en negrillas; indica el sitio de corte de la peptidasa de la señal. La secuencia señal, de aproximadamente 20 aminoácidos de longitud, posee 8-12 residuos centrales hidrofóbicos flanqueados por uno o mas residuos básicos. El flanco con más cargas positivas se orienta hacia el citosol. La secuencia señal N-terminal se remueve en la mayoría de las proteínas de secreción. Experimento que demuestra la importación co-traducción de polipéptidos al lúmen del RE microsomas: pequeñas vesículas del RE que resultan de la ruptura de las células y que pueden purificarse por centrifugación. observación: las proteínas sintetizadas in vitro y posteriormente mezcladas con microsomas conservan la secuencia señal y son degradadas con tripsina. (proteína de secreción) *(notar que el mecanismo de importación difiere del descripto en los precursores mitocondriales). observación: las proteínas sintetizadas en presencia de microsomas se importan al lúmen de los microsomas, la secuencia señal ha sido removida, y son resistentes al tratamiento con tripsina. Lodish et al, MCB 2004 Experimento que demuestra la localización intravesicular de las proteínas de secreción Marcado radioactivo de proteínas en células vivas. Incubación con metionina-35S ~ 24h Homogeneizado de las células y purificación de los microsomas Se separan dos fracciones iguales, una fracción es incubada con detergente. Ambas fracciones son incubadas con proteasa. Luego se analiza la integridad de las proteínas de secreción radioactivas por SDS-PAGE/autoradiografia. Lodish et al, MCB 2004 Un complejo ribonucleoproteico (SRP) es esencial para asociar al polipéptido naciente a la membrana del RE El SRP es un complejo de seis proteínas asociadas a una molécula de RNA (~ 300 nucleótidos). El dominio Alu interacciona con el ribosoma y detiene la elongación del polipéptido precursor. El dominio S interacciona con la secuencia señal, el ribosoma y el receptor SR. Al formarse el complejo SRP/SP/ribosoma, p54 adopta una conformación que le permite unir GTP. ER membrane La interacción del SRP y el receptor SR en el RE es coordinada por la unión previa de GTP en ambos. el receptor del SRP (SR) es un heterodímero Halic & Beckmann, COSB 2005 Modelo de transferencia y translocación del polipéptido precursor al RE La elongación de la cadena polipeptídica en el ribosoma impulsa su translocación a través del canal del translocon. La SRP se asocia al ribosoma y al péptido señal (2) y luego transfiere el complejo a un receptor localizado en el RE (3). El canal del ribosoma es alineado con el canal del translocon y comienza la translocación (4). La hidrólisis del GTP induce la disociación del SRP y su receptor y libera la secuencia señal, la cual es posicionada en el canal. Comenzada la translocación, la secuencia señal es clivada por una peptidasa del lumen del RE (5). Concluida la translocación (6), el ribosoma se disocia del RE (7) y el polipéptido se pliega en el lúmen del RE (8). Lodish et al, MCB 2004 Resúmen de las características de secuencias proteicas que confieren localización subcelular Target Organelle Usual Signal Signal Location Removal* within Protein Endoplasmic reticulum N-terminal (+) Mitochondrion N-terminal (+) Chloroplast N-terminal (+) Peroxisome C-terminal ( ) Internal ( ) Nucleus Nature of Signal "Core" of 6 12 mostly hydrophobic amino acids, often preceded by one or more basic amino acids 3 5 nonconsecutive Arg or Lys residues, often with Ser and Thr; no Glu or Asp residues . 25-50 residues long. No common sequence motifs; generally rich in Ser, Thr, and small hydrophobic amino acid residues and poor in Glu and Asp residues. 25-50 residues long. Usually Ser-Lys-Leu at extreme Cterminus One cluster of 5 basic amino acids, or two smaller clusters of basic residues separated by ? 10 amino acids * Indicates whether signal sequence usually is (+) or is not ( ) removed after a protein enters its target organelle. These signals direct the protein from the cytosol into the matrix space of the mitochondrion or the corresponding stroma of the chloroplast; other signals discussed in the text redirect proteins into other subcompartments of these organelles. Lodish et al, MCB 2004 El reconocimiento de las señales de localización puede depender de características de la secuencia primaria, secundaria o terciaria NLS, SKL son ejemplos de señales dependientes de la secuencia primaria. La secuencia señal de proteínas de mitocondrias y del RE depende principalmente de las caracterísitcas de la estructura secundaria. Algunas NLS y la secuencia señal de localización a lisosomas son conformacionales. secuencia señal de importación a mitocondrias Alberts et al, MBC 2002 Las proteínas de andamiaje o “scaffolding” contribuyen a concentrar proteínas relacionadas funcionalmente en subcompartimientos del citosol cAMP PKA AKAP79 asocia a las enzimas PKA y PKC con diferentes receptores en la membrana plasmática. la proteína PSD-95 contribuye a concentrar diversas proteínas en la post-sinapsis.